云南省犬细小病毒VP2基因检测分析
2015-03-06郑龙龙朱玲云宋春莲高利波毕峻龙
郑龙龙 ,朱玲云,宋春莲,高利波,毕峻龙,徐 英
(1.云南农业大学动物科学技术学院,云南昆明 650201;2.楚雄州动物疫病预防控制中心,云南楚雄 675000;3.云南农业职业技术学院,云南昆明 650031 )
云南省犬细小病毒VP2基因检测分析
郑龙龙1,朱玲云1,宋春莲1,高利波1,毕峻龙2,徐 英3*
(1.云南农业大学动物科学技术学院,云南昆明 650201;2.楚雄州动物疫病预防控制中心,云南楚雄 675000;3.云南农业职业技术学院,云南昆明 650031 )
为了解云南省犬细小病毒VP2基因分子流行变异情况,对来源于云南省7个地级市13个犬细小病毒样品进行了VP2基因的扩增和序列分析,并与相关参考序列进行了遗传进化比对分析。结果表明,13个样品中9个基因型为CPV-2a,3个基因型为CPV-2b,1个基因型为CPV-2。氨基酸序列变异主要集中在297位-518位氨基酸处,其中324位和440位氨基酸变异在犬细小病毒进化过程中扮演着重要角色。遗传进化分析显示10个样品序列形成了一个相对于国内外参考序列独立的遗传进化分支,提示云南省犬细小病毒以CPV-2a 基因型流行为主。
犬细小病毒;VP2基因;基因型;遗传进化
犬细小病毒病(Canine parvovirus disease,CPVD)是由犬细小病毒(Canine parvovirus disease,CPV)引起的一种急性、烈性传染病,是当前对犬类危害最大的传染病之一。该病能够引起犬类白细胞减少、剧烈呕吐、出血性肠炎等多种症状。犬细小病毒属于细小病毒科细小病毒属成员,编码2个结构蛋白(VP1和VP2),其中VP2蛋白可诱导机体产生中和抗体,是保护性颗粒抗原。VP2蛋白中一些具有生物学意义的主要抗原位点的变异导致了病毒抗原特性和宿主范围的改变。CPV-2于20世纪70年代后期被发现不久之后,CPV-2逐渐被新的抗原变异株CPV2-a和CPV2-b取代[1-3]。近年来,另外一种新的变异样品序列CPV2-c被证实在欧美国家广泛存在[4],国内学者于2010年发现并证实CPV-2C变异样品序列也存在于我国[5-6]。随着频繁的贸易交流,犬细小病毒不断出现新的变异样品序列,流行病学呈现复杂态势,导致免疫失败现象时有发生,为犬细小病毒病的预防和控制带来了很大困难。由于目前几乎没有关于犬细小病毒云南株流行样品序列基因型的相关报道。本研究通过对细小病毒云南分离株VP2基因进行遗传进化和氨基酸位点突变的分析,调查云南省犬细小病毒分子流行变异情况,为该病的防控提供一定的理论参考依据。
1 材料与方法
1.1 材料
1.1.1 病料来源 采自云南省不同地区发生疑似犬细小病毒犬场及宠物医院病犬粪便,样品相关信息见表1。

表1 样品相关信息Table 1 Related information of sample gene sequence
1.1.2 主要试剂 DNA聚合酶、pMDTM18-T载体、限制性内切酶BsrⅠ、HindⅢ等分子生物学试剂为宝生物工程(大连)有限公司产品;其他试剂由云南农业大学动物科学技术学院动物传染病实验室提供。
1.1.3 仪器设备 移液器、超净工作台、高速冷冻离心机、恒温水浴锅、PCR仪、电泳槽、凝胶电泳成像仪等设备由云南农业大学动物科学技术学院动物传染病实验室提供。
1.2 方法
1.2.1 引物设计与合成 VP2序列全长1 755 bp,编码584个氨基酸,参考 GenBank中犬细小病毒VP2基因参考序列,即M19296、M24003、EU659121、GU569947和GU569948等序列,应用Oligo6.0软件,设计用于扩增CPV VP2基因的一对引物其中上游引物:5′-ACC ATG TAG ACT AAC ACA TAC-3′,下游引物:5′-ACA TAT TAG CAG CAT CAG-3′,扩增片段长度为741 bp。
1.2.2 VP2片段的扩增及克隆 使用DNA提取试剂盒提取组织样品总DNA,根据PCR试剂说明书优化反应体系及条件,其中10×PCR buffer 2.5 μL,dNTP Mixture 1 μL,上游和下游引物分别0.5 μL,TaqDNA聚合酶0.5 μL,模板1 μL,加水补足25 μL。进行PCR扩增后,将扩增产物和上样缓冲液混合后,用15 g/L的琼脂糖凝胶进行电泳,20 min后观察结果。 将阳性PCR产物经切胶回收后与pMD18-T Vector进行连接反应,连接产物转化进入DH5α感受态细胞,挑取单克隆菌株后,扩大培养,提取质粒,并使用限制性内切酶进行酶切鉴定。
1.2.3 序列测定与分析 将鉴定为阳性的菌种进行测序分析,使用DNA Star和MEGA 4.0等生物学软件分别对所获得基因序列进行位点突变和遗传进化分析,并与国内外参考序列进行比较并绘制遗传进化树(表2)。
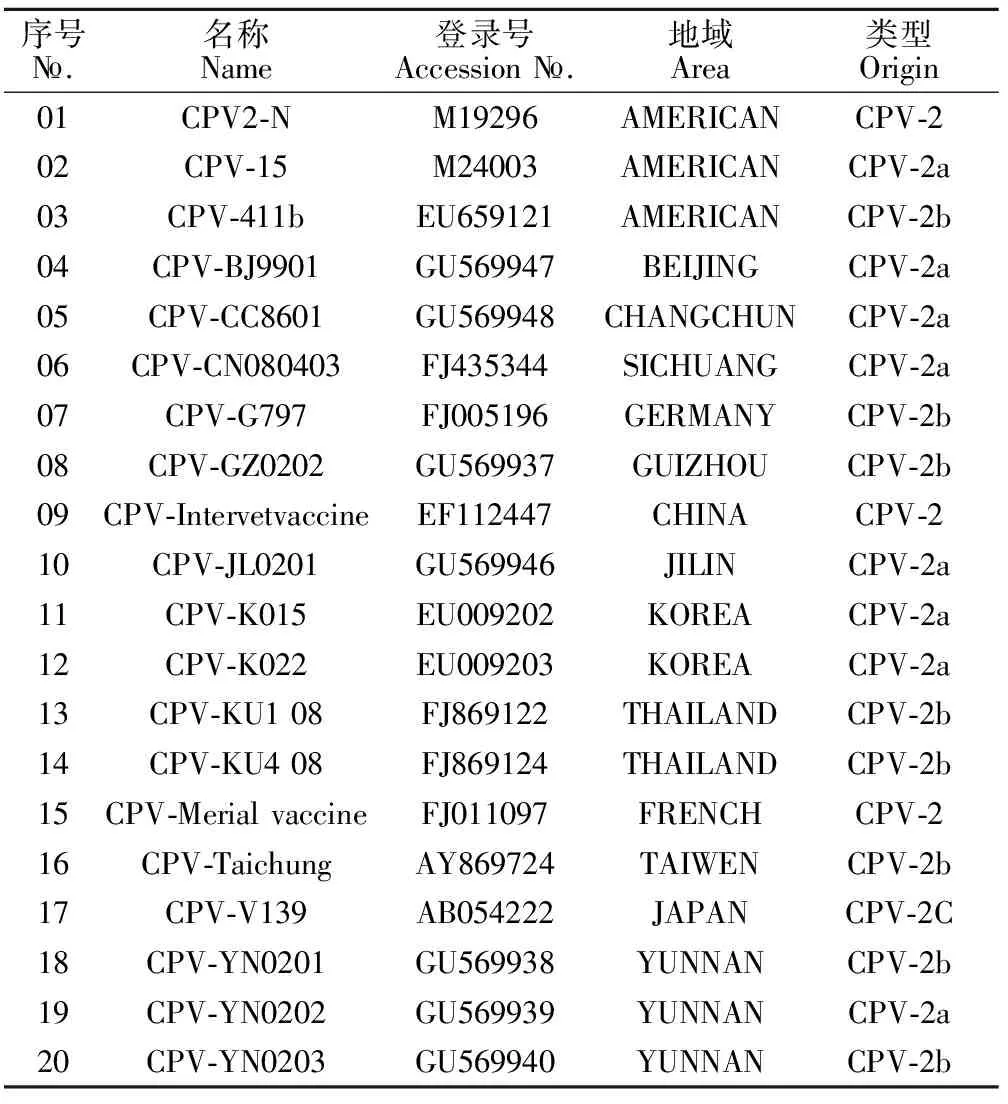
表2 分析所用的CPV参考序列及其来源Table 2 CPV strains used in this study and their origins
2 结果
2.1 VP2基因的扩增与分析
样品DNA经PCR扩增后,经15 g/L的琼脂糖凝胶进行电泳检测,经凝胶成像系统观察后,发现阳性对照及样品均在DNA Maker DL 700 bp附近处有一条带,与预期741 bp相符(图1)。

图1 VP2基因PCR扩增结果Fig.1 PCR results of VP2 gene
2.2 VP2基因重组质粒酶切鉴定
通过DNA Star 6.0分析,在pMDTM18-T载体上选择HindⅢ,在目的片段第572位选择BsrⅠ进行双酶切,预期目的片段为2 834 bp和599 bp或3 237 bp和196 bp。将切胶回收纯化的CPV VP2基因的PCR扩增产物(741 bp),连接、转化到感受态细胞DH5α中,经过培养后提取重组质粒进行酶切鉴定。重组质粒经过HindⅢ和BsrⅠ双酶切后得到两个大小约为2 834 bp和599 bp的片段,酶切结果与预期相符,表明成功构建了重组质粒(图2)。

图2 重组质粒酶切鉴定Fig.2 Identification of recombinant plasmids digested with BsrⅠ and HindⅢ
2.3 VP2序列测定与分析
测序结果显示,本试验获得的13个VP2基因包含着741个碱基,编码247个氨基酸。对所获得的基因序列使用生物学软件DNA Star与其他参考序列进行核苷酸同源分析(表3)。结果表明,于2012年获得的5株VP2基因的核苷酸同源性为99.5%~100%;于2013年获得的8株VP2基因的核苷酸同源性为96.8%~100%;2012年序列和2013年样品序列P2基因的核苷酸同源性98.8%~99.2%;2012序列与参考序列的核苷酸同源性为98.7%~99.5%,与疫苗序列EF112447核苷酸同源性为98.5%~98.8%;2013年序列与参考序列的核苷酸同源性为98.8%~99.75%,与疫苗序列EF112447核苷酸同源性为98.7%~99.6%。与2012年获得的5株VP2基因的蛋白同源性为99.6%~100%;与2013年获得的8株VP2基因的蛋白同源性为97.2%~100%;2012年序列和2013年序列VP2基因的蛋白同源性为98.8%~99.2%;2012年序列与参考序列的蛋白同源性为97.6%~99.2%,与疫苗序列EF112447蛋白同源性为97.2%~97.6%。2013年序列与参考序列的蛋白同源性为97.2%~100%,与疫苗EF112447蛋白同源性为96.8%~99.2%。
2.4 VP2基因氨基酸突变分析
将实验获得的13个VP2氨基酸序列和部分欧美、亚洲及我国样品序列的相应氨基酸序列进行比较(表4)。云南VP2氨基酸序列变异主要集中在297位~518位氨基酸处,其中297、300、305、324、375、386、426、427、440和518位点存在氨基酸替换。2012YN-1、2012YN-2、2012YN-3、2012YN-4、2012YN-5、2013YN-6、2013YN-10、2013YN-11、2013YN-12为CPV-2a型,2013YN-7株为CPV2型,2013YN-8和2013YN-9株为CPV-2b型,CPV-2a型是云南省2012年-2013年的主要流行序列。
2.5 VP2基因遗传进化树分析
将所得到的13个2012年-2013年云南流行序列VP2基因与参考序列进行遗传分析后绘制进化树,如图3所示。本试验所得病毒序列2013YN-7、2013YN-8和2013YN-9序列和其他云南毒株序列亲源关系较远;2012YN-1、2012YN-2、2012YN-3、2012YN-4、2012YN-5 2013YN-6、2013YN-10、2013YN-11、2013YN-12、2013YN-13相对于其他参考序列形成了一个独立分支。

表3 不同序列VP2序列氨基酸同源性Table 3 Sequence similarities of vp2 gene from difficult strains on nucleotide acid and amino acid levels %

表4 VP2基因片段中发生置换的氨基酸Table 4 The substitutions of the amino acids in the VP2 gene

图3 CPV VP2基因遗传进化分析Fig.3 Phylogenetic relationship of CPV VP2 gene
3 讨论
VP2基因全长1 755 bp,编码584个氨基酸,它的关键位点的变异决定着犬细小病毒基因型。CPV-2a和CPV-2b 基因型序列VP2蛋白的在87、300、305位点氨基酸与CPV-2基因型不同[7]。CPV-2a和CPV-2b序列只在VP2基因的第426位氨基酸不同,其中426位点Asp是CPV-2b型序列特有的。在CPV-2a、CPV-2b基因型的基础上,VP2蛋白的第300位点发生了G-D替换,由此产生了CPV-2c(a)和CPV-2c(b)两个新的突变基因型[8],因此,本研究扩增的741 bp VP2基因片段主要包括了决定犬细小病毒基因型的关键位点,可以根据位点的变异来判断云南流行株的基因型。
尽管CPV广泛存在于世界,它的基因型在不同的国家和地区具有很大差异。近年来,在欧洲的许多国家,如英国、德国和意大利,CPV-2b型 和 CPV-2c型逐渐超过CPV-2a型,成为CPV优势流行基因型;CPV-2b型和 CPV-2c型在北美地区同许多欧洲国家一样是CPV的优势基因型;而在南美,以CPV-2c型为CPV主要流行基因型[9-10]。CPV-2a是亚洲和澳大利亚的CPV主要优势基因型[11-13]。Zhang R等[14-15]通过分析2006年-2009年的CPV序列,证明我国CPV主要流行CPV-2a基因型。将云南13个CPV VP2的氨基酸序列使用生物学软件进行氨基酸位点变异分析后,结果表明2012YN-1、2012YN-2、2012YN-3、2012YN-4、2012YN-5、2013YN-6、2013YN-10、2013YN-11、2013YN-12基因型为CPV2a,2013YN-7基因型为CPV2,2013YN-8和2013YN-9基因型是CPV2b,CPV-2a
流行程度超过CPV-2b,是当前云南省CPV主要流行基因型。本试验所获得的VP2氨基酸序列变异主要集中在VP2基因297位~518位氨基酸处,其中97、300、305、324、375、386、426、427、440、518位点存在氨基酸替换,2012YN-1、2012YN-2、2012YN-3、2012YN-4、2012YN-5、2013YN-6、2013YN-10、2013YN-11、2013YN-12、2013YN-13的324位氨基酸由Tyr变成Ile位点,同324位点临近的323位点对病毒具有重要的生物意义,该位点影响着病毒和犬运载蛋白受体的结合及感染宿主的范围[16],2012YN-1、2012YN-3、2013YN-6、2013YN-11、2013YN-12、2013YN-13的440氨基酸位点由Thr变成Ala,相关研究表明了该位点在细小病毒进化过程中扮演着重要角色。所得到的13个2012年-2013年云南流行VP2基因序列与国内外VP2参考序列绘制遗传进化树,云南流行毒2013YN-9和2013YN-8样品序列和其他云南毒亲源关系较远,2012YN-1、2012YN-2、2012YN-3、2012YN-4、2012YN-5、2013YN-6、2013YN-10、2013YN-11、2013YN-12、2013YN-13形成了一个独立分支,提示:犬细小病毒云南流行基因型在云南本地的流行过程中形成了相对独立于其他参考序列的云南分支。
本研究通过对云南省犬细小病毒序列进行基因型和遗传进行分析,表明了当前云南省流行的病毒基因型主要有 CPV-2a、CPV-2b和CPV-2,其中以CPV-2a占主要地位,初步了解云南省犬细小病毒VP2基因序列遗传进化趋势,为该病的预防和控制提供了一定的理论基础和参考依据。
[1] Marina G C, Maximiliano W, Lorena B, et al. Study of canine parvovirus evolution: comparative analysis of full-length VP2 gene sequences from Argentina and international field strains[J].Virus Genes,2012,44:32-39.
[2] Decaro N, Desario C, Amorisco F, et al. Detection of a canine parvovirus type 2c with a non-coding mutation and its implications for molecular characterisation[J].Vet J,2013,196:555-557.
[3] 鞠厚斌,周锦萍,刘 健,等.上海地区犬细小病毒的分离及VP2基因序列分析[J].中国动物传染病学报,2013,21(1):37-41.
[4] Calderon M G, Romanutti C, Antuono D, et al. Evolution of canine parvovirus in Argentina between years 2003 and 2010: CPV2c has become the predominant variant affecting the domestic dog population[J].Virus Res,2011,157:106-110.
[5] 张仁舟,杨松涛,冯 昊,等.中国国内首次检测到犬细小病毒CPV-2c[J].中国病原生物学杂志,2010,54(2):246-249.
[6] 姜燕霞,张淑玲,王金良,等.犬细小病毒JN株的分离鉴定及VP2基因序列分析[J].动物医学进展,2013,34(5):83-86.
[7] Hoelzer K, Shackelton L A, Parrish C R, et al. Phylogenetic analysis reveals the emergence, evolution and dispersal of carnivore parvoviruses[J]. J Gen Virol,2008,89(9):2280-2289.
[8] Martella V, Cavalli A, Pratelli A, et al. A canine parvovirus mutant is spreading in Italy[J].J Clin Microbiol,2004,42:1333-1336.
[9] Hong C, Decaro N, Desario C, et al. Occurrence of canine parvovirus type 2c in the United States[J].J Vet Diagn Invest,2007,19:535-539.
[10] Perez R, Francia L, Romero V, et al. First detection of canine parvovirus type 2c in South America[J].Vet Microbiol,2007,124:147-152.
[11] Kang B K, Song D S, Lee C S, et al. Prevalence and genetic characterization of canine parvoviruses in Korea[J].Virus Genes,2008,36:127-133.
[12] Meers J, Kyaw-Tanner M, Bensink Z, et al. Genetic analysis of canine parvovirus from dogs in Australia[J].Aust Vet J,2007,85:392-396.
[13] Nandi S, Chidri S, Kumar M, et al. Occurrence of canine parvovirus type 2c in the dogs with haemorrhagic enteritis in India[J].Res Vet Sci,2010,88:169-171.
[14] 李世静,嵇辛勤,主 性,等.犬细小病毒VP2基因测序分析[J].动物医学进展,2013,34(6):96-100.
[15] Zhang R, Yang S, Zhang W, et al. Phylogenetic analysis of the VP2 gene of canine parvoviruses circulating in China[J].Virus Genes,2010,40:397-402.
[16] Jaime Aldaz, Juan García-Díaz, Lucía Calleros, et al. High local genetic diversity of canine parvovirus from Ecuador[J].Vet Microbiol,2013,166(1-2):214-219.
Detection and Analysis of VP2 Gene of Canine Parvovirus in Yunnan Province
ZHENG Long-long1,ZHU Ling-yun1,SONG Chun-lian1,GAO Li-bo1,BI Jun-long2,XU Ying3
(1.CollegeofAnimalScienceandTechnology,YunnanAgriculturalUniversity,Kunming,Yunnan,650201,China; 2.ChuxiongProvinceCenterforDiseaseControlandPrevention,Chuxiong,Yunnan,675000,China; 3.YunnanVocationalandTechnicalCollegeofAgriculture,Kunming,Yunnan,650031,China)
In order to investigate the prevalence and variation of VP2 gene of CPV in Yunnan province, the gene amplification and sequence analysis were performed for thirteen strains of canine parvovirus, comprising of 9 strains of CPV-2a, 3 strains of CPV-2b and 1 strain of CPV-2. It was shown that most of the amino acid variations occurred within the region of position 297-518. A high level of substitution at 324 and 440 sites was associated with the evolution of antigenic variants in circulating parvovirus types. Based on the phylogenetic analysis on the amino acid level of the VP2 proteins, the thirteen strains of CPVs were grouped into 3 clusters. Nine of them belonged to CPV-2a, three belonged to CPV-2b, and one belonged to CPV-2. The result showed that CPV-2a was the main prevalent strain in Yunnan province and formed the independent branch.
Canine parvovirus;VP2 gene;typing;genetic evolution
2014-09-28
云南省公益性关键技术研究开发计划子项目(2013CH001)
郑龙龙(1988-),男,山西运城人,硕士研究生,主要从事动物传染病学与分子流行病学研究。*
S852.659.2
:A
:1007-5038(2015)01-0066-05
