新功能生物元器件的筛选、设计与功能鉴定
2015-03-02周志华赵宗保牛国清中国科学院上海生命科学研究院植物生理生态研究所中国科学院合成生物学重点实验室上海000中国科学院大连物理化学研究所大连60中国科学院微生物研究所北京000
周志华赵宗保牛国清( 中国科学院上海生命科学研究院植物生理生态研究所中国科学院合成生物学重点实验室,上海 000; 中国科学院大连物理化学研究所,大连60; 中国科学院微生物研究所,北京 000)
新功能生物元器件的筛选、设计与功能鉴定
周志华1赵宗保2牛国清3
(1 中国科学院上海生命科学研究院植物生理生态研究所中国科学院合成生物学重点实验室,上海 200032;2 中国科学院大连物理化学研究所,大连
116023;3 中国科学院微生物研究所,北京 100101)
周志华,上海生命科学院植物生理生态研究所研究员,课题组长。研究方向:活性萜类化合物的合成途径及其关键生物元件的解析与功能鉴定;重要萜类天然化合物于真菌底盘细胞中的异源合成与途径优化;真菌底盘细胞的基因组编辑及适配性优化。
E-mail:zhouzhihua@sippe.ac.cn

作者简介
赵宗保,研究员,中国科学院大连化学物理研究所生物技术部副主任。主要从事能源生物技术和化学生物学领域的探索性、前瞻性研究和技术创新。
E-mail:zhaozb@dicp.ac.cn

作者简介
牛国清,博士,中国科学院微生物研究所副研究员。研究方向为微生物次级代谢产物的生物合成与调控。
E-mail:Niugq@im.ac.cn发展在环境、资源、能源等方面面临的若干重大挑战提供新技术与方法 。合成生物学旨在将工程原理应用到生物系统来改变生物技术 。在短短不到十年的时间里,合成生物学领域已经产生了很多技术应用,为药品生产 、生物基产品的生产 和基因治疗 提供了新的途径。
生物元件或器件、装置和系统是合成生物学的三大基本要素。生物元件是指遗传系统中最简单、最基本的生物积块(BioBrick),是具有特定功能的氨基酸或者核苷酸序列,可以在更大规模的设计中与其他元件进一步组合成具有特定生物学功能的生物学装置(Device) 。2003年,美国麻省理工大学相关合成生物学实验室就成立了标准生物元件登记库(Registry of Standard Biological Parts)(http://partsregistry.org/),专门收集各种满足标准化条件的生物元件。全世界的科学家都可以提交自己设计的模块供其他人获取,以设计更加复杂的系统。目前标准生物元件登记库有15 000多个生物元件(http://partsregistry.org/cgi/ partsdb/Statistics.cgi),既包括启动子、转录单元、质粒骨架、接合转移元件、转座子、蛋白质编码区等DNA序列,也包括核糖体绑定位点和终止子等RNA序列以及蛋白质结构域。标准生物元件登记库中,每一个生物学元件,如启动子、核糖体结合位点(RBS)都以载体形式保存。
虽然越来越多的生物元件被发现,但很多生物元件都没有被详细定性。如标准生物元件库中的元件大部分是由iGEM参赛者提供的,其中很多都没有经过系统的定性,也无法保证品质。生物元件的性能会随不同的底盘细胞类型、不同的实验条件而改变,而不是总表现相同的功能,且发挥功能的时间也不总是相同的 。2010年12月12日,在夏威夷举行的生物技术工业组织环太平洋地区会议上,合成生物学工程研究中心的创建人Drew Endy和Vivek Mutalik发起了国际发展生物技术开放基金,由美国国家自然基金(NSF)、劳伦斯伯克利国家实验室(LBNL)和生物砖基金(BBF)联合支持,用于开发新的生物元件并对已经存在的元件进行详细定性描述。Drew Endy、Vivek Mutalik以及生物砖基金的执行主席Holly Million强调该开放基金的目的是开发可免费使用的标准DNA元件,推动合成生物学的研究进程,缩短研发周期并降低研发成本。
为了使合成生物学像其他工程学设计那样,可以在电脑帮助的模拟设计下构建新的生物系统,英国学者创建了一个标准虚拟生物元件库(SVPs) ,为合成生物学提供模块化建模元件库。华盛顿大学的研究者创建了一个标准生物元件知识库(SBPkb),查询和检索生物元件用于合成生物学研究和应用 。SBPkb将标准生物元件知识库中所有元件的信息转换成可以利用合成生物学元件语义框架进行运算的信息。该框架称作为合成生物学公开语言语义(SBOL-semantic),是合成生物学公开语言的一部分,用于描述通用的合成生物学实体,其目的是提高所有生物元件的分散和交换。
Endy D. Foundations for engineering biology. Nature, 2005, 438: 449-453.
Benner S A, Sismour A M. Synthetic biology. Nat Rev Genet, 2005, 6 (7): 533-543.
Paddon C J, Keasling J D. Semi-synthetic artemisinin: a model for the use of synthetic biology in pharmaceutical development. Nature Reviews Microbiology, 2014, 12(5):355-367.
Ajikumar P K, Xiao W H, Tyo K E, et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science, 2010, 330:70-74.
Basu S, Gerchman Y, Collins C H, et al. A synthetic multicellular system for programmed pattern formation. Nature, 2005, 434: 1130-1134.
Lu T K, Collins J J. Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy. Proc Natl Acad Sci USA, 2009, 106: 4629-4634.
Purnick P E, Weiss R. The second wave of synthetic biology: from modules to systems. Nat Rev Mol Cell Biol, 2009, 10 (6): 410-422.
Shetty R P, Endy D, Knight T F. Engineering BioBrick vectors from BioBrick parts. J Bio Eng, 2008, 2: 5.
Kwok R. Five hard truths for synthetic biology. Nature, 2010, 463(7279):288-290.
与自然界巨大的生物元件资源相比,MIT标准生物学元件登记库中的现有库存仅仅是沧海一粟。现阶段如何鉴定与建立更多的通用性生物元件依然是合成生物学的重要任务 。2010年,我国“973”计划启动合成生物学重大项目,“新功能人造生物器件的构建与集成”、“抗逆元器件构建和机理研究”和“生物固氮与抗逆模块的人工设计与优化”等项目关注生物元器件的设计、构建与改造。生物元件可以从自然界分离出来,也可以对天然生物元件进行修饰、重组和改造后获得具有新功能的生物元器件。
1 新功能元器件的筛选与功能鉴定
1.1 基于基因组或转录组信息挖掘与功能鉴定生物元器件
生物元件主要来源于自然界,因此从自然界中分离是获得生物元件的主要途径之一。以植物重要次生代谢产物合成途径关键生物元件的筛选与功能鉴定为例,虽然合成生物学的核心在于设计,但是对于结构多样而合成途径未知的天然化合物,合成途径的设计还需要向自然界学习,从其在自然界中的生物合成途径中获得启发。在大规模基因组测序前,从自然界中分离生物元件主要是运用传统的分子克隆方法或基于保守序列的PCR方法。
近十年来,随着DNA测序技术效率的提高与成本的大幅下降,越来越多可生产重要天然产物的植物或微生物的基
Cooling M T, Rouilly V, Misirli G, et al. Standard virtual biological parts: a repository of modular modeling components for synthetic biology. Bioinformatics, 2010, 26(7):925-931.
Michal G, Cesar R, Deepak C, et al. Standard biological parts knowledgebase. PLoS One, 2011, 6(2): e17005.
Canton B, Labno A, Endy D. Refinement and standardization of synthetic biological parts and devices. Nat Biotechnol, 2008, 26 (7):787-793.
Hezari M,Lewis N G,Croteau R.Purification and characterization of taxa-4(5),11(12)-diene synthase from Pacific yew(Taxus brevifolia) that catalyzes the first committed step of taxol biosynthesis.Archives of Biochemistry and Biophysics,1995,322(2):437-444
Zhou Y J,Gao W, Rong Q,et.al.Modular pathway engineering of diterpenoid synthases and the mevalonic zcid pathway for miltiradiene production,Journal of the American Chemical Society,2012,134:3234-3241.
Guo J, Zhou Y J, Hillwigc M L, et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(29):12108-12113.
Shang Y, Ma Y, Zhou Y, et al. Biosynthesis, regulation, and domestication of bitterness in cucumber. Science, 2014, 346:1084-1088.
Tansakul P, Shibuya M, Kushiro T, et al. Dammarenediol-II synthase, the first dedicated enzyme for ginsenoside biosynthesis, in Panax ginseng. FEBS Lett, 2006, 580:5143-5149.
Han J Y, Kim H J, Kwon Y S, et al. The Cyt P450 enzyme CYP716A47 catalyzes the formation of protopanaxadiol from dammarenediol-II during ginsenoside biosynthesis in Panax ginseng. Plant Cell Physiol, 2011, 52:2062-2073.因组或转录组获得解析。通过生物信息学预测,直接从基因组或转录组数据中获得各种的序列信息,为合成生物学提供丰富的信息资源。DNA或cDNA数据的积累为天然产物的生物合成途径的解析奠定了重要基础,但对许多合成途径更复杂的天然产物而言,它们的合成途径仅仅被鉴定了其中的几个步骤,仍然期待大量的研究投入。如抗癌药物紫杉醇在红豆杉中的合成极其复杂,从二萜化合物前体GGPP开始到终产物紫杉醇包含了19步酶促反应。1995年,Hezari M等克隆 并鉴定了催化GGPP生成紫杉二烯taxadiene的合成酶,揭示了第一步反应。经过多年的努力,各国科学家还解析了后续步骤中涉及的8个细胞色素P450、5个酰基/芳香基转移酶以及1个氨基酸变位酶,而剩余的5个基因仍有待研究 。另外,我国科学家在2009年基于cDNA芯片的功能基因组学挖掘获得了催化GGPP生成丹参酮前体次丹参酮二烯(miltiradiene)的合成酶SmCPS和SmKSL,2013年又解析了催化次丹参酮二烯到弥罗松酚(ferruginol)的细胞色素P450 CYP76AH1。但最终生成丹参酮还涉及多步未知的酶促反应。最近,我国科学家在解析黄瓜中的三萜化合物葫芦素的合成途径也取得了重大进展 。结合基因组学和生物化学方法,解析了葫芦素碳合成途径中的9个编码基因并鉴定了其中4个酶的功能(包括催化第一步反应的葫芦二烯醇合酶Bi、中间步骤涉及的2个细胞色素P450以及最后步骤的乙酰基转移酶),其他基因的功能仍待鉴定。
基于人参cDNA数据的分析,韩国与日本科学家首先于2006年克隆并鉴定了达玛稀二醇合酶(DDS) ,2011年韩国科学家克隆与鉴定细胞色素P450 (CYP716A47)是催化达玛稀二醇转化为原人参二醇(PPD)的关键酶 ,2012年又进一步鉴定了催化原人参二醇产生原人参三醇(PPT)的细胞色素P450(CYP716A53v2) ,但这些细胞色素P450的还原酶以及将PPD和PPT转化为人参皂苷的糖基转移酶尚没有被报道。虽然国内外研究机构已经公开了13个人参属植物(包括人参、西洋参和三七)的转录组数据,但由于采用第二代测序技术(如solexa),获得的读长为80~150bp,传统的方法仅能拼接到少数的基因全长序列。中国科学院合成生物学重点实验室 采用短reads的优化拼接方法,大大提高了拼接基因的序列长度及完整ORF的比例。从人参属植物的cDNA数据的重拼接数据中,获得512个UDP-糖基转移酶基因(长度>1000bp),并通过设计引物从人参cDNA样品中克隆了其中80多个UDP-糖基转移酶基因。同样的方法,还克隆了2个烟酰胺腺嘌呤二核苷酸(NADPH)-细胞色素P450还原酶,功能鉴定表明这2个细胞色素P450还原酶与CYP716A47或/和CYP716A53v2可以高效地催化DM或/和PPD的羟基化反应(PCT/CN2015/073550)。
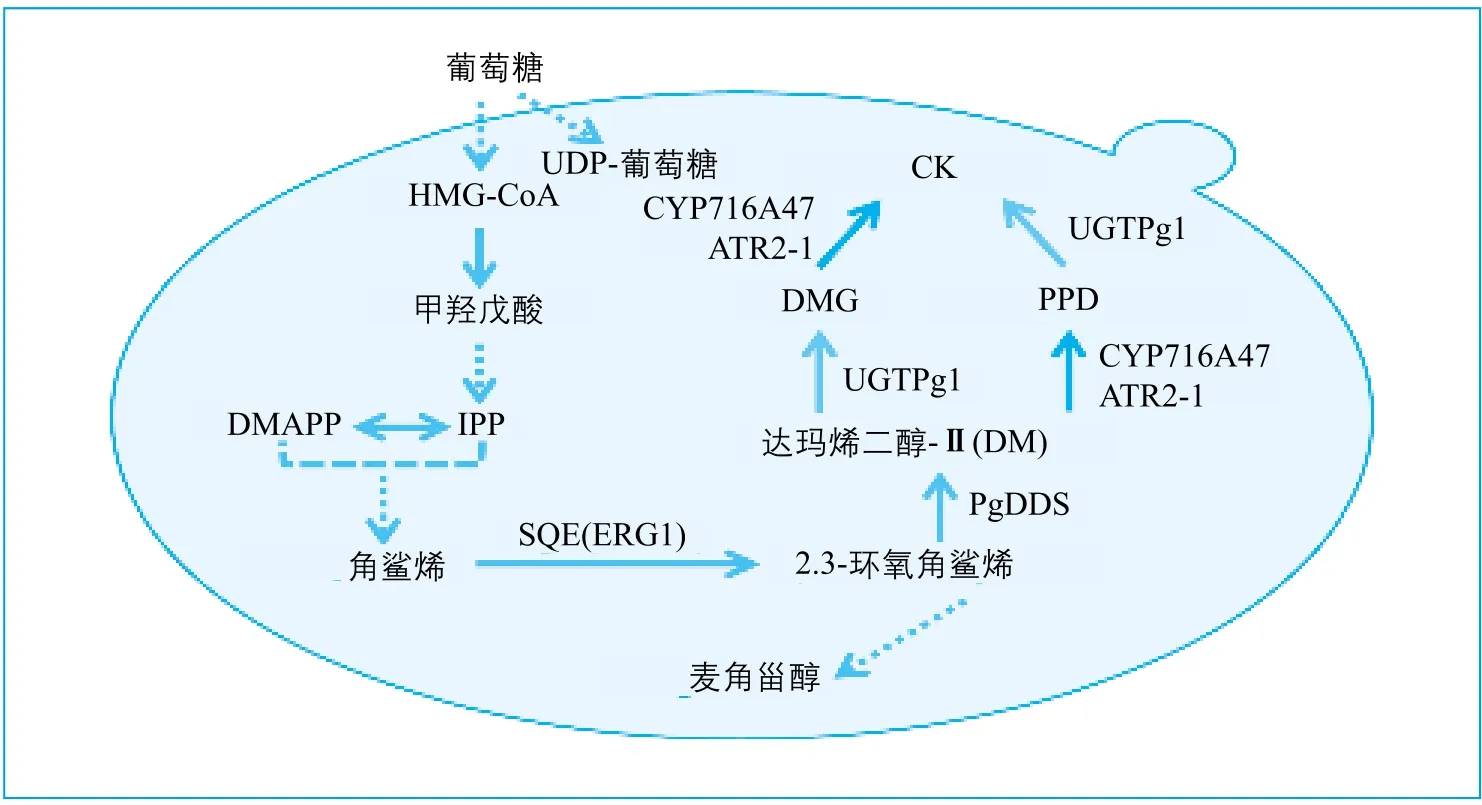
图1 基于人参UDP-糖基转移酶的筛选与鉴定确定了人参皂苷CK两条潜在合成途径
Han J Y, Hwang H S, Choi S W, et al. The Cyt P450 enzyme CYP716A47 catalyzes the formation of protopanaxadiol from dammarenediol-II during ginsenoside biosynthesis in Panax ginseng. Plant Cell Physiol, 2012, 53:1535-1545.
Wei Y, Zhou H, Zhang J, et al. Insight into dominant cellulolytic bacteria from two biogas digesters and their glycoside hydrolase genes. PLoS One, 2015, 10(6):e0129921.
Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast. Cell Research, 2014, 24:770-773.
Wang P, Wei Y, Fan Y, et al. Production of bioactive ginsenosides Rh2 and Rg3 by metabolically engineered yeasts. Metab Eng, 2015, 29: 97-105.
We i W, Wa n g P, We i Y, e t a l. Characterizations of Panax ginseng UDP-glycosyltransferases catalyzing protopanaxatriol and biosyntheses of bioactive ginsenosides F1 and Rh1 in metabolically engineered yeasts. Molecular Plant, 10.1016/j.molp.2015.05.010.
Niu G, Li L, Wei J, et al. Cloning, heterologous expression, and characterization of the gene cluster required for gougerotin biosynthesis. Chemistry & Biology, 2013, 20(1):34-44.
Wei J, Tian Y, Niu G, et al. GouR, a TetR-family transcriptional regulator, coordinates the biosynthesis and export of gougerotin in Streptomyces graminearus. Applied and Environmental Microbiology, 2014, 80(2): 714-722.
Du D, Zhu Y, Wei J, et al. Improvement of gougerotin and nikkomycin production by engineering their biosynthetic gene clusters. Applied Microbiology and Biotechnology, 2013, 97(14): 6383-6396.
笔者实验室表达了从人参中克隆得到的80多个UDP-糖基转移酶基因并获得了65个表达产物(UDP-糖基转移酶的初提物),确定了其中20个UDP-糖基转移酶在糖基供体UDP-葡萄糖存在的条件下对已知的人参皂苷前体或皂苷具有转糖基催化作用~(PCT/ CN2013/088819),其他UDP-糖基转移酶的底物(糖基受体)与糖基供体还有待进一步研究。对这20个UDP-糖基转移酶的酶学特性进行分析后,发现这些糖基转移酶具有较强位点(或空间)特异性~(图1)。
UGTPg1和UGTPg45分别特异性催化原人参二醇及其皂苷的C-20S、C-3位的羟基糖基化 ,UGTPg100特异性催化原人参三醇及其皂苷的C-6位的羟基糖基化 ,UGTPg29则可以进一步在Rh2的3位糖基上再延伸一个葡萄糖基生成人参皂苷Rg3。笔者实验室还发现了一些氨基酸序列相似性较高的糖基转移酶,其具有不同功能或活性,基于这些酶的序列比对及定点突变分析,鉴定了多个决定糖基转移酶活性或底物特异性的关键氨基酸残基 。
1.2 通过基因组文库克隆与基因簇的分析功能鉴定生物元器件
链霉菌是天然产物的重要来源之一,参与这些次级代谢产物合成的基因一般以成簇的形式排列,对这些基因的克隆与鉴定常常依赖于基因组文库的构建。禾粟链霉菌(Streptomyces graminearus)产生的核苷肽类抗生素谷氏菌素,具有抗细菌、支原体及抗病毒和肿瘤等活性。中国科学院微生物研究所牛国清副研究员基于HPLC、MS和NMR确定了谷氏菌素的化学结构,并通过筛选禾粟链霉菌fosmid文库克隆了完整的谷氏菌素生物合成基因簇,并在异源的底盘链霉菌中成功表达该基因簇。通过功能鉴定,已经确定生物合成基因簇中含有1个调控基因gouR和14个结构基因(gouA-N),通过体内遗传(基因敲除与互补)和中间积累产物的化学分析,对部分结构基因的功能进行了鉴定。基于这些实验结果和生物信息学分析,其生物合成途径推测如下:胞嘧啶首先与尿苷二磷酸葡糖醛酸(UDP-glucuronic acid)相连形成胞嘧啶葡糖醛酸 (cytosylglucuronic acid,CGA),CGA经氧化、胺化形成4氨基CGA;随后活化的丝氨酸和肌氨酸依次与4氨基CGA相连,形成云南霉素;云南霉素的葡糖醛酸C-6位经氨化形成谷氏菌素(图2)。此外,对调控基因gouR进行深入研究,发现GouR通过协调谷氏菌素的合成和外排调控谷氏菌素的产生~。
1.3 利用元基因组技术挖掘与功能鉴定新功能生物元器件
Ettwing K F, Butler M K, Le Paslier D, et al. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria. Nature, 2010, 464: 543-548.
Chu X, He H, Guo C, et al. Identification of two novel esterases from a marine metagenomic library derived from South China Sea. Appl Microbiol Biotechnol, 2008, 80:615-625.
Jeon J H, Kim J T, Kim Y J, et al. Cloning and characterization of a new cold-active lipase from a deep-sea sediment metagenome. Appl Microbiol Biotechnol, 2009, 81:865-874.
Waschkowitz T, Rockstroh S, Daniel R. Isolation and characterization of metalloproteases with a novel domain structure by construction and screening of metagenomic libraries. Appl Environ Microbiol, 2009, 75:2506-2516.
Simon C, Herath J, Rockstroh S, et al. Rapid identification of genes encoding DNA polymerases by function-based screening of metagenomic libraries derived from glacial ice. Appl Environ Microbiol, 2009, 75:2964-2968.
Geng A, Zou G, Yan X, et al. Expression and characterization of a novel metagenome-derived cellulase Exo2b and its application to improve cellulase activity in Trichoderma reesei. Applied Microbiology and Biotechnology, 2012, 96(4):951-962.
Yan X, Geng A, Zhang J, et al. Discovery of (hemi-)cellulase genes in a metagenomic library from a biogas digester using 454 pyro-sequencing. Applied Microbiology and Biotechnology, 2013, 97:8173-8182.
随着微生物分子生态学技术的发展,使人们认识到自然界中的大部分微生物是不可培养的,可培养的微生物仅占自然界中实际存在微生物的1%,这些未培养微生物中必然蕴藏着巨大的生物元件资源。近年发展起来的元基因组技术,可以让人们走出纯培养微生物的局限,从自然界,特别是极端环境中获得更丰富的生物资源,包括合成生物学所需要的各种生物元件。利用元基因组
Taylor A I, Pinheiro V B, Smola M J, et al . Catalysts from synthetic genetic polymers. Nature, 2015, 518(7539):427-430.
Xiong P, Wang M, Zhou X, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability. Nature Communications, 2014, 5:5330.
de Graef M R, Alexeeva S, Snoep J L, et al. The steady-state internal redox state (NADH/NAD) reflects the external redox state and is correlated with catabolic adaptation in Escherichia coli. J Bacteriol, 1999, 181: 2351-2357.
Mampel J, Buescher J M, Meurer G, et al. Coping with complexity in metabolic engineering. Trends Biotechnol, 2013, 31: 52-60.
Ji D B, Wang L, Hou S H, et al. Creation of bioorthogonal redox systems depending on nicotinamide flucytosine dinucleotide. J Am Chem Soc, 2011, 133: 20857-20862.
Ji D B, Wang L, Liu W J, et al. Synthesis of NAD analogs to develop bioorthogonal redox system. Sci Chin Chem, 2013, 56:296-300.
Hou S H, Ji D B, Liu W J, et al. Identification of malic enzyme mutants depending on 1,2,3-triazole moiety-containing nicotinamide adenine dinucleotide analogs. Bioorg Med Chem Lett, 2014, 24:1307-1309.
王磊. 基于烟酰胺胞嘧啶二核苷酸的代谢电路研究. 中国科学院大连化学物理研究所博士学位论文, 2014.技术,一方面通过测序可以获得未培养生物中的基因信息,获得许多未知的生物元件信息;另外一方面可以通过功能筛选,获得许多具有各种用途的功能基因。如对河底沉积物的富集培养物元基因组测序后,找到了以NO2-为电子受体来氧化甲烷的新代谢途径 。目前各国科学家利用元基因组技术,从未培养生物中分离获得许多有重要工业应用价值的酶基因,如酯酶基因 、脂肪酶基因 、蛋白酶基因 ,以及从冰川冰的元基因组文库中筛选到的DNA聚合酶I基因 。事实证明,从环境中未培养微生物的元基因组中,基于序列筛选或元基因组文库的功能筛选,可以挖掘到大量新的生物元件。
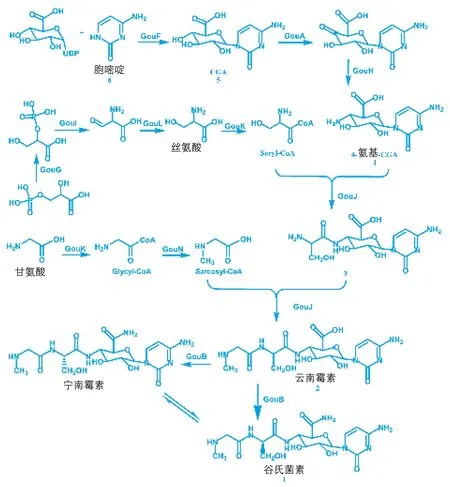
图2 禾粟链霉菌中谷氏菌素的生物合成途径
中国科学院合成生物学重点实验室在环境微生物元基因组研究过程中建立了基于(元)基因文库的糖基水解酶功能筛选平台 ,并以454焦磷酸测序技术获得的两个读长较短(约230bp)的厌氧沼气发酵微生物群落元基因组数据为研究对象,利用优化的短序列拼接方法对上述元基因组454短序列进行拼接,得到了167个较长的含有木质纤维素酶基因的contigs(>1kb),其代表了整个厌氧沼气发酵群落中优势的糖基水解酶。在此基础上,设计引物验证了其中50个contigs拼接的准确性,并利用这些引物对沼气发酵群落的fosmid文库进行了筛选,找到了含有这些contigs的阳性克隆。对11个阳性克隆进行测序,发现有些fosmid可以被拼接成更大的contig(Contig76N21和Contig76E20的长度分别达到80kb和60kb) 。在这些大的contig中,发现了含有多个纤维素降解相关基因的基因簇。如在Contig76N21和Contig76E20中,除了纤维素降解酶之外,还有一些调节纤维素酶基因表达的调控因子和编码ABC转运蛋白的基因成簇排列,这些大的基因簇应该来源于发酵群落中优势的纤维素菌,可能在纤维素的高效降解过程中发挥重要作用 。同时,通过这些优势纤维素酶基因的分析,仅发现小部分基因编码形成纤维小体所需的dockerin结构域,这说明所研究的厌氧沼气发酵群落中纤维小体并不是优势的纤维素降解系统。此外,GH5家族是纤维素降解中很重要的一种糖基水解酶,对拼接得到的4个具有全长序列的GH5家族基因在大肠杆菌中进行了表达。其中Contig8188-GH5编码的糖基水解酶具有较高的甘露糖酶酶活(2625U/mg蛋白),其最适温度和最适pH分别为60℃和 6.0,因此具有一定的工业应用潜力。这种优化的序列拼接方法也同样适用于高通量测序仪solexa等来源的短读长元基因组reads(150~300bp)的拼接,适用于从各种环境中挖掘天然产物合成基因簇。
2 生物元件的设计与改造
人工设计与合成或改造生物元件将为合成生物学提供物质基础与新思路。通过数学模型预测、设计生物器件或生物系统,是合成生物学的核心目标。为实现这一目标,有必要对现有生物元件的结构与功能进行优化与改造。通过解析单个功能元件以及互作的功能元件复合体的分子结构,揭示生物分子的相互作用模式,特别是负责催化与调控的蛋白质元件以及这两者之间的结构与功能的关系。在此基础上优化改造各种功能元件,使设计的生物器件或系统达到最优。2014年12月1日,《Nature》杂志公布了剑桥大学研究团队的最新研究成果。研究人员利用人造遗传物质合成出世界上第一个人工酶,合成的酶(XNAzymes)自然界中并不存在,但可以在实验室中引起化学反应 。2014年11月,《Nature Communications》杂志发表了中国科学技术大学生命科学学院教授刘海燕研究组在蛋白质分子设计方法方面的研究成果 。蛋白质是由不同类型的氨基酸按照特定顺序串联成的长链状生物大分子,很多蛋白质分子需要在空间折叠成稳定的三维结构才能发挥功能。目前能够折叠成稳定三维结构的蛋白质几乎都是天然蛋白质,其氨基酸序列是自然进化形成的。而蛋白质设计是人工指定蛋白质的氨基酸序列,使其具有预期的三维结构和功能。刘海燕研究组建立了一种全新的统计能量函数用于蛋白质设计,不仅检测效率高,还能通过实验筛选对设计做出改进。基于此方法,研究组以3种不同天然蛋白质的空间结构为设计目标,获得了4个稳定折叠的人工蛋白质。这种蛋白质从头设计的新途径,可以保证设计的蛋白质空间结构与预定目标高度吻合,为蛋白质结构功能的设计与改造提供了新工具。
Hasty J, McMillen D, Collins J J. Engineered gene circuits. Nature, 2002, 420 (14): 224-230.
Michalowski C B, Short M D, Little J W. Sequence tolerance of the phage PRM promoter: implications for evolution of gene regulatory circuitry. J Bacteriol, 2004, 186: 7899-7999.
Elowitz M B, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403: 335-338.
Basu S, Mehreja R, Thiberge S, et al. Spatiotemporal control of gene expression with pulse-generating networks. Proc Natl Acad Sci USA, 2004, 101(17): 6355-6360.
Chang D T H, Huang C Y, Wu C Y, et al. YPA: an integrated repository of promoter features in Saccharomyces cerevisiae. Nucleic Acids Res, 2011, 39:D 647-652.
Hammer K, Mijakovic I, Jensen P R. Synthetic promoter libraries-tuning of gene expression. Trends Biotechnol, 2006, 24(2):53-55.
Hartner F S, Ruth C, Langenegger D, et al. Promoter library designed for fine-tuned gene expression in Pichia pastoris. Nucleic Acids Res, 2008, 36 (12): e76.
Qin X L, Qian J C, Yao G F, et al. GAP promoter library for fine-tuning gene expression in Pichia pastoris. Appl Environ Microbiol, 2011, 77(11):3600-3608.
2.1 人工氧化还原体系的设计、构建和功能鉴定
氧化还原反应是最常见的代谢反应类型之一,其中绝大部分通过辅因子依赖型氧化还原酶催化实现。由于辅因子还广泛参与胞内其他生物学过程,人工代谢途径改造扰动辅因子水平的生物学效应尚难以预测 。设计构建基于辅因子的正交体系,是避免人工代谢体系与天然代谢体系相互干扰、降低系统复杂度、提高调控代谢网络有效性的新策略 。中国科学院大连化学物理研究所赵宗保课题组 在前期研究生物正交氧化还原体系所取得成果的基础上,拓展NAD类似物的结构空间,并以与萜类或聚酮合成代谢相关的氧化还原酶为目标,创建依赖于NAD类似物的氧化还原代谢步骤,从而为选择性调控氧化还原代谢提供新元件和新工具。课题组完成了22个NAD类似物的化学合成和结构鉴定(图3),测定了部分类似物与苹果酸酶突变体ME L310R/Q401C以及ME-L310K/ L404S的活性,发现类似物NBrCD、NMeCD和NClCD等可与苹果酸酶突变体形成较好的活性配对 。
亚磷酸脱氢酶(PTDH)催化亚磷酸转化为磷酸同时将NAD还原为NADH,该反应在水溶液中不可逆。因此,利用PTDH有可能为细胞提供一种新的还原力(能量)供给途径。笔者课题组全合成了来源于Pseudomonas stutzeri WM88 的PTDH基因,将其克隆到载体pET24b 和pUC18。依据PTDH与NAD结合的特点,选择构建了系列双位点饱和突变体表达文库N145/M153、Q172/K177、A212/L217、I150/M153、E175/K177 和T214/L217,筛选得到可有效利用NCD的突变体PTDH(L151V/D213C/ T214P/L217P)。将来源于Ralstonia sp. R4506的PTDH基因克隆到载体pUC18,得到表达质粒pUC-R4506 (Amp抗性,Lac启动子)。构建了PTDHR4506(I151R)系列饱和突变体表达文库,筛选获得突变体PTDHR4506(I151R/E213D/I218F),其利用NCD的偏好性比野生型PTDH提高了1600倍,为高效、高选择性产生NCDH奠定了基础。
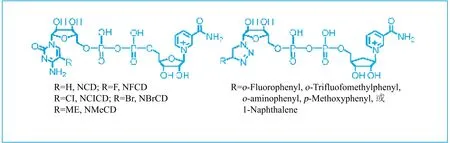
图3 NAD类似物的合成
以E. coli为宿主,在其胞内表达核苷转运蛋白AtNDT2、可利用NCD的突变酶ME L310R/Q401C和PTDH L151V/D213Q,构建了模型能量传递调控体系。当培养基中存在亚磷酸、NCD和亚磷酸盐时,工程菌株能氧化利用亚磷酸盐产生NCDH,推动胞内丙酮酸还原羧化生产苹果酸,苹果酸产量提高35%(图4)。这是世界首次实现耦合胞外能量源,选择性推动胞内物质代谢的研究实例 。这些结果表明,NAD类似物有可能作为非天然的能量载体,而与NAD类似物匹配的氧化还原酶可作为选择性控制能量传递的模块,在合成生物学研究中发挥独特的作用。
2.2 启动子改造和文库构建
启动子是合成生物学工程技术中非常重要的调控元件之一,各种遗传线路 、基因开关 、基因振荡 和细胞群体系统脉冲发生器 等功能的发挥大都需要通过启动子的调控来实现。同时,异源合成途径在底盘细胞中的适配性的优化也需要通过调节途径中关键酶的启动子强度来实现。目前,合成生物学常用的底盘细胞自身可用的启动子极其有限,因此需要对现有的启动子进行改造获得新的启动子。蛋白定向改造技术日益成熟,已经应用于启动子的改造。
通过定向改造技术,建立包括各种不同强度启动子的文库,是实现~基因精确调控的有利遗传工具。目前,已经对毕赤酵母和酿酒酵母中的启动子进行了改造,通过构建启动子文库,得到了一系列不同强度的启动子元件。在工程化的产番茄红素菌株的优化过程中,人们发现番茄红素产量与脱氧磷酸木酮糖酶合酶表达水平成线性增长关系:脱氧磷酸木酮糖酶合酶参与的反应是限速步骤,最大番茄红素的产量需要最适的脱氧磷酸木酮糖酶合酶表达水平 。因此,需要分析不同强度启动子下脱氧磷酸木酮糖酶合酶表达量与番茄红素产量的相关性。为此,研究者利用易错PCR方法对酿酒酵母的TEF1启动子进行改造,建立了突变启动子库,得到了一系列强度不同的突变启动子 。在此基础上,Nevoigt等 进一步对TEF1突变库的11个启动子特性进行了研究。研究表明,这些突变启动子的活性是野生型活性的8%~120%。
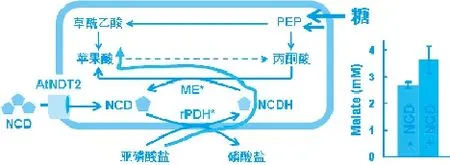
图4 模型能量传递调控体系
除了基因的组成型表达以外,建立一个易于控制、可进行条件诱导或抑制的基因表达盒也常应用于合成生物学研究与工程中。通过启动子的改造,得到具有特定调节特性的启动子将有望执行基因表达的复杂调控,并突破由于诱导剂的毒性、高价格和介导的多效性等影响诱导型启动子应用的限制。德国科学家应用随机突变和多级流式细胞筛选,改造与筛选酿酒酵母的氧响应性启动子DAN1的突变子,获得了两个在非严谨厌氧条件下可诱导的突变子。这使得酵母发酵中基因表达的诱导可以通过细胞生长过程中氧气的消耗来完成,而且,无论是在严格厌氧还是微厌氧环境下,突变启动子的表达量远远高于野生型启动子的DAN1。
利用模块化的组件构建具有可预测行为的工程化基因网络是合成生物学的主要目标之一 。然而,由于缺乏合适的组件和构建网络通常需要大量反复测试工作,使得构建预期功能的基因网络受到阻碍。美国波士顿大学学者发表了一种利用启动子文库结合杂交模型用于指导预测酿酒酵母基因网络构建的方法。利用芯片模型耦合了由多元化组件组成的文库,这些多元化组件是通过随机非必需序列合成的。他们在酿酒酵母中合成了调节启动子文库,并利用该文库构建具有各种预期输入输出特性的前馈环网络,通过该研究证实了上述方法的有效性。为了拓宽该方法的应用范围,他们合成了一个具有计时器功能的基因网络,并可通过组件的选择进行更改。运
Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering. Proc Natl Acad Sci USA, 2005, 102 (36): 12678-12683.
Nevoigt E, Kohnke J, Fischer C R, et al. Engineering of promoter replacement cassettes for fine-tuning of gene expression in Saccharomyces cerevisiae. Appl Environ Microbiol, 2006, 72 (8): 5266-5273.
Nevoigt E, Fischer C, Mucha O, et al. Engineering promoter regulation. Biotechnol Bioeng, 2007, 96(3):550-558.
Guido N J, Wang X, Adalsteinsson D, et al. A bottom-up approach to gene regulation. Nature, 2006, 439: 856-860.
Ellis T, Wang X, Collins J J. Diversitybased, model-guided construction of synthetic gene networks with predicted functions. Nat Biotechnol, 2009, 27(5): 465-471.
Cai Y, Wilson M L, Peccoud J. GenoCAD for iGEM: a grammatical approach to the design of standard-compliant constructs. Nucleic Acids Res, 2010, 38(8):2637-2644.
Rialle S, Felicori L, Dias-Lopes C, et al. BioNetCAD: design, simulation and experimental validation of synthetic biochemical networks. Bioinformatics, 2010, 26(18):2298-2304.用该基因网络控制酵母沉淀的时间,表明该设计的即插即用性质,可方便应用到生物技术产业中 。
随着合成生物学的发展,具有相似功能的生物元件会越来越多,那么如何对这些元件进行挑选,以达到系统或者装置的设计目标,是一个非常重要的问题。一般的方法,是利用这些元件分别合成目标序列,然后来比较它们的功能。但是,在目前的条件下,由于元件的数量越来越多,合成的步骤也越来越复杂,往往不可能针对所有候选元件进行实验 。因此,利用计算机辅助设计(computer-aided design,CAD)来帮助进行生物元件的选择,已成为合成生物学一个重要的研究方向。Ellis等 就是通过构建数学模型,然后根据计算结果来选择适当强度的启动子,从而合成了所需的基因回路,达到了预期的实验目标。目前,已经有一些计算机软件来帮助建立数学模型,然后根据生物元件的生物学数据(动力学参数和启动子强度等)来预测合成的系统是否符合设计的目标,从而帮助生物元件的选择,计算机辅助设计大大加快了合成生物学的研究速度 。
[本研究得到合成生物学“973”项目“新功能人造生物器件的构建与集成”课题3“新功能人造生物器件的构建”(2012CB721103)的资助。]
■ 反馈服务编码 W3618
10.3969/j.issn.1674-0319.2015.06.007

合成生物学与基因工程、代谢工程最显著的区别,在于能够将大量生物元器件进行快速、随意的组装,而实现这一目标的前提,是将生物元件或器件进行详细地功能表征与标准化。目前合成生物学面临的主要挑战之一,是可用的生物元件和模块(器件)种类局限、数量不足、表征描述不清楚、通用性程度不高。为此,有必要在功能元件的挖掘、表征、设计、改造与标准化方面开展系统研究。文章对重要天然产物合成途径的生物元件进行了挖掘与功能表征,并对氧化还原代谢的功能元件进行了设计与改造。
合成生物学是按照一定的规律和已有的信息设计和构建新的生物元件、装置和系统,或者重新设计已有的天然生物系统(http://syntheticbiology. org/)。合成生物学的核心在于设计、改造、重建或制造生物器件、生物反应系统、代谢途径与过程,乃至创造具有生命活动能力的细胞和生物个体,为生命起源、生物进化、复杂疾病、干细胞等生物学难题的研究开拓新的蹊径,为解决人类
