hsa-miR-342-3p生物信息学分析
2015-02-23马占兵焦海燕霍正浩
马占兵,耿 芝,焦海燕,2,霍正浩,2*
(1.宁夏医科大学基础医学院医学遗传系与细胞生物学系,银川 750004;
2.宁夏回族自治区生殖与遗传重点实验室, 银川 750004)
hsa-miR-342-3p生物信息学分析
马占兵1,2,耿 芝1,焦海燕1,2,霍正浩1,2*
(1.宁夏医科大学基础医学院医学遗传系与细胞生物学系,银川 750004;
2.宁夏回族自治区生殖与遗传重点实验室, 银川 750004)
摘要:通过生物信息学方法预测hsa-miR-342-3p靶基因及其功能机制。检索PubMed 有关hsa-miR-342-3p的研究报道并进行功能分析;检索miRBase获取hsa-miR-342-3p序列;通过TargetScan, Pictar 和PITA数据库预测靶基因并取交集,对其进行组织和疾病特异性表达谱分析、功能富集分析(GO enrichment analysis)、信号转导通路富集分析(Pathway enrichment analysis)和蛋白质相互作用网络分析(PPI analysis)。结果发现: hsa-miR-342-3p序列在多物种间具有高度保守性; hsa-miR-342-3p在肾脏组织和急性淋巴细胞性白血病、乳腺癌疾病中表达水平较高(RPM≥1 000);预测得到14个hsa-miR-342-3p靶基因;靶基因分子功能分别富集于转化生长因子活性、DNA结合和蛋白激酶激活等(P<0.05); hsa-miR-342-3p靶基因GO生物学过程主要集中于大分子代谢抑制,肺部组织发育、呼吸系统发育及管状组织发育建成(P<0.05);细胞信号通路主要富集于TGF-Beta信号通路、细胞因子、受体作用信号通路及前列腺疾病信号通路(P<0.01)。hsa-miR-342-3p在体内分布广泛,预测的靶向TGF-Beta信号通路可能在疾病发生中发挥重要调控作用,是具有潜在研究价值的生物学靶标。
关键词:hsa-miR-342-3p;靶基因;基因本体;信号通路;生物信息学
微小RNA(microRNA,miRNA)是一类长度约在22个核苷酸(nt)左右,具有转录后水平调控功能的内源性非编码单链小RNA分子[1]。研究证明,miRNA在转录或转录后水平上调节一部分信号分子参与细胞发育、分化、增殖凋亡和新陈代谢等多种生理过程[2]。miRNA-342-3p是一个被为广泛研究的微小RNA分子,通过文献分析表明其主要在腺癌、肝癌、肺癌、乳腺癌及结肠癌等癌症中发挥抑癌作用[3-6]。hsa-miR-342-3p分子可能与细胞周期调控、DNA损伤修复等有关,但其具体作用靶标和信号通路,尚待深入研究。目前,miRNA靶标预测算法均需要运用到序列互补配对特性,热力学稳定性,及进化保守性等特征,联合使用不同算法的靶标预测数据库交叉预测可有效降低预测的假阳性率[7]。miRNA作用功能复杂,单一miRNA可有多个靶基因,目前研究预测每个 miRNA 将可能会调控 200 个左右的基因,而多个miRNAs也可交叉调控单一靶标。因此,有效预测miRNA靶基因并对其进行系统生物学分析是研究其作用机制的重要环节之一[8]。基于多算法交叉的靶标预测,结合靶标基因组织表达特异性等信息,可快速而有效地获取靶基因信号通路和调控网络等重要信息,并有可能挖掘得到潜在的生物学功能信息。本研究通过靶标预测,数据整理和信息分析,获得相关重要信息,以期为进一步验证hsa-miR-342-3p功能提供数据支持,为探索hsa-miR-342-3p在癌症中的作用机制和生物学功能提供可能的理论依据。
1资料与方法
1.1 hsa-miR-342-3p特异性表达谱分析
利用Human MiRNA Expression Database (http://bioinfo.life.hust.edu.cn/smallRNA/index.php)在线工具检索获得hsa-miR-342-3p在不同组织和疾病中的RPM(Reads Per Million reads)数据,获得hsa-miR-342-3p在不同组织和疾病中特异性表达丰度信息,为靶基因组织和疾病定位及功能研究提供更多信息。
1.2 靶基因数据集获得
利用 miRBase (http://www.mirbase.org/) 在线工具获得hsa-miR-342-3p碱基序列、染色体定位和不同物种序列比对信息。分别使用TargetScan (http://www.targetscan.org)、Pictar(http://pictar.mdc-berlin.de/)和PITA(http://genie.weizmann.ac.il/)三种数据库获得hsa-miR-342-3p靶基因预测结果,提取靶基因GenBank ID或基因Symbol,并将其统一映射为GenBank ID,获取三种预测结果的交集作为进一步分析的靶基因数据集。
1.3 靶基因功能及表达分析
检索PubMed 关于hsa-miR-342-3p的相关研究,同时,提交靶基因至GeneCards(http://www.genecards.org/)数据库,获取靶基因功能信息。使用Human-Bodymap (http://www.ebi.ac.uk/arrayexpress/experiments/E-MTAB-513/)在线数据库获得靶基因在疾病和组织中的特异性表达信息。
1.4 富集分析
基因本体GO(Gene Ontology)和代谢通路KEGG富集分析采用Bioconductor GOstats包完成[9]。GOstats作为Bioconductor项目重要的基因功能注释和富集软件,可完成基因产物所有的注释,也可通过查询单一GO术语得到具有这个注释的所有基因产物。通过该软件获得靶基因的GO号及其层次分类注释信息,包括细胞组分(Cellular component), 细胞分子功能(Molecular function, MF)和生物学过程(Biological process, BP)等。利用KEGG数据库完成通路富集分析,对预测出的相关靶基因进行信号通路富集分析,并计算OR值和P值。
1.5 蛋白质互作网络分析
将预测靶蛋白名称列表提交至STRING(http://string-db.org/)蛋白质互作分析数据库[10],选择物种为HumanSpecies,其它参数默认,执行蛋白质相互作用网络分析。
1.6 统计学分析
GO分析以高频蛋白编码基因为背景基因,采用超几何分布方法计算P值,以P<0.05为相对于背景具有统计学意义阈值,得到基因集合在GO类别上的富集信息。KEGG富集的P值采用DAVID数据库附带的Fisher Exact Test软件计算P值,以P<0.05为显著性阈值,得到基因集合相对于背景具有统计学意义的信号通路。上述统计分析和计算均在Bioconductor3.0[11]平台上完成。
2结果分析
2.1 hsa-miR-342-3p同源性分析
人类成熟hsa-miR-342-3p序列号为MIMAT0000753,位于14q32.2。通过对小鼠、大鼠、黑猩猩等5个哺乳物种的miR-342-3p序列分析,发现人类成熟hsa-miR-342-3p分子“42-ucucacacagaaaucgcacccgu-63”23个碱基在各个物种间高度保守,见表1。
表1不同物种中hsa-miR-342-3p的保守序列
Table 1Conserved sequence of hsa-miR-342-3p in different species

物种miRNA名称保守序列人hsa-miR-342-3p42-ucucacacagaaaucgcacccgu-63小鼠mmu-miR-342-3p61-ucucacacagaaaucgcacccgu-83大鼠rno-miR-34261-ucucacacagaaaucgcacccgu-83黑猩猩ptr-miR-34260-ucucacacagaaaucgcacccgu-82猕猴mml-miR-34261-ucucacacagaaaucgcacccgu-83
2.2 hsa-miR-342-3p表达谱结果
Jing Gong 等[12]研究指出,人体不同组织样本和疾病中大约70%的成熟 miRNAs 低表达或不表达 (RPM<1), 只有大约9% 的已知 miRNAs 处于相对高水平表达 (RPM≥100)。如图1所示,利用HMED数据库检索,发现hsa-miR-342-3p在很多不同组织和疾病样本中都高表达,特别是在急性淋巴细胞性白血病(ALL)、乳腺肿瘤(Breast tumor)及肾脏(Kidney)表达水平较高(RPM≥1 000)。

图1 hsa-miR-342-3p 在不同组织和疾病中的平均表达水平Fig.1 Average expression level of hsa-miR-342-3p gene in different tissues and diseases
2.3 hsa-miR-342-3p靶基因预测
采用miRNA靶基因数据库TargetScan、Pictar和PITA预测hsa-miR-342-3p靶基因,结果分别得到155、55和1 950个靶基因,取交集后共得到14个靶基因,如图2所示, 靶基因信息如表2所示。

图2 不同数据库预测所得靶基因交集Fig.2 Intersection of prediction target genes using different database

序号GENBANK登录号基因名称基因官方名称1NM_000945proteinphosphatase3(formerly2B),regulatorysubunitB,alphaisoformPPP3R12NM_001005609ectodysplasinAeda3NM_001204bonemorphogeneticproteinreceptor,typeII(serine/threoninekinase)bmpr24NM_001429E1Abindingproteinp300EP3005NM_001546inhibitorofDNAbinding4,dominantnegativehelix-loop-helixproteinid46NM_002442musashihomolog1(Drosophila)MSI17NM_003339ubiquitin-conjugatingenzymeE2D2(UBC4/5homolog,yeast)ube2d28NM_006206platelet-derivedgrowthfactorreceptor,alphapolypeptidepdgfra9NM_006549calcium/calmodulin-dependentproteinkinasekinase2,betacamkk210NM_017519ATrichinteractivedomain1B(SWI1-like)ARID1B11NM_019106septin3SEPT312NM_033296Mof4familyassociatedprotein1Mrfap113NM_173822familywithsequencesimilarity126,memberBFAM126B14NM_182398ribosomalproteinS6kinase,90kDa,polypeptide5RPS6KA5
2.4 靶基因功能研究
使用Human-Bodymap测序数据平台,将靶基因名称列表提交至服务器,分析得到靶基因在人体不同组织的特异表达谱。如图3所示,SEPT3基因只在脑、淋巴结和睾丸中表达,且在脑中高表达;MSI1基因只在脑、结肠、卵巢、前列腺和睾丸组织中表达;EDA基因在脑组织、白细胞、肝脏和肌肉中不表达;RPS6KA5基因在脂肪组织、肾脏、肝脏中不表达。ID4在白细胞中不表达;MRFAP1、UBE2D2、PPP3R1、BMPR2、EP300、CAMKK2、ARID1B、FAM126B基因具有广谱性表达特性。
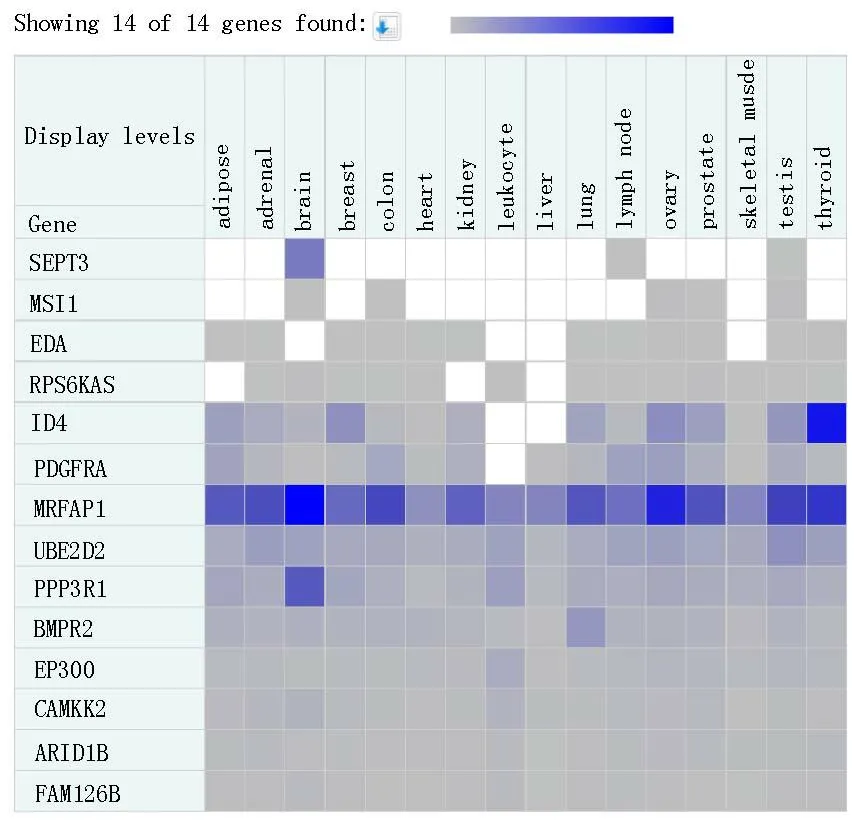
图3 靶基因在正常人体组织的特异性表达分布Fig.3 Specific expression of target genes in normal human tissues
2.5 GO富集分析
将上述14个靶基因的Gene IDs分别投射至GO两大应用功能上,成功显著映射到8个GO术语上,结果发现其中14个基因全部具有GO分子功能注释信息,其功能主要富集于转化生长因子活性、DNA结合、核染色质结合以及蛋白激酶活性等(P<0.005);14个基因全部具有生物学过程注释信息,显著富集于肺部发育、呼吸系统发育、管状组织发育及正向调控生物大分子代谢等(P<0.005),结果分别见表3和表4。

表3 预测靶基因(部分)GO分子功能分类结果(P<0.005)
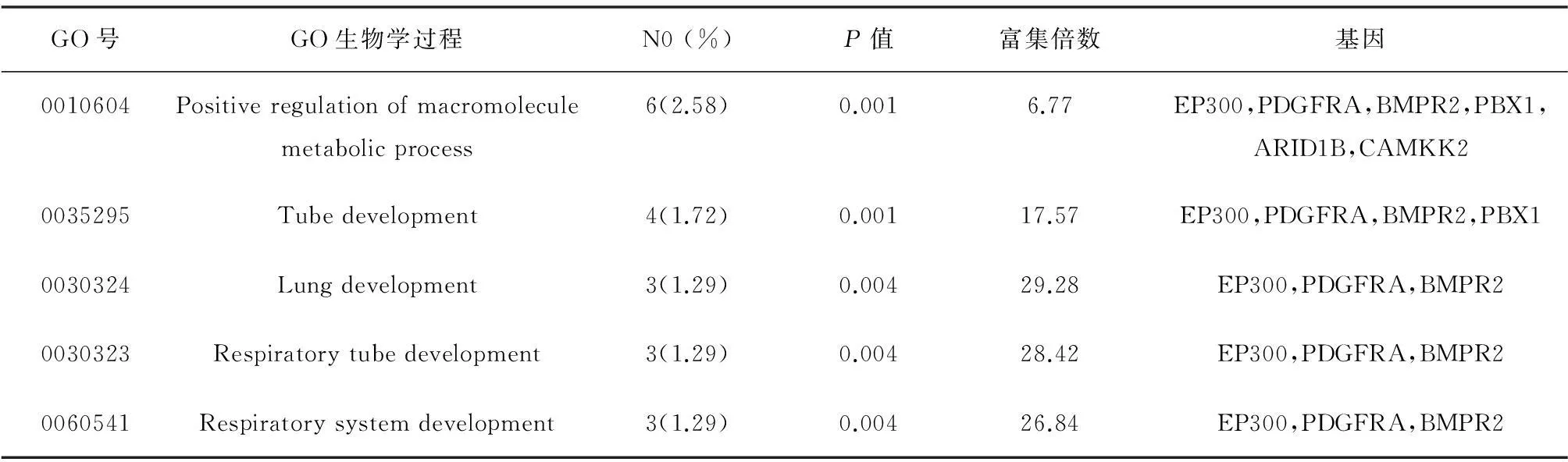
表4 预测靶基因GO生物学过程分类结果(P<0.005)
2.6 代谢通路富集分析
在GO注释分类的基础上,利用已有生物通路数据,对基因集合中的14个基因进行生物通路富集分析。如图4结果显示,在经典通路数据库KEGG中hsa-miR-342-3p预测靶基因集合显著富集于TGF-beta信号通路(P=0.000),可能由膜上BMPRII受体介导信号转导,下游通路主要有ID4蛋白负调控核转录因子及由EP300蛋白完成细胞周期G1期调控。此外,还显著富集于细胞因子及受体相互作用网络(P=0.006)以及前列腺癌疾病通路(P=0.008),结果见表5。
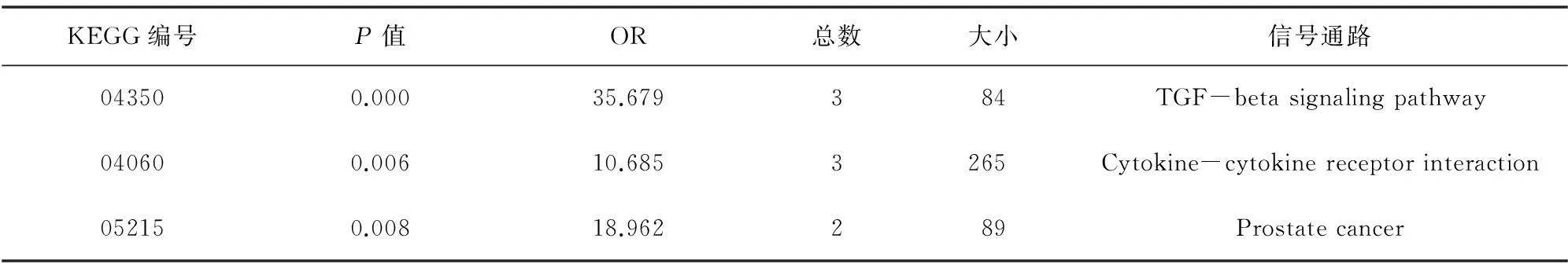
表5 预测靶基因KEGG 通路数据库富集分析结果(P<0.05)

图4 KEGG显著富集的信号通路Fig.4 Significant enrichment of signal pathway using KEGG
2.7 蛋白质相互作用网络分析
预测得到hsa-miR-342-3p靶基因总数14个,根据STRING蛋白质相互作用数据的信息可以构建蛋白质互作网络,根据该网络拓扑中度的重要性对靶基因进行排序。其中排在前五位的分别是“PPP3R1”,“PDGFRA”,“MSI1”,“RPS6KA5”,“EP300”。结果如图5所示。

图5 靶基因蛋白质互作网络Fig.5 Interaction network of target protein
3讨论
miRNA在许多生物学过程中发挥重要调控作用,人类三分之一基因由其调控,特别的,miRNA表达谱异常与癌症疾病发生密切相关[13-14]。hsa-miR-342-3p序列在多物种间高度保守,且在多种组织和疾病中广谱表达,提示其可能具有广泛的生物学功能。采用信息学方法,系统分析靶标基因功能和信号通路,对丰富和深入研究hsa-miR-342-3p疾病调控机制具有重要意义。
本文交叉预测得到hsa-miR-342-3p靶基因总共有14个,尚未见文献报道证实其相互作用。有关hsa-miR-342-3p表达谱研究已表明其在乳腺癌、肾癌等癌症中高表达。hsa-miR-342-3p靶基因GO注释主要富集在转化生长因子活性、DNA结合、蛋白激酶活性等分子功能调控和呼吸系统发育、管状组织建成等生物学过程中。KEGG可对某一miRNA诸多靶基因所涉及信号通路进行整合富集分析,本研究KEGG通路分析发现hsa-miR-342-3p靶基因功能显著富集于TGF-β信号通路、细胞因子信号通路以及前列腺癌疾病通路上。上述结果提示hsa-miR-342-3p不仅在脉管组织器官发育中起重要调控作用,而且可能通过靶向骨形态发生蛋白受体2(BMRRII)而转导参与TGF-β信号通路,并通过下游ID4蛋白和EP300蛋白参与DNA损伤修复和细胞周期调控,其具体作用机制有待进一步验证。胞内转化生长因子-β(Transforming growth factor-β,TGF-β)信号通路在胚胎的早期发育、免疫炎性反应、肿瘤以及机体代谢平衡中都扮演着非常重要的角色[15]。机体内TGF-β信号通路的激活受到非常精确的调控,研究其分子机制具有非常重要的基础和应用意义。富集分析发现hsa-miR-342-3p靶向TGF-β信号通路组成蛋白,提示hsa-miR-342-3p可能通过调控TGF-β信号通路而参与肿瘤发生发展。
近年来研究发现miRNA广泛参与了癌症信号通路的调节,且与癌细胞的凋亡、转移和浸润密切相关[16,17]。hsa-miR-342-3p可能作为肿瘤抑制因子和激酶调控因子而参与癌症病程。Wang等的研究表明hsa-miR-342-3p通过靶向DNA甲基化转移酶DNMT而显著抑制裸鼠结直肠癌细胞的生长与转移[18],Li等研究发现hsa-miR-342-3p的过度表达可抑制宫颈癌细胞系的增殖、迁移和侵袭[19]。Crippa E等研究发现过表达hsa-miR-342-3p可显著降低ID4蛋白而提高乳腺癌抑制基因BRCA1的表达,抑制乳腺癌发展[20]。Ryan JL研究发现在EB(人类疱疹病毒病)感染人胃癌细胞后,会引发细胞周期调节因子ID4、血管生成因子HIF1a以及DNA修复因子BRCA1协同变化[21]。上述研究提示我们预测得到的hsa-miR-342-3p靶标ID4及下游分子响应网络的存在。众多miRNA受肿瘤抑制基因TP53激活而发挥转录后调控作用[22]。hsa-miR-342-3p靶标HIF1a和肿瘤抑制蛋白TP53存在密切相互作用,因此, hsa-miR-342-3p是否靶向ID4蛋白并协同HIF1a和TP53调控癌症信号通路值得进一步实验证实和深入研究。
4结论
通过生物信息学方法对hsa-miR-342-3p进行了系统的分析,结果显示hsa-miR-342-3p在多种组织和疾病中表达,靶基因分布具有组织特异性,并可能通过调控多个靶基因而参与TGF-beta信号通路调节,在机体的多种生理、病理过程中发挥重要作用。hsa-miR-342-3p可作为结直肠癌、胰腺癌、乳腺癌等管状组织相关癌症的生物标志性分子[23],具有重要的研究价值。本文筛选预测得到的14个候选基因,为我们后续进一步探索hsa-miR-342-3p与癌症机制方面提供了初步的研究方向。
参考文献
[1]BARTEL D P.MicroRNAs:genomics,biogenesis,mechanism and function[J].Cell,2004,116(2):281-297.
[2]CHENG A M,BYROM M W,SHELTON J, et al. Antisense inhibition of human miRNAs and indications for an involvement of miRNA in cell growth and apoptosis[J].Nucleic Acids Research, 2005, 33(4): 1290-1297.
[3]ZHAO L, ZHANG Y.hsa-miR-342-3p affects hepatocellular carcinoma cell proliferation via regulating NF-κB pathway [J]. Biochemical and Biophysical Research Communications, 2015, 457(3):370-377.
[4]LOWERY A J, MILLER N, DEVANEY A, et al. MicroRNA signatures predict oestrogen receptor, progesterone receptor and HER2/neu receptor status in breast cancer[J].Breast Cancer Research,2009,11(3):R27.
[5]XIE X, LIU H, WANG M, et al.hsa-miR-342-3p targets RAP2B to suppress proliferation and invasion of non-small cell lung cancer cells[J].Tumour Biology, 2015, 36(7):5031-5038.
[6]TAO K,YANG J,GUO Z,et al.Prognostic value of miR-221-3p, hsa-miR-342-3p and miR-491-5p expression in colon cancer [J].American Journal of Translational Research,2014,6(4):391-401.
[7]OULAS A, KARATHANASIS N, LOULOUPI A, et al. Prediction of miRNA targets[J].Methods in Molecular Biology, 2015, 1269:207-229.
[8]WEI S, XU H, KUANG Y. Systematic enrichment analysis of microRNA expression profiling studies in endometriosis [J].Iranian Journal of Basic Medical Sciences,2015,18(5):423-432.
[9]FALCON S,GENTLEMAN R. Using GOstats to test gene lists for GOterm association[J].Bioinformatics, 2007, 23(2):257-365.
[10]SZKLARCZYK D, FRANCESCHINI A, WYDER S,et al. STRING v10:protein-protein interaction networks, integrated over the tree of life[J].Nucleic Acids Research, 2015, 43(Database issue):D447-D452.
[11]GENTLEMAN R C, CAREY V J, BATES D M,et al. Bioconductor:open software development for computational biology and bioinformatics[J].Genome Biology,2004,5(10):R80.
[12]GONG J,WU Y,ZHANG X,et al. Comprehensive analysis of human small RNA sequencing data provides insights into expression profiles and miRNA editing[J].RNA Biology, 2014,11(11):1375-1385.
[13]LEWIS B P,BURGE C B,BARTEL D P. Conserved seed pairing,often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J].Cell,2005,120(1):15-20.
[14]LU J, GETZ G, MISKA E A, et al. MicroRNA expression profiles classify human cancers [J].Nature,2005,435:834-838.
[15]MEULMEESTER E, TEN DIJKE P.The dynamic roles of TGF-β in cancer [J].The Journal of Pathology,2011,223(2):205-223.
[16]LYNAM-LENNON N,MAHER S G,REYNOLDS J V. The roles of microRNA in cancer and apoptosis[J].Biological Reviews of the Cambridge Philosophical Society, 2009, 84(1): 55-71.
[17]TSUCHYIA S,OKUNO Y,TSUJIMOTO G. MicroRNA:biogenetic and functional mechanisms and involvements incell differentiation and cancer[J].Journal of Pharmacological Sciences, 2006, 101(4): 267-270.
[18]WANG H,WU J,MENG X, et al. Microrna-342 inhibits colorectal cancer cell proliferation and invasion by directly targeting DNA methyltransferase 1[J].Carcinogenesis, 2011, 32(7):1033-1042.
[19]LI Xuri,CHU Huijun,LÜ Teng, et al. Mir-342-3p suppresses proliferation, migration and invasion by targeting foxm1 in human cervical cancer[J]. FEBS Letters, 2014, 588:3298-3307.
[20]CRIPPA E,LUSA L,CECCOL D E,et al. miR-342 regulates BRCA1 expression through modulation of ID4 in breast cancer[J].PLoS One, 2014, 9(1):e87039.
[21]RYAN J L, JONES R J,KENNEY S C,et al.Epstein-Barr virus-specific methylation of human genes in gastric cancer cells [J].Infectious Agents and Cancer, 2010, 5:27.
[22]NAVARRO F,LIEBERMAN J.miR-34 and p53: New insights into a complex functional relationship[J].PLoS One, 2015,10(7):e0132767.
[23]LEE K H,LEE J K,CHOI D W,et al. Postoperative prognosis prediction of pancreatic cancer with seven microRNAs[J].Pancreas, 2015, 44(5):764-772.
Bioinformatics analysis of hsa-miR-342-3p
MA Zhanbing1, 2,GENG Zhi1,JIAO Haiyan1, 2,HUO Zhenghao1, 2*
(1.DepartmentofMedicalGeneticsandCellBiology,SchoolofBasicMedicalScience,NingxiaMedicalUniversity,Yinchuan750004,China;
2.KeyLabofReproductionandGeneticsofNingxiaHuiAutonomousRegion,Yinchuan750004,China)
Abstract:To predict the target genes of the hsa-miR-342-3p and its function by bioinformatics analysis. All relevant studies of hsa-miR-342-3p were searched in PubMed and reviewed. The sequence of hsa-miR-342-3p was obtained from miRBase. TargetScan, Pictar and PITA were used to do intersection dataset of the target genes of hsa-miR-342-3p. The bioinformatics analysis of the target genes of hsa-miR-342-3p involved tissue and disease specific expression profile analysis,enrichment (gene ontology, GO), signal transduction pathway enrichment and protein interaction network analyses. The results showed that hsa-miR-342-3p sequences were highly conserved in various species. hsa-miR-342-3p had relatively high expression in kidney and disease of ALL, breast cancer (RPM≥1 000). There were 14 target genes of hsa-miR-342-3p identified. GO analyses showed that hsa-miR-342-3p target genes were enriched in growth factor activity, DNA binding and protein kinase activity (GO molecular function,P<0.05),and enriched in positive regulation of macromolecule metabolic process, lung development, tube development and respiratory tube development (GO biology process,P<0.05); Pathway analyses showed that the target genes set mainly located in TGF-Beta signaling pathway, cytokine-cytokine receptor interaction signaling pathway and prostate disease pathway (P<0.01).The conclusion is hsa-miR-342-3p may be involved in the diseases via TGF-Beta signaling pathway, which might be a potential biological marker for further investigation.
Keywords:Hsa-miR-342-3p; Target genes; Gene ontology; Pathway; Bioinformatics
中图分类号:Q343.1
文献标志码:A
文章编号:1672-5565(2015)04-212-08
doi:10.3969/j.issn.1672-5565.2015.04.02
作者简介:马占兵,男,助教,研究方向:复杂疾病遗传;E-mail:mabing_516@163.com.*通信作者:霍正浩,男,教授,研究方向:人类群体遗传学; E-mail:huozhh@163.com.
基金项目:宁夏医科大学2014年度校级科研项目(XM201401)。
收稿日期:2015-10-31;修回日期:2015-11-15.
