与肿瘤相关的计算microRNA组学研究综述
2015-02-23李大鹏廖之君
李大鹏,鞠 颖,廖之君,邹 权
(1.秦皇岛市第四医院肿瘤内科,河北 秦皇岛 066000;
2.厦门大学计算机科学系,福建 厦门 361005;
3.天津大学计算机科学与技术学院,天津 300072;
4.福建医科大学生物化学与分子生物学系,福州 350108)
与肿瘤相关的计算microRNA组学研究综述
李大鹏1,鞠颖2*,廖之君3,4,邹权3
(1.秦皇岛市第四医院肿瘤内科,河北 秦皇岛 066000;
2.厦门大学计算机科学系,福建 厦门 361005;
3.天津大学计算机科学与技术学院,天津 300072;
4.福建医科大学生物化学与分子生物学系,福州 350108)
摘要:癌症基因组学研究一直都是肿瘤遗传学研究的重要课题。目前已有许多研究致力于寻找与肿瘤相关的基因。而调控基因表达的miRNA在肿瘤基因组研究中还有很大的研究空间。本文从系统生物学的视角,综述了与肿瘤相关的致病miRNA寻找、表达差异和isomiR的研究现状;最后给出了对近期miRNA组学热点的评述和未来的研究方向。
关键词:癌症;miRNA;系统生物学;生物网络
恶性肿瘤(俗称癌症)已经超过心血管和脑血管疾病,成为了我国居民第一大死亡原因。根据我国的《肿瘤登记年报》和《中国卫生统计年鉴》的数据,我国每年新发癌症病例约350万,因癌症死亡病例约250万。全国每6分钟就有一人被确诊为癌症,每天有近万人成为癌症患者,平均每7~8人就有一人死于癌症。医学和医疗水平都在提高,心脑血管等疾病的治疗效果逐年得到改善,然而癌症的诊断、治疗和预防都没有明显进步,使得目前癌症成为了死亡第一大因素,而且新增癌症患者的数量仍然呈每年递增的态势。
1研究背景
在癌症研究的科研投入方面,1971年美国政府就宣布了向癌症宣战(War on Cancer),2015年初刚刚宣布了癌症基因组图谱计划完成,该计划共投资2.75亿美元[1]。我国的投入力度也日渐加强。由于存在巨大的需求和丰富的临床资源,我国在癌症研究方面已有引领世界的潜力和趋势,尤其是在鼻咽癌、肝癌、胃癌等地区性癌症的研究上,我国已走在世界的前列。对于癌症等疾病的研究,正在由细胞水平走向分子水平,而基因芯片和下一代测序技术都为分子水平的研究提供了数据支持。这些数据有利于探寻肿瘤遗传易感性和分子标志物,研究肿瘤发生、发展的分子机制,和发展肿瘤个性化诊断治疗新方法[2]。
随着医疗事业的发展,现代医学越来越依靠信息科学为其做数据分析与决策参考。目前,医学和生命科学的研究焦点都集中在癌症的产生、治疗和遗传因素分析。已有大量的研究表明:癌症的发病和各种基因的差异表达存在着密切的关系。人类生理系统中活跃的基因多达2万余个,基因的差异表达类型也有多种多样,包括不同基因在表达量上的差异,同一种基因在可变表达模式上的差异,以及基因中存在的多态性差异等。因此,有必要通过信息学的手段,融合已有的分子生物学和生物医学的先验知识,指导癌症与正常人在基因层面的差异化分析。更进一步,通过综合遗传、统计、人工智能和临床等交叉学科知识,建立不同肿瘤的危险度评价、药物疗效评价模型,构建个体诊断模式,制定常见恶性肿瘤的防治对策和措施,探索诊疗新方法,为肿瘤的早期诊断和治疗提供依据。
已有大量研究表明,许多疾病在分子水平上都与基因表达水平有关,这其中不仅有常见的遗传病,还有包括多种癌症的复杂疾病。这些复杂疾病虽然不具有显著的遗传现象,但是在DNA层面的差异性说明了这些复杂疾病具有遗传因素。最新的研究认为恶性肿瘤的确诊应该综合医学图像信息和基因表达信息[3]。而肿瘤药物的设计,则需要更多的依靠基因表达信息和相关基因的通路知识[4,5]。
肿瘤基因组图谱(TCGA)计划由美国国家癌症研究中心(National Cancer Institute , NCI)和国家人类基因组研究中心(National Human Genome Research Institute, NHGRI)于2006年联合启动的项目,第一阶段为期三年,耗资1亿美元,研究的癌症类型是多形成性成胶质细胞瘤(GBM)和卵巢癌,并于2008年在Nature发表了GBM的研究成果。2009年9月,再投资2.75亿美元,针对20余种癌症进行大规模实验,目前总计36种癌症。TCGA利用以大规模测序为主的基因组分析技术,通过广泛的合作,理解癌症的分子机制,提高人们对癌症发病分子基础的科学认识及提高诊断、治疗和预防癌症的能力。TCGA的目标是完成一套完整的与所有癌症基因组改变相关的“图谱”。除了美国的TCGA计划以外,英国的sanger研究中心也建立了癌症基因组计划(Cancer Genome Project,CPG);还有全球性的国际癌症基因组组织(International Cancer Genome Consortium,ICGC)。这几个大规模的计划和组织提供了许多癌症患者在各种水平上的基因表达和测序数据,这些珍贵的数据有望引领全世界的科研工作者解决和攻克癌症的预防和治疗,并建立个性化的癌症诊疗手段[6,7]。
肿瘤免疫治疗是肿瘤遗传学研究最璀璨的硕果。2014年,施贵宝公司和默沙东公司(美国两家大制药公司)研发的抗PD-1抗体药被批准使用治疗黑色素瘤,使这个原来平均生存期不到一年的恶性肿瘤,变的可控。PD-1通过特异的调高被肿瘤抑制住的病人自身的免疫系统来杀死肿瘤细胞,比起化疗、放疗和靶向治疗,对肿瘤的长期疗效有着更好的效果。 我国科研工作者也在癌症的致病基因方面取得了瞩目的成果。曹雪涛院士发现了在活体中Fas信号对肺癌细胞的生长具有关键的促进作用,该研究为炎症促进癌症的产生和免疫逃避理论提供了支持[8]。北京基因组研究所的刘江研究员利用系统生物学的方法发现了一个可以用于诊断肾癌的蛋白质SPOP,并有望通过阻断该基因的表达达到治疗肾癌的目的[9]。复旦大学的樊嘉教授发现CD151与肝小细胞癌的癌转移侵袭有关联,是癌转移的生物标记;而CD151/c-Met是一个新型的预后指标,它还有可能成为一个新的治疗靶位[10]。
癌症作为复杂疾病,不仅在编码蛋白的基因表达层面存在表达差异,越来越多的研究发现miRNA的差异表达也与多种癌症存在着密切的关系。
2miRNA与肿瘤相关的计算问题
2.1 肿瘤和miRNA的关系
microRNA(简称miRNA)的致病特征是新兴的研究热点,越来越多的证据显示miRNA在癌症的发病、治疗和预测中扮演了重要的角色[11,12]。在不同肿瘤组织中特定miRNA表达谱分析不仅有利于阐明肿瘤的发生发展机制,更为肿瘤的诊断和治疗提供了理论依据[13,14]。
全基因组关联分析证明许多人类miRNA基因位于癌症相关的基因组区域,还有发现表明miRNA的绝对表达水平在肿瘤组织中显著下降,与mRNA的表达谱相比,miRNA表达谱更有利于癌症分类[15,16]。这是因为miRNA作为生物网络的中心节点可以影响多个靶标基因,因此miRNA也被当作生物标记(Biomarker)来协助癌症的早期诊断[17]。
2008年,研究发现miR-21的过量表达与肿瘤患者的存活率密切相关[18],这也开辟了利用miRNA的表达谱信息对肿瘤的前期预测与后期治疗。同期研究发现miR-373和miR-520c的上调能加速乳腺癌的转移[19]。而let-7g、miR-181b、miR-200c、miR-21及miR-17-92基因簇等在结直肠癌中高表达,其中miR-17-92基因簇在微卫星稳定性结直肠癌细胞中上调,miR-21能下调抑癌基因Pdcd4,从而促进结直肠癌细胞侵袭转移[20]。近期研究发现,let-7和miR-34不但具有抑制肿瘤的功能,两者结合使用可以能够更好的阻止癌基因表达,抑制癌细胞增殖和侵袭,相关成果已进入临床试验阶段[21]。
我国的科研工作者也在miRNA指导癌症研究方面做出了突出的贡献。哈尔滨医科大学的杨宝峰教授用实验证实了miR-1的失调能导致心脏病的发生[22]。哈尔滨工业大学的王亚东教授课题组建立了首个miRNA与疾病的数据库miR2Disease,该数据库收录了2 884 对miRNA与疾病关系,涉及到346个miRNA和132种疾病[23]。而随后北京大学的崔庆华教授建立的HMDD数据库收集了10 368条miRNA与疾病的关系,其中包括了572个miRNA和378种疾病[24]。这其中的大部分疾病都与肿瘤相关。然而,这两个数据库的主要数据来源是文献挖掘。研究者发现,事实上还有大量的miRNA与疾病的关系有待挖掘和验证[25,26]。于是,利用系统生物学的方法,基于已有的miRNA之间、疾病之间和miRNA与疾病之间的知识,预测未知的miRNA与疾病以及癌症之间的联系成为了近期的研究热点。
2.2 基于网络的miRNA与肿瘤关系预测
传统的克隆定位方法在研究基因与表型关系中取得了巨大的成功,尤其是在作物育种方面,可以通过亲代的杂交和回交,经历几代育种过滤后,找出影响表型的基因座位置。然而这种方法很难用于人类疾病的研究。由于人类基因组庞大、人类繁衍周期长、子代之间以及与亲代之间无法杂交回交等因素,使得生物学家和医学研究者更倾向于使用生物信息学辅助预测基因与疾病的关联。
对于基因与疾病的关联网络,目前已有较深入的研究[27],但编码基因的功能相似度计算方式无法直接应用于miRNA上。根据相似度计算时考虑的网络拓扑结构,目前的方法可以分为局部相似性计算和全局相似性计算。
局部相似性计算提出的较早,一般只改进miRNA之间的功能相似性计算方法。哈尔滨工业大学的蒋庆华老师利用靶基因集的重合度计算miRNA的功能相似性,从而构建了miRNA-疾病的布尔关系网络[28],如图1所示。黑龙江大学的玄萍副教授提出了使用k近邻的策略计算miRNA,同时将靶标重合信息结合起来,取得了更好的效果[29]。哈尔滨商业大学的韩柯副教授将已有的miRNA与疾病的关系融入到miRNA的功能相似度计算中,进一步提高了预测精度[30]。
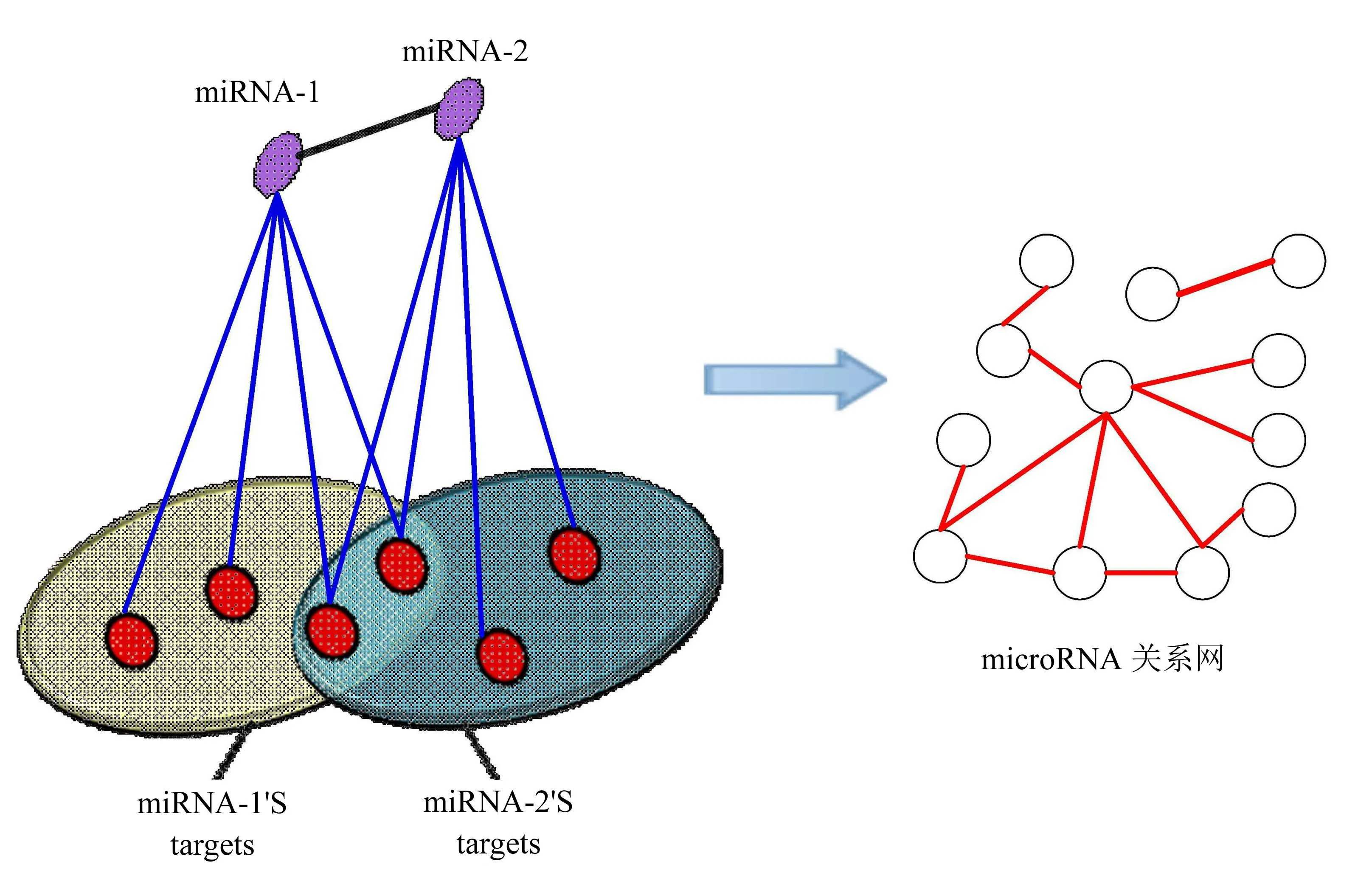
图1 基于共同靶基因的miRNA关系网构造方法Fig.1 Construction of miRNA-miRNA network based on co-targets
全局相似性能更好地考虑到已有知识带来的计算偏差,具有更好地统计解释性。中国科学院数学与系统科学研究院的闫桂英研究员就提出了利用随机游走策略评估潜在的miRNA与疾病间的关系[31];她们还进一步使用半监督学习方法提高了预测准确率[32]。哈尔滨医科大学的李霞教授在随机游走策略的基础上考虑了蛋白质蛋白质相互作用关系对miRNA靶标基因集重合度的计算影响,提高了预测精度[33]。
以上的研究将miRNA家族和成熟体等同看待,没有考虑到成熟体的多样性导致靶标基因存在的差异。因而,未来的研究有必要考虑isomiR的差异性对疾病关系预测带来的影响。
2.3 isomiR与肿瘤
对于同一条miRNA前体,表达出多样性的成熟体被称为isomiR,如图2所示。自从2008年Morin正式提出了isomiR的概念后[34],已有多项研究表明isomiR的表达具有细胞、组织和发育的特异性,并且多种疾病的致病机制也与它们有关[35,36]。isomiR不仅可以成为疾病诊断或治疗的生物标记或靶标,而且对RNA干扰技术和细胞重编程也有重要的指导意义[37]。
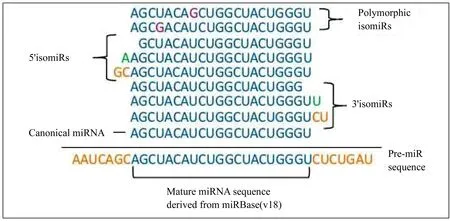
图2 isomiR表达示意图Fig.2 Diagram of isomiR expression
在isomiR的研究中发现了臂转换(Arm switching)现象与肿瘤有密切的联系[38]。臂转换现象表示了miRNA前体中优势成熟体位点的动态选择,或者是3’端和5’端表达比率发生动态的改变。通常isomiR表达谱在正常人中是保守的,暗示了isomiR表达谱不是随机产生,而是受到了严格的调控。然而,在癌细胞甚至癌症患者的癌旁组织中,特定的miRNA成熟体位点可能产生异常的isomiR表达谱。研究者需要检测到这种异常,并进而分析出该isomiR的异样表达是否与癌细胞的病变机理存在着关系。目前国际上相关研究较少,台湾国立阳明大学的王学伟教授[39,40]和南京邮电大学的郭丽博士[41-45]在isomiR的表达特异和进化分析上作了部分基础性工作。在软件和信息学方法的研究上面,还主要以对RNASeq的数据分析为主,部分RNASeq分析软件已经集成了isomiR现象的检测功能[46]。
不但一条miRNA前体可以表达出多种不同的成熟体isomiR,同一个成熟体也可以由不同的前体剪切而来[47]。因此,成熟体isomiR层面和前体层面在研究miRNA时属于完全不同的视角。而以往的研究多把miRNA的前体和成熟体一一对应,进行功能研究的时候不加以区分。正是考虑到这种差异在过去的研究中被忽略,需要提出用基于网络的方法研究miRNA前体和家族层面对各种癌症的影响,进而指导成熟体isomiR层面与癌症的关联,这些都是未来值得研究的课题。
3目前存在的问题与未来研究方向
癌症一直是医学研究的热点问题。目前已从多种组学的角度对各种癌症进行了深入的研究,建立了多模态的数据库,而且研究者已对多组不同癌症的人群进行了全基因组关联分析,然而在编码基因和miRNA家族层面始终没有找到关键性突破。绝大多数研究者归咎于肿瘤细胞产生的复杂机理。2006年,RNA的干扰机制获得了诺贝尔奖之后,一段时间内研究人员寄希望于通过研究miRNA和其他非编码RNA来解决癌症的诊疗。然而,近十年来miRNA以被研究的较为深入,却没有从根本上推动癌症诊疗的向前发展;而其他非编码RNA,尤其是近期成为热点的长非编码RNA(long non-coding RNA,lncRNA)还存在大量的未知[48,49],研究者发现对lncRNA的研究难度远远高于miRNA[50,51]。
迄今还缺少在isomiR层面对癌症的系统研究和全基因组水平的关联分析。目前的isomiR水平上的癌症分析还主要以统计手段为主,缺乏多isomiR之间的影响、相互作用和疾病关联分析。考虑到全基因组水平isomiR庞大的数量,未来有必要用miRNA-疾病网络指导关键miRNA和isomiR的选择,以期进一步从多isomiR协同作用的角度对癌症的基因遗传分析做出解释。
参考文献
[1]HEIDI L. End of cancer atlas prompts rethink[J]. Nature, 2015, 517: 128-129.
[2]KRISTENSEN V, LINGJRDE O, RUSSNES H,etal. Principles and methods of integrative genomic analyses in cancer[J]. Nature Reviews Cancer, 2014, 14(5): 299-313.
[3]LIN Dongdong,CAO Hongbao, CALHOUN V D,etal. Sparse models for correlative and integrative analysis of imaging and genetic data[J]. Journal of Neuroscience Methods, 2014, 237(30): 69-78.
[4]ADAMSON P, HOUGHTON P, PERILONGO G,etal. Drug discovery in paediatric oncology: roadblocks to progress[J]. Nature Reviews Clinical Oncology, 2014, 11(12):732-739
[5]DONG Y, BATRA J, ANAND K,etal. Transforming the future of treatment for ovarian cancer[J]. Clinical & Experimental Pharmacology, 2014, 4(3): 1000157.
[6]HAYDEN E. Personalized cancer therapy gets closer[J]. Nature, 2009, 458(7235):131.
[7]CHRIS E. Using patient data for personalized cancer treatments[J]. Communications of the ACM,2014, 57(4): 13-15
[8]ZHANG Y, LIU Q, ZHANG M,etal. Fas signal promotes lung cancer growth by recruiting myeloid-derived suppressor cells via cancer cell-derived PGE2[J]. The Journal of Immunology, 2009, 182(6): 3801-3808.
[9]LI G, CI W, KARMAKAR S,etal. SPOP promotes tumorigenesis by acting as a key regulatory hub in kidney cancer[J]. Cancer Cell, 2014, 25(4): 455-468.
[10]KE A W, SHI G M, ZHOU J,etal. Role of overexpression of CD151 and/or c-Met in predicting prognosis of hepatocellular carcinoma[J]. Hepatology, 2009, 49(2): 491-503.
[11]SQUADRITO M, ETZRODT M, DE PALMA M,etal. MiRNA-mediated control of macrophages and its implications for cancer[J]. Trends in Immunology, 2013, 34(7): 350-359.
[12]SHI H, WU Y, ZENG Z,etal. A Discussion of MicroRNAs in Cancers[J]. Current Bioinformatics, 2014, 9(5): 453-462.
[13]WANG Q, WEI L, GUAN X,etal. Briefing in family characteristics of microRNAs and their applications in cancer research[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2014, 1844(1): 191-197.
[14]ZOU Q,MAO Y,HU L,etal. miRClassify:An advanced web server for miRNA family classification and annotation[J].Computers in Biology and Medicine, 2014, 45: 157-160.
[15]LU J, GETZ G, MISKA E,etal. MiRNA expression profiles classify human cancers[J]. Nature, 2005, 435(7043): 834-838.
[16]CHANG H, LI S, HO M,etal. Comprehensive analysis of miRNAs in breast cancer[J]. BMC Genomics, 2012, 13(Suppl 7): S18.
[17]SCHWARZENBACH H,NISHIDA N,CALIN G A,etal.Clinical relevance of circulating cell-free miRNAs in cancer[J].Nature Reviews Clinical Oncology, 2014, 11(3): 145-156.
[18]LAWRIE C, GAL S, DUNLOP H,etal. Detection of elevated levels of tumour-associated miRNAs in serum of patients with diffuse large B-cell lymphoma[J]. British Journal of Haematology, 2008, 141(5): 672-675.
[19]HUANG Q, GUMIREDDY K, SCHRIER M,etal. The miRNAs miR-373 and miR-520c promote tumour invasion and metastasis[J].Nature Cell Biology, 2008, 10(2): 202-210.
[20]ASANGANI I, RASHEED S, NIKOLOVA D,etal. MiRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer[J].Oncogene, 2007, 27(15): 2128-2136.
[21]KASINSKI A L, KELNAR K, STAHLHUT C,etal. A combinatorial miRNA therapeutics approach to suppressing non-small cell lung cancer[J].Oncogene, 2015, 34(27): 3547-3555.
[22]YANG B, LIN H, XIAO J,etal. The muscle-specific miRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2[J].Nature Medicine, 2007, 13(4): 486-491.
[23]JIANG Q, WANG Y, HAO Y,etal. miR2Disease: a manually curated database for miRNA deregulation in human disease[J].Nucleic Acids Research, 2009, 37(suppl 1): D98-D104.
[24]LI Y, QIU C, TU J,etal. HMDD v2.0: a database for experimentally supported human miRNA and disease associations[J].Nucleic Acids Research, 2014, 42(Database issue):D1070-D1074.
[25]ZOU Q, LI J, HONG Q,etal.Prediction of MicroRNA-Disease Associations Based on Social Network Analysis Methods[J].BioMed Research International,2015,2015:1-9.
[26]CHEN K, ZOU Q, PENG Z,etal. Hierarchical Identification of MicroRNA Families for Biomedical Applications[J].Journal of Computational and Theoretical Nanoscience,2014,11(3): 883-887.
[27]ZOU Q, LI J, WANG C,etal. Approaches for recognizing disease genes based on network[J]. BioMed Research International, 2014(1), 2014:1-10.
[28]JIANG Q, HAO Y, WANG G,etal.Prioritization of disease miRNAs through a human phenome-miRNAome network[J].BMC Systems Biology,2010, 4(Suppl 1):S2.
[29]XUAN P, HAN K, GUO M,etal. Prediction of miRNAs associated with human diseases based on weighted k most similar neighbors[J].PloS One, 2013, 8(8): e70204.
[30]HAN K, XUAN P, DING J,etal. Prediction of disease-related miRNAs by incorporating functional similarity and common association information[J].Genetics and Molecular Research, 2013, 13(1): 2009-2019.
[31]CHEN X, LIU M, YAN G. RWRMDA: predicting novel human miRNA-disease associations[J]. Molecular BioSystems,2012,8(10):2792-2798.
[32]CHEN X, YAN G. Semi-supervised learning for potential human miRNA-disease associations inference[J].Scientific Reports,2014, 4:5501.
[33]SHI H, XU J, ZHANG G,etal. Walking the interactome to identify human miRNA-disease associations through the functional link between miRNA targets and disease genes[J].BMC Systems Biology, 2013, 7(1): 101.
[34]MORIN R, O’CONNOR M, GRIFFITH M,etal. Application of massively parallel sequencing to miRNA profiling and discovery in human embryonic stem cells[J].Genome Research,2008,18(4): 610-621.
[35]FERNANDEZ-VALVERDE S, TAFT R, MATTICK J. Dynamic isomiR regulation in Drosophila development[J]. RNA, 2010, 16(10): 1881-1888.
[36]ZHOU H, ARCILA M, LI Z,etal. Deep annotation of mouse iso-miR and iso-moR variation[J]. Nucleic Acids Research, 2012, 40(13): 5864-5875.
[37]谢兆辉,李学贵,许禔森.miRNA的异构体——isomiR[J].中国生物化学与生物物理学报,2014,30(8):739-745.
XIE Zhaohui,LI Xuegui,XU Tisen.The Isoforms of miRNA——isomiR[J].Chinese Journal of Biochemistry and Molecular Biology,2014, 30(8): 739-745.
[38]LI S, LIAO Y, HO M,etal. miRNA arm selection and isomiR distribution in gastric cancer[J].BMC Genomics, 2012, 13(Suppl 1): S13.
[39]CHENG W, CHUNG I, HUANG T,etal. YM500: a small RNA sequencing(smRNA-seq)database for miRNA research[J]. Nucleic Acids Research, 2013, 41(D1): D285-D294.
[40]CHENG W, CHUNG I, TSAI C,etal. YM500v2: a small RNA sequencing(smRNA-seq)database for human cancer miRNome research[J]. Nucleic Acids Research, 2014:43(D1):1-6.
[41]GUO L, ZHANG H, ZHAO Y,etal. In-Depth Exploration of miRNA: A new approach to study miRNA at the miRNA/isomiR Levels[J]. Current Bioinformatics, 2014, 9(5): 522-530.
[42]GUO L, LI H, LIANG T,etal. Consistent isomiR expression patterns and 3′ addition events in miRNA gene clusters and families implicate functional and evolutionary relationships[J]. Molecular Biology Reports, 2012, 39(6): 6699-6706.
[43]GUO L, LU Z. Global expression analysis of miRNA gene cluster and family based on isomiRs from deep sequencing data[J]. Computational Biology and Chemistry, 2010, 34(3): 165-171.
[44]GUO L, YANG Q, LU J,etal. A comprehensive survey of miRNA repertoire and 3′ addition events in the placentas of patients with pre-eclampsia from high-throughput sequencing[J]. PloS One, 2011, 6(6): e21072.
[45]GUO L, ZHANG H, ZHAO Y,etal. Selected isomiR expression profiles via arm switching?[J]. Gene,2014, 533(1): 149-155.
[46]SABLOK G, MILEV I, MINKOV G,etal. isomiRex: Web-based identification of miRNAs, isomiR variations and differential expression using next-generation sequencing datasets[J]. FEBS Letters, 2013, 587(16): 2629-2634.
[47]WANG M, WANG W, ZHANG P,etal. Discrimination of the expression of paralogous miRNA precursors that share the same major mature form[J]. PLoS One, 2014, 9(3): e90591.
[48]HUANG Y, LIU N, WANG J,etal. Regulatory long non-coding RNA and its functions[J]. Journal of Physiology and Biochemistry, 2012, 68(4): 611-618.
[49]WANG C, WEI L, GUO M,etal. Computational approaches in detecting non-coding RNA[J]. Current Genomics, 2013, 14(6): 371.
[50]WANG Y, CHEN L, CHEN B,etal. Mammalian ncRNA-disease repository: a global view of ncRNA-mediated disease network[J].Cell Death & Disease, 2013, 4(8): e765.
[51]JIANG Q, WANG J, WU X,etal. LncRNA2Target: a database for differentially expressed genes after lncRNA knockdown or overexpression[J].Nucleic Acids Research,2015, 43(D1):D193-D196.
Review on computational microRNomics related to tumor research
LI Dapeng1, JU Ying2*, LIAO Zhijun3,4, ZOU Quan3
(1.DepartmentofInternalMedicine-Oncology,TheFourthHospitalinQinhuangdao,Qinhuangdao,QinhuangdaoHebei066000,China;
2.DepartmentofComputerScience,XiamenUniversity,XiamenFujian361005,China;
3.SchoolofComputerScienceandTechnology,TianjinUniversity,Tianjin300072,China;
4.DepartmentofBiochemistryandMolecularBiology,FujianMedicalUniversity,Fuzhou350108,China)
Abstract:Cancer genomics is an essential topic in the tumor genetics research. Lots of works have been done for identifying oncogenes. However, the regulatory miRNAs remains to be further studied.In this paper, we focused on onco-miRNAs, and reviewed the cancer related miRNA detection, expression and isomiRs from the view of system biology. Fimally we summarized the recent hot topics on microRNomics.
Keywords:Cancer; miRNA; System biology; Biological network
中图分类号:TP802
文献标志码:A
文章编号:1672-5565(2015)04-225-06
doi:10.3969/j.issn.1672-5565.2015.04.04
作者简介:李大鹏,男,主治医师,研究方向:肿瘤信息学和肿瘤诊治;E-mail:dapeng3529908@sina.com.*通信作者:鞠颖,女,副教授,研究方向:生物信息学和生物医学工程;E-mail:yju@xmu.edu.cn.
基金项目:国家自然科学基金(61370010);秦皇岛市科技支撑计划项目(NO.20152A217)。
收稿日期:2015-09-24;修回日期:2015-11-09.
