铜绿丽金龟雌虫触角全长cDNA文库的构建及质量分析
2015-02-14李克斌曹雅忠仵均祥
赵 莹, 尹 姣, 李克斌, 曹雅忠, 仵均祥
(1.农业部西北黄土高原作物有害生物综合治理重点实验室,西北农林科技大学植物保护学院,杨凌 712100;2.中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193)
实验方法与技术
铜绿丽金龟雌虫触角全长cDNA文库的构建及质量分析
赵 莹1,2, 尹 姣2, 李克斌2, 曹雅忠2, 仵均祥1*
(1.农业部西北黄土高原作物有害生物综合治理重点实验室,西北农林科技大学植物保护学院,杨凌 712100;2.中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193)
为了研究铜绿丽金龟(Anomala corpulentaMotschulsky)成虫嗅觉相关蛋白的功能,本文以铜绿丽金龟雌成虫触角为原始材料提取总RNA,利用SMART技术构建了铜绿丽金龟雌虫触角全长cDNA文库。经测定,原始文库滴度为6.74×106pfu/mL,扩增后的文库滴度为1.08×1010pfu/mL,文库重组率为98.98%。随机挑取24个单克隆,通过菌液PCR检测得知文库插入片段的大小在0.5~2.0 kb之间,片段平均长度为1.0 kb左右。经过菌液PCR并测序筛选cDNA文库,本试验获得了两条编码气味结合蛋白的全长基因序列AcorOBP1和AcorOBP2。以上数据表明,已获得高质量的铜绿丽金龟触角全长cDNA文库,并筛选、克隆获得了气味结合蛋白基因序列。这为进一步研究触角内气味结合蛋白的功能奠定了基础。
铜绿丽金龟; 触角; 全长cDNA文库
金龟子属于鞘翅目、金龟总科,是我国农林生产中的重要害虫,目前全世界已知约2万种,中国已报道的约1 800种[1]。其幼虫蛴螬为害多种植物,主要取食植物地下根、茎部分,生活隐蔽,分布广,食量大,食性杂,难以防治,是我国地下害虫中的最大类群。统计数据表明,植物受害地下部分中86%是由蛴螬为害造成的[2]。蛴螬咬断幼苗根茎部,影响农作物幼苗的生长,造成产量和质量的下降。例如,蛴螬为害花生致使花生果实秕仁、空壳率增加,花生田大面积减产甚至绝收[3]。金龟子成虫主要在地上活动,取食林木、果树以及农作物的叶片、花蕾等。铜绿丽金龟(Anomala corpulenta)是我国金龟子中的重要优势种群[4],主要取食榆树、杨树、柳树、樱花、葡萄、海棠、杏、银杏木、月季等植物叶片,为害严重时,导致植物仅留叶脉,食光叶片后又迁移到其他苗木上继续为害[5],造成极大的经济损失,严重影响农林果业的发展。
全长cDNA文库的构建、测序和功能注释是了解生物结构和功能的重要方法之一[6],在分离、克隆、筛选新基因以及基因功能研究等方面具有重要作用。SMART(Switching Mechanism At 5′end of the RNA Transcript)技术由美国Clontech公司推出[7],是目前研究技术比较成熟,应用最为广泛的全长cDNA文库构建方法。SMART技术在反转录合成第一链cDNA时,事先加入3′末端带Oligo(dG)的SMART引物,以mRNA为模板在反转录酶(Power ScriptTMReverse Transcriptase)的作用下合成全长第一链cDNA;此外,在目的基因与载体连接阶段,仅仅对目的ds cDNA进行SfiⅠ单酶切,就可以得到两个黏性末端不同的cDNA,这样目的片段就能够定向插入特定的载体中,大大提高了文库的阳性克隆率[8]。SMART法构建文库需要的mRNA量少[9-10],且操作简便、快速,降低了mRNA降解的几率,最终获得具有较高全长序列比例的cDNA文库。
触角是昆虫感受外界环境的主要器官,在昆虫寻找食物、寻求配偶或选择生殖场所的过程中起到至关重要的作用。金龟子触角上的嗅觉感器居多,主要为板形感器和锥形感器;对化学气味具有选择性,而且雌雄两性间常常存在嗅觉差异[11]。研究发现,大多数昆虫的气味结合蛋白在表达时显示几乎完全的性不关联[12-15];但是有些昆虫雌虫对植物挥发物更敏感,这可能是为了更有利于雌虫选择产卵寄主[16]。另外,雌虫在种群繁衍中担负着生殖后代的生物本能。所以我们选择了构建铜绿丽金龟雌虫触角全长cDNA文库,这样一方面可能一次性永久保留更多的基因资源,另一方面为进一步顺利筛选、克隆以及表达嗅觉蛋白,从分子水平深入研究铜绿丽金龟嗅觉机制奠定基础。
1 材料和方法
1.1 试验材料
1.1.1 供试虫源
试虫采集地点为河南省洛阳市郊,用灯光诱捕法捕捉铜绿丽金龟成虫,在室内将雌虫与雄虫分开,在冰上取其触角,放入1.5 mL无菌离心管中,每管收集10对触角,在液氮中迅速冷冻,最后将离心管放入-80℃超低温冰箱中冷藏备用。
1.1.2 主要试剂及来源
RNeasy Plus Mini Kit购自Qiagen公司。SMARTTMcDNA Library Construction Kit(Cat.No. 634901)购自美国Clontech公司,噬菌体包装试剂盒MaxPlaxTMLambda Packaging Extracts为美国Epicentre试剂盒,二甲苯青购自Sigma公司,LATaq酶、二甲基亚砜(DMSO)购自TaKaRa公司,抗生素、X-gal、IPTG购自上海生工生物工程有限公司。引物合成与PCR产物测序由上海生工生物工程有限公司完成。
1.1.3 培养基配制
90 mm LB/MgSO4平板、150 mm LB/MgSO4平板:在100 mL LB液体培养基中加入1.5 g琼脂粉,再加入1 m L浓度为1 mol/L的MgSO4溶液,混匀后高温灭菌。灭菌结束后将培养基平铺于若干个直径为90 mm(简称为LB平板A)或150 mm(简称为LB平板B)的无菌平板中,超净台凝固后,在4℃条件下保存。
LB/Amp平板:在100 mL LB液体培养基中加入1.5 g琼脂粉,高温灭菌,待培养基温度降至50℃时加入100μL浓度为50 mg/mL的Amp,迅速混合均匀后将培养基平铺于若干个直径为90 mm无菌平板中,超净台凝固后,在4℃条件下保存(简称为LB平板C)。
1.2 试验方法
1.2.1 总RNA提取
用无水乙醇燃烧处理研钵、研杵之后用液氮迅速冷却。从80℃超低温冰箱中取出100对雌虫触角放入液氮中备用。将触角放入含有液氮的研钵中,迅速研磨直至触角呈粉末状。按照Qiagen公司RNeasy Plus Mini Kit说明书要求提取总RNA。取2μL样品,用1.1%琼脂糖凝胶电泳检测总RNA质量,并用核酸蛋白分析仪检测其浓度。
1.2.2 双链cDNA合成
按照SMARTTMcDNA Library Construction Kit试剂盒说明书,以3μL总RNA为模板,以SMRATⅣ寡聚核苷酸和CDSⅢ/3′-PCR为引物,在SMART ScribeTMMMLV反转录酶的作用下反转录合成cDNA第一链。
再以cDNA为模板,在CDSⅢ/3′-PCR引物和5′-PCR引物引导下,经过LD-PCR(long distance polymerase chain reaction),合成双链cDNA(ds cDNA)。反应程序为95℃1 min;95℃15 s、68℃6 min,20个循环;结束后4℃保存。取2μL产物,用1.1%琼脂糖凝胶电泳检测扩增效率,其余产物在-20℃保存。
1.2.3 蛋白酶K消化
在0.5 mL离心管中加入50μL ds cDNA和2μL蛋白酶K,混匀后45℃孵育20 min;孵育结束后加入50μL去离子水,混匀,再加入100μL体积比为25∶24∶1的苯酚∶氯仿∶异戊醇,轻柔颠倒,14 000 r/min离心5 min;将上清液转入新的离心管中,加入100μL体积比为24∶1的氯仿∶异戊醇,轻柔颠倒,14 000 r/min离心5 min;取上清液转入新的离心管中,加入3 mol/L醋酸钠10μL、20μg/μL糖原1.3μL、95%乙醇260μL,14 000 r/min离心20 min,弃上清液,用80%乙醇100μL洗涤沉淀,待沉淀干燥后,加入79μL去离子水溶解沉淀。
1.2.4SfiⅠ酶切
将已获得的ds cDNA进行SfiⅠ酶切,酶切后加入2μL 1%的二甲苯青,为后续试验做准备。
1.2.5 cDNA分级分离
按试剂盒说明书要求准备16个1.5 mL离心管。将CHROMA SPIN-400分级分离柱内基质摇匀,使凝胶基质完全悬浮,去除柱子内的气泡,移走底盖使柱内缓冲液流尽。沿柱子内壁加入700μL圆柱缓冲液,待缓冲液排尽后,加入用二甲苯青染过色的cDNA样品,静置1 min,使样品完全吸附在基质表面,用100μL圆柱缓冲液洗涤柱子,待无缓冲液流出后,加入600μL圆柱缓冲液,按每管1滴收集样品。每管取3μL样品,用1.1%琼脂糖凝胶电泳检测。收集前4管有ds cDNA条带的原样品液,汇集于一支新的离心管中,加入1/10回收样品液体积的3 mol/L醋酸钠、20μg/μL糖原1.3μL和2.5倍回收样品液体积的95%乙醇,在-20℃条件下过夜浓缩,离心,弃上清液,用7μL去离子水溶解沉淀。
1.2.6 cDNA与λTriplE×2噬菌体载体连接
为了获得cDNA与载体的最优连接比例,使用3个比例的连接体系,16℃过夜连接。反应体系如表1所示。
1.2.7 噬菌体包装
室温解冻噬菌体包装提取物,向连接产物中加入25μL噬菌体包装提取物,30℃孵育90 min,反应结束后再加入25μL噬菌体包装提取物,30℃孵育90 min。孵育结束后分别加入1×λ稀释缓冲液500μL、氯仿25μL,涡旋混匀,将噬菌体溶液保存在4℃冰箱中。
1.2.8 文库质量的鉴定
未扩增文库滴度:过夜培养E.coliXL1-Blue菌,A600达到2.0后,离心弃上清液,用7.5 mL浓度为10 mmol/L的MgSO4溶液重悬菌体。噬菌体溶液用1×λ稀释缓冲液稀释,比例分别为1∶5、1∶10、1∶15、1∶20,分别取1μL稀释后的噬菌体溶液与200μL重悬菌液,混匀,37℃培养10 min,加入2 mL融化的顶层琼脂,混匀后平铺于LB平板A,37℃过夜培养。
文库滴度(pfu/mL)=(噬菌斑数×稀释倍数× 103)/涂板噬菌体体积(μL)
重组率测定:过夜培养E.coliXL1-Blue菌,离心收集菌体,用5 mL浓度为10 mmol/L的MgSO4溶液重悬菌体。添加X-gal与IPTG到融化的顶层琼脂中,每2 mL融化的顶层琼脂使用X-gal(100 mmol/L)、IPTG(100 mmol/L)各50μL,将细菌培养物、包装后噬菌体溶液和2 m L融化的顶层琼脂混匀,快速平铺于LB平板A上,37℃培养6~18 h,直至显蓝斑。
1.2.9 扩增文库的建立
根据未扩增文库滴度估算文库扩增时所需LB平板B的数目。例如,150 mm平板有(6~7)×104个克隆,则文库1×106个克隆则需要20个平板。
过夜培养E.coliXL1-Blue菌,A600达到2.0后,离心弃上清液,用7.5 mL浓度为10 mmol/L的MgSO4溶液重悬菌体。将细菌培养物、包装后噬菌体溶液和4 mL融化的顶层琼脂三者混匀,平铺于LB平板B,37℃培养6~18 h。向长出噬菌斑的平板中加入12 mL 1×λ稀释缓冲液,4℃冰箱中过夜放置。取出冰箱中的平板,50 r/min室温孵育1 h,将平板内的噬菌体溶液收集到50 mL离心管中,加入10 mL氯仿,涡旋震荡2 min,7 000 r/min离心10 min,收集上清液到新的50 mL离心管中,4℃保存扩增后文库(长期保存文库则需要加终浓度为7%的DMSO,并保存在-70℃超低温冰箱)。
1.2.10 噬菌体文库向质粒文库的转化
过夜培养E.coliBM 25.8菌,A600达1.1~1.4时,加入100μL浓度为1 mol/L的MgCl2溶液。将500μL细菌培养物与200μL噬菌体溶液混匀,31℃孵育1 h;再加入1 mL LB培养基,31℃,225 r/min条件下培养1 h。取1~10μL感染细胞悬浮液涂于LB平板C,31℃过夜培养。
1.2.11 插入片段大小检测及序列筛选
插入片段大小检测:随机挑取24个单菌落,以载体上已知片段为引物序列,进行菌液PCR。反应程序为:94℃4 min;94℃45 s,58℃45 s,72℃1 min,共30个循环;72℃10 min。反应结束后取3μL产物用1.1%琼脂糖凝胶电泳检测,其余产物-20℃保存。依据此步骤,将PCR获得的产物送生工公司测序,根据反馈的信息,在GenBank上筛选目的基因片段。引物序列为:
上游引物:5′-CTCGGGAAGCGCGCCATTGTGTTGGT-3′;下游引物:5′-ATACGACTCACTATAGGGCGAATTGGCC-3′。
2 结果与分析
2.1 铜绿丽金龟触角总RNA的提取质量检测
1.1 %的琼脂糖凝胶电泳检测结果如图1所示。28 S与18 S条带清晰,而5 S无明显条带,且拖带现象不明显。经核酸蛋白分析仪检测,A260/A280值为2.16,A260/A230值为2.42,总RNA浓度为395 ng/μL,说明总RNA质量很高,可用于cDNA文库的构建。
2.2 双链的合成
如图2、图3所示,反转录合成的第一链cDNA与LD-PCR后形成的ds cDNA均为弥散的条带,其中有几条比较明显的条带。

图1 铜绿丽金龟触角总RNAFig.1 The total RNA from the antennae ofAnomala corpulenta

图2 铜绿丽金龟触角cDNA第一链Fig.2 First-strand cDNA from the antennae ofAnomala corpulenta
2.3 cDNA分级分离检测
经过柱层析分离,共收集了16管cDNA片段样品,电泳检测如图4所示,1~6管几乎没有ds cDNA,7~10号管有明显大于500 bp的ds cDNA条带,11~16管无明显ds cDNA条带。收集7~10管cDNA样品到新的离心管中,用于后续连接试验。
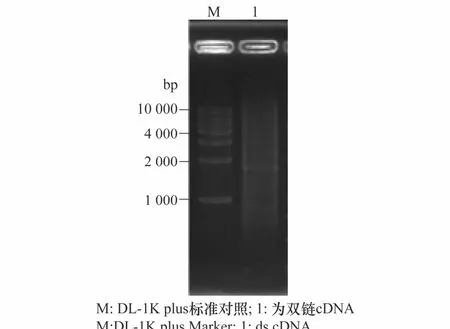
图3 铜绿丽金龟触角ds cDNAFig.3 The ds cDNA from the antennae ofAnomala corpulenta
2.4 cDNA文库的质量鉴定
2.4.1 文库库容量及重组率的检测
根据SMART试剂盒提供的测定方法,测得原始文库的滴度为6.74×106pfu/m L。扩增之后文库的滴度为1.08×1010pfu/m L。蓝白斑筛选得克隆重组率为98.98%,是高质量的cDNA文库。
2.4.2 文库插入片段大小检测
从LB平板C上随机挑取了24个单菌落,经1.1%琼脂糖凝胶电泳检测,结果如图5所示,插入片段分布在500~2 000 bp范围内,其中以500~1 000 bp大小的片段为主。

图4 铜绿丽金龟触角双链cDNA分级图Fig.4 Separation of ds cDNA from the antennae ofAnomala corpulenta

图5 铜绿丽金龟触角cDNA文库部分PCR扩增的电泳结果Fig.5 PCR detection of partial clones of cDNA library from antennae ofAnomala corpulenta
2.5 筛选结果
根据测序公司反馈的序列信息,在GenBank上利用BLAST筛选目的片段。经序列比对,获得了铜绿丽金龟气味结合蛋白(odorant binding proteins,OBPs)基因全长序列,并推导获得氨基酸序列。如图6所示。推导的氨基酸序列位于核苷酸序列之下,方框标注的为保守的半胱氨酸位点。OBP1全长608 bp,开放阅读框长度为405 bp,编码135个氨基酸,其中前19个氨基酸残基为预测的信号肽,预测分子量大小为14.816 ku,等电点为4.43。OBP2全长602 bp,开放阅读框长度为459 bp,编码153个氨基酸,其中前20个氨基酸残基为预测的信号肽,预测分子量大小为12.906 ku,等电点为4.33。昆虫的气味结合蛋白OBPs普遍具有6个保守的半胱氨酸位点,基本结构模型为Cys-X(20 66)-Cys-X(3)-Cys-X(21 43)-Cys-X(8 14)-Cys-X(8 6)-Cys,X代表每个半胱氨酸之间间隔的氨基酸残基数,其中第2个半胱氨酸与第3个半胱氨酸之间总是间隔3个氨基酸残基,第5个半胱氨酸与第6个半胱氨酸之间总是间隔8个氨基酸残基。根据两个OBPs的半胱氨酸位点的数量和位置,可知这两个蛋白的氨基酸序列属于经典模型OBPs。

图6 OBP1和OBP2核苷酸序列及推导的氨基酸序列Fig.6 Nucleotide and deduced amino acid sequence of OBP1 and OBP2
通过http:∥us.expasy.org/cgi-bin/protscale.pl,对已获得的气味结合蛋白开放阅读框进行在线疏水性分析。从图7中我们可以看出,两条基因序列编码的氨基酸N-端都有一段疏水的氨基酸残基,即信号肽,而且这两条氨基酸序列都能形成明显的疏水腔。

图7 AcorOBP1和AcorOBP2氨基酸序列疏水性分析Fig.7 Hydrophobic analysis for the deduced amino acid sequences of the AcorOBP1 and AcorOBP2
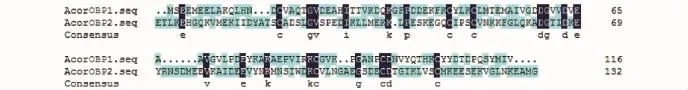
图8 铜绿丽金龟OBPs序列联配Fig.8 Alignment of amino acid sequences of AcorOBPs
对获得的铜绿丽金龟OBPs两条氨基酸序列进行序列联配,如图8所示,序列间相似度很低,氨基酸序列一致性仅为16.4%,蛋白之间的分化度很高。此外,铜绿丽金龟OBPs与其他金龟子OBPs进行序列联配(图9),发现铜绿丽金龟OBP1与其他金龟子OBP1或PBP1的同源性较高,序列间相似度均在80%以上,表明在不同种之间有较高的保守性。而铜绿丽金龟OBP2分化程度较高,与小栗鳃金龟OBP2的相似度为88%,与大黑鳃金龟OBP2的相似度为54%,与暗黑鳃金龟的相似度为53%。
通过MEGA 4.0构建了部分鞘翅目昆虫OBPs的系统发育树(图10),1 000次重复结果,结果表明34个OBPs分成2个大的分支。OBP1与OBP2序列同源性较低,在进化树中两者位于不同的两个分支中,并且距离较远,这表明他们属于不同的亚群,并且可能在昆虫中所行使的功能不同。

图10 铜绿丽金龟OBPs与其他鞘翅目昆虫OBPs的系统发育树Fig.10 Phylogenetic tree of AcorOBPs and other OBPs from Coleoptera insects
3 结论与讨论
3.1 关于总RNA的提取
文库构建的质量决定文库应用的价值,要想构建好的文库,最关键的步骤就是总RNA的提取[12]。在本次试验中分别采用Trizol试剂法和试剂盒(RNeasy Plus Mini Kit购自Qiagen)两种方法提取铜绿丽金龟雌虫触角总RNA。无论哪种方法在初始研磨阶段都要进行RNA酶的灭活,以免造成试验过程中总RNA的降解[18]。用1.1%琼脂糖凝胶电泳检测两种方法的结果,发现Trizol试剂法提取试虫触角总RNA 5S条带明显,且18S、28S有拖带现象,降解严重;而采用试剂盒法提取总RNA几乎看不到5S RNA,降解不明显。核酸蛋白分析仪检测总RNA浓度,结果表明,第二种方法获得的总RNA浓度远远小于第一种方法,这表明采用第二种方法提取总RNA时需要准备充足的原始材料。比对两种方法的A260/A280值、A260/A230值,Trizol法提取的RNA两个比值均小于1.5,这可能是由于组织裂解不够充分,对杂质的去除不够彻底[19];试剂盒法提取的总RNA两个比值均大于2.0,表明提取的总RNA比较纯,基本无蛋白和其他杂质污染[19]。采用Qiagen公司的RNeasy Plus Mini Kit提取总RNA操作简单、快捷(只需1 h)且DNA污染少,虽然成本较高,但是比较适合少量RNA的提取[20],因此本试验采用该法提取铜绿丽金龟触角总RNA。此外,在选择建库起始材料时,要特别注意了解试虫及其触角的采集时间。本文建库的目的在于获取铜绿丽金龟触角嗅觉相关蛋白,因此,在铜绿丽金龟成虫寻找食物、配偶、产卵寄主盛期采集,且触角的采集要迅捷,可保障所建文库中具有较丰富的相关目的蛋白。
3.2 关于后续工作
自20世纪70年代中期首例cDNA克隆问世以来,构建cDNA文库已成为研究功能基因组学的基本手段之一[21]。构建cDNA文库可以保存濒危珍稀生物资源,也可用于分离全长基因进行基因功能研究。根据一般文库的构建标准:未扩增文库滴度达1.0×106pfu/mL,试剂盒所提供的对照片段重组率达80%以上,所建文库即为有效文库。我们利用Qiagen公司的RNeasy Plus Mini Kit提取到了高质量的总RNA,运用SMART技术构建了铜绿丽金龟雌虫触角cDNA文库,经检测得原始文库的滴度为6.74×106pfu/m L,文库的重组率为98.98%,通过随机PCR扩增得知插入片段大小在500~2 000 bp范围内,且获得了两种不同的OBP基因编码序列,这些数据表明已获得高质量的铜绿丽金龟雌虫触角cDNA文库。本文仅进行了铜绿丽金龟雌虫cDNA文库的构建,由于雌雄两性间常常存在嗅觉差异,大多数昆虫气味结合蛋白的表达与两性存在不关联现象[11-15]。因此,在后续工作中将以雄虫相关试验作为对比补充,逐渐完善铜绿丽金龟触角嗅觉相关蛋白的分析,为后期进一步筛选、克隆以及表达更多嗅觉蛋白,从分子水平全面研究解析铜绿丽金龟嗅觉机制奠定坚实基础。
[1] 刘广瑞,章有为,王瑞,等.中国北方常见金龟子彩色图鉴[M].北京:中国林业出版社,1997.
[2] 姚庆学,张勇,丁岩.金龟子防治研究的回顾与展望[J].东北林业大学学报,2003,31(3):64 66.
[3] 王丙丽,王洪亮.花生田蛴螬的成灾原因及综合治理[J].河南农业科学,2006(7):66 68.
[4] 罗宗秀,李克斌,曹雅忠,等.河南部分地区花生田地下害虫发生情况调查[J].植物保护,2009,35(2):104 108.
[5] 费仁雷,李克斌,肖春,等.铜绿丽金龟发生为害特点及防治[C]∥吴孔明.植保科技创新与病虫防控专业化——中国植物保护学会2011年学术年会论文集.北京:中国农业科学技术出版社,2011.
[6] 晏慧君,黄兴奇,程在全.cDNA文库构建策略及其分析研究进展[J].云南农业大学学报,2006,21(1):1 6.
[7] Endege W O,Steinmann K E,Boardman L A,et al.Representative cDNA libraries and their utility in gene expression profiling[J].Biotechniques,1999,26:542 551.
[8] Zhu Y Y,Machleder E M,Chenchik A,et al.Reverse transcriptase template switching:A SMARTTMapproach for fulllength cDNA library construction[J].Biotechniques,2001,30(4):892 897.
[9] Wellenreuther R,Schupp I,Poustka A,et al.SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones[J].BMCGenomics,2004,5(1):36.
[10]Schuler G D.Pieces of the puzzle:expressed sequence tags and the catalog of human genes[J].Journal of Molecular Medicine,1997,75(10):694 698.
[11]孙凡,王广利,赵奎军.金龟子嗅感器及嗅觉机理的研究进展[J].东北农业大学学报,2007,38(3):411 415.
[12]Györgyi T K,Roby-Shemkovitz A J,Lerner M R.Characterization and cDNA cloning of the pheromone-binding protein from the tobacco hornworm,Manduca sexta:a tissue-specific developmentally regulated protein[J].Proceedings of the National Academy of Sciences,1988,85(24):9851 9855.
[13]Steinbrecht R A,Laue M,Ziegelberger G.Immunolocalization of pheromone-binding protein and general odorant-binding protein in olfactory sensilla of the silk moths Antheraea and Bombyx[J].Cell and Tissue Research,1995,282(2):203 217.
[14]Zhang S,Maida R,Steinbrecht R A.Immunolocalization of odorant-binding proteins in noctuid moths(Insecta,Lepidoptera)[J].Chemical Senses,2001,26(7):885 896.
[15]Callahan F E,Vogt R G,Tucker M L,et al.High level expression of“male specific”pheromone binding proteins(PBPs)in the antennae of female noctuiid moths[J].Insect Biochemistry and Molecular Biology,2000,30(6):507 514.
[16]陈展册,苏丽,戈峰,等.绿盲蝽对性信息素类似物和植物挥发物的触角电位反应[J].昆虫学报,2010(1):47 54.
[17]赵双宜,吴耀荣,夏光敏.介绍一种简单高效的植物总RNA提取方法[J].遗传,2002,24(3):337 338.
[18]Woodhead M,Taylor M A,Davies H V,et al.Isolation of RNA from blackcurrant(RibesnigrumL.)fruit[J].Molecular Biotechnology,1997,7(1):1 4.
[19]张雅琼,郭华春.魔芋叶片中4种总RNA提取方法的比较[J].分子植物育种,2010,8(1):196 200.
[20]赵小兰,苏晓华,赵梁军.多花蔷薇总RNA提取方法[J].中国生物工程杂志,2005,25(9):89 93.
[21]Hofstetter H,Schamböck A,Van Den Berg J,et al.Specific excision of the inserted DNA segment from hybrid plasmids constructed by the poly(d A)·poly(d T)method[J].Biochimica et Biophysica Acta(BBA)-Nucleic Acids and Protein Synthesis,1976,454(3):587 591.
Construction and quality analysis of a full-length cDNA library from antennae ofAnomala corpulentaMotschulsky
Zhao Ying1,2,Yin Jiao2,Li Kebin2,Cao Yazhong2,Wu Junxiang1
(1.Key Laboratory of Integrated Pest Management on Crops in Northwest Loess Plateau,Ministry of
Agriculture,College of Plant Protection,Northwest A&F University,Yangling712100,China;
2.State Key Laboratory for Biology of Plant Disease and Insect Pests,Institute of Plant
Protection,Chinese Academy of Agricultural Sciences,Beijing100193,China)
In order to study the function of the olfactory associated proteins inAnomala corpulentaMotschulsky,a full-length cDNA library was constructed based on SMART technique.Total RNA was extracted from the female antennae ofA.corpulenta.The titer of primary library was6.74×106pfu/mL,and the recombinant rate was 98.98%.The amplified library showed a titer of 1.08×1010pfu/mL.Twenty-four monoclonal colonies had been picked randomly and detected by PCR.The results showed that the sizes of amplified fragments were 0.5-2.0 kb,with an average of 1.0 kb.By PCR and sequencing,this study obtained two full-length gene sequencesAcorOBP1 andAcorOBP2 that coding odorant binding proteins.These experiment data showed that a high-quality full-length cDNA library of the female antennae ofA.corpulentahad been constructed successfully,and also have screened and cloned the odorant binding protein gene sequences,which laying solid foundation for further research on the function of odorant binding proteins.
Anomala corpulenta; antennae; full-length cDNA library
Q 78.5
A
10.3969/j.issn.0529 1542.2015.01.021
2014 01 26
2014 03 01
公益性行业(农业)科研专项(201003025)
*通信作者 E-mail:junxw@nwsuaf.edu.cn
