儿童与成人慢性乙型肝炎患者乙型肝炎病毒Core基因区准种特征及正选择压力差异分析
2015-02-04邓海君黄勇黄爱龙龙泉鑫
邓海君,黄勇,2,黄爱龙,龙泉鑫
儿童与成人慢性乙型肝炎患者乙型肝炎病毒Core基因区准种特征及正选择压力差异分析
邓海君1,黄勇1,2,黄爱龙1,龙泉鑫1
1. 重庆医科大学,感染性疾病分子生物学教育部重点实验室,重庆渝中 400016;2. 重庆医科大学附属第二医院检验科,重庆渝中 400010
儿童与成人慢性乙型肝炎患者的临床特征差异明显。乙型肝炎病毒(Hepatitis B virus, HBV)病毒准种特征与其致病特性紧密相连,HBV病毒Core 基因区富含免疫表位,该区域的准种特征直接反映病毒变异与病毒应对宿主免疫压力间的动态过程。文章通过扩增170名儿童慢性乙型肝炎患者及121名成人慢性乙型肝炎患者病毒Core基因区,按照病毒基因型以及病毒e抗原(Hepatitis B virus e antigen, HBeAg)状态进行分组,使用序列复杂度、多样性、非同义突变率(Non-synonymous substitution ratio,)、同义突变率(Synonymous substitution ratios ,)等指标衡量不同组别之间的病毒准种特征;使用不同模型计算不同组别中受到正选择压力的位点,进一步结合HBV Core基因区免疫表位信息,进行正选择位点的定位分析。结果发现,儿童乙型肝炎病毒患者体内病毒Core基因区序列复杂性和多样性低于成人患者,且前者Core基因区正选择位点个数显著低于后者,这说明儿童慢性乙型肝炎患者体内病毒受到的选择压力低于成人患者。在儿童及成人慢性感染病人组中,HBeAg阳性病人体内病毒受到的选择压力低于HBeAg阴性病人。儿童及成人慢性感染患者体内病毒存在13个正选择位点,大多数正选择位点位于已知的抗原表位上。本研究从分子进化角度揭示了儿童与成人慢性乙型肝炎病例体内病毒Core基因区序列准种差异,为两类病人显著不同的临床表征提供了群体遗传学的解释。
乙型肝炎病毒;Core基因区;儿童病例;正选择压力
乙型肝炎病毒(Hepatitis B virus, HBV)感染仍然是一个重要的公共卫生问题,全世界大约有3.5亿人口感染HBV。HBV感染的临床症状多样,从无症状携带者、急慢性肝炎到肝纤维化、肝癌以及肝衰竭[1],严重威胁人类健康。
乙型肝炎病毒是一个部分闭合的环状双链DNA病毒,但与其他DNA病毒显著不同的是,HBV病毒在复制过程依赖逆转录酶产生的前基因组RNA(Pregenomic RNA)完成,而逆转录酶缺乏校正活性(Proof-reading capacity),故病患体内HBV病毒以一种异质性的种群形式存在(Quasispecies)。众多研究证实HBV准种特征与其药物反应性[2,3]、疾病进程[4~6]等重要临床特征紧密相关。特定病理阶段病毒准种特征反映的是病毒对特定免疫压力的适应。细胞毒性T淋巴细胞(Cytotoxic T lymphocytes, CTL)在宿主清除HBV感染中起主要作用[7],而HBV Core 区域的表位(Epitope)是主要的CTL识别靶区域[8,9],且之前的研究证实HBV Core基因区具有最高的碱基取代率(Substitution rates)[10],故HBV Core区病毒准种特征可能是病毒对宿主免疫压力适应情况的最直接反应。儿童慢性乙型肝炎感染患者与成人患者相比,大部分病例e抗原呈阳性状态[11,12],大部分病患处于以高的病毒滴度与正常的肝功能为特征的免疫耐受期[13],且儿童慢性乙型肝炎患者具有更多的HBV特异性的T淋巴细胞[14],这些特征提示儿童慢性乙型肝炎患者与成人患者体内病毒准种特征可能存在较大差异。本研究收集了儿童慢性HBV感染患者及成人慢性HBV感染患者的血清标本,通过PCR扩增的方法获得Core基因区序列,通过一系列分子进化手段获得两病人群体在Core基因区的准种差异,以期从群体遗传学角度为不同慢乙肝感染的临床表征提供解释。
1 材料和方法
1.1 研究对象
随机选取2011~2013年间在重庆医科大学附属儿童医院、附属第一医院及附属第二医院就诊的170例慢性HBV感染儿童患者及121例成人患者血清样本作为研究对象。所有患者血清均为乙型肝炎病毒表面抗原阳性(Hepatitis B virus surface antigen, HBsAg),乙型肝炎病毒表面抗体阴性(Hepatitis B virus surface antibody, HBsAb),并经血清学等检查排除甲型肝炎病毒(Hepatitis A virus, HAV)、丙型肝炎病毒(Hepatitis C virus, HCV)、丁型肝炎病毒(Hepatitis D virus, HDV)、戊型肝炎病毒(Hepatitis E virus, HEV)及人免疫缺陷病毒(Human immunodeficiency virus, HIV)重叠感染。
1.2 巢氏PCR扩增HBV Core基因区
取200 μL患者血清,利用Qiagen公司的QIAamp DNA Blood Mini kit按照说明书要求提取血清中的病毒DNA。根据GenBank已公布的HBV基因组序列,针对Core基因区两段保守区域设计两对引物,分两轮巢氏PCR扩增HBV Core基因,引物序列信息见表1。PCR扩展方法参考文献[15]。

表1 HBV Core基因巢氏PCR扩增引物
1.3 HBV Core基因PCR产物测序、基因分型及比对分析
PCR扩增阳性产物由Invitrogen公司直接测序。临床样本测序结果采用NCBI Genotyping工具(http: //www.ncbi.nlm.nih.gov/projects/genotyping/formpage.cgi)进行基因分型。从GenBank中下载784条B基因型及1289条C基因型HBV全基因组序列,采用ClustalW软件分别进行比对分析,计算B基因型及C基因型每个核苷酸位点的多样性,以各位点比率最高的核苷酸作为该位点的一致性序列,采用ClustalW分析软件,将临床样本测序结果分别与B、C基因型一致性序列进行比对和分析。
1.4 Core基因病毒准种复杂度分析
采用ClustalW软件将每组中的核苷酸和氨基酸序列进行比对,使用MEGA 6软件计算序列两两之间的核苷酸进化距离(Nucleotide distance)和氨基酸进化距离(Amino acid distance)。利用KaKs_Calculator软件,采用GMYN方法计算序列两两之间的非同义突变率(Non-synonymous substitution ratio,)、同义突变率(Synonymous substitution ratios,)和/。用香农熵(Shannon entropy,)来评估位点复杂度。香农熵计算公式如下:
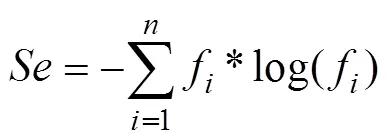
其中,代表每个位点碱基种类,f代表碱基的频率。
1.5 Core基因区正选择位点分析
采用ClustalW软件,将临床样本测序结果进行序列比对,利用MEGA 6软件包中的Neighbor- Joining(NJ)方法构建系统进化树,采用Bootstrap方法,重复1000次,检验系统进化树的可靠性。然后采用PAML软件中的Codeml包进行正选择位点分析,对每对模型之间的对数似然比(Log likelihood values, LLR)采用似然比检验法(Likelihood ratio test, LRT)进行假设检验(=2),评估氨基酸位点是否受到正选择压力的作用。选择M0、M1、M2、M7和M8等5种模型,其中M0用于评估Neighbor-Joining法构建的系统进化树的分支长度,同时该长度作为其余模型使用的进化树长度的初始值。使用两对模型进行比较:M1 vs. M2和M7 vs. M8,其中,M2和M8检测均使用贝叶斯方法检测正选择位点,选用贝叶斯经验检验(Bayes empirical Bayes, BEB)为正选择位点鉴定的结果。
1.6 统计学分析
采用SPSS 17.0进行统计学分析,连续性变量参数资料的比较采用Wilcoxon-Mann-Whitney检验。离散变量资料率的比较采用卡方检验或Fisher精确检验。定义< 0.05为差异有统计学意义。
2 结果与分析
2.1 病例基本临床信息
病人临床信息如表2所示,共收集到儿童病例170例,成人病例121例。儿童病例Alanine transaminase, ALT)水平显著低于成人病例(53.451± 63.714 vs. 105.967±96.242,<0.001),病毒DNA水平高于成人病例(6.770±2.091 vs. 6.324±1.356,=0.001),儿童病例显著呈现免疫耐受状态。两组病人的e抗原构成比并不具有统计学差异,从而避免患者e抗原状态构成比影响实验结果。
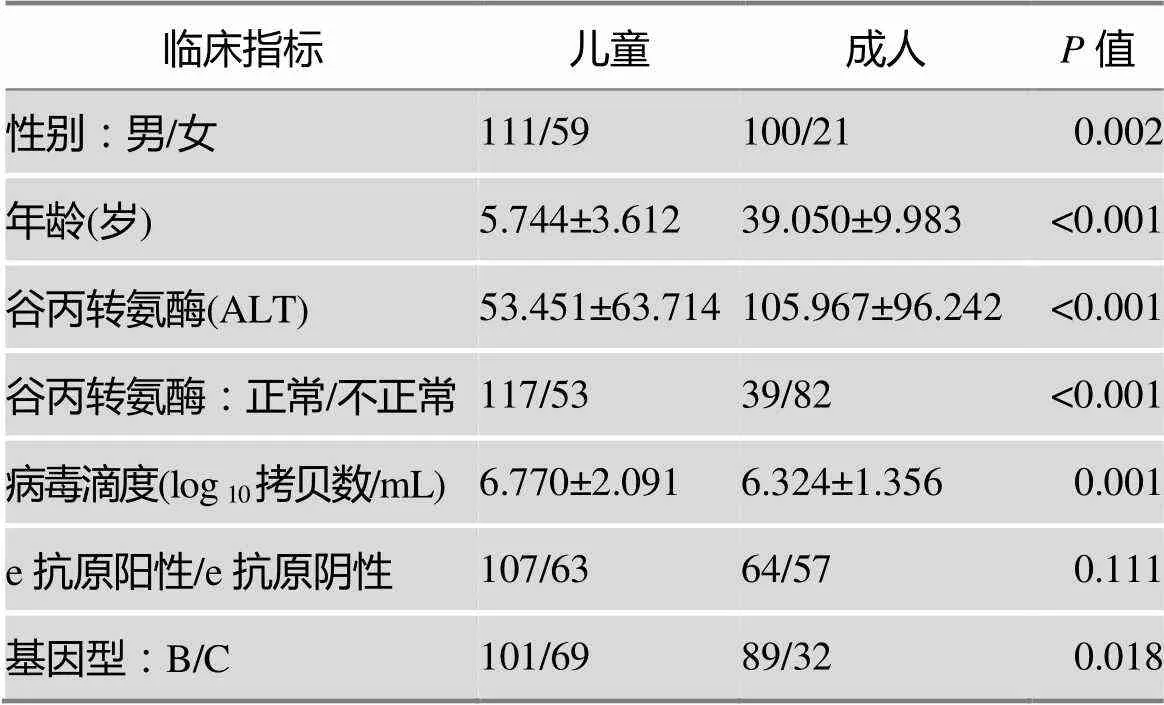
表2 本研究所招募的慢性乙型肝炎病人临床信息
2.2 儿童HBV慢性感染病例和成人HBV慢性感染病例Core基因区异质性分析
比较儿童慢性乙型肝炎及成人慢性乙型肝炎患者体内病毒Core基因区序列,发现两组病人病毒序列的复杂度在该区域并无统计学差异(>0.05)。比较两组病人序列多样性,发现无论在核酸水平还是在蛋白水平,儿童组序列平均遗传距离(Mean genetic distance)均小于成人组(<0.001);儿童组序列的非同义突变也小于成人组非同义突变数值,而两组病人的并不存在统计学差异,儿童组/也低于成人组,说明在选择压力没有放松的情况下,成人组显然受到了更强的正选择压力(表3)。
考虑到不同基因型病毒序列之间存在较大的差异,为了排除群体序列复杂度和多样性的计算中因为基因型差异带来的假阳性结果,本文按照基因型进行分组。同时,考虑到e抗原状态是一个重要的HBV慢性感染患者临床指标,且e抗原状态与患者体内病毒准种变迁密切相关,因此本文进一步按照病人e抗原状态分组,比较不同e抗原状态下成人慢性感染病例和儿童慢性感染病例体内病毒进化差异。可以看出,在各分组病例中,成人病例病毒准种的、、核苷酸平均遗传距离、氨基酸平均遗传距离等数值都显著高于儿童病例,只有基因B型e抗原阳性病例中,儿童与成人病例体内病毒序列复杂度并不存在显著差异(>0.05)。HBeAg阴性病例病毒Core基因相对于对应HBeAg阳性病例病毒Core基因承受了更高的选择压力。
2.3 不同病例群体体内病毒Core基因区正选择位点统计
各组中正选择位点检测结果见表4。似然比检测分析发现,对于B基因型患者,儿童组e抗原阳性患者中,正选择模型M2与接近中性模型M1进行比较,LRT值在=2水平不显著,且未发现有正选择位点;M8(β和ω)模型与M7(β)模型比较,LRT在df=2水平同样不显著,没有发现正选择位点。在B基因型患者中,儿童e抗原阴性患者和成人e抗原阴性患者M2 vs. M1和M8 vs. M7比较LRT均差异显著,分别检测出8个和5个正选择位点。成人e抗原阳性B基因型型患者中,M2 vs. M1和M8 vs. M7比较LRT差异不显著,但M8模型仍检测出4个正选择位点(P5、P130、P135和G153)。在C基因型患者中,儿童e抗原阴性患者M2 vs. M1和M8 vs. M7的LRT检验均差异不显著,且未发现正选择位点;儿童e抗原阳性患者M2 vs. M1和M8 vs. M7的LRT检验也差异不显著,未检测到正选择位点。在成人C基因型患者中,e抗原阳性患者的M2 vs. M1和M8 vs. M7比较LRT检测不显著,但检测到P130一个正选择位点;成人e抗原阴性C基因型患者组M2 vs. M1和M8 vs. M7比较LRT检验差异显著,检测到4个正选择位点(L84、P130、Q151和S155)。对所有正选择位点在各组中的频率分析发现,正选择位点的突变频率都较高(图1)。
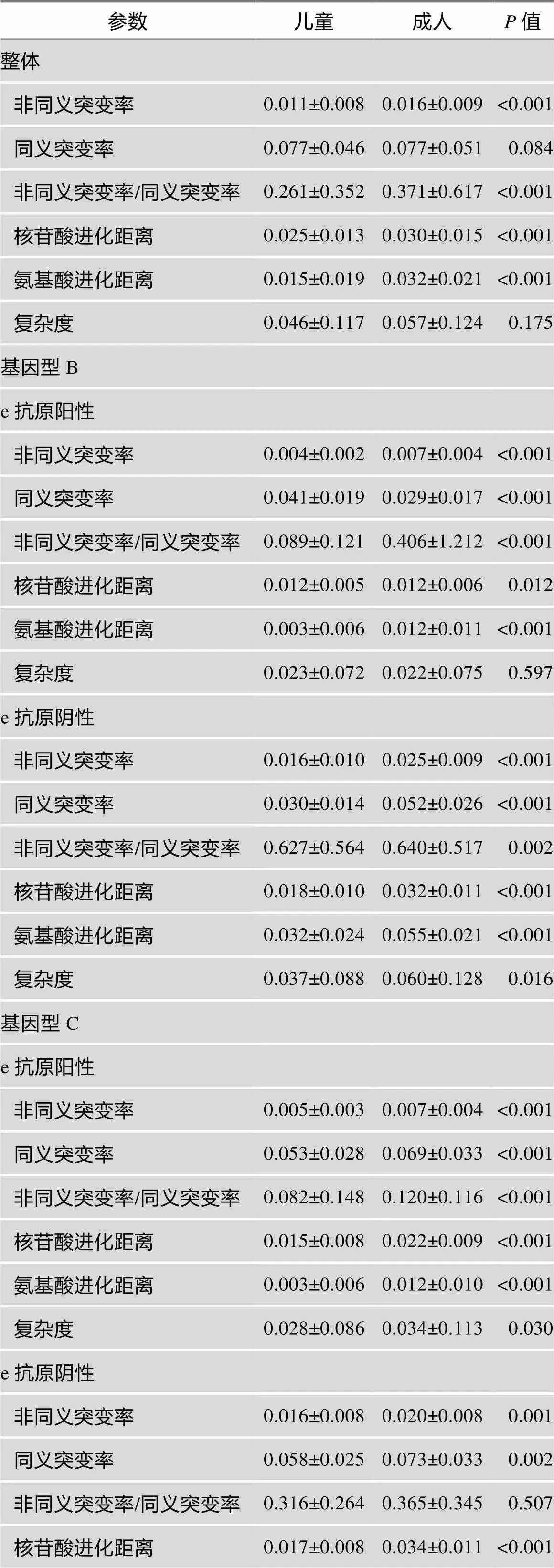
表3 儿童及成人患者体内病毒Core基因区准种复杂度及多样性比较
注:*:<0.05;**:<0.01;***:<0.001。
在同一基因型病例中,e抗原阴性患者群体中正选择位点数明显多于e抗原阳性患者群体;除基因B型e抗原阴性患者群体外,其他3类患者群体中,成人病例中检出的正选择位点个数高于儿童病例。这提示e抗原阴性状态以及较高年龄患者体内HBV Core基因区承受了更高的选择压力。
按照病毒基因型,将不同患者群体正选择位点进行“交汇”分析,结果如图2所示。对于基因B型病毒序列而言,P135位点作为一个在e抗原阳性成人患者、e抗原阴性成人患者以及e抗原阴性儿童患者中都存在的正选择位点,可能是一个重要的宿主免疫识别位点。

图1 不同组别病人体内HBV病毒Core基因正选择位点突变频率展示
图中横坐标各点表示本研究中筛选得到的正选择位点,阴影方框表示该位点在特定组别病人中被确定为正选择位点,且阴影深度表示该位点突变率,颜色越深,表示该位点突变率越高。
2.4 不同病人群体体内HBV Core基因区正选择位点免疫表位特性分析
对各病例群体的承受正选择位点的氨基酸进行免疫表位的分析,结果见表5,免疫表位信息根据文献来确认[16,17]。与文献报道的确定HBV Core基因区免疫表位进行对比,发现大多数正选择位点都位于相应的免疫表位中。在B基因型乙型肝炎病毒感染样本中,共筛选得到10个正选择位点,其中6个位点位于已知抗原表位区段,2个位点(S49和I97)与已知抗原表位邻接。在C基因型样本中,共筛选得到4个正选择位点,其中3个位点位于已知抗原表位区段。在各病例群体中筛选得到的正选择位点,均超过60%的位点处于已验证免疫表位之中,再次证明这些正选择位点所受的选择压力往往是来自宿主免疫系统。
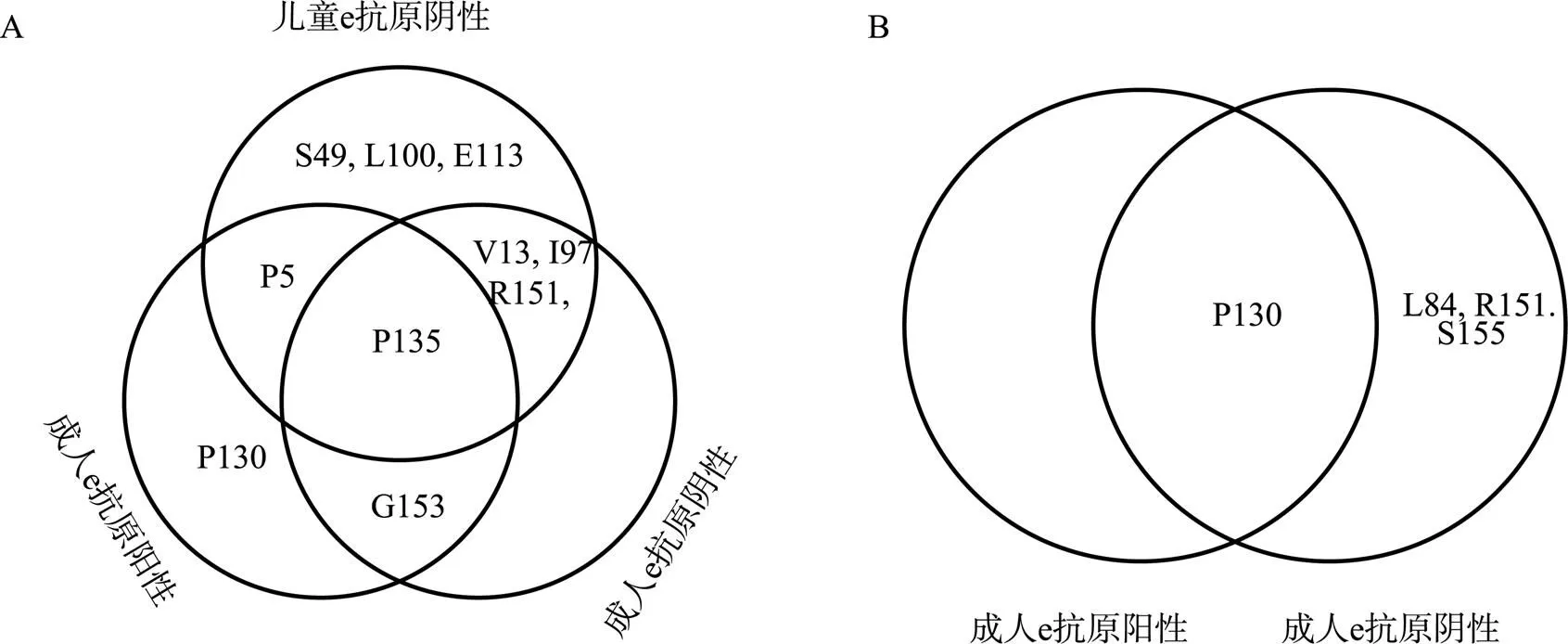
图2 不同基因型HBV病毒正选择位点“交汇”分析
A:B基因型正选择位点;B:C基因型正选择位点。
3 讨 论
儿童慢性乙型肝炎患者在临床表征上与成人慢性乙型肝炎患者区别较大区别,本文对两患者群体体内病毒基因组Core基因区的准种特征进行描述,并使用基于密码子的最大似然法分析了该基因区的正选择压力。研究结果揭示儿童与成人患者在Core基因区的准种特征存在显著差异,两群体的该区域序列具有显著不同的多样性:成人患者群体序列多样性显著高于儿童患者群体。病毒的遗传多样性主要是受宿主免疫系统选择压力和病毒群体规模影响[18,19]。即使在儿童组病毒群体规模高于成人病例(入组的儿童患者数多于成人患者数,且儿童病毒载量高于成人组病毒载量),通过计算得到的Core区域序列多样性仍然是成人患者组高于儿童患者组,这充分证明了成人慢性乙型肝炎患者经受了更高的免疫选择压力。强免疫压力环境下病毒准种呈现高的复杂度与多样性在急性肝衰竭病人(Acute-on-chronic liver failure, ACLF)与普通慢性乙型肝炎患者比较研究中也有发现。
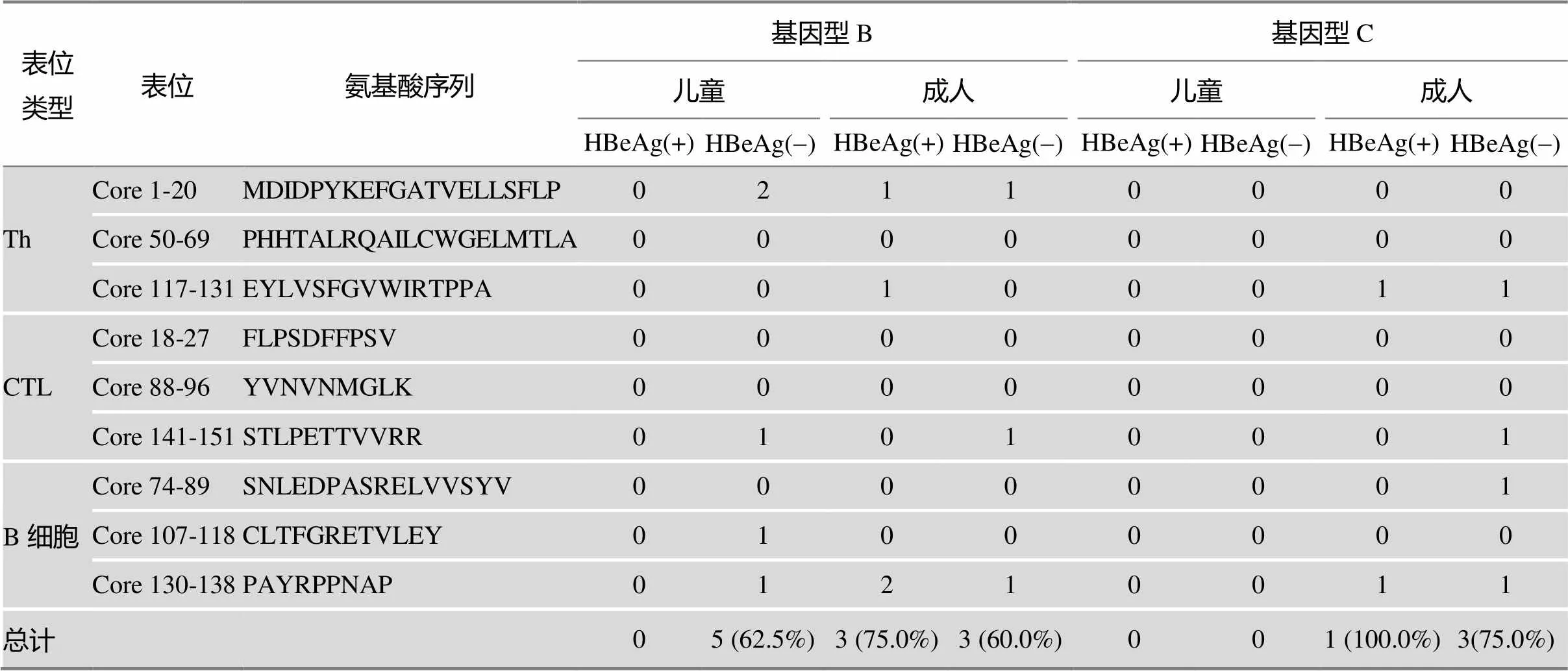
表5 不同组别慢乙肝感染病例体内病毒Core基因区正选择位点免疫表位特性分析
注:“总计”表示特定病人群中位于免疫表位的正选择位点个数,位于不同免疫表位的同一个正选择位点只计算一次;百分比表示特定病人群中位于免疫表位的正选择位点的百分比。
不同基因型病毒序列拥有不同的正选择位点。基因B型和基因C型病毒Core基因区分别具有10个和4个正选择位点。只有少数位点在两种不同基因型乙型肝炎病毒都被正选择压力筛选,可能与乙型肝炎病毒流行所呈现的地域特性有关,这与之前文献报道一致[20]。
本文进一步将各组患者人Core基因区正选择位点进行分析,成人e抗原阴性患者Core基因区存在显著多的正选择位点(M2模型)。这说明儿童患者体内病毒Core基因区域更多的处于中性进化(Natural evolution),而成人患者体内病毒该区域则更多的受正选择压力的影响,再次证明成人患者体内病毒受更大的免疫选择压力影响。更为重要的是,大多数的这些正选择位点都位于HLA-限制性表位之中,可能导致相关表位与对应HLA分子结合力下降。这些处于表位中的正选择位点的存在,可能对于病毒逃逸宿主免疫监视(Immune surveillance),保持自身存活非常重要。另外一种解释是,HLA限制表位发生突变后,会打破免疫耐受,促使发生T 淋巴细胞介导的组织免疫损伤。这也部分解释儿童往往处于免疫耐受阶段,即以高的病毒DNA滴度和正常的肝功能水平为体现,而成人处于免疫激活阶段(通常具有肝功能损伤表现)。另外,在e抗原阴性患者中筛选得到的正选择位点显著高于e抗原阳性患者。类似的现象在之前的纵向研究中也有报道[21],推测可能是因为HBeAg血清学转换前后CD8+ T细胞被激活后引起的选择压力增强[22]。这些正选择位点的出现往往影响HBV相关表位肽与HLA分子结合进而引起T细胞识别位点发生改变[23]。此外,在P135位点在多组B基因型病毒感染患者群中被确定为正选择位点,提示该位点可能对于宿主免疫识别的重要作用。有文献显示,P135所在PPAY结构域的突变可能导致新生HBV核衣壳的装配出现障碍[24]。
通过使用分子进化手段,本文首次从群体遗传的角度阐述了儿童慢性乙型肝炎患者与成人慢性乙型肝炎患者在病毒Core基因区的差异,结果证实成人慢性乙型肝炎病患经受了更高的免疫选择压力;成人患者群体体内病毒受宿主免疫压力筛选,获得了更多的位于表位中的正选择位点,一方面有利于病毒逃避宿主免疫监视,获得更多存活机会,另一方面也可能会打破免疫耐受,发生肝脏组织免疫损伤。
[1] Lee WM. Hepatitis B virus infection, 1997, 337(24): 1733–1745.
[2] Peveling-Oberhag J, Herrmann E, Kronenberger B, Farnik H, Susser S, Sarrazin C, Zeuzem S, Hofmann WP. Dynamicsof hepatitis B virus quasispecies heterogeneity and virologicresponse in patients receiving low-to-moderate genetic barrier nucleoside analogs, 2013, 20(4): 234–239.
[3] Rodriguez C, Chevaliez S, Bensadoun P, Pawlotsky JM. Characterization of the dynamics of hepatitis B virus resistance to adefovir by ultra-deep pyrosequencing, 2013, 58(3): 890–901.
[4] Li F, Zhang D, Li Y, Jiang D, Luo S, Du N, Chen W, Deng L, Zeng C. Whole genome characterization of hepatitis B virus quasispecies with massively parallel pyrosequencing, 2014, 21(3): 280–287.
[5] Chen L, Zheng CX, Lin MH, Huang ZX, Chen RH, Li QG, Li Q, Chen P. Distinct quasispecies characteristics and positive selection within precore/core gene in hepatitis B virus HBV associated acute-on-chronic liver failure, 2013, 28(6): 1040–1046.
[6] Cheng Y, Guindon S, Rodrigo A, Wee LY, Inoue M, Thompson AJV, Locarnini S, Lim SG. Cumulative viral evolutionary changes in chronic hepatitis B virus infection precedes hepatitis B e antigen seroconversion, 2013, 62(9): 1347–1355.
[7] Gehring AJ, Sun DX, Kennedy PTF, Nolte-'t Hoen E, Lim SG, Wasser S, Selden C, Maini MK, Davis DM, Nassal M, Bertoletti A. The level of viral antigen presented by hepatocytes influences CD8 T-cell function, 2007, 81(6): 2940–2949.
[8] Osiowy C, Giles E, Tanaka Y, Mizokami M, Minuk GY. Molecular evolution of hepatitis B virus over 25 years, 2006, 80(21): 10307–10314.
[9] Wang HY, Chien MH, Huang HP, Chang HC, Wu CC, Chen PJ, Chang MH, Chen DS. Distinct hepatitis B virus dynamics in the immunotolerant and early immunoclearance phases, 2010, 84(7): 3454–3463.
[10] van de Klundert MA, Cremer J, Kootstra NA, Boot HJ, Zaaijer HL. Comparison of the hepatitis B virus core, surface and polymerase gene substitution rates in chronically infected patients, 2012, 19(2): e34–e40.
[11] Popalis C, Yeung LTF, Ling SC, Ng V, Roberts EA. Chronic hepatitis B virus (HBV) infection in children: 25 years' experience, 2013, 20(4): e20–e26.
[12] Paganelli M, Stephenne X, Sokal EM. Chronic hepatitis B in children and adolescents, 2012, 57(4): 885–896.
[13] Ni YH. Natural history of hepatitis B virus infection: pediatric perspective, 2011, 46(1): 1–8.
[14] Kennedy PTF, Sandalova E, Jo J, Gill U, Ushiro-Lumb I, Tan AT, Naik S, Foster GR, Bertoletti A. Preserved T-cell function in children and young adults with immune-tolerant chronic hepatitis B, 2012, 143(3): 637–645.
[15] Huang Y, Deng HJ, Peng Z, Huang Y, Long QX, Huang AL. Mutations of Basal core promoter and precore regions in hepatitis B virus genotypes B and C, 2015, 15(1): e23034.
[16] Desmond CP, Bartholomeusz A, Gaudieri S, Revill PA, Lewin SR. A systematic review of T-cell epitopes in hepatitis B virus: identification, genotypic variation and relevance to antiviral therapeutics, 2008, 13(2): 161–175.
[17] Akarca US, Lok ASF. Naturally occurring hepatitis B virus core gene mutations, 1995, 22(1): 50–60.
[18] Stumpf MPH, Pybus OG. Genetic diversity and models of viral evolution for the hepatitis C virus, 2002, 214(2): 143–152.
[19] Domingo E, Gomez J. Quasispecies and its impact on viral hepatitis, 2007, 127(2): 131–150.
[20] Xu Z, Wu GH, Li FF, Bai J, Xing WJ, Zhang DK, Zeng CQ. Positive selection signals of hepatitis B virus and their association with disease stages and viral genotypes, 2013, 19: 176–187.
[21] Lim SG, Cheng Y, Guindon S, Seet BL, Lee LY, Hu PZ, Wasser S, Peter FJ, Tan T, Goode M, Rodrigo AG. Viral quasi-species evolution during hepatitis Be antigen seroconversion, 2007, 133(3): 951–958.
[22] Rehermann B, Lau D, Hoofnagle JH, Chisari FV. Cytotoxic T lymphocyte responsiveness after resolution of chronic hepatitis B virus infection, 1996, 97(7): 1655–1665.
[23] Bertoletti A, Costanzo A, Chisari FV, Levrero M, Artini M, Sette A, Penna A, Giuberti T, Fiaccadori F, Ferrari C. Cytotoxic T lymphocyte response to a wild type hepatitis B virus epitope in patients chronically infected by variant viruses carrying substitutions within the epitope, 1994, 180(3): 933–943.
[24] Garcia ML, Reynolds TD, Mothes W, Robek MD. Functional characterization of the putative hepatitis B virus core protein late domain using retrovirus chimeras, 2013, 8(8): e72845.
(责任编委: 岑山)
Distinct quasispecies characteristics and positive selection within the core gene in chronic hepatitis B virus infected child and adult patients
Haijun Deng1, Yong Huang1,2, Ailong Huang1, Quanxin Long1
There are significant differences in clinical characteristics between chronic hepatitis B virus infected (CHB) child and adult patients. Viral quasispecies characteristics are associated with its pathogenic properties. For hepatitis B virus (HBV), its core region is the main immune recognition region for its enriched epitopes. In our study, we discuss the quasispecies characteristics and positive selection within core gene within chronic HBV infected child and adult patients. By analyzing 170 core gene sequences from child CHB patients and 121 core genes sequences from adult CHB patients, quasispecies characteristics were described by sequence complexity, diversity, non-synonymous substitution ratio () and synonymous substitution ratios (). In addition, positive selection sites were also determined by bioinformatics tools. Then, all these parameters were compared between child and adult CHB patient groups. Compared with child patients, adult patients with CHB showed distinct quasispecies characteristics within the core region, had a higher sequence complexity and diversity and more positive selection sites, suggesting that the adult CHB patients had a higher immune selection pressure on the HBV core gene. Reduced selection pressure on the HBV core gene in hepatitis B e antigen (HBeAg)-positive CHB patients than HBeAg negative CHB patients were observed in both adult and child patient groups. The majority of the screened positive selection sites lay within human leukocyte antigens (HLA)-restricted epitopes. In conclusion, this study analyzed the quasispecies characteristics discrepancy between child and adult patients with CHB, and revealed the possible reason for the distinct clinical characteristics in the perspective of population genetics.
hepatitis B virus; core region; child patients; positive selection
2015-02-02;
2015-03-20
十二五国家传染病重大专项(编号:2013ZX10002002)和重庆市科委基础与前沿研究计划(编号:cstc2013jcyjC10002)资助
邓海君,硕士研究生,专业方向:乙型肝炎病毒准种特征分析。E-mail: 164943607@qq.com
龙泉鑫,助理研究员,研究方向:乙型肝炎病毒准种特征分析。E-mail: longquanxin@gmail.com
10.16288/j.yczz.15-060
2015-4-13 9:26:01
http://www.cnki.net/kcms/detail/11.1913.R.20150413.0926.001.html
