长链非编码RNA UCA1在胃癌组织中的表达及其效应分析
2015-01-18滕卫军郑晓娟华宏军林峰
滕卫军 郑晓娟 华宏军 林峰
长链非编码RNA UCA1在胃癌组织中的表达及其效应分析
滕卫军 郑晓娟 华宏军 林峰
目的钼分析胃癌组织特征长链非编码RNA(lncRNA)表达谱,并探讨长链非编码RNA UCA1在胃癌中的表达及效应。方法钼采用基因芯片检测6例胃癌患者的肿瘤组织及癌旁正常组织LncRNA表达水平,分析胃癌组织特征lncRNA表达谱,进一步通过定量PCR检测40例胃癌及其对应的癌旁正常组织中UCA1的表达并分析淋巴结转移、肿块大小、细胞分化程度及病理分期等临床病理特征的关系。在此基础上,利用siRNA下调AGS胃癌细胞内UCA1表达水平,利用CCK-8试剂盒检测UCA1干预对胃癌细胞增殖能力的影响。结果 LncRNA基因芯片检测到胃癌组织1 021条差异>2倍的lncRNA(P<0.05),定量PCR验证明显差异表达的UCA1在胃癌组织中的表达显著高于正常组织(P<0.01)。此外,临床病理相关分析显示,UCA1表达水平还与胃癌的细胞分化程度及病理分期相关。分化低的胃癌组织中UCA1表达水平明显高于中、高分化的胃癌组织(P<0.05);病理分期Ⅲ/Ⅳ期胃癌组织的UCA1表达水平明显高于Ⅰ/Ⅱ期的标本(P<0.05)。此外,下调UCA1胃癌细胞表达可明显抑制细胞增殖。结论 异常表达的lncRNA参与了胃癌的发生、发展,其中肿瘤组织上调表达的UCA1是胃癌重要的促癌基因。
胃癌 长链非编码RNA 细胞增殖
胃癌是我国最常见的恶性肿瘤之一,其发病机制尚未完全阐明。从分子水平阐明胃癌发生、发展机制,将为寻求新的胃癌标记物及有效的生物治疗靶点提供新的思路。长链非编码RNA(long non-coding RNA,lncRNA)是一类转录本长度超过200nt的RNA,它们本身并不编码蛋白,而是以RNA的形式在多种层面上调控基因的表达水平[1-4]。lncRNA在转录水平、转录后水平以及表观遗传等多个层面参与了X染色体沉默、基因组印记以及染色质修饰、转录激活、转录干扰、核内运输等多种重要的调控过程[1-4]。近年来人们开始关注lncRNA在各种生物学过程以及疾病中所起到的作用,尤其是癌症[5-6]。有研究表明lncRNA与肿瘤细胞增殖、侵袭转移及复发高度相关[7],但目前关于lncRNA在胃癌中的研究尚停留在起步阶段。现我们采用lncRNA基因芯片检测胃癌组织及癌旁正常组织lncRNA的表达水平,筛选胃癌组织特征lncRNA表达谱,在此基础上选择胃癌组织上调最明显的UCA1(LINC00178),通过定量PCR进行验证,并通过体外干预实验,分析其对胃癌细胞增殖的影响。
1 材料和方法
1.1 实验标本 40例胃癌组织及配对癌旁正常组织标本来自2008-2012年在两家医院行手术治疗的胃癌患者。患者术前均未行放、化疗,年龄42~80岁,中位年龄65岁;组织病理类型的判断参照WHO胃癌的病理学分类标准,TNM分期参照UICC/AJCC 2002年标准。每例标本留取肿瘤组织及对应的距肿瘤5cm以上的癌旁正常胃黏膜组织(经组织病理切片检测证实),离体后10min内置于液氮中保存待做后续分析。
1.2 材料和试剂 Trizol试剂购自美国Invitrogen公司;DNase I、RNase-free试剂购自美国Thermo公司;ReverTra AceRqPCR RT Kit、SYBRRGreen Realtime PCR Master Mix等购自日本Toyobo公司;CCK-8(Cell Counting Kit-8)细胞增殖/毒性检测试剂盒购自日本同仁研究所;人胃癌AGS细胞购自中科院上海细胞库;Arraystar Human LncRNA Microarray V3.0芯片购自上海康成生物技术有限公司;LncRNAsiRNA和Negative Control由上海吉玛公司合成;引物由上海英骏生物技术有限公司合成。
1.3 实验方法
1.3.1 总RNA的提取 取液氮保存的胃癌组织及癌旁正常胃黏膜组织标本,在液氮中碾碎至粉状,按Trizol试剂说明书提取总RNA,DEPC处理过的蒸馏水溶解。为去除可能污染的DNA,抽提的RNA溶液按DNase I说明书步骤进一步纯化。采用NanoDrop2000超微量分光光度计(美国Thermo公司)检测RNA溶液OD260/OD280,计算RNA浓度和纯度,OD260/OD280比值>1.8方可用于检测。1.0%琼脂糖变性凝胶电泳检测RNA的完整性。
1.3.2 胃癌组织lncRNA表达谱检测 抽提6例胃癌患者的肿瘤组织及癌旁正常组织的RNA,根据Arraystar Human lncRNA Microarray V3.0芯片检测的说明书,检测30 586条lncRNA的表达,选择差异倍数>2,P<0.05的lncRNA,采用非监督等级聚类方法分析胃癌组织特征lncRNA表达谱。
1.3.3 lncRNA UCA1表达分析 以GAPDH作为内参,分析UCA1表达。相关分析的引物序列见表1。2μg总RNA按照ReverTraAcerTqPCR RT Kit说明书进行逆转录。以20μl反应体系进行real-time PCR。定量PCR检测反应体系包括:1μl RT产物,1×SYBR Green IMaster mix,10μmol/L特异前向引物、10μmol/L特异反向引物。real-time PCR条件为:95℃ 5min后,94℃ 15s,60℃1min,72℃30s,40个循环。所有样品做3复孔。realtime PCR使用Bio-Rad CFX96仪器进行。根据待测标本的Ct值,以GAPDH为内参照,采用定量PCR中的相对定量法,以N=2-ΔΔCt表示标本中CXCL14相对表达量,其中△△Ct=(CtUCA1-CtGAPDH)肿瘤-(CtUCA1-Ct-GAPDH)正常。

表1 定量PCR检测相关引物序列
1.3.4 细胞增殖与活性检测 AGS胃癌细胞接种于含10%胎牛血清的RPMI-1640培养基中,在CO2体积分数为5%,37℃条件下培养、传代。每2~3d传代1次,取对数生长期的细胞用于实验。为分析UCA1干预对细胞增殖与活性的影响,我们进一步采用siRNA干预胃癌细胞UCA1表达,1×105细胞/100μl接种96孔板,以20、40、80nM等浓度的 UCA1siRNA,按照 Lipofectamine2000转染试剂说明书转染细胞,同时设未转染空白对照及阴性对照。参照CCK-8试剂盒说明书,用酶标仪测定在450nm处的OD值,检测细胞相对活性=实验组OD值/空白对照组OD值。
1.4 统计学处理 采用SPSS 17.0统计软件,测得计量资料采用中位数(四分位间距)表示,UCA1在胃癌及其配对癌旁组织表达水平比较采用配对的非参数Wilcoxon符号秩检验;胃癌组织UCA1相对表达水平与胃癌临床病理指标之间的关系采用Mann-Whitney U检验(两组)和Kruskal-Wallis H检验(多组)进行分析。
2 结果
2.1 胃癌组织lncRNA表达谱分析 6例胃癌组织及其配对的癌旁正常组织30 586条lncRNA中,与癌旁正常组织相比,1 021条lncRNA在胃癌组织的表达差异倍数>2(P<0.05),其中611条lncRNA明显上调,410条lncRNA明显下调。上调和下调表达最明显的10条lncRNA见表2。
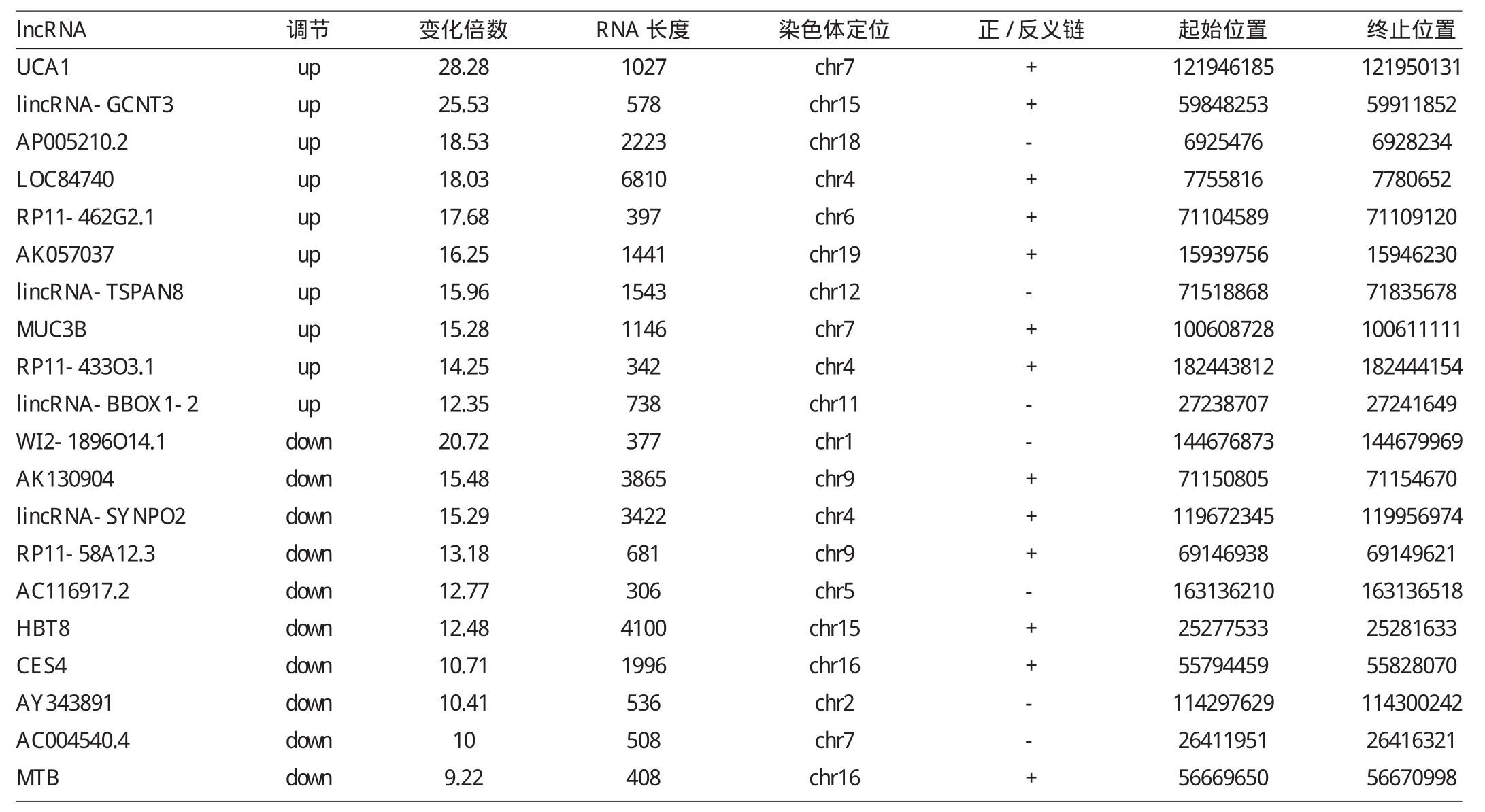
表2 胃癌组织差异表达最明显的lncRNA
2.2 UCA1表达水平在胃癌组织中表达的验证及其与胃癌临床病理特征的关系 在上述芯片筛选的胃癌组织特征lncRNA表达谱中,UCA1(LINC00178)上调表达最明显,与癌旁正常对照比较,胃癌组织中表达上调达28.28倍。采用定量PCR分析对40例胃癌及其配对癌旁组织UCA1表达水平也进一步证实芯片筛选的结果(图1),采用配对样本的Wilcoxon符号秩检验其差异有统计学意义(P<0.01)。

图1 UCA1在胃癌及其配对癌旁组织中的表达
进一步分析UCA1表达水平与胃癌临床病理特征的关系显示,UCA1的表达水平在不同细胞分化程度及病理分期存在统计学差异,低分化胃癌组织的UCA1表达水平明显高于中高分化的标本(P<0.05);病理分期Ⅲ/Ⅳ期胃癌组织的UCA1表达水平明显高于Ⅰ/Ⅱ期的水平(P<0.05)。UCA1的表达水平和性别、年龄、肿块位置、肿块大小、肿块侵袭深度及淋巴结转移情况无关(均P>0.05),见表3。
2.3 下调UCA1表达对胃癌细胞增殖的影响 见图2。由图2可见,与空白对照、阴性对照比较,20、40及80nM浓度的UCA1 siRNA干预实验组细胞增殖均明显受到抑制(均P<0.05)。
3 讨论
在后基因组时代,RNA组学研究逐渐成为热点:基因组超过98%的转录产物为不编码蛋白的非编码RNA,lncRNA是一类转录本长度超过200nt的RNA,它们本身并不编码蛋白,起初lncRNA被认为是基因组转录的“噪音”,近年越来越多的研究表明,lncRNA可在表观遗传学、转录及转录后水平调控机体生理、病理过程而受到关注。lncRNA通常分为5类:即正义(Sense)、反义(Antisense)、双向(Bidirectional)、基因内(Intronic)及基因间(Intergenic)lncRNA[8]。目前发现lncRNA与很多的临床意义。
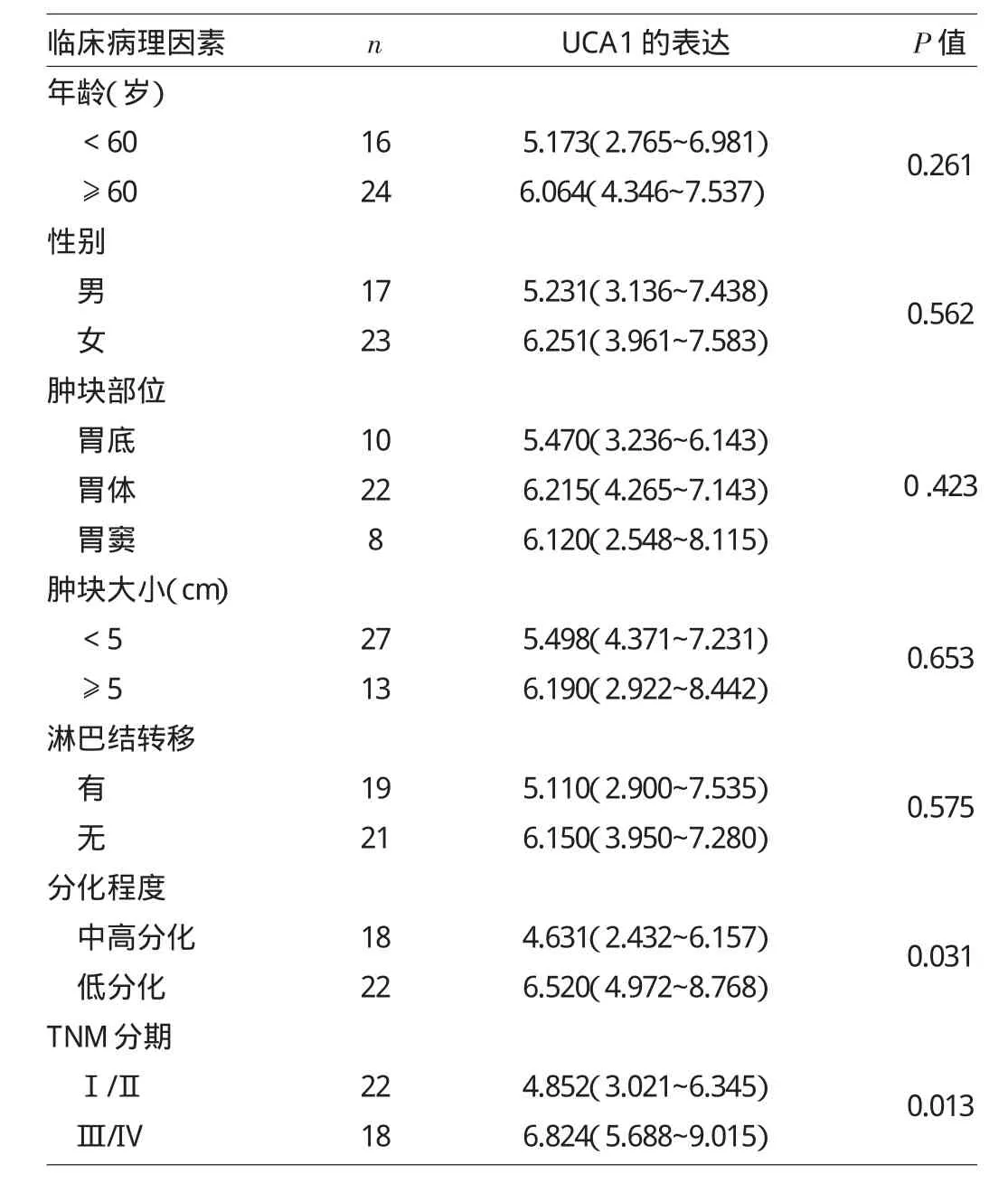
表3 UCA1表达水平与胃癌临床病理特征的关系
本研究利用lncRNA基因芯片检测6例胃癌组织及其配对的癌旁正常组织lncRNA的表达水平,发现了1 021条在胃癌组织异常表达lncRNAs,其中lincRNABBOX1-2、CR594506及UCA1等lncRNA已被证实[18]。利用(http://www.ebiomed.org/ncFANs/or http://www.noncode.org/ncFANs/)提供的ncFANs(non-coding RNA Function Annotation Server)网络界面分析也显示,多条lncRNA也与肿瘤发生密切相关[19-20]。定量PCR分析也进一步验证了上调最明显的UCA1(LINC00178)长链非编码肿瘤的发生、发展密切相关,已成为肿瘤研究的新方向[8-9]。

图2 UCA1siRNA干预对AGS胃癌细胞细胞增殖的影响(A:siRNA干预胃癌细胞对UCA1表达影响;B:UCA1 siRNA干预对AGS胃癌细胞增殖的影响。与空白对照及阴性对照比较,*P<0.05)
近年来的研究表明,lncRNA在肿瘤中非常敏感,是肿瘤特异标记物,同时,lncRNA也可以作为肿瘤治疗的分子靶点[10-12]。例如,lncRNA与结肠直肠癌的发生、发展密切相关,其可通过DNA甲基化、组蛋白修饰等多种途径参与结肠直肠癌的发生、发展过程[13-14]。HULC已经被鉴定出在肝癌及肠癌肝转移的组织中特异性高表达,是临床肝癌的诊断和预后判断的生物标记[10]。在原发乳腺癌肿瘤中HOTAIR表达增高,并且HOTAIR表达水平是原发乳腺癌肿瘤转移和患者死亡的独立的预后因素[11]。而且,siRNA干扰HOTAIR表达,能够明显抑制肿瘤侵袭和转移,表明HOTAIR可以作为诊断和治疗的重要靶点[11]。此外,lncRNA的表达与肿瘤的化疗敏感性有关,沉默lncRAN表达能显著增加顺铂或甲烯氧四环素诱导的细胞凋亡,从而增加化疗敏感性[12]。但LncRNA与胃癌相关的研究尚为数不多。Cao等[15]利用GEO数据库分析,发现胃癌组织71条上调、17条下调表达的lncRNA。Lin等[16]利用LncRNA芯片检测也发现了胃癌组织2 621条差异表达的lncRNA。Song等[17]也发现了135个差异表达lncRNA,并探讨了H19及uc001lsz RNA,提示其和胃癌的发生、发展有密切的关系。
UCA1属于基因间lncRNA,长度2 314bp,定位于19号染色体p13.12区域。已经证实,UCA1上调表达可影响大肠癌细胞增殖、凋亡和细胞周期分布[21],并与黑色素瘤的转移相关[22]。最近Li等[23]也发现,食管鳞状细胞癌UCA1过表达与预后不良相关。本研究分析UCA1表达水平与胃癌临床病理特征的关系显示,UCA1的表达水平与细胞分化程度及病理分期相关,而且,下调UCA1胃癌细胞表达可明显抑制细胞增殖,进一步提示UCA1也是胃癌重要的促癌基因。已经证实,UCA1可通过mTOR-STAT3/microRNA143途径上调己糖激酶2(hexokinase 2)促进肿瘤细胞糖代谢[24],并可通过抑制p27(Kip1)表达促进乳腺肿瘤生长[25]。此外,UCA1还可通过CREB及PI3-K依赖的途径调控膀胱癌细胞周期分布[26]。UCA1在胃癌发生、发展中的机制有待于进一步研究。
[1]Gong C,Maquat L E.lncRNAs transactivate STAU1-mediated m RNA decay by dup lexing with 3'UTRs via Alu elements[J].Nature,2011,470(7333):284-288.
[2]Mercer TR,DingerM E,Mattick JS.Long non-coding RNAs:insights into functions[J].Nature reviews Genetics,2009,10(3): 155-159.
[3]Wang K C,Chang H Y.Molecu larmechanisms of long noncod ing RNAs[J].Molecularce ll,2011,43(6):904-914.
[4]Wilusz JE,Sunwoo H,Spector D L.Long noncod ing RNAs:functional surp rises from the RNA world[J].Genes&development, 2009,23(13):1494-1504.
[5]Silva J M,Perez D S,Pritchett J R,et al.Identification of long stress-induced non-cod ing transcrip ts that have altered exp ression in cancer[J].Genom ics,2010,95(6):355-362.
[6]Huarte M,Rinn J L.Large non-cod ing RNAs:m issing links in cancer?[J].Hum anmoleculargenetics,2010,19(R2):R152-161.
[7]Tsai M C,Sp itale R C,Chang H Y.Long intergenic noncod ing RNAs:new links in cancer p rog ression[J].Cancer research,2011, 71(1):3-7.
[8]Ponting C P,O liver P L,Reik W.Evolution and functions of long noncod ing RNAs[J].Cell,2009,136(4):629-641.
[9]Harries LW.Long non-cod ing RNAs and hum an d isease[J].Biochem ica lSociety transactions,2012,40(4):902-906.
[10]Wang J,Liu X,Wu H,etal.CREB up-regulates long non-cod ing RNA,HULC exp ression through interaction with m icroRNA-372 in liver cancer[J].Nuc leic acids research,2010,38(16):5366-5383.
[11]Kogo R,Shimamura T,Mimori K,et al.Long noncoding RNA HOTAIR regulates polycomb-dependent chromatin modification and is associated with poor p rognosis in colorectalcancers [J].Cancer research,2011,71(20):6320-6326.
[12]Zhu Y,Yu M,LiZ,etal.identified long noncoding RNA,enhances human b ladder tumor grow th,invasion,and survival[J].Urology 2011,77(2):510-515.
[13]Yang F,Huo XS,Yuan SX,etal.Rep ression of the long noncoding RNA-LET by histone deacetylase 3 contributes to hypoxia-m ed iatedm etastasis[J].Mo lecularce ll,2013,49(6):1083-1096.
[14]QiP,Xu M D,NiS J,etal.Low exp ression o f LOC285194 is associated w ith poor p rognosis in colorectal cancer[J].Journalof translationalm ed icine,2013,11:122.
[15]CaoW J,Wu H L,He B S,etal.Analysis of long non-cod ing RNA exp ression p rofiles in gastric cancer[J].World journal of gastroenterology:WJG,2013,19(23):3658-3664.
[16]Lin X C,Zhu Y,Chen W B,et al.Integ rated analysis of long non-cod ing RNAs and m RNA exp ression p rofiles reveals the potentialro le o f lncRNAs in gastric cancer pathogenesis[J].International journalofoncology,2014,45(2):619-628.
[17]Song H,Sun W,Ye G,et al.Long non-cod ing RNA exp ression p rofile in human gastric cancer and its c linicalsignificances[J]. Journalof translationalmed icine,2013,11:225.
[18]GuW,Gao T,Sun Y,etal.LncRNA exp ression p rofile reveals the potential role of lncRNAs in gastric carcinogenesis[J].Cancer, 15(3):1-10.biomarkers:section A ofDiseasemarkers,2015,15 (3):1-10.
[19]Liao Q,Liu C,Yuan X,et al.Large-scale p rediction of long non-coding RNA functions in a coding-non-cod ing gene co-exp ression network[J].Nuc leic acids research,2011,39(9): 3864-3878.
[20]Liao Q,Xiao H,Bu D,etal.ncFANs:a web server for functional annotation of long non-coding RNAs[J].Nuc leic acids research,2011,39(Web Server issue):W118-124.
[21]Han Y,Yang YN,Yuan HH,etal.UCA1,a long non-coding RNA up-regulated in colorectal cancer in fluences cell p roliferation, apop tosis and cellcyc le d istribution[J].Patho logy,2014,46(5): 396-401.
[22]Tian Y,Zhang X,Hao Y,et al.Potential roles of abnormally exp ressed long noncod ing RNA UCA1 and Malat-1 in m etastasis o fme lanoma[J].Me lanoma research,2014,24(4):335-341.
[23]Li J Y,Ma X,Zhang C B.Overexp ression of long non-cod ing RNA UCA1 p red icts a poor p rognosis in patients w ith esophagealsquamous ce llcarcinoma[J].Internationa l journalof c linicaland experimentalpatho logy,2014,7(11):7938-7944.
[24]Li Z,Li X,Wu S,et a l.Long non-cod ing RNA UCA1 p rom otes g lycolysis by up regu lating hexokinase 2 through the m TOR-STAT3/m ic roRNA143 pathway[J].Cancer sc ience,2014, 105(8):951-955.
[25]Huang J,Zhou N,Watabe K,etal.Long non-cod ing RNA UCA1 p romotes b reast tumorg row th by supp ression ofp27(Kip1)[J]. Celldeath&d isease,2014,5:e1008.
[26]Yang C,LiX,Wang Y,etal.Long non-cod ing RNA UCA1 regulated cellcyc le d istribution via CREB through PI3-K dependent pathway in b laddercarcinoma cells[J].Gene 2012,496(1):8-16.
Expression of long non-coding RNA UCA1 in gastric carcinoma tissue
TENG Weijun,ZHENG Xiaojuan,HUA Hongjun,et al.De-partment of Gastroenterology,Jinhua Central Hospital,Jinhua 321000,China
Objective To investigate the p rofiles of long non-cod ing RNA(lncRNA)exp ression in gastric cancer tissue.Methods The exp ression of lncRNAs was detected w ith m icroarray assay in 6 sam p les ofgastric tumor and matched adjacent tissues.The p rofiles of lncRNAs in gastric cancer tissues were identified.The UCA1 exp ression was evaluated by real-time reverse transcrip tion polymerase chain reaction(RT-PCR)in 40 gastric tumor sam p les and matched ad jacent tissues. The correlations of UCA1 exp ression w ith lym ph node metastasis,tumor size,celld ifferentiation,pathologicalstage were further analyzed.Tumor cellp roliferation was assessed follow ing siRNA knockdown of UCA1 by using the CCK 8 kits.Results A total of1 021 lncRNA exp ressed in gastric tumor sam p les>2 folds thanmatched ad jacent tissues were identified(P<0.05)using m icroarray assay.RT-PCR showed that the exp ression of UCA1 was significantly highly in gastric cancer than that in ad jacent tissues(P<0.0001).High UCA1 exp ression was associated w ith celldifferentiation and pathologicalstage ofgastric cancer.UCA1 exp ression in low-d ifferentiated gastric cancerwas significantly higher than that in w ith high-d ifferentiated cancer(P=0.031).In add ition,UCA1 level in stageⅢ/Ⅳgastric cancerwas significantly higher than thatw ith stage I/IIcancer(P=0.013).Furthermore, inhibition of UCA exp ression supp ressed the p roliferation of gastric cancer cells.Conclusion Abnormalexp ression of LncRNA may be involved in the development of gastric cancer.Up-exp ression of UCA1 in tumor tissue m ight act as an oncogene and p romote the developmentofgastric cancer.
Gastric cancer Long non-cod ing RNA Cellp roliferation
2014-12-22)
(本文编辑:沈昱平)
金华市科技局重点课题项目(2012-3-006);台州市科技局项目(1301ky40)
321000金华市中心医院消化内科(滕卫军、郑晓娟、华宏军);台州市第一人民医院普外科(林峰)
林峰,E-mail:linfeng2316@sina.com
