大肠杆菌araE基因对典型苯丙素类化合物转运过程的影响
2015-01-06周景文李江华
王 奎, 周景文, 李江华*
(1.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122;2.江南大学 生物工程学院,江苏 无锡214122)
大肠杆菌araE基因对典型苯丙素类化合物转运过程的影响
王 奎1,2, 周景文1,2, 李江华*1,2
(1.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122;2.江南大学 生物工程学院,江苏 无锡214122)
为了研究阿拉伯糖转运蛋白对大肠杆菌转运典型苯丙类素化合物的能力,采用基因过量表达和基因沉默技术,实现对蛋白质表达量进行控制,从而研究AraE对苯丙类素化合物的转运过程机制。同时,用定量PCR技术在转录水平考察并验证了araE基因的过量表达水平和基因抑制程度。通过测定菌体的生长动力学参数,衡量菌体对典型苯丙素类化合物的转运能力。结果表明,大肠杆菌araE基因只参与了大肠杆菌对白藜芦醇的转运,对其他种类的苯丙素类化合物作用不明显。
araE;大肠杆菌;苯丙素类化合物;基因沉默;定量PCR;转运
苯丙素类化合物(phenylpropanoids)是自然界种类最多的多酚类植物次级代谢产物,广泛存在于植物界中,目前发现的种类已超过8 000多种。苯丙素类化合物是天然存在的一类苯环与三个直链碳连接构成的酚类化合物,主要包括简单苯丙素、香豆素、木脂素、木质素类和黄酮类[1]。苯丙素类化合物具有广泛的生理功能。在植物体中,苯丙素类化合物主要参与调节植物生长、抵抗紫外伤害、抵抗病毒、信号转导和清除活性氧自由基[2-3]。在生物体内,苯丙素类化合物还具有广泛的药学价值,具有较强的抑菌、抗炎、抗氧化、抗肿瘤、免疫调节、抗癌、抗病毒、降血糖、抗辐射和降血脂等多种生物学作用[4-6]。近年来,由于苯丙素类化合物的广泛应用,其市场每年增长30%以上。大部分天然苯丙素类化合物在植物体内含量极低,提取法成本高昂且受季节限制较为严重[7]。此外,由于其具有复杂的化学结构,用化学方法合成较困难。这些因素导致了市场上的苯丙素类化合物价格较高,部分甚至处于供不应求的状态[8]。微生物具有生长速度快、适应力强、遗传操作简单等特点,用微生物法合成苯丙素类化合物已经吸引越来越多的科研人员关注。目前许多研究人员利用酿酒酵母 (Saccharomyces cerevisiae)和大肠杆菌(Escherichia coli)等为宿主菌进行苯丙素类化合物的合成[9-11]。
大肠杆菌作为常用的工程菌,遗传背景清晰,分子操作技术完善,是生物技术生产平台的首选,很早就有人通过改造大肠杆菌相关代谢途径生产苯丙素类化合物[12]。目前通过基因工程、代谢工程等技术手段已实现了多种苯丙素类化合物在大肠杆菌中的合成,如柚皮素、白藜芦醇等[13]。但是由于菌体无法快速地将苯丙素类化合物转运出胞外,造成大量的苯丙素类产物在胞内大量积累,对工程菌生长造成毒害作用,极大的限制了产量的进一步提高。因此,特异性或者非特异性的转运通道蛋白对提高产量具有着重要的作用[14],尤其是单向通道蛋白,可以降低胞内积累的苯丙素类化合物的含量,减少菌体的代谢负担,促进苯丙素类化合物的合成。
AraE转运蛋白是由Macpherson等在1981年首次鉴定出来的,最初鉴定的功能是一类阿拉伯糖低亲和力高转运能力的H+协同转运蛋白。后来的研究发现,AraE的功能不仅仅局限于转运阿拉伯糖,对其他的糖类如半乳糖、木糖、核糖和海藻糖等都有一定的转运效果[15]。Yu等在酿酒酵母中表达大肠杆菌的非特异性阿拉伯糖转运蛋白AraE能够使白藜芦醇的产量提高2.44倍[16]。也有研究表明,在酿酒酵母中共同表达大肠杆菌来源的araE基因和其他来源的TAL、4CL、STS基因能够使白藜芦醇的产量从1.27 mg/L提升到3.1 mg/L[17]。在工程菌大肠杆菌(BW27784)中协同表达4CL、STS和araE基因,可使白藜芦醇产量达到100 mg/L[18]。但是目前还未有阿拉伯糖转运蛋白AraE在大肠杆菌内对苯丙素类化合物转运能力的报道。AraE转运蛋白在酿酒酵母中对白藜芦醇有转运作用是因为白藜芦醇和阿拉伯糖相似都含有多个羟基[16]。作者选取具有相似羟基的两种苯丙素类化合物芦丁和柚皮素,用以观察AraE蛋白对其他种类的苯丙素类化合物是否同样有转运作用。藜芦醇、芦丁和柚皮素的结构见图1。
1 材料与方法
1.1 材料
1.1.1 菌种和质粒 大肠杆菌 E.coli BL21(DE3):宿主菌株;大肠杆菌E.coli JM109:质粒构建菌株;基因操作所用质粒 pMD19-T Simple vector:购自TaKaRa公司;基因表达所用的质粒pRSFDuet-1和pACYCDuet-1:购自Novagen公司。

图1 所选3种苯丙素类化合物结构Fig.1 Structure of 3 phenylpropanoids
1.1.2 培养基
1)大肠杆菌LB培养基:NaCl 10 g/L,胰蛋白胨20 g/L,酵母浸粉10 g/L,固体培养基添加琼脂20 g/L;阳性转化子筛选培养基:LB培养基中分别加入500 μg/mL氨苄青霉素 (质粒 pMD19-T Simple vector)、340 μg/mL氯霉素 (质粒pACYCDuet-1)或100 μg/mL卡那霉素(质粒pRSF-PTasRNA)。
2)菌株生长曲线测定培养基:M9培养基(13.3 g/L M9CA Broth,0.2g/dL 葡 萄 糖 ,1 mmol/L MgSO4,0.5 μg/mL Thiamine·HCl,过滤除菌),1.0 g/L芦丁或者柚皮素或者白藜芦醇 (DMSO溶解),100 μg/mL卡那霉素或者 340 μg/mL氯霉素,IPTG(0,100,200,400 μmol/L)[19]。
1.1.3 酶、引物、DNA Marker及相关试剂盒pMD18-T Simple Vector、LATaq、DNA Marker、Solution I连接酶、大肠杆菌感受态制备试剂盒、质粒小量抽提试剂盒、胶回收试剂盒、细菌总DNA提取试剂盒以及各种内切酶均购自TaKaRa公司。本研究中所用的引物合成和测序工作均由生工生物工程(上海)有限公司完成。
1.2 araE过量表达重组菌的构建
以大肠杆菌基因组为模板,引物P1和P2扩增获得araE基因片段,使用限制性内切酶位点EcoRI和HindIII,连入具有氯霉素抗性的pACYCDuet-1。化学法转入大肠杆菌E.coli BL21 (DE3),用引物P3和P4进行菌落PCR验证。构建好的菌株命名为E.coli BL21(DE3)/pACYC-araE。对照菌株为含有空载体pACYCDueT-1的E.coli BL21,命名为E.coli BL21(pACYC),并进行酶切验证。
1.3 基因沉默菌的构建
利用购自Agilent公司的QuikChange II XL定点突变试剂盒将pRSFDuet-1上的XhoI去掉,即将序列CTCGAG突变成序列CTAGAG,突变后的质粒命名为pRSFDuet-1(Mut)。基因沉默质粒特有的PTasRNA序列包含的trc启动子、lac操纵子、38 bp末端序列和 stem-loop结构的 rrnB终止子通过GeneScript公司合成[20]。将合成好的PTasRNA序列和pRSFDuet-1(Mut)用内切酶PfoI和BamHI双酶切后所获得的片段连接即为基因沉默质粒,命名为pRSF-PTasRNA。
araE基因的asRNA序列(-40到130 bp)用引物P5和P6,见表1。以E.coli BL21(DE3)基因组为模板进行扩增。片段进行胶回收后与pMD19-T Simple vector连接。将连接体系用化学转化法的方法转入E.coli JM109感受态,并用通用引物进行菌落PCR验证。提取阳性转化子质粒送上海生工进行基因测序,测序正确质粒命名为pMD19-T-ΔaraE。
将 pMD19-T-ΔaraE和 pRSF-PTasRNA用XhoI和NcoI酶进行双酶切,胶回收目的片段和酶切后的质粒连接并转化,使用验证引物P7和P8进行菌落PCR验证。提取连接成功的质粒通过化学转化法转入E.coli BL21(DE3)感受态,构建好的菌株命名为E.coli BL21 (DE3)/pRSF-asRNA-araE。对照菌株用含有空载体pRSF-asRNA的E.coli BL21,命名为E.coli BL21(pRSF-asRNA),见图2。

图2 沉默质粒pRSF-asRNA-araE构建过程Fig.2 Construction of silencing araE plasmid pRSF-asRNA-araE
1.4 定量PCR方法
E.coli BL21(DE3)的RNA提取以及cDNA文库的建立,分别参照天根公司的RNAprep pure Cell/ Bateria Kit和Takara公司反转录试剂盒PrimeScript○RRT reagent Kit。参照Takara公司的SYBR Premix Ex Taq试剂盒和LightCycler Real Time PCR扩增仪进行qPCR反应。根据NCBI公布的araE的基因序列,由Beacon Designer 7.0设计用于qPCR的引物P9和P10,内参基因为16s DNA,引物为P11和P12。按照以下程序进行qPCR反应:95℃预变性40 s,95℃变性5 s,57℃延伸30 s,持续40个循环,50℃冷却30 s,每个基因做3次平行以保证数据的重复性。用LightCycler分析绝对定量及相对定量数据。

表1 构建表达载体时使用的引物序列Table 1 Oligonucleotides sequence used in vectors construction
1.5 SDS-PAGE
挑取E.coli BL21(pACYC-araE)单菌落接种到含有340 μg/mL氯霉素的M9培养基,30℃、200 r/min活化10 h。按照1%的接种体积分数接种到含有340 μg/mL氯霉素的M9培养基,30℃培养4 h,加入终浓度100 μmol/L的IPTG,诱导4 h。离心收集1 mg菌体,加入60 μL含有Benzonase的裂解液(裂解液需用1×PBS稀释),30℃、200 r/min振荡30 min。离心收集沉淀,用裂解液清洗沉淀3次,加入60 μL 20%SDS溶液,用枪头吹吸充分重悬。离心后取10 μL上清液,加入10 μL上样缓冲液,99℃加热10 min。采用12 g/dL分离胶,5 g/dL浓缩胶进行聚丙烯酰胺凝胶电泳(SDS-PAGE),鉴定蛋白质是否表达。
1.6 重组菌生长动力学参数测定方法
挑取E.coli BL21 (pACYC-araE)或E.coli BL21(pRSF-asRNA-araE)单菌落接种到含有340 μg/mL氯霉素的M9培养基,37℃、200 r/min活化10 h。按照1%的接种体积分数接种到过量表达菌株生长曲线测定培养基中。将接种后的培养基分装至96孔板中,每孔200 μL,置于预热至30℃的96孔板振荡器上,900次/min振荡培养,每隔1小时用酶标仪测量OD600直至生长至稳定期[21]。将获得的数据通过软件OriginPro 8.5通过公式拟合得出菌株在不同条件下的最大比生长速率(μmax)[22]。

其中,μmax为最大比生长速率;tmax为μ达到最大值时的时间;tL为延迟期的时间。
2 结果与分析
2.1 araE基因的转录水平分析
通过对araE基因过量表达和基因沉默控制阿拉伯糖转运蛋白AraE在细胞内的含量。过量表达可以提高转录水平,进而提高蛋白质含量。基因沉默是通过表达一段asRNA序列,导致基因转录失败,进而导致蛋白质含量的减少。定量PCR结果显示,在100 μmol/L IPTG诱导下,基因沉默导致菌体内araE基因mRNA水平下降了10倍,而过量表达使菌株体内araE基因mRNA水平上调了12倍,见图3。

图3 不同菌株araE基因转录水平定量PCR验证Fig.3 Relative expression levels of araE gene in different strains
2.2 araE基因过量表达的SDS-PAGE验证
对菌株E.coli BL21(pACYC-araE)不可溶性蛋白进行SDS-PAGE电泳鉴定。图4显示,所构建的重组菌株E.coli BL21(pACYC-araE)在相对分子质量约为47 000处明显比对照菌表达量大,与预期相符,说明AraE蛋白在大肠杆菌中实现了过量表达。
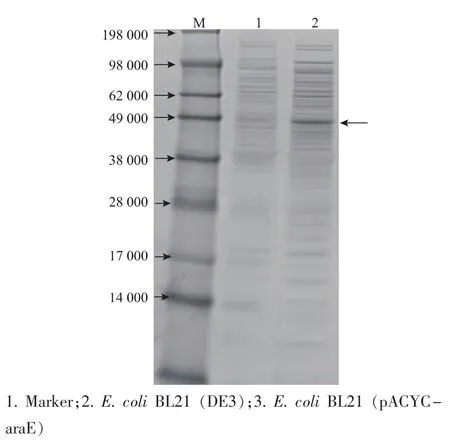
图4 AraE在大肠杆菌中表达的SDS-PAGEFig.4 SDS-PAGE analysis of recombinant AraE in E.coli BL21(DE3)
2.3 araE过量表达重组菌株对不同种类苯丙素类化合物的转运能力
E.coli BL21(DE3)在M9培养基中的μmax达到0.52 h-1,添加400 μmol/L IPTG后,μmax下降到0.45 h-1。基因过量表达菌株E.coli BL21(DE3)/pACYC-araE在1.0 g/L芦丁环境中,通过不同浓度的IPTG(0、100、200、400 μmol/L)来控制其表达量,发现其最大比生长速率分别为0.24、0.21、0.17、0.13 h-1;在1.0 g/L柚皮素的环境中,其最大比生长速率分别为0.21、0.19、0.17、0.16 h-1;在1.0 g/L白藜芦醇的环境中,其最大比生长速率分别为0.13、0.17、0.21、0.29 h-1,见图5。

图5 araE过量表达菌株在不同浓度IPTG诱导后在不同种类苯丙素类化合物刺激下最大比生长速率Fig.5 μmaxof E.coli BL21 (DE3)/pACYC-araE in the 1 g/L rutin,naringenin or resveratrol and induced by different concentrations of IPTG
对过量表达菌株E.coli BL21(DE3)/pACYC-araE在三种不同物质刺激下的生长曲线的研究发现,随着IPTG浓度的增加,也即AraE蛋白质表达量的增加,发现E.coli BL21 (DE3)/pACYC-araE菌株在芦丁和柚皮素的刺激下的最大比生长速率都略微下降,下降幅度和原始菌株随着IPTG浓度增加下降的幅度差不多,说明AraE蛋白表达量的增加对于提高大肠杆菌在芦丁或者柚皮素的环境中的生长速率没有作用,也即说明AraE蛋白对于芦丁或者柚皮素没有转运作用。但是E.coli BL21(DE3)/pACYC-araE在白藜芦醇的环境中随着IPTG浓度的增加,也即AraE蛋白表达量的增加,最大比生长速率也增加,说明AraE蛋白能够有效地提升大肠杆菌在白藜芦醇中的生长速率,也即AraE蛋白在大肠杆菌中也能转运白藜芦醇。这与先前的研究酿酒酵母中AraE蛋白能够转运白藜芦醇结果相一致[16,23]。
2.4 araE基因沉默菌株对不同种类苯丙素类化合物的转运能力
基因沉默菌株 E.coli BL21 (DE3)/pRSF-asRNA-araE在1.0 g/L芦丁环境中,通过不同浓度的IPTG(0、100、200和400 μmol/L)来控制其表达量,发现其最大比生长速率分别为0.24、0.22、0.19、0.14 h-1;在1.0 g/L柚皮素的环境中,其最大比生长速率分别为0.21、0.18、0.16、0.14 h-1;在1.0 g/L白藜芦醇的环境中,其最大比生长速率分别为0.13、0.08、0.04、0.02 h-1,见图6。
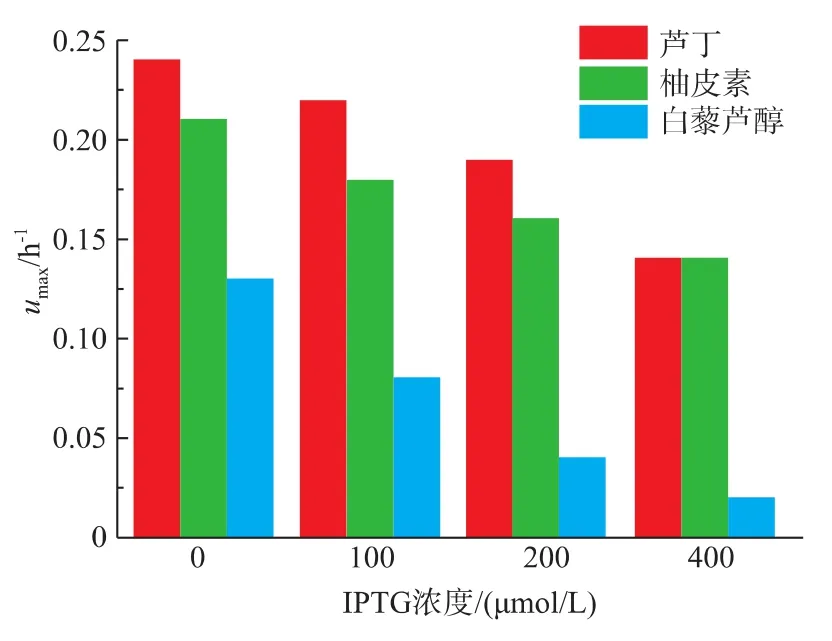
图6 araE基因沉默菌株在不同浓度IPTG诱导后在不同种类苯丙素类化合物刺激下最大比生长速率Fig.6 μmaxof E.coli BL21 (DE3)/pRSF-asRNA-araE in the 1 g/L rutin,naringenin or resveratrol and induced by different concentrations of IPTG
对于基因沉默菌株E.coli BL21(DE3)/pRSF-asRNA-araE生长曲线的研究发现,AraE蛋白质表达量的降低能够降低大肠杆菌对于白藜芦醇的耐受,而对于芦丁和柚皮素没有很明显的效果。这个结果和基因过量表达的结果相符,从两方面说明了AraE蛋白在大肠杆菌能够转运白藜芦醇,而对芦丁和柚皮素没有转运效果[24]。由于白藜芦醇和AraE蛋白底物都具有多个羟基,并且都具有一定的极性,所以AraE对于白藜芦醇也具有一定的转运效果[25]。
3 结语
随着苯丙素类化合物对肿瘤、抗氧化等方面的研究深入,市场对其需求日益增加[26]。目前关于苯丙素类化合物合成研究也日益加快,在大肠杆菌以及酿酒酵母中都已成功构建苯丙素类化合物的合成途径[27],所以解决高浓度苯丙素类化合物在胞内的积累问题迫在眉睫,急需找到苯丙素类化合物的高效转运蛋白,降低胞内的产物含量,降低代谢负担,实现高效的苯丙素类化合物生产[28]。
作者考察了AarE通道蛋白对3种典型苯丙素类化合物的转运情况,通过蛋白质表达水平的差异,实现通道蛋白转运能力的控制,结果首次发现了大肠杆菌中的araE基因能够提升大肠杆菌对白藜芦醇的耐受能力,该研究对国内开发使用大肠杆菌生产白藜芦醇有一定的指导意义。但是关于转运机理还需进一步研究,而不仅仅局限识别羟基基团,同时对芦丁和柚皮素的转运蛋白也还需进一步研究。
作者所采用的基因沉默手段,解决了蛋白质表达水平控制方面的难题。目前国内的研究多数通过基因敲除手段实现酶的表达量降低,所需的实验操作繁琐,费时长,而且在一些菌种不易实现。采用在转录水平上的mRNA调控,可以方便快速的抑制目的基因的表达,实现代谢流的调控,对今后的相关研究有借鉴作用。
[1]Cushnie T,Lamb A J.Antimicrobial activity of flavonoids[J].International Journal of Antimicrobial Agents,2005,26(5):343-356.
[2]Agati G,Brunetti C,Di Ferdinando M,et al.Functional roles of flavonoids in photoprotection:New evidence,lessons from the past[J].Plant Physiology and Biochemistry,2013,72:35-45.
[3]尤新.食品抗氧化剂与人体健康[J].食品与生物技术学报,2006,25(2):1-5. YOU Xin.Food Anti-Oxidants and Their Inflence to Human Health[J].Journal of Food Science and Biotechnology,2006,25(2):1-5.(in Chinese)
[4]Ferreyra M L F,Rius S P,Casati P.Flavonoids:biosynthesis,biological functions,and biotechnological applications[J].Frontiers in Plant Science,2012,3(222):1-15.
[5]陈丛瑾,王琪,李欣.黄酮类化合物抗氧化和抑菌生物活性研究进展[J].中国药房,2011,22(35):3346-3348.CHEN Congjin,WANG Qi,LI Xin.The progress of antioxidant and bacteriostasis bioactive of flavonoids[J].Chinese Pharmacy,2011,22(35):3346-3348.(in Chinese)
[6]马兵钢,郝青南,马超.大豆异黄酮分离检测及保健功效的研究[J].食品与生物技术学报,2008,27(6):69-72. MA Binggang,HAO Qingnan,MA Chao.The methods of separation and detection for soybean isoflavones and hygienical functions of soybean isoflavones[J].Journal of Food Science and Biotechnology,2008,27(6):69-72.(in Chinese)
[7]于涛,钱和.膜分离技术在提取银杏叶黄酮类化合物中的应用[J].无锡轻工大学学报,2004,23(6):55-58. YU Tao,QIAN He.The application of membrane technology in extracting flavones compounds in ginkgo leaves[J].Journal of Wuxi University of Light Industry,2004,23(6):55-58.(in Chinese)
[8]Fowler Z L,Koffas M A.Biosynthesis and biotechnological production of flavanones:current state and perspectives[J].Applied Microbiology and Biotechnology,2009,83(5):799-808.
[9]Leonard E,Yan Y,Fowler Z L,et al.Strain improvement of recombinant Escherichia coli for efficient production of plant flavonoids[J].Molecular Pharmaceutics,2008,5(2):257-265.
[10]Yan Y,Kohli A,Koffas M A.Biosynthesis of natural flavanones in Saccharomyces cerevisiae[J].Applied and Environmental Microbiology,2005,71(9):5610-5613.
[11]Zhou J,Du G,Chen J.Novel fermentation processes for manufacturing plant natural products[J].Current Opinion in Biotechnology,2014,25:17-23.
[12]Horinouchi S.Combinatorial biosynthesis of plant medicinal polyketides by microorganisms[J].Current Opinion in Chemical Biology,2009,13(2):197-204.
[13]Pandey R P,Malla S,Simkhada D,et al.Production of 3-O-xylosyl quercetin in Escherichia coli[J].Applied Microbiology and Biotechnology,2013,97(5):1889-1901.
[14]Yazaki K.Transporters of secondary metabolites[J].Current Opinion in Plant Biology,2005,8(3):301-307.
[15]Kolodrubetz D,Schleif R.L-arabinose transport systems in Escherichia coli K-12[J].Journal of Bacteriology,1981,148(2):472-479.
[16]Wang Y,Halls C,Zhang J,et al.Stepwise increase of resveratrol biosynthesis in yeast Saccharomyces cerevisiae by metabolic engineering[J].Metabolic Engineering,2011,13(5):455-463.
[17]Jeandet P,Delaunois B,Aziz A,et al.Metabolic engineering of yeast and plants for the production of the biologically active hydroxystilbene,resveratrol[J].Journal of Biomedicine and Biotechnology,2012,2012(1):1-14.
[18]Watts K T,Lee P C,Schmidt-Dannert C.Biosynthesis of plant-specific stilbene polyketides in metabolically engineered Escherichia coli[J].BMC Biotechnology,2006,6(1):22.
[19]Watts K T,Lee P C,Schmidt‐Dannert C.Exploring recombinant flavonoid biosynthesis in metabolically engineered Escherichia coli[J].Chembiochem,2004,5(4):500-507.
[20]Nakashima N,Tamura T,Good L.Paired termini stabilize antisense RNAs and enhance conditional gene silencing in Escherichia coli[J].Nucleic Acids Research,2006,34(20):138-138.
[21]Elowitz M B,Surette M G,Wolf P-E,et al.Protein mobility in the cytoplasm of Escherichia coli[J].Journal of Bacteriology,1999,181(1):197-203.
[22]宋健,林建群,金燕,等.以比生长速率时间曲线为基础的生物群体生长数学模型[J].微生物学通报,2007,34(5):836-838. SONG Jian,LIN Jian Qun,JIN Yan,et al.A new population growth model based on the time dependent changes of the specific growth rate[J].Microbiology China,2007,34(5):836-838.(in Chinese)
[23]Jeandet P,Vasserot Y,Chastang T,et al.Engineering microbial cells for the biosynthesis of natural compounds of pharmaceutical significance[J].BioMed Research International,2013,20(3):56-61.
[24]Khlebnikov A,Datsenko K A,Skaug T,et al.Homogeneous expression of the PBAD promoter in Escherichia coli by constitutive expression of the low-affinity high-capacity AraE transporter[J].Microbiology,2001,147(12):3241-3247.
[25]Buschke N,Schafer R,Becker J,et al.Metabolic engineering of industrial platform microorganisms for biorefinery applications-optimization of substrate spectrum and process robustness by rational and evolutive strategies[J].Bioresource Technology,2013,135:544-554.
[26]丁利君,苏桂良.微波辅助提取蜈蚣草黄酮及其抗氧化[J].食品与生物技术学报,2009,28(5):79-85.DING Lijun,SU Guiliang.Extraction of flavonoids from ladder brake with microwave and its antioxidative activity[J].Journal of Food Science and Biotechnology,2009,28(5):79-85.(in Chinese)
[27]Wu J,Du G,Zhou J,et al.Metabolic engineering of Escherichia coli for (2 S)-pinocembrin production from glucose by a modular metabolic strategy[J].Metabolic Engineering,2013,16:48-55.
[28]Wu J,Liu P,Fan Y,et al.Multivariate modular metabolic engineering of Escherichia coli to produce resveratrol from l-tyrosine[J]. Journal of Biotechnology,2013,167(4):404-411.
Identification of araE Gene Associated with Response to Different Phenylpropanoids by Gene Silencing in Escherichia coli BL21
WANG Kui1,2, ZHOU Jingwen1,2, LI Jianghua*1,2
(1.Key Laboratory ofIndustrialBiotechnology,Jiangnan University,Wuxi214122,China;2.Schoolof Biotechnology,Jiangnan University,Wuxi 214122,China)
AraE is an important unidirectional transport channel protein that could transport some compounds outside the cell.E.coli is a common host-bacterial to produce phenylpropanoids,and the growth was inihibited by phenylpropanoids.In order to investigate the transport ability of AraE on phenylpropanoids in E.coli,the gene overexpression and silencing routes was used in this study to regulate the expression of AraE.The level of overexpression and silencing was confirmed by qPCR and SDS-PAGE.The transport ability of phenylpropanoids was measured by detection of the recombinant bacterial growth kinetics parameter (μmax)in different typical phenylpropanoids(rutin,naringenin and resveratrol).The result showed that AraE could only transport resveratrol in E.coli.
araE,E.coli,phenylpropanoids,gene silencing,qPCR,transport
Q 786
A
1673—1689(2015)08—0856—08
2014-03-05
国家自然科学基金面上项目(31370130)。
*通信作者:李江华(1966—),男,江西九江人,工学博士,教授,博士研究生导师,主要从事酶技术、发酵过程优化与控制方面的研究。E-mail:lijianghua@jiangnan.edu.cn
