利用TAIL-PCR技术克隆拟南芥抗旱相关的csm2基因
2014-12-31陈光朗孙成磊阳立波曹树青
程 丹, 陈光朗, 孙成磊, 阳立波, 曹树青
(合肥工业大学 生物与食品工程学院,安徽 合肥 230009)
0 引 言
水分不足是限制农业发展的重要因子,提高植物自身抗旱性和水分利用效率来发展农业具有重要的意义。研究表明,植物的抗旱性是由多基因控制的,不同作物和不同品种适应干旱的方式是多种多样的[1]。探讨作物的抗旱机理、提高水分利用效率、改良作物的抗旱性是目前农业科学倍受关注的研究内容[2]。培育耐旱作物的主要途径有:将野生耐旱植物驯化成作物的传统育种;利用组织培养和诱变生物技术产生突变表型进行培育;基因工程培育等。利用正向遗传学途径,筛选抗旱突变体并分离抗旱主效基因是培育耐旱作物品种的主要方法[3]。本文利用甘露醇模拟干旱环境,筛选出一株抗旱突变株,并利用TAIL-PCR克隆出其抗旱csm2基因。该研究为进一步阐明干旱胁迫的分子机理、提高植物的抗旱能力具有重要的理论意义和潜在的应用价值。
1 材料与方法
1.1 实验材料
拟南芥(Arabidopsis)生态型为Columbia,突变体库均来自美国拟南芥种质资源中心,实验采用2种方法培养植株[4]。
1.2 实验方法
1.2.1 拟南芥抗旱突变体的筛选
以确定浓度的甘露醇配制MS培养基进行筛选。在培养皿中央点野生型种子作为对照,其余部分点突变体种子,水平放置培养。定期观察并拍照,若有能生长的突变体幼苗,作为候选突变体,将之移栽到土壤中培养[5]。
1.2.2 拟南芥抗旱突变体的鉴定
分别将突变体和野生型种子点种于含有400mmol/L甘露醇的MS培养基中,培养皿封口后放入4℃冰箱中春化2d后置于培养室中培养,观察生长情况。
1.2.3 基因克隆
利用CTAB法提取鉴定过的突变株的基因组DNA[6]。利用TAIL-PCR技术克隆突变体突变基因[7-8]。取100ng的突变体基因组 DNA,TDNA插入区侧翼序列利用特异引物(SP)、随机引物(AD)序列以及PCR反应程序进行TAILPCR扩增。本实验主要采用1组巢式引物LexA2、LexA3、LexA6,1个随机引物 AD2,利用TAIL-PCR技术对T-DNA侧翼序列进行了扩增,得到1条750bp左右的片段,将所获得的序列与TAIR中的已知基因进行同源性分析。结果发现,T-DNA插入片段的侧翼序列属于拟南芥的At1g07530基因。
1.2.4 验证 TAIL-PCR结果
突变体和野生型总RNA的提取和纯化参照改良的Trizol法[9]。纯化后的RNA以Oligo(dT)为引物进行反转录合成cDNA,再以合成的cDNA为模板,验证TAIL-PCR结果的可靠性,并运用RT-PCR分析突变体和野生型体内cDNA中At1g07530基因的表达水平。
2 结果与分析
2.1 拟南芥抗旱突变体的筛选及鉴定
在400mmol/L甘露醇条件下,野生型均不能发芽,但突变体可正常发芽,表明筛选出的csm2-1突变体为抗旱突变体,如图1所示。
将野生型和csm2-1突变体分别点在MS培养基和400mmol/L甘露醇条件下的MS培养基中,发现在 MS培养基中,csm2-1突变体和野生型的生长无显著差异,而在400mmol/L甘露醇条件下,csm2-1根长显著长于野生型,且其发芽率也显著高于野生型,如图2所示。
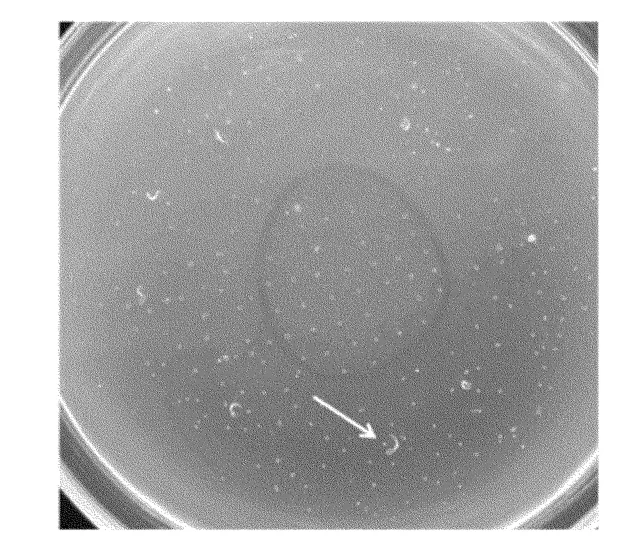
图1 甘露醇浓度为400mmol/L时筛选的候选突变体

图2 突变体的表型鉴定
2.2 基因克隆
利用TAIR-PCR技术对T-DNA侧翼序列进行扩增,得到1条750bp左右的片段,如图3所示,将所获得的序列与TAIR中的已知基因进行同源性分析。结果发现,克隆的基因片段中有666bp碱基连续与拟南芥At1g07530基因序列完全吻合,且T-DNA插入片段的侧翼序列在At1g07530基因内部的1 520bp处。
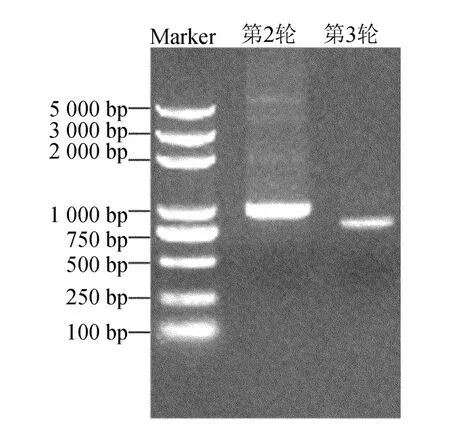
图3 突变体TAIL-PCR结果
2.3 TAIL-PCR结果的验证
为验证TAIL-PCR结果,设计了1对位于TDNA插入点位置两侧的引物F和R,并结合TAIL-PCR的特异引物之一LexA6进行PCR鉴定。以野生型和突变体的基因组DNA为模板,分别利用引物F和R、LexA6和R进行PCR扩增,如图4所示,野生型可以扩增出符合预期大小(1 050bp)的目的P7段(泳道1),而在突变体中扩增出目的条带(泳道4),由于野生型未插入TDNA片段,因而LexA6在野生型中不存在结合靶序列,相反在突变体中存在LexA6结合靶序列,因此LexA6与R间的片段未能扩增得到(泳道3),而在突变体中得到了相应的片段(980bp)。这 说 明 在 突 变 体csm2-1 中 的At1g07530基因的确存在 T-DNA 插入,即TAIL-PCR结果真实可靠[10]。
为进一步确定csm2-1的干旱耐受性是由于T-DNA插入导致At1g07530基因失活所致,进行了反转录PCR(RT-PCR),结果表明,在突变体中At1g07530基因没有表达,而在野生型中,该基因可以正常表达,如图5所示。这说明突变体的干旱耐受表型确实是由于At1g07530基因的缺失引起的。
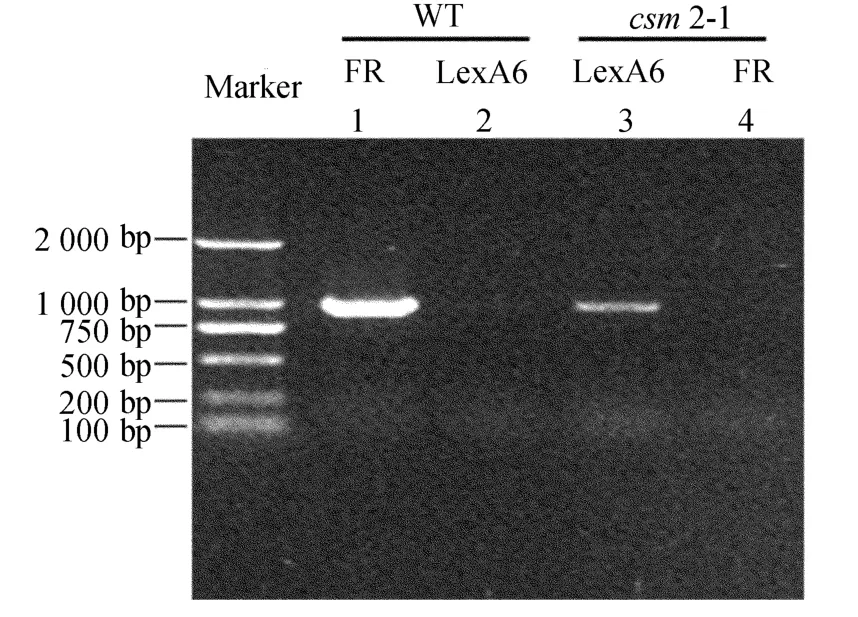
图4 T-DNA插入位置
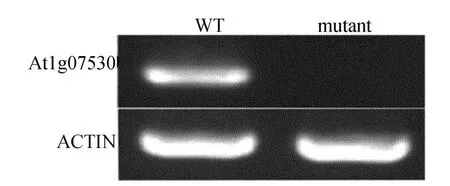
图5 RT-PCR验证At1g07530基因表达情况
3 讨 论
本实验分离出的抗旱突变体csm2-1与野生型相比表现出显著的抗旱性,通过鉴定确定csm2-1是抗旱突变体。利用TAIL-PCR克隆出csm2的抗旱基因,并通过PCR及RT-PCR等方法证实了突变体是由于T-DNA插入At1g07530基因,导致该基因不能表达所致。
对环境胁迫响应基因的分离鉴定及其相应的信号转导途径的研究是探讨植物潜化抗逆机制的重要方法之一,利用转基因等相关技术能够有效提高作物抵御环境胁迫的能力[11-12]。但是csm2基因在拟南芥信号传递中的功能还需进一步解析,其对干旱胁迫信号转导及其应答胁迫的分子机制将是进一步研究的重点。
[1] 张 彤,齐 麟.植物抗旱机理研究进展[J].湖北农业科学,2005(4):107-110.
[2] 喻方圆,锡 增.植物逆境生理研究进展[J].世界林业研究,2003,16(5):6-11.
[3] 王建革,苏晓华,张冰玉.植物抗旱研究工作中的问题与方法初探[J].中国农学报,2004,20(3):93-96.
[4] Shinozaki K,Yamaguchi-Shinozaki K,Seki M.Regulatory network of gene expression in the drought and cold stress responses[J].Curr Opin Plant Biol,2003,6(5):410-417.
[5] Murashige T,Skoog F.A revised medium for rapid growth and bioassays with tobacco tissue cultures[J].Physiol Plant,1962,15:473-497.
[6] 张娇娇,江 力,杨 杰,等.拟南芥抗旱突变体的筛选和鉴定[J].合肥工业大学学报:自然科学版,2012,35(4):531-535.
[7] 安鑫龙,董金皋,韩建民.应用CTAB法提取玉米大斑病菌DNA[J].河北农业大学学报,2001,24(1):38-41.
[8] Liu Y G,Whitter R F.Thermal asymmetric interlaced PCR:automatable amplification and sequencing of insert end fragments from P1and YAC clones for chromosome walking[J].Genomics,1995,25:674-681.
[9] 罗丽娟,施季森.一种DNA侧翼序列分离技术:TAIL-PCR[J].南京林业大学学报:自然科学版,2003,27(4):87-90.
[10] Sternberg D,Mandels G R.Induction of cellulolytic enzymes in Trichoderma reesei by sophorose[J].J Bacteriol,1979,139:761-769.
[11] 刘 浩,苏 凡,王棚涛.利用TAIL-PCR方法克隆拟南芥干旱主效基因NCED3[J].河南大学学报:自然科学版,2009,39(6):621-625.
[12] 刘 强,张 勇,陈受宜.干旱、高盐及低温诱导的植物蛋白激酶基因[J].科学通报,2000,45(6):561-566.
