基于线粒体Cyt b和COI基因的中国林蛙系统发生关系
2014-12-29杨宝田
周 瑜,杨宝田
林蛙又称棕色蛙(Brown Frog),隶属于无尾目(Anura)蛙科(Ranidae)林蛙属(Rana),主要分布于欧亚大陆温带地区至中国、印度一带和北美南部地区[1]。中国的林蛙物种资源丰富,包括中国林蛙种组(Rana chensinensis species group)、长肢林蛙种组(Rana longicrus species group)和黑龙江林蛙种组(Rana amurensis species group)[2]。中国分布的林蛙属物种约有17个,包括近年发表的新种徂徕林蛙(Rana culaiensis)[3]、猫儿山林蛙(Rana maoershanensis)[4]和 Rana jiemuxiensis[5]。
由于形态特征的相似性,林蛙属各物种的识别较为困难[6]。近年来,随着DNA序列分析技术的迅速发展,其在物种和个体识别、亲缘关系鉴定以及分子进化和系统发育关系分析等诸多方面显示了极大的优势,从而得到了广泛的应用。由于母系遗传和不存在重组过程的特点,线粒体DNA在很多动物研究中被证明能够很好地展现物种系统发生关系[7]。在对林蛙属物种的遗传变异和系统发生关系研究中,线粒体DNA细胞色素b(Cyt b)基因都被视为很好的指示序列[8]。细胞色素氧化酶I(COI)基因作为DNA条形码标识序列,在两栖类物种鉴定方面也得到广泛关注[9-10]。中国林蛙属物种关系复杂,前人的相关研究主要集中在对某一个种组内部的系统发生关系的分析,包括近年新发现物种在内的较全面的系统发生关系有待进一步研究。
本研究应用Cyt b基因和COI基因序列分析对林蛙属物种进行研究,评价相关物种的有效性,全面分析中国分布林蛙属物种的系统发生关系。
1 材料与方法
1.1 样本收集、基因组提取与PCR扩增
本研究所收集的中国分布的林蛙属物种样本及来源见表1。样本从野外采集经鉴定后取肌肉或肝脏组织置于75%乙醇保存备用。采用SDS/蛋白酶K裂解,酚-氯仿抽提法提取组织中的总DNA[11]。

R .kunyuensis SYNU07050112 Mt.Kunyu,Shandong,China KF020609 KF020624 SYNU07050130 Mt.Kunyu,Shandong,China KF020610 KF020625 R.chensinensis KIZRD05SHX01 Huxian,Shaanxi JF939080 JN984335 SYNU-hld1 Huludao,Liaoning KF020612 KF020627 R.dybowskii KIZ070423448 Huangnihe,Jilin JF939078 JF939116 SYNU07040020 Huanren,Liaoning Unpublished Unpublished SYNU13070055 Xiuyan,Liaoning Unpublished Unpublished SYNU13070053 Mt.Guanmenshan,Liaoning Unpublished Unpublished R.huanrensis SYNU040006 Huanren,Liaoning JF939072 JN984545 SYNU07040035 Huanren,Liaoning Unpublished KF204668 R.kukunoris CJ06102001 Qinghai Lake,Qinghai JF939073 JN984213 Outgroup Pelophylax nigromaculatus AB043889 Lithobates catesbeianus AB761267
PCR 扩增引物参考 Zhou 等(2012)[12],Tanaka-Ueno 等(1999)[13],Che 等(2012)[9](表 2)。PCR 扩增反应总体积为25μl。反应条件:94℃预变性5min,然后进行94℃变性30s、46℃退火30s、72℃延伸60s,35个循环,最后72℃延伸5min。PCR产物由北京六合华大基因有限公司进行双向测序,以确保序列的准确性。
1.2 DNA序列数据处理和系统发育分析
所测得序列用DNAStar 7.0进行双向测序结果的拼接及序列校对,并结合由Yan等(2011)[5]发表的数据资源,使用ClustalX 1.81软件[14]进行多重序列比对。将两个基因片段合并一起进行分析。p-distance计算使用 MEGA5.1 软件[15]。
以GenBank数据库中黑斑侧褶蛙(Pelophylax nigromaculatus)和美国牛蛙(Lithobates catesbeianus)为外群[16-17],采用最大似然法(maximum likelihood,ML)和贝叶斯法(Bayesian inference,BI)分别对线粒体基因构建分子系统树。用RAxML 7.0.4软件[18]执行rapid-hill-climbing运算法则构建ML树,并进行1000次拟重复抽样的bootstrap分析,以检验各节点的置信度。用MrBayes 3.1.2[19]软件构建贝叶斯树,以随机树为起始树,4条马尔可夫链运行1000万代,每1000代抽样并保存数据一次,舍弃老化25%样本后,根据剩余样本构建合意树。分析过程中对两个基因片段分别按第一密码子、第二密码子和第三密码子使用独立模型进行分析。ML树采用GTRCAT模型,BI树的最佳碱基替换模型由 MrModeltest 2.3[20]软件分析得出表3。

表2 引物序列

表3 使用MrModeltest软件分析得到的各分区数据ACI碱基替代模型
2 结果
2.1 序列分析
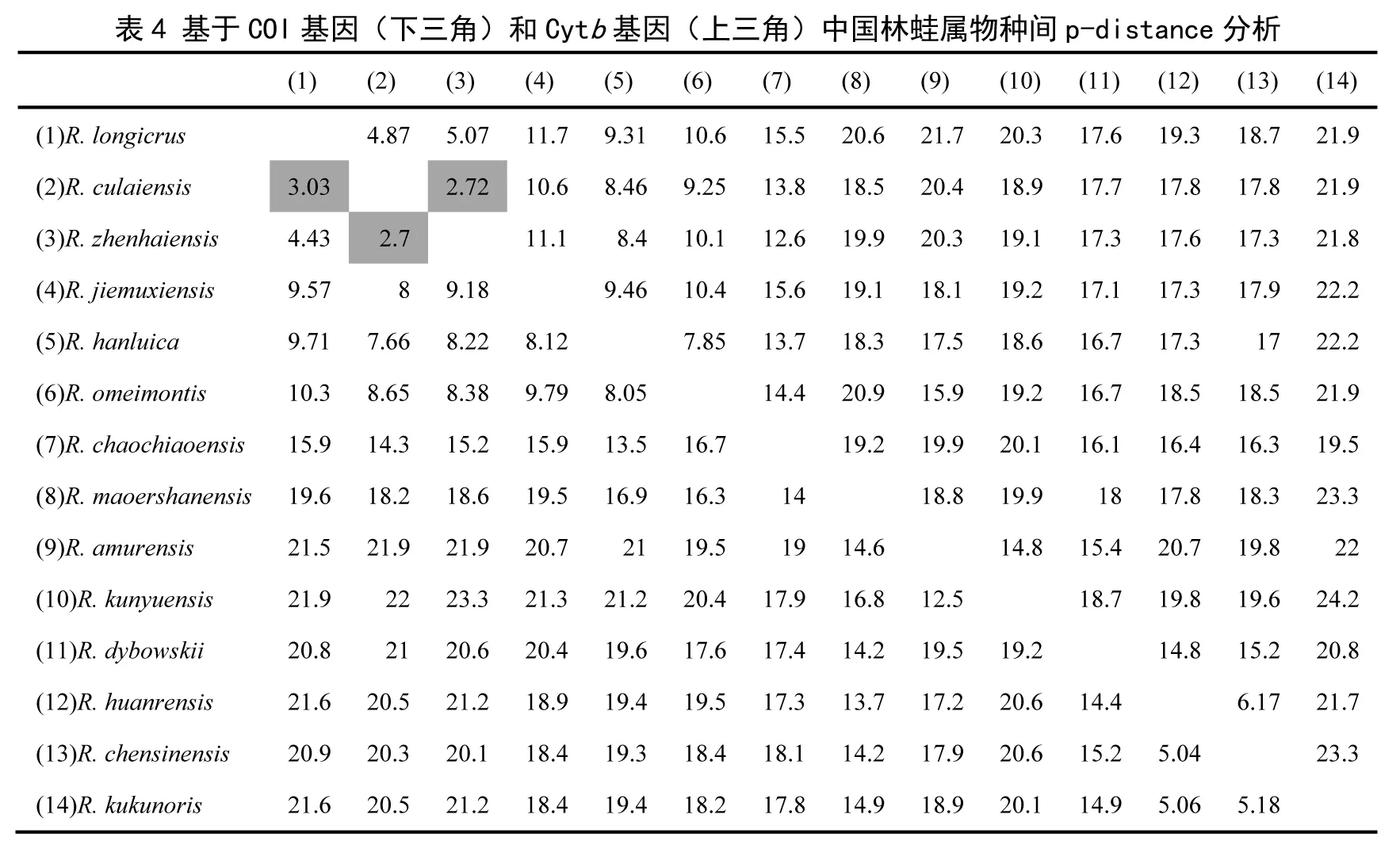
实验测得Cyt b基因片段长度≥629bp,COI基因片段长度~560bp。Kartavtsev和 Lee(2006)[21]基于大量样本分析了Cyt b基因片段与COI基因片段在种内、近缘物种之间、属内各物种间及属间的p-distance变化范围。其中近缘物种间的p-distance值为Cyt b:5.52±1.34,COI:4.91±0.83。为了探讨中国分布的林蛙物种种间关系,我们也对Cyt b基因和COI基因片段序列数据进行了种间p-distance分析(表4)。分析结果显示,除徂徕林蛙与镇海林蛙之间的p-distance(Cytb:2.7;COI:2.7)和徂徕林蛙与长肢林蛙COI基因间p-distance(COI:3)较低外,其余中国分布林蛙属物种种间p-distance大于近缘物种之间p-distance(Cytb:4.9~24.2;COI:4.4 ~23.3)。徂徕林蛙与长肢林蛙 p-distance值介于 Kartavtsev和 Lee(2006)[21]分析的种内与相近物种之间。猫儿山林蛙与其他林蛙属物种的p-distance结果显示,猫儿山林蛙与长肢林蛙种组物种关系较近,最近为昭觉林蛙。
2.2 系统发育树构建
分析构建的ML树和BI树的拓扑结构基本一致(图1)。从图1中可以看出:(1)除徂徕林蛙(Bayesian posterior probabilities,PP=0.73;bootstrap values,BS=0.79)外,其他林蛙物种的单系性均得到了肯定(PP=1.0;BS=1.0);(2)3个林蛙种组中黑龙江林蛙种组与中国林蛙种组的单系性得到支持(PP=1.0;BS=1.0),除猫儿山林蛙以外的长肢林蛙种组单系性也得到支持(PP=1.0;BS=1.0);(3)系统发育树还显示猫儿山林蛙与黑龙江林蛙种组构成姊妹群,但支持度较低(PP=0.52;BS=0.28)。
ML树与BI树结构的不同之处为:(1)ML树显示3个种组间关系为中国林蛙种组与黑龙江林蛙种组加猫儿山林蛙组成的群系首先构成姊妹群,再与长肢林蛙种组合并,而在BI树中这3个并列支系构成了多歧树;(2)在长肢林蛙种组中,ML树由徂徕林蛙、镇海林蛙和长肢林蛙构成的支系先与R.jiemuxiensis结合,再与由峨眉林蛙和寒露林蛙构成的支系结合。BI系统树则为R.jiemuxiensis与上述两个支系构成并系的多歧树。
?

图1 基于线粒体COI基因和Cyt b基因联合分析的ML树(图左)和贝叶斯树(图右)
3 讨论
3.1 物种有效性评价
基于COI和Cyt b基因片段的林蛙属物种间遗传距离分析结果表明,除徂徕林蛙与镇海林蛙间p-distance值较小外,其余林蛙物种间p-distance值均大于Kartavtsev and Lee(2006)界定的种间p-distance值。系统发育树也证实,除徂徕林蛙外,其余林蛙物种的单系性得到支持。所以研究认为徂徕林蛙的物种有效性和分类地位有待进一步全面与镇海林蛙和长肢林蛙进行联合评估,包括几个新种在内的其余林蛙物种有效性均得到肯定。
猫儿山林蛙为Lu等(2007)[7]基于形态学分析发表的林蛙属新物种,与长肢林蛙种组物种形态更为相似。Yang等(2010)[22]基于线粒体16SrRNA基因片段对其分析结果显示,猫儿山林蛙与中国林蛙互为姊妹群。Yan等(2011)[5]对长肢林蛙种组物种进行全面分析,结果显示采自猫儿山地区的形态近似猫儿山林蛙的物种为寒露林蛙,并认为猫儿山林蛙为寒露林蛙的同物异名。我们的研究结果表明虽然猫儿山林蛙在系统发育树中的地位没有得到完全解决,但猫儿山林蛙与其他林蛙物种较大的p-distance(Cyt b:15.9-23.3;COI:13.7-19.6)和系统发育树均证明其并不是寒露林蛙的同物异名物种,猫儿山林蛙为有效物种。
3.2 系统发生关系
中国林蛙种组的单系性得到支持。Zhou等(2012)[12]详细研究了由中国林蛙、高原林蛙和桓仁林蛙组成的中国林蛙复合群(Rana chensinensis species complex),中国林蛙复合群与东北林蛙构成姊妹支系。长肢林蛙种组的单系成立,但其内部物种的系统发生关系复杂。本研究分析结果得到长肢林蛙种组由(1)昭觉林蛙、(2)峨眉林蛙、(3)寒露林蛙、(4)R.jiemuxiensis和(5)徂徕林蛙、镇海林蛙和长肢林蛙5个亚支系构成,但5个亚支系间的系统发生关系没能解决,有待进一步研究。黑龙江林蛙种组的单系性被支持,由黑龙江林蛙与昆嵛林蛙构成的姊妹群组成。
猫儿山林蛙在系统发育树中的地位虽与黑龙江林蛙种组较近,但是较大的p-distance值和系统树中较低的节点支持度均表明猫儿山不属于任何一个种组。其系统地位有待结合更大量的物种样本和序列数据进一步分析。
4 结论
(1)尽管猫儿山林蛙的系统发生地位没有得到完全解决,但其并不是寒露林蛙的同物异名物种,即猫儿山林蛙为有效物种。(2)中国林蛙种组、黑龙江林蛙种组和长肢林蛙种组的单系性得到肯定,长肢林蛙种组物种的系统发生关系有待进一步研究确定。
[1]Frost D.R.Amphibian Species of the World 5.6,an Online Reference.2013;Available from:http://research.amnh.org/herpetology/amphibia/index.php.
[2]费梁,等.中国动物志(两栖纲:无尾目,蛙科)[M].北京:科学出版社,2009:967-1645.
[3]Li P.,Lu Y.,Li A.A new species of brown frog from Bohai,China[J].Asiatic Herpetological Research,2008(11):62-70.
[4]Lu Y.,Li P.,Jiang D.A new species of Rana(Anura,Ranidae)from China[J].Acta Zootaxonomica Sinica,2007(32):792-801.
[5]Yan F.,Jiang K.,Chen H.,et al.Matrilineal History of the Rana longicrus Species Group(Rana ,Ranidae,Anura)and the Description of a New Species from Hunan,Southern China[J].Asian Herpetological Research,2011,2(2):61-71.
[6]Che J.,Pang J.,Zhao E.,et al.Phylogenetic relationships of the Chinese brown frogs(Genus Rana)inferred from partial mitochondrial 12Sand 16SrRNA gene sequences[J].Zoological Science,2007(24):71-80.
[7]SacconeC.,GiorgiI C.D.,Gissi C.,et al.Evolutionary genomics in Metazoa:the mitochondrial DNA as amodel system[J].Gene,1999(238):195-209.
[8]Leach A.D.,Fujita M.K.Bayesian species delimitation in West African forest geckos(Hemidactylus fasciatus)[J].Proceedings of the Royal Society B:Biological Sciences,2010,277(1697):3071-3077.
[9]Che J.,Chen H.M.,Yang J.X.,et al.Universal COI primers for DNA barcoding amphibians[J].Molecular Ecology Resources,2012,12(2):247-258.
[10]Jeong T.J.,Jun J.,Han S.,et al.DNA barcode reference data for the Korean herpetofauna and 4 their applications[J].Molecular Ecology Resources,2013,DOI:10.1111/1755-0998.12055.
[11]Sambrook J.,Fritsch E.F.,Maniatis T.Molecular cloning:a laboratory manual[M].New York:Cold spring harbor laboratory press 1989(2).
[12]Zhou W.,Wen Y.,Fu J.,et al.Speciation in the Rana chensinensis species complex and its relationship to the uplift of the Qinghai-Tibetan Plateau[J].Molecular Ecology,2012,21(4):960-973.
[13]Tanaka-ueno T.,Matsui M.,Wu G.et al.Identity of Rana chensinensis from other brown frogs as assessed by mitochondrial cytochrome b sequences[J].Copeia,1999,99(1):187-190.
[14]Thompson J.D.,Gibson T.J.,Plewniak F.,et al.The ClustalX windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997(25):4876-4882.
[15]Tamura K.,Dudley J.,Nei M.,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007(24):1596-1599.
[16]Lin Y.,Tao B.,Fang X.,et al.The complete mitochondrial genome of Lithobates catesbeianus(Anura:Ranidae)[J].Mitochondrial DNA,2013(07):1-2.
[17]Sumida M.Complete Nucleotide Sequence and Gene Rearrangement of the Mitochondrial Genome of the Japanese Pond FrogRana nigromaculata[J].Genes and Genetic Systems,2001(76):311-325.
[18]Stamatakis A.,Hoover P.,Rorgemont J.A rapid bootstrap algorithm for the RAxML Web servers[J].Systematic Biology 2008(57):758-771.
[19]Ronouist F.,Huelsenbeck J.P.MRBAYES 3:Bayesian phylogenetic inference under mixed models[J].Bioinformatics,2003(19):1572-1574.
[20]Nylander J.A.A.MrModeltest v2.Program distributed by the author,in Evolutionary Biology Centre,Uppsala University,2004.
[21]Kartavtsev Y.P.,Lee J.S.Analysis of Nucleotide Diversity at the Cytochrome b and Cytochrome Oxidase 1 Genes at the Population,Species,and Genus Levels[J].Russian Journal of Genetics,2006(42):341-362.
[22]Yang B.,Lu Y.,Li P.Discussion on validity of Rana maoershanensis based on partial sequence of 16SrRNA gene[J].A-sian Herpetological Research,2010,1(2):97-102.
