梯度金黄色葡萄球菌DNA杂交分子信标激发荧光初探
2014-12-25聂静苑彭新凯何剪太
李 乐 , 聂静苑 , 彭新凯 , 李 林 , 何剪太
(1.中南大学 卫生部肝肠胆外科研究中心,湖南 长沙410008;2. 长沙市食品质量安全监督检测中心,湖南长沙 410013)
近年来,病原菌在全球范围内的肆掠,造成了广泛的关注。数千种病原菌已经被证实对人类具有明显的危害作用,包括感染、食物中毒以及各种免疫反应。
金黄色葡萄球菌是一种葡萄状排列的革兰阳性球菌,该细菌能够造成人类皮肤感染、肺炎、脑膜炎以及严重的食物中毒等状况。有报道显示20%的人群长期携带该种细菌[1]。当前在食品检测以及医疗领域,金黄色葡萄球菌的检测一般都基于常规的生化培养方法,时间3~5 d。因此如何减少检测时间成为摆在食品监督以及医疗卫生领域的一大难题。
近几年,各种关于金黄色葡萄球菌在临床医疗以及食品安全领域快速检测的新方法不绝于耳,相关基础研究工作也在不断的推进,包括基于纳米颗粒的免疫层析法[2],表面增强生物传感器[3]核苷酸链式反应结合酶免反应检测金黄色葡萄球菌的肠毒素A基因[4],环介导恒温扩增法检测金黄色葡萄球菌,以及商业化API试剂盒以及金黄色葡萄球菌测试片等实验室基础研究工作。虽然这些新型的方法较传统的方法能够缩短时间,但由于该类方法的重复性较差,阻碍了其在现实工作中的推广。因此,研究并开发一种能够运用到实际工作中检测金黄色葡萄球菌的检测体系,已成为摆在各类专业技术人员面前的新课题和迫切需要。
分子信标是一种特异的DNA序列结构其中茎部结构由两个互补序列结合杂交而成。一个荧光基团标记在一条序列的一头,而另一头则标记一个淬灭基团。由于荧光基团与淬灭基团的距离很近,荧光信号被淬灭而不被激发。这种特殊的基因机构在很多领域具有运用[5-14]。在作者的研究中,拟借用这样的独特结构,借助ATMND染料结合在核苷酸缺失位点中,同时,利用ATMND互补的C碱基能够淬灭该染料的荧光而暂时失去荧光信号的特点,设计出新奇的检测方法。这种方法针对金黄色葡萄球菌序列并结合分子信标的环状互补序列,同时在分子信标的茎部设计基因缺失位点,让ATMND染料能够嵌入且被互补的C碱基淬灭。由于ATMND染料能够自动插入缺失位点,并被C碱基淬灭,因此当杂交目标序列被混入探针溶液后,茎环结构的构象将发生根本改变,分子信标变成打开状态。ATMND染料也远离了C碱基,荧光信号被重新恢复。因此通过检测荧光强度就能客观反应目标DNA的存在。这种利用ATMND无标记探针方法已经成功的在腺苷、Pb2+、UO2、Hg2+的检测[15-18]中有所使用。当然,这样的独特的结构与设计方法与传统的分子信标方法检测比较,大幅地降低了检测成本。作者将不同浓度的金黄色葡萄球菌DNA与设计独特的分子信标探针进行杂交,以获得金黄色葡萄球菌DNA与分子信标探针荧光恢复的比例状况,同时验证该分子信标探针以及结合体系与对金黄色葡萄球菌DNA是否存在相关特异性。为将来该方法真正用于金黄色葡萄球菌的检测工作中奠定坚实的基础。
1 材料与方法
1.1 材料
1.1.1 试剂 ATMND:美国RYAN科技公司产品;细菌DNA提取试剂盒:鼎国生物产品;半固体培养基:广东环凯生物技术有限公司产品;脑心浸液:广东环凯生物技术有限公司产品;DNA序列由上海生工生物有限公司合成,序列可以参看表1。其他的试剂均为分析纯试剂并未进行其他纯化和改变。所有的实验用水均使用双蒸水进行。

表1 DNA序列列表Table 1 Sequences of oligonucleotides used in this work
1.1.2 菌株及培养基 阳性标准菌株为金黄色葡萄球菌 (ATCC6538),阴性菌株为阪崎肠杆菌(ATCC2900)、大肠埃希菌(ATCC25922)、铜绿假单胞菌(ATCC27853)、单核细胞增生李斯特菌(CMCC(B)54002)、沙门氏菌(CMCC(B)50115)。 所有的细菌均保存于4℃,半固体培养基中。所有的菌种复苏以及增菌培养基均由脑心浸液培养。在整个实验过程中所有设计细菌分离和DNA提取步骤均在生物安全柜中进行,废物处理均为高压灭菌锅121℃灭菌20分钟后丢弃。
1.1.3 主要仪器 Mastercycler:EPPEDORF公司产品;荧光分光光度计:F7000,日立公司产品。
1.2 实验原理
作者设计了能与金黄色葡萄球菌的目标DNA杂交的含有脱碱基位点的茎-环结构,从图1可知,在缺乏目标DNA时,ATMND与茎-环结构的脱碱基位点紧密结合,从而抑制其荧光信号。当金黄色葡萄球菌黄色葡萄球菌目标DNA加入到溶液中,其与茎-环结构DNA杂交,ATMND从D-SPACER位点释放,从而使被猝灭的荧光信号重新恢复。这种基于ATMND染料的茎环结构基因缺失位点探针与金黄色葡萄球菌的特异DNA目标序列的检测效果。这种探针对于金黄色葡萄球菌DNA特异性目标序列具有相当的敏感性并能够使得荧光复原的效果。

图1 ATMND荧光分子信标基因探针与黄色葡萄球菌目标DNA结合释放荧光染料激发荧光示意图Fig.1 Scheme of motivated fluorescence by StaphylococcusaureusDNA hybrid with stem-loop structure probe with ATMND
1.3 实验方法
1.3.1 不同梯度金黄色葡萄球菌DNA与分子信标杂交,荧光强度恢复实验 在离心管内分别加入360 μL 缓 冲 液 (10 mmol/L HEPES pH 7.0,100 mmol/L NaCl和 1 mmol/L EDTA),24 μL ATMND(1 μmol/L),36 μL 茎环结构的 DNA(10 μmol/L)以及不同体积的 3,6,9,12,15,18,21,24 μL 的金黄色葡萄球菌特异性目标DNA序列(10 μmol/L),即金黄色葡萄球菌特异性目标DNA序列终浓度分别为7.09×10-2,1.41×10-1,2.10×10-1,2.78×10-1,3.45×10-1,4.11×10-1,4.76×10-1,5.41×10-1μmol/L。 漩涡震荡后,用MASTERCYCLER加热器对溶液进行70℃10 min变性处理,再逐步30 min内冷却到5℃。然后将混合液转移到石英比色皿中,并持续控制在5℃测定。5 min后,用荧光分光光度计检测荧光强度ex/em=358/405 nm。
1.3.2 标准菌株DNA与分子信标探针验证该体系的特异性 在离心管内分别加入360 μL缓冲液(10 mmol/L HEPES pH 7.0,100 mmol/L NaCl和 1 mmol/L EDTA),24 μL ATMND (1 μmol/L),36 μL茎环结构的DNA (10 μmol/L)以及不同标准菌株(金黄色葡萄球菌,阪崎肠杆菌、大肠埃希菌、铜绿假单胞菌、单核细胞增生李斯特菌、沙门氏菌)的DNA 各 6 μL (10 μmol/L)。 漩 涡 震 荡 后 , 用MASTERCYCLER加热器对溶液进行70℃10 min变性处理,再逐步30 min内冷却到5℃。然后将混合液转移到石英比色皿中,并持续控制在5℃测定。5 min后,用荧光分光光度计检测荧光强度ex/em=358/405 nm,方法参考1.2.1。
2 结 果
2.1 金黄色葡萄球菌目标DNA的量与荧光强度的关系的探讨
为了进一步评估分子信标探针的实用效果,在相同的条件下,对金黄色葡萄球菌目标DNA的量与荧光强度的关系进行研究,金黄色葡萄球菌DNA的量从 3~24 μL(10 μmol/L)逐渐增加,随着目标DNA的量的增加,荧光强度增大,从金黄色葡萄球菌DNA 3 μmol/L的荧光强度10一直上升至金黄色葡萄球菌DNA 24 μL的990。其荧光强度随着目标DNA的浓度增高而增强。如图2所示。即在本实验体系中金黄色葡萄球菌特异性目标DNA序列终浓度的检测下限为7.09×10-2μmol/L。
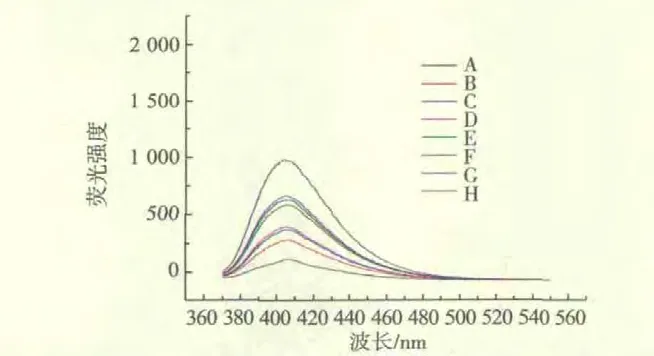
图2 金黄色葡萄球菌目标DNA的量与荧光强度的关系Fig.2 Relationship between fluorescence enhancement rateand StaphylococcusaureustargetDNA concentrations
2.2 茎环结构无标记探针与实际细菌DNA序列结合的荧光效果的测定
随后,在相同的条件下,对不同种类的标准菌株DNA进行了探讨,以验证其分子信标探针以及体系对对金黄色葡萄球菌DNA的特异性。标准金黄色葡萄球菌菌株和其它标准阴性对照菌株DNA也同时进行了荧光检测比较,如图3所示。当标准金黄色葡萄球菌的DNA被加入并与探针杂交后,ATMND从缺失位点释放,且产生明显的荧光恢复信号。而其它阴性对照菌株DNA对这种探针具有不敏感或者是荧光信号恢复不强等效果。包括大肠埃希氏菌、单核细胞增生李斯特菌、阪崎肠杆菌、铜绿假单胞菌以及沙门氏菌荧光激发不明显。作者验证了该分子信标探针以及实验体系对于金黄色葡萄球菌标准菌株DNA具有一定的敏感性。为将来的实际检测鉴定了坚实的理论和数据基础。
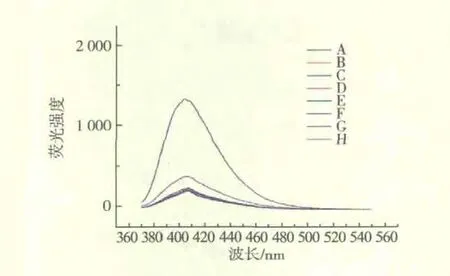
图3 荧光分光光度计检测茎环结构无标记探针与实际细菌DNA序列结合的荧光效果Fig.3 Fluorescence spectra of stem-loop structure probhybrid with standard strains DNA
3 讨 论
这种基于缺失DNA位点的分子信标探针能够很好的利用ATMND染料的特点,结合特异性金黄色葡萄球菌目标DNA序列,通过茎-环结构的构象改变,重新激发ATMND荧光。由于荧光的强度客观反应出目标细菌DNA量的多少,因此,这种独特的检测探针在测试金黄色葡萄球菌DNA的有无或者半定量上具有一定的特点和先进性。当然,实验过程中ATMND染料具有高温随意进出缺失位点的特殊性,因此整个过程需要持续在5℃的温度下进行。最后,与传统的生物化学培养方法相比,这种技术还是具有明显的先进性,包括在时间效率以及成本估算中占有一定额优势。今后,将继续利用这种方法在未来的实际运用,包括更复杂的食品和临床样本环境体系,或者无需DNA提取的条件下进行基础研究和测定工作。
[1]Kluytmans J A J W,van BelkumA,Verbrugh H A,et al.Nasal carriage of Staphylococcus aureus:epidemiology,underlying mechanisms,and associated risks.Clin.Microbiol.Rev[J].1997,10:505-520.
[2]Huang S H.Gold nanoparticle-based immunochromatographic assay for the detection of Staphylococcus aureus[J].Sens Actuators B:Chem,2007,127:335-340.
[3]Chang T C,Huang S H.Evaluation of a latex agglutination test for rapid identification of Staphylococcus aureus from foods[J].J Food Prot,1993,56:759-762.
[4]Aitichou M,Henkens R,Sultana A M,et al.Detection of Staphylococcus aureus enterotoxin A and B genes with PCR-EIA and a hand-held electrochemical sensor[J].Mol Cell Probes,2004,18:373-377.
[5]Cao Z h,Suljak,Steven W,et al.Molecular Beacon Aptamers for Protein Monitoring in Real-Time and in Homogeneous Solutions[J].Curr Proteomics,2005,2:31-40.
[6]Cho E J,Rajendran M,Ellington A D,Aptamers as Emerging Probes for Macromolecular Sensing[J].Advanced Concepts in Fluorescence SensingTopics in Fluorescence Spectroscopy,2005,10:127-155.
[7]Swensen J S,Xiao Y,Ferguson B S,et al.Real-time monitoring of cocaine in undiluted blood serum via a microfluidic,electrochemicalaptamer-based sensor[J].J Am Chem Soc,2009,131:4262-4266.
[8]Freeman R,Finder T,WillnerI.Multiplexed analysis of Hg2+and Ag+ions by nucleic acid functionalized CdSe/ZnS quantum dots and their use for logic gate operations[J].Angew Chem Int Ed,2009,48:7818-7821.
[9]Zuo X,Xiao Y,Plaxco KW.High specificity, electrochemical sandwich assays based on singleaptamer sequences and suitable for the direct detection of small-molecule targets in blood and other complex matrices[J].J Am Chem Soc,2009,131:6944-6945.
[10]Rhee W J,Santangelo P J,Jo H,et al.Target accessibility and signal specificity in live-cell detection of BMP-4 mRNA using molecular beacons[J].Nucleic Acids Res 2008,36(5):e30.
[11]Silverman A P,Kool E T.Quenched probes for highly specific detection of cellular RNAs[J].Trends Biotechnol,2005,23:225-230.
[12]Wu SM,Tian Z Q, Zhang Z L,et al.Direct fluorescence in situ hybridization (FISH) in Escherichia coli with a target-specific quantum dot-based molecular beacon[J].Biosens Bioelectron,2010,26:491-496.
[13]Rai V,Nyine Y T,Hapuarachchi H C,et al.Electrochemically amplified molecular beacon biosensor for ultrasensitive DNA sequence-specific detection of Legionella sp[J].Biosens Bioelectron,2012,32:133-140.
[14]Sandhya S,Chen W,Mulchandani A.Molecular beacons:a real-time polymerase chain reaction assay for detecting Escherichia coli from fresh produce and water.Anal.Chim[J].Acta,2008,614:208-212.
[15]Wang K,Tang Z,Yang C J,et al.Molecular Engineering of DNA:Molecular Beacons[J].Angew Chem,2009,48:856-870.
[16]Xiang Y,Wang Z,Xing H,et al.Label-Free Fluorescent Functional DNA Sensors Using Unmodified DNA:A Vacant Site Approach[J].Anal Chem,2010,82:4122-4129.
[17]Sato Y,Nishizawa S,Teramae N.Label-free molecular beacon system based on DNAs containingabasic site and fluorescent ligands that bind abasic sites[J].Chem Eur J,2011,17:11650-11656.
[18]Su Y X,Gao S,Kang L,et al.Establishment of real-time quantitative PCR basedmethods for detection of Staphylococcus aureus in food[J].Bull Acad Mil Med Sci,2010,34:25-29.
