厌氧氨氧化耦合脱氮系统中反硝化细菌研究
2014-12-14王春香刘常敬郑林雪北京工业大学建筑工程学院北京100124
王春香,刘常敬,郑林雪,李 军 (北京工业大学建筑工程学院,北京 100124)
厌氧氨氧化耦合脱氮系统中反硝化细菌研究
王春香,刘常敬,郑林雪,李 军*(北京工业大学建筑工程学院,北京 100124)
采用聚合酶链式反应、分子克隆等分子生物学方法,通过构建nirS克隆文库研究了厌氧氨氧化耦合异养反硝化脱氮系统中的反硝化微生物.进行同源性分析和系统发育树构建,结果表明,从nirS克隆文库中随机挑选的100个克隆子中有51个阳性克隆子,共分为12个操作单元.经比对发现这些微生物主要为变形菌门细菌和未知菌类,其中 β-proteobacteria占据绝对优势且有较多种类,少部分属于γ-proteobacteria.
厌氧氨氧化;反硝化菌;聚合酶链式反应(PCR);nirS克隆文库
厌氧氨氧化(ANAMMOX)工艺是由荷兰Delft大学开发的生物脱氮新技术[1],厌氧氨氧化反应器对氮的去除主要是利用厌氧氨氧化细菌作用,以氨氮为电子受体,将亚硝酸盐氮还原为氮气.反硝化作用的概念是由Gayon等[2]于 1882 年提出的,是指硝酸根和亚硝酸根通过微生物的异养还原作用,变成一氧化氮,一氧化二氮,或最终还原成氮气的过程,在废水处理系统中反硝化作用已经发展成为一个有效去除氮素的步骤.异养的反硝化过程需要碳源,研究发现,不但小分子的有机物,如甲醇、甲醛等可以作为反硝化过程的能量物质,一些复杂的大分子有机物也能够被反硝化细菌所利用.根据这一特性,可以利用反硝化细菌来降解废水中的复杂有机物.反硝化作用主要是由反硝化细菌引起的,反硝化细菌具有降解各种有机污染物的能力,包括芳香族和脂肪烃类[3].然而大部分细菌是不可培养或难以培养的,使得分离培养方法研究反硝化细菌往往不够全面,利用分子生物学方法来认识反硝化菌将更具有科学性.
反应器以苯酚作为有机碳源,酚等有机物虽然对微生物有抑制作用,但可以作为电子供体用于反硝化[4-6],反硝化降解酚类有机物就会解除其毒性抑制作用.Toh等[7]在用ANAMMOX工艺处理焦化废水的研究中证实了反硝化菌的存在.
本实验利用PCR、分子克隆等分子生物学方法,研究了以苯酚为电子供体的厌氧氨氧化耦合异养脱氮系统内的反硝化菌的种类.
1 材料与方法
1.1 采样
污泥取自厌氧氨氧化耦合异养反应器底部.试验采用 UASB生物膜反应器,有效容积为10L,反应器内温度维持在 25℃左右,水力停留时间为1.5h,进水pH值控制在7.8左右,反应器长期稳定运行.反应器的进出水指标如图 1所示.-N、-N去除率高达 100%,TN去除率为 87.51%,异养反硝化对 TN去除贡献率达到13.28%.

图1 进、出水-N、-N和-N含量变化Fig.1 Variation of -N, -N and -N during the operation
1.2 细菌总DNA提取
泥样在1.5mL离心管中静沉10min后舍去上清液,15000r/min高速离心 5min后,去除上层清液,取0.3g用于总DNA提取,剩余污泥冷冻备用.采用Ezup柱式基因组DNA提取试剂盒(上海生工)提取后,取 5µL用 1.2%琼脂糖凝胶检测.其余置于-20℃储存备用.
1.3 聚合酶链式反应(PCR)
将提取的细菌总 DNA作为 PCR的模板,PCR反应在 MyiQ Real-time PCR扩增仪(美国,Bio-Rad)上进行,采用反硝化菌特异性引物对nirS1F和nirS6R[8].nirS1F和nirS6R引物序列分别为:
nirS1F:5’-CCT A(C/T)T GGC CGC C (A/G)C A (A/G)T-3’
nirS6R:5’-CGT TGA ACT T (A/G)C CGG T-3’
采用25µL反应体系:10x PCR Buffer, 2.5µL;dNTPs (各 2.5mmol/L),1.0µL; MgCl2(25mmol/L),1.5µL;nirS1F(20µmol/L),0.5µL;nirS6R(20µmol/L),0.5µL;Taq酶(5U,Takara)0.3µL; DNA 1.0µL,加灭菌超纯水到25µL.
PCR 反应条件:95℃预变性 6min;95℃变性30s,60℃退火40s,72℃延伸1min,10个循环;95℃变性 30s,55℃退火 40s,72℃延伸 1min,25个循环;72℃延伸7min;4℃保持.
1.4 克隆、转化和nirS克隆文库的建立
PCR产物采用 SanPrep 柱式 DNA 胶回收试剂盒(上海生工)进行纯化后,与pMD18-T载体(Takara)连接.连接体系(总体积 10µL):PCR 产物3µL;pMD18-T Vector 1µL;Solution I 5µL;ddwater 1µL.将连接产物转入 DH5α感受态细胞中,用含X-Gal、IPTG、Amp的LB培养基37℃培养16h后,进行蓝白班筛选.从克隆文库中随机挑选 100个白斑,用pMD18-T载体引物RV-M和M13-47作为PCR引物,验证筛选的克隆插入的片段大小是否正确.
M13-47:5’-GAGCGGATAACAATTTCAC ACAGG-3’;
RV-M:5’-CGCCAGGGTTTTCCCAGTCAC GAC-3’.
片段大小正确的PCR产物用 Hha I (Takara,Japan)限制性内切酶进行酶切.Hha I 酶切体系(10µL)为:Hha I,0.5µL;10×PCR Buffer,1µL;PCR产物5µL;ddwater,3.5µL.酶切反应于37℃进行4h,酶切产物用 3%的琼脂糖凝胶电泳检测,然后在凝胶成像系统中成像,并根据凝胶成像结果中的酶切带型划分操作单元(OTU).基因文库中每个OTU选择一个代表菌株送往上海生工生物公司测序.
2 结果与讨论
2.1 细菌总DNA提取
污泥样品提取的总 DNA经琼脂糖凝胶电泳检测,DNA提取电泳图如图 2,可见所提取的 DNA片段分子量大小在 23kbp左右,属于比较完整的细菌总 DNA,适用于下一步PCR扩增.

图2 细菌总DNA提取电泳Fig.2 Electrophoresis of the DNA extracts
2.2 PCR扩增及产物纯化
PCR扩增结果如图 3所示.用反硝化细菌nirS基因特异性引物扩增所产生的DNA片段大小为890bp,符合预期的长度,但有非特异性扩增,需要进行切胶纯化.图4为PCR扩增产物切胶纯化后的结果.

图3 反硝化细菌PCR扩增电泳图Fig.3 Electrophoresis of the PCR productions

图4 反硝化细菌PCR扩增产物纯化Fig.4 Electrophoresis of the PCR productions after purification
2.3 反硝化细菌nirS克隆文库
在构建的反硝化细菌nirS克隆文库中,随机挑选100个进行PCR,共得到51个阳性克隆子.图5为部分结果.
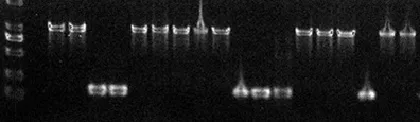
图5 克隆子的PCR电泳Fig.5 Electrophoresis of the PCR results of clones
2.4 反硝化菌的酶切和序列测定
采用Hha I限制性内切酶对51个阳性克隆子进行酶切分型,部分酶切结果如图5所示.共得到12个OTUs,每个OUT所包含的克隆子数量不一样,从1到21个不等.

图6 HhaI酶切图谱Fig.6 Restriction patterns of denitrifying bacteria digested by Hha I
对12个OUTs的代表性克隆子进行克隆测序,将测序所得基因序列输入到NCBI网站,利用BLAST程序与数据库中已有的序列进行比对鉴定,发现大部分克隆子比对后与最近物种的相似度为 77%~94%.这是由于 nirS基因序列较长(达900bp左右).因此比对到的与最相似物种的相似度并没有像普通 16SrDNA比对结果那么高(200bp左右的16SrDNA比对的相似度一般都高达 99%左右.根据莫旭华等[9]的群落结构计算方法和系统学分析,该文库的库容大小为 90%,Shannon–Weiner指数为 2.02, Simpson指数为0.79,丰富度为3.05.

表1 样品克隆测序比对结果Table 1 Results of the best march to known species of the clones
表1所示为克隆序列测序比对后的信息.从表中可以看出比对的结果主要为变形菌门细菌和未知菌类.其中β-proteobacteria占据绝对优势且有较多种类,其 OTUs和克隆丰富度分别为58.3%和 62.7%.少部分属于 γ-proteobacteria的Pseudomonas stutzeri(恶臭假单胞菌属)和盐单胞菌属(Halomonas sp.),其 OTUs和克隆丰富度分别为16.7%和7.8%.说明变形杆菌在该脱氮系统中是最主要的菌群[10],还有部分未培养的环境样品,其OTUs和克隆丰富度分别为16.7%和9.8%,证实了传统纯化分离、培养等微生物学分析测定技术难于真实准确的反映出反应器运行过程中微生物的群落构成及数量,分子生物学技术可以检测出未培养菌属和未知菌属,优势显而易见.此外,还有一个克隆在数据库中比对后没有找到已知的相近物种,未能得到详细信息,相关研究也得出类似结论[11-12].
郑平等[13]和Heylen等[14-15]指出,各种环境中最常见的反硝化细菌都属于变形菌纲.在污水中,nirS反硝化菌属多是 β-proteobacteria和γ-proteobacteria[16].在本研究中,比对得到的菌属大 部 分 归 属 于 Proteobacteria 门,βproteobacteria纲数量上占有绝对优势,包括Comamonadaceae、Azoarcus tolulyticus、Thauera aromatica和 Cupriavidus.少部分属于γ-proteobacteria,包括 Pseudomonas stutzeri 和Halomonas sp.,研究结果具有一致性.这些细菌不仅具有反硝化作用,而且为异养型细菌,对有机碳源苯酚的降解有重要作用.对于苯酚生物降解的研究,国外起步比较早,己经分离得到的许多苯酚降解菌中就包括 Pseudomonas[17-21].β-proteobacteria纲中的 Azoarcus和 Thauera属的大多数成员都与反硝化条件下芳香族化合物[22-23]的降解有关.Anders等[24]在1995年发现,在厌氧条件下 Thauera aromatica能够利用phenol、benzoate等作为唯一碳源生长来降解污染物.此外,Manefield等[25]使用RNA-SIP 技术证明 Thauera sp.和Acidovorax[26]是苯酚的主要降解菌.1997年,Hinteregger等[27]发现在鼓泡塔反应器中Halomonas sp.可完全降解盐含酚废水中的苯酚.Comamonadaceae属细菌具有降解芳香族化合物的能力也已被报道[28-29].本研究中,苯酚浓度 18.82mg/L的条件下,耦合反应器能长期稳定运行.异养反硝化对 TN去除贡献率达到13.28%,可以证明 Proteobacteria对于系统的脱氮和苯酚的降解有很大贡献.
王弘宇等[30]发现生物陶粒脱氮反应区内假单胞菌(Pseudomonas sp.)对氮素的去除率达到 43.90%.李佳霖等[31]研究长江口邻近海域沉积物中通过微生物的反硝化途径去除的氮约为初级生产消耗无机氮量的1/3.以上数据说明,反硝化细菌对于污水生物处理系统内氮素的去除有很大的贡献.本研究中,反硝化菌对 TN去除贡献率达到 13.28%,与以上研究结果具有相似性.
2.5 构建系统发育树
对获得的克隆和相应的最相近已知物种构建系统发育树,结果如图7所示.从系统发育树可以看出,大部分的阳性克隆子和已知的反硝化细菌具有较高的相似性,也就说明了在本研究中的反硝化菌基本由普通的反硝化菌构成.同时也有大量分离的克隆子不与目前已知的 nirS 基因相似,属于未知菌属,说明水处理中的反硝化群落需要进一步研究.
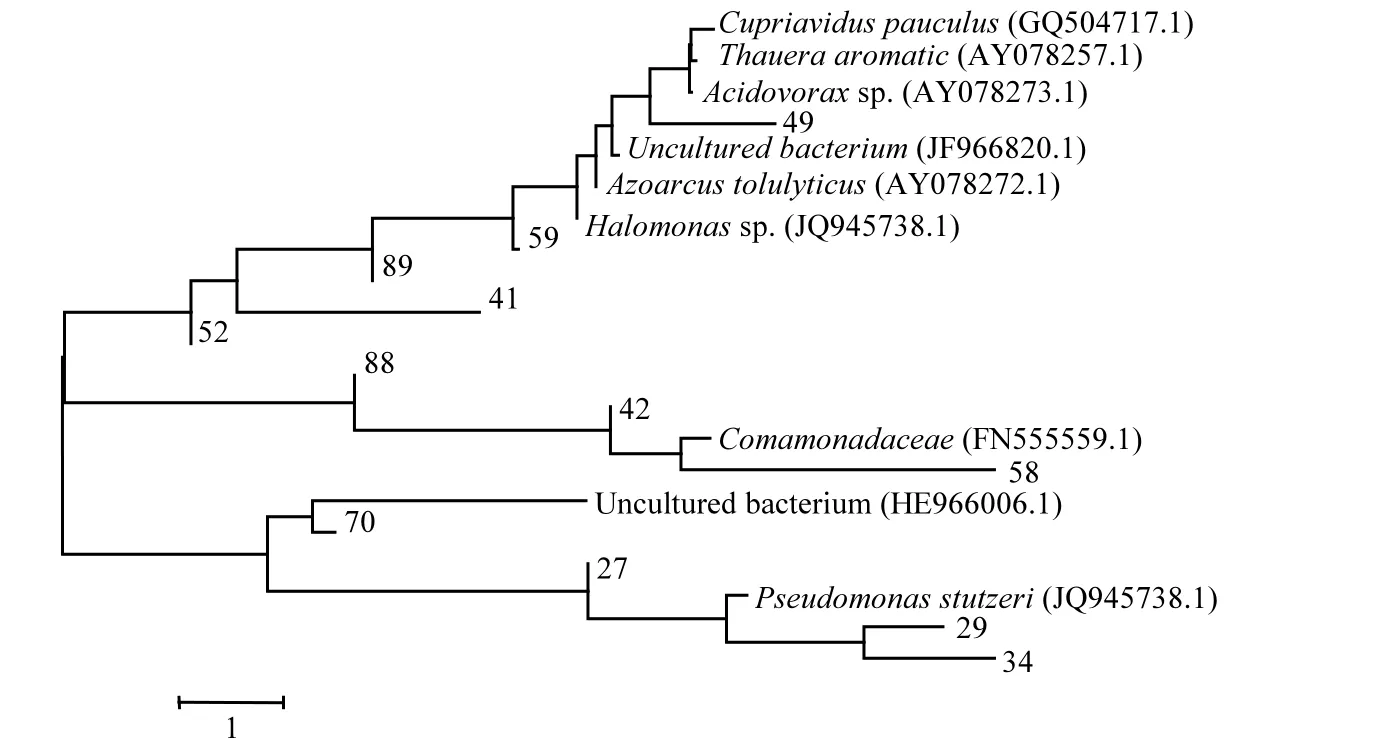
图7 反硝化细菌系统发育分析Fig.7 The phylogenetic analysis results of the denitrifying bacteria
3 结论
3.1 反应器所得克隆子比照后菌属主要为变形菌门细菌和未知菌类.其中 β-proteobacteria占据绝对优势且有较多种类,其OTUs和克隆丰富度分别为58.3%和62.7%.少部分属于 γ-proteobacteria,其OTUs和克隆丰富度分别为16.7%和7.8%.说明变形杆菌在该脱氮系统中是最主要的菌群.还有部分未培养的环境样品,其OTUs和克隆丰富度分别为16.7%和9.8%.
3.2 反应器中的优势反硝化菌为变形菌纲细菌,对于系统的氮的去除和苯酚的降解有很大贡献,对TN去除贡献率达到13.28%.
[1]Mulder A, Graaf A A, Robertson L A, et al. Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor [J].FEMS Microbiology Ecology, 1995,16(3):177-184.
[2]Gayon U, Dupetit G. Sur la fermentation des nitrates [J]. CR Acad.Sci., 1882,95:644-646.
[3]Holliger C, Zehnder A J B. Anaerobic biodegradation of hydrocarbons [J]. Current Opinion in Biotechnology, 1996,7(3):326-330.
[4]Zhu J H, Lin J P, Zhang B, et al. Simultaneous removal of phenol and nitrate in an anaerobic bioreactor [J]. Journal of Environmental Engineering, 2006,132(9):1073-1077.
[5]Maranon E, Vazquez I, Rodriguez J, et al. Coke wastewater treatment by a three-step activated sludge system [J]. Water, Air,and Soil Pollution, 2008,192(1-4):155-164.
[6]Ramos A F, Gomez M A, Hontoria E, et al. Biological nitrogen and phenol removal from saline industrial wastewater by submerged fixed-film reactor [J]. Journal of Hazardous Materials,2007,142(1):175-183.
[7]Toh S, Ashbolt N. Adaptation of anaerobic ammonium-oxidising consortium to synthetic coke-ovens wastewater [J]. Applied Microbiology and Biotechnology, 2002,59(2/3):344-352.
[8]Braker G, Zhou J, Wu L, et al. Nitrite reductase genes (nirK and nirS)as functional markers to investigate diversity of denitrifying bacteria in Pacific Northwest marine sediment communities [J].Applied and Environmental Microbiology, 2000,66(5):2096-2104.
[9]莫旭华,麻 威,史荣久,等.氮肥对小麦田土壤 nirS型反硝化细菌多样性的影响 [J]. 微生物学报, 2009(9):1203-1208.
[10]Yu Z, Mohn W W. Bacterial diversity and community structure in an aerated lagoon revealed by ribosomal intergenic spacer analyses and 16S ribosomal DNA sequencing [J]. Applied and Environmental Microbiology, 2001,67(4):1565-1574.
[11]Chon K, Kim Y, Chang N I, et al. Evaluating wastewater stabilizing constructed wetland, through diversity and abundance of the nitrite reductase gene, with regard to nitrogen control [J]. Desalination, 2010,264(3):201-205.
[12]方 芳,王淑梅,冯翠杰,等.好氧条件下复合生物膜-活性污泥反应器中的反硝化菌群结构 [J]. 生态学杂志, 2011,30(3):430-437.
[13]郑 平,徐向阳,胡宝兰.新型生物脱氮理论与技术 [M]. 北京:科学出版社, 2004.
[14]Heylen K, Gevers D, Vanparys B, et al. The indicies of nirS and nirK and their genetic heterogeneity in cultivated denitrifiers [J].Environmental Microbiology, 2006,8:2012-2021.
[15]Heylen K, Vanparys B, Gevers D, et al. Nitric oxide reductase(norB)gene sequence analysis reveals discrepancies with nitrite reductase (nir)gene phylogeny in cultivated denitrifiers [J].Environmental Microbiology, 2007,9:1072-1077.
[16]Lee H W, Lee S Y, Lee J W, et al. Molecular characterization of microbial community in nitrate-removing activated sludge [J].FEMS Microbiology Ecology, 2002,41(2):85−94.
[17]Kotresha D, Vidyasagar G M. Isolation and characterisation of phenol-degrading Pseudomonas aeruginosa MTCC 4996 [J].World Journal of Microbiology and Biotechnology, 2008,24(4):541-547.
[18]Kukor J J, Olsen R H. Molecular cloning, characterization, and regulation of a Pseudomonas pickettii PKO1 gene encoding phenol hydroxylase and expression of the gene in Pseudomonas aeruginosa PAO1c [J]. Journal of Bacteriology, 1990,172(8):4624-4630.
[19]Pawlowski K, Kunze R, De Vries S, et al. Isolation of total, poly(A)and polysomal RNA from plant tissues [M]//Plant molecular biology manual. Springer Netherlands, 1994:231-243.
[20]Hamdy M K, Sherrer E L, Randles C I, et al. Some characteristics of a phenol-oxidizing Pseudomonas [J]. Appl. Microbiol.,1956,4(2):71.
[21]Feist C F, Hegeman G D. Phenol and benzoate metabolism by Pseudomonas putida: regulation of tangential pathways [J].Journal of Bacteriology, 1969,100(2):869-877.
[22]Mechichi T, Stackebrandt E, Gad'on N, et al. Phylogenetic and metabolic diversity of bacteria degrading aromatic compounds under denitrifying conditions, and description of Thauera phenylacetica sp. nov., Thauera aminoaromatica sp. nov., and Azoarcus buckelii sp. nov [J]. Archives of Microbiology, 2002,178(1):26-35.
[23]Zhou J, Fries M R, Chee-Sanford J C, et al. Phylogenetic analyses of a new group of denitrifiers capable of anaerobic growth on toluene and description of Azoarcus tolulyticus sp. nov[J]. International Journal of Systematic Bacteriology, 1995,45(3):500-506.
[24]Anders H J, Kaetzke A, KÄMPFER P, et al. Taxonomic position of aromatic-degrading denitrifying pseudomonad strains K 172 and KB 740 and their description as new members of the genera Thauera, as Thauera aromatica sp. nov., and Azoarcus, as Azoarcus evansii sp. nov., respectively, members of the beta subclass of the Proteobacteria [J]. International Journal of Systematic Bacteriology, 1995,45(2):327-333.
[25]Manefield M, Whiteley A S, Griffiths R I, et al. RNA stable isotope probing, a novel means of linking microbial community function to phylogeny [J]. Applied and Environmental Microbiology, 2002,68(11):5367-5373.
[26]Manefield M, Griffiths R I, Leigh M B, et al. Functional and compositional comparison of two activated sludge communities remediating coking effluent [J]. Environmental Microbiology,2005,7(5):715-722.
[27]Hinteregger C, Streichsbier F. Halomonas sp., a moderately halophilic strain, for biotreatment of saline phenolic waste-water[J]. Biotechnology Letters, 1997,19(11):1099-1102.
[28]Boon N, Goris J, De Vos P, et al. Genetic diversity among 3-chloroaniline-and aniline-degrading strains of the Comamonadaceae [J]. Applied and Environmental Microbiology,2001,67(3):1107-1115.
[29]Khan S T, Horiba Y, Yamamoto M, et al. Members of the family Comamonadaceae as primary poly (3- hydroxybutyrate - co- 3 -hydroxyvalerate)-degrading denitrifiers in activated sludge as revealed by a polyphasic approach [J]. Applied and Environmental Microbiology, 2002, 68(7):3206-3214.
[30]王弘宇,马 放,杨 开,等.两株异养硝化细菌的氨氮去除特性[J]. 中国环境科学, 2009,29(1):47-52.
[31]李佳霖,白 洁,高会旺,等.长江口海域夏季沉积物反硝化细菌数量及反硝化作用 [J]. 中国环境科学, 2009,29(7):756-761.
Denitrifying bacteria of anaerobic ammonium-oxidizing denitrifying system.
WANG Chun-xiang, LIU Changjing, ZHENG Lin-xue, LI Jun*(Key Laboratory of Beijing for Water Quality Science and Water Environment Recovery Engineering, Beijing University of Technology, Beijing 100124, China). China Environmental Science,2014,24(7):1878~1883
Denitrifying microorganisms in the anaerobic ammonium oxidation coupling heterotrophic denitrification system were investigated. The methods used were PCR and molecular cloning. By building nirS clone libraries, homology analysis and constructing system Phylogenetic tree, 51clones were assigned to 12 OTUs. By BLAST alignment found these microbes were mainly proteobacteria and uncultured environment samples. Proteobacteria were mainly β-Proteobacteria and γ-Proteobacteria.
anaerobic ammonium-oxidizing (ANAMMOX);denitrifying bacteria;polymerase chain reaction (PCR);nirS clone libraries
X172
A
1000-6923(2014)07-1878-06
2013-10-25
国家自然科学基金(51078008);北京市自然科学基金(8122005);北京市教委重点项目(KZ201110005008)
* 责任作者, 教授, jglijun@bjut.edu.cn
王春香(1988-),女,山东菏泽人,北京工业大学硕士研究生,研究方向污水生物处理.
