塔克拉玛干沙漠生物结皮中几种藻类的系统发育分析
2014-11-12王丹龚春霞苟亚峰周路朱军保高剑峰
王丹,龚春霞,苟亚峰,周路,朱军保,高剑峰
(石河子大学生命科学学院,新疆 石河子832000)
据统计,2011年中国北方沙漠化土地达37.59万km2,其中轻度沙漠化土地占33.80%,中度沙漠化土地占22.84%,重度沙漠化土地占22.16%,严重沙漠化土地面积占21.21%,沙漠化已成为人类所面临的严重的环境问题之一[1]。在沙漠化修复的研究中,现阶段固沙的主要生物技术方法为防风林及草带的建立[2],与此同时微生物在沙漠化治理的研究中也得到了初步探讨。潘惠霞等[3]在新疆古尔班通古特沙漠的结皮中分离到了一株寡营养细菌,该菌株可产生大量胞外粘多糖,将该菌株的培养液喷洒到流沙表面后,可形成6 mm厚的粘合性的沙层。邹碧莹和张元翼[4]在地中海沙漠化生态系统的研究中发现,植物种子萌发期加入丛枝菌根真菌与固氮根瘤菌不仅能协助植被在沙漠化区域的建立,同时还可以增加土壤肥力与质量。尽管沙漠化区域微生物的研究展示了一定的应用潜力,而相对于沙漠化区域的大面积分布,对沙漠化生态系统中微生物的研究还处于起步阶段[5-8]。
生物结皮通常是由各种光合及非光合生物组成的,包括蓝藻、地衣、绿藻、苔藓、真菌及一些微小原生动物。这些栖居在土壤表层的生物往往生活在极端恶劣的气候环境条件下。例如位于新疆准噶尔盆地腹地的古尔班通古特沙漠年平均降水量不超过150 mm,在沙漠腹地仅有70~100 mm,年均温为6~10℃,极端温度为40℃以上,均有结皮生物[9-10]。Evans和Ratledge[11]一致认为结皮生物群落在沙漠中对固定土壤和养分循环发挥着重要的作用。
目前,李芳芳等[12]用18S r RNA基因对古尔班通古特沙漠分离的绿藻进行了系统学研究,赵建成等[9]对该沙漠生物结皮进行了研究,结果表明,该沙漠中有很多常见的单细胞绿藻,例如小球藻、胶球藻、链带藻和衣藻等。Lewis和Lewis[13]的研究结果表明在结皮生物中的绿藻主要包含三大类群,绿藻纲、共球藻纲和轮藻纲。
塔克拉玛干沙漠是世界第二大流动沙漠,研究其生物结皮及微生物组成显得尤为重要,因为沙漠化严重威胁塔克拉玛干沙漠南缘和北缘的绿洲农业发展。围绕塔克拉玛干沙漠生态环境已经有很多学者进行了有关研究报道,但主要集中在沙危害分异规律、沙漠腹地人工绿地、沙漠自然植被光合特性、耗水量、根生态以及土壤种子库的研究等方面[2-7],而关于微生物的研究仅见关于其数量在结皮中的分布研究[13],特别是结合分子生物学方法对微生物群落结构特征的研究更为少见。张丙昌等[14]对古尔班通古特沙漠中绿藻的区系组成、生态分布特点和结皮不同发育阶段绿藻种类组成的动态变化进行了研究,李芳芳等[12]运用分子生物学18S r RNA基因对古尔班通古特沙漠分离的绿藻进行了系统学研究。本研究运用形态学和分子生物学相结合的方法对塔克拉玛干沙漠生物结皮中几种藻类进行研究,拟建立一个适合分离纯化和鉴定沙漠微藻的方法,为后续塔克拉玛干沙漠微藻的研究工作提供理论依据。
1 材料与方法
1.1 自然概况
中国新疆塔里木盆地中央的塔克拉玛干沙漠,气候属于典型的大陆性暖温带干旱荒漠气候,年平均气温9~11.2℃,最热月份8月的极端最高气温为43.2℃,最冷月份1月的极端最低气温为-19.3℃,年降水量60~80 mm,年内分配不均衡,多集中在春、夏两季,且年际变率大。沿着古尔班通古特沙漠公路附近采集沙样(N 37°36.732′~N 40°47.377′,E 80°23.442′~E 84°17.638′),多流动沙丘上基本无植物生长,偶见沙米(Agriophyllum squarrosum)及花棒(Hedysarum scoparium),盖度小于1%。
1.2 采样
采样时,用无藻铲采集沙样表层及其以下至10 cm深,收集于无菌密封袋。每次采样后用70%酒精擦拭采样工具,以防交叉污染。
本实验藻种均分离自塔克拉玛干沙漠采集沙样的混合培养藻液。表1列出了2011年11月19日从塔克拉玛干沙漠分离的6株微藻的沙样采集地点。
1.3 沙漠微藻的混合培养、分离与纯化
1.3.1 混合培养 称取5 g采集的沙样于灭菌的装有30 m L培养液的50 m L三角瓶中,充分混匀,置于光照培养箱,光照强度为2500 lx,光暗周期为12 h/12 h,温度为23℃的条件下培养1~2周,随时观察杂菌污染情况,如杂菌过多,则终止培养。
1.3.2 分离与纯化 采用平板法和96孔板有限稀释法分离纯化上述混合培养的藻液。分离过程中定期镜检培养藻液,直到1000倍镜下观察的微藻形态一致即纯化成功,然后将已纯化成功的微藻进行逐级扩大培养。

表1 塔克拉玛干沙漠采样地的分布概况Table 1 Sampling locations of algae strains used in present study in the Taklimakan desert
1.4 分离纯化微藻的分子鉴定
1.4.1 DNA提取 取对数生长期OD680的藻液400~600 m L,3000 r/min离心8 min,收集沉淀,用液氮研磨至粉末置于1.5 m L离心管中;用TIANGEN植物基因组DNA提取试剂盒提取微藻基因组DNA。
1.4.2 序列扩增及测序 用于扩增绿藻的5.8S-ITS的引物序列为:上游5′-TTCTTAGTTGGTGGGTTGCCT-3′,下游5′-TTTCATCTTTCCCTCACGGTA-3′[15]。
用于扩增真核绿藻的18S r DNA的引物序列为:上游5′-ACCTGGTTGATCCTGCCAGTAG-3′,下游5′-ACCTTGTTACGACTTCTCCTTCCTCC-3′[16]。
用于扩增16S r DNA的原核生物通用引物序列为:上游5′-AGAGTTTGATCCTGGCTCAG-3′,下游5′-AAGGAGGTGATCCAGCC-3′[17]。
PCR反应体系为20μL,dd H2O:12.4μL,Buffer:2.0μL,MgCl2:1.2μL,d NTP:1.2μL,引物1:1.0μL,引物2:1.0μL,DNA模板:1.0μL,Taq酶:0.2μL。PCR反应体系扩增条件:94℃预变性5 min;94℃变性1 min,60℃退火30 s,72℃延伸90 s,30个循环;72℃延伸5 min,扩增结果用1%的琼脂糖进行电泳检测,凝胶成像系统记录实验结果。将PCR产物用TIANGEN普通琼脂糖凝胶DNA回收试剂盒回收后再用p MD18-T载体连接试剂盒(Ta KaRa)进行连接,转入大肠杆菌DH5a,由北京六合华大基因科技股份有限公司进行测序。
1.5 系统发育分析
将测得序列提交至GeneBank,利用Blast搜索引擎中的nucleotide blast获得所测序列的同源序列,用Gene-Doc软件进行多序列对位分析,MEGA 4.1软件进行序列分析,并采用邻接法 (NJ)建树,Kimu-ra 2-parameter计算遗传距离值,重复1000次计算bootstrap值。
2 结果与分析
2.1 5株藻种的形态学特征
从塔克拉玛干沙漠6个采样地分离得到6株微藻。在光学显微镜100倍油镜下观察其细胞形态。其特征为TLD2A1单细胞球形,色素体杯状,占细胞的一半或稍多,细胞直径3~6μm。TLD2B细胞呈椭圆或卵形,两端略为变尖,眼点位于细胞中部,细胞宽3~5μm,长7~9μm。TLD5A1植物体呈中空而薄的裂片状,生长后期漂浮,呈褐色或深褐色,藻丝呈强烈卷曲或有时平行排列,细胞呈球形或近球形。TLD6B单细胞或几个细胞聚在一起,呈球形,细胞壁薄,细胞直径5~10μm。TLD7A-2游动的单细胞,细胞呈椭圆形,细胞宽8~12μm,长10~13μm。TLD7B-5单细胞,形态多呈月牙状,细胞长3~9μm,宽2~4μm,两顶端直线距离3~7μm。
根据其形态特征参照文献[18]进行检索分类,经鉴定表明TLD2A1、TLD6B的初步分类地位为绿藻门、绿藻纲、绿球藻目;TLD2B、TLD7A-2初步分类为绿藻门,绿藻纲,团藻目;TLD7B-5初步分类为绿藻门、绿藻纲;TLD5A-1初步分类为蓝藻门(经石河子大学高剑峰教授加以鉴定)。
2.2 序列分析
2.2.1 5株绿藻的序列分析 依据形态观察TLD2A1、TLD2B、TLD6B、TLD7A-2和TLD7B-5均为绿藻门,用18S r DNA保守区引物分别扩增了5株微藻的18S r DNA序列,其长度分别为1767,1759,1766,1764和2657 bp;TLD2A1、TLD7A-2、TLD7B-5三株藻5.8S r DNA-ITS的扩增产物长度分别为1539,1437,2428 bp,与NCBI已知的数据库进行比较,根据比对同源性相近藻的18S r DNA全序列及普通小球藻(Chlorella vulgaris)(AB237642)r DNA序列,确定5.8S r DNA-ITS(包括ITS1、ITS2和5.8S r DNA区域)序列长度分别为650,564和672 bp。数据提交至GenBank。其中TLD7B-5的18S核糖体r DNA测序后发现其长度比另外2株绿藻长很多,将其在Blast比对后发现有2个内含子,去除内含子后与各序列于NCBI Blastn比对并进行核酸数据库同源性分析(表2)。TLD2A1、TLD2B、TLD6B、TLD7A-2、TLD7B-5的18S r DNA序列G+C含量分别为49.7%,49.5%,49.8%,48.0%和48.2%。

表2 6株沙漠微藻核酸序列与NCBI核酸数据库同源性比对分析Table 2 Blast of the nucleotide sequence of 6 desert algae strains
2.2.2 16S r DNA序列分析 依据形态观察TLD5A1为蓝藻门,用16S r DNA引物序列扩增了TLD5A1的16S r DNA序列,长度为1481 bp。数据提交至GenBank,将测得序列与GenBank中7个序列及外类群集胞藻的16S r RNA基因序列进行排列对齐,排序后共有1443 bp位点,比较了TLD5A1的16S rDNA和GenBank中7个序列的碱基组成发现,TLD5A1的GC含量为54.7%,其他7个序列的G+C含量均在53.9%~55.0%之间。其序列于NCBI Blastn比对并进行核酸数据库同源性分析(表2)。
2.3 系统发育分析
2.3.1 绿藻系统发育分析 将分离藻株中5株绿藻的18S r DNA和其中3株的5.8S r DNA-ITS序列于Blast同源比对分析,下载与其同源性相对较高的序列5~9个,全部输入GeneDoc软件,比对后将序列开始和结尾长短不一的片段删减整齐,比对结果输入到MEGA 4.1软件中,选取的核苷酸置换模型为Kimura双参数模型(Kimura 2-parameter),自展支持分析重复1000次。得到的18S r DNA和5.8S r DNA的NJ树(图1和图2)。

图1 基于18S r DNA基因序列的邻接树Fig.1 NJ tree based on sequences of 18S rDNA gene节点处数字代表1000次重复自展分析所得到支持率(%)The numbers at the nodes represent bootstrap support of 1000 replicates(%).下同The same below.
5株绿藻的18S r DNA系统发育树分析表明,实验所分离的5株绿藻可以分别在3个大分支内,其中1个类群可归于小球藻科,以TLD2A1、TLD6B为代表,它们的18S r DNA序列与普通小球藻藻株的相似性最高,为99.89%。TLD2B和TLD7A-2分别与衣藻科的9个物种聚为一大分支,其中TLD2B与咸胞藻(Brachiomonas sp.)聚为一支相似性为98.16%,自展支持率为91%,TLD7A-2与Chlamydomonas sp聚为一支相似性为97.65%,自展支持率为72%,TLD7B-5的18S r DNA 与绿藻门,绿藻纲环藻目的Selenastraceae、栅藻科(Scenedesmaceae)、环藻目Sphaeropleales三科物种聚为一支,其中与绿球藻(Tetranephris brasiliensis)同源性最高为98.86%。
TLD2A1、TLD7A-2、TLD7B-5三株绿藻的5.8S r DNA-ITS系统发育分析,结合他们的18S r DNA数据表明,TLD2A1与Chlorella sp.为同一属,TLD7A-2的5.8S r DNA-ITS与莱茵衣藻(Lobochlamys segnis)、衣藻(Chlamydomonas inflexa)、南极衣藻(Chlamy domonas sp.)聚为一支,与南极衣藻相似性最高为72.94%。TLD7B-5的5.8S r DNA-ITS与环藻目科(Selenastraceae)的月牙藻(Pseudokirchneriella sp.)聚为一支,相似性最高,为87.31%,自展支持率低于50%,这与其18S r DNA数据有些差异。

图2 基于5.8S r DNA-ITS基因序列的邻接树Fig.2 NJ tree based on sequences of 5.8S rDNA-ITSgene
2.3.2 1株蓝藻系统发育分析 将TLD5A1的16S r DNA序列于Blast同源比对分析,下载与其同源性相对较高的序列7个,全部输入GeneDoc软件,比对后将序列开始和结尾长短不一的片段删减整齐,比对结果输入到MEGA 4.1软件中,选取的核苷酸置换模型为Kimura双参数模型(Kimura 2-parameter),自展支持分析重复1000次,得到的16S r DNA的NJ树见图3。
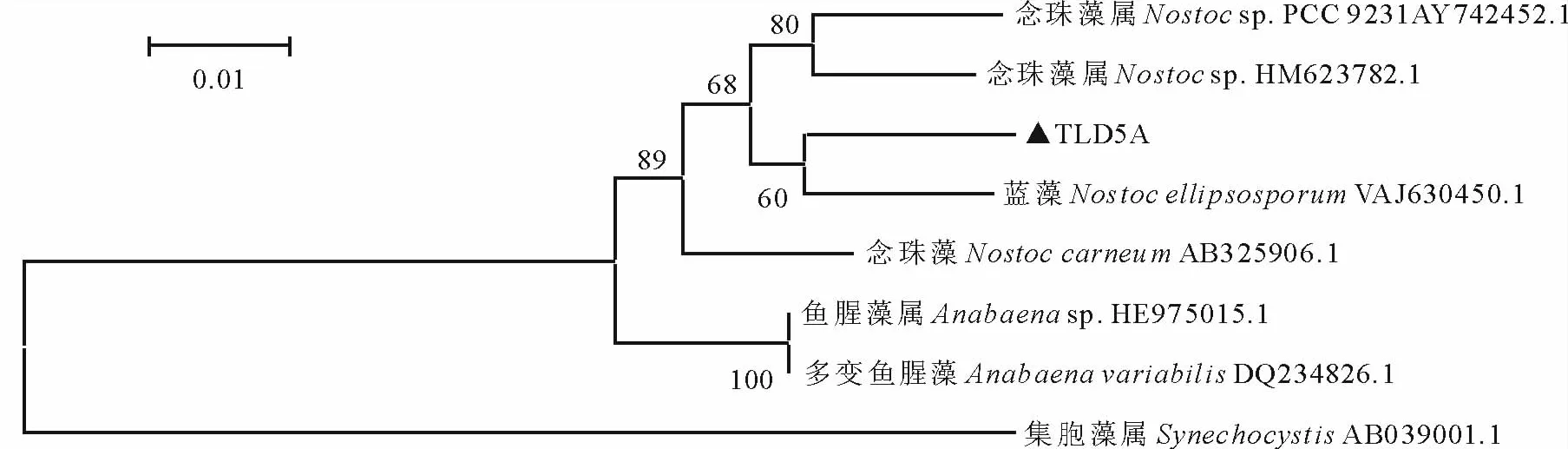
图3 基于16S r DNA-ITS基因序列的邻接树Fig.3 NJ tree based on sequences of 16S r DNA-ITSgene
它的16S r DNA序列与念珠藻科的物种聚为一支,其中与念珠藻属的蓝藻(Nostoc ellipsosporum)相似性最高,为97%,有37个碱基不同,NJ树也与其聚为一支,自展支持率为60%。
3 讨论
3.1 鉴定
塔克拉玛干沙漠的藻类研究相关报道较少见,目前国内对藻类结皮的研究,主要为形态学鉴定,这种方法费时并且需要鉴定者有丰富的分类经验,很多形态学特征会随着生长环境和生长阶段的改变而变化。随着分子生物学技术的发展,5.8S r DNA、18S r DNA、16S r DNA等序列分析方法在物种鉴定中已经普遍使用[15-17]。本研究先采用传统的形态鉴定,再结合现代分子生物学方法对分离藻株进行分析,这样较大程度地保证了结果的准确性。本研究从塔克拉玛干沙漠通过形态学和分子学鉴定分离得到了5株绿藻,其中3株隶属于绿藻纲,2株为共球藻纲。在沙漠中的绿藻主要包含三大类群,绿藻纲、共球藻纲和轮藻纲[19-20],因此推断这些沙漠绿藻均起源于淡水藻,当然这有待更多实验研究的验证。
3.2 结论
依据文献报道,97%~99%全序列相似的可判定为一个属。形态学和18S r DNA数据结合分析表明,实验分离的5株绿藻均鉴定到科,分别为衣藻科(TLD2B,TLD7A-2)、小球藻科(TLD2A1、TLD6B)、环藻科(TLD7B-5),5.8S rDNA-ITS序列进化速度快,变异丰富,适合种内鉴定,TLD2A1的5.8S r DNA与18S r DNA数据结果一致,与小球藻亲缘关系最近,TLD7B-5的5.8S r DNA-ITS与18S结果有一定差异,可能是因为GenBank中物种序列不全所造成的。
5株绿藻的18S r DNA序列GC含量为48.0%~49.8%,包含在与它们同源的30个类群GC含量之间(48.0%~50.0%),差异为1.8%。同时对3株绿藻的5.8S r DNA-ITS序列的碱基构成进行分析,TLD2A1同其小球藻属的物种比对发现GC含量均在57.1%~58.4%之间,差异不足1.4%。TLD7A-2的GC含量为44.2%,远小于其他4个相近物种的GC含量(50.2%~52.0%),可能和TLD7B-5类似,由于数据库中物种序列不完全所导致的。发现18S r DNA序列GC含量没有明显差异,而5.8S r DNA-ITS序列不同科属之间的GC含量差异明显,并且同一科属其GC含量也可能有所不同(如TLD7A-2)。TLD5A1的GC含量为54.7%,Blast比对后与其同源性较高的其他7个序列的GC含量均在53.9%~55.0%之间,这与Li和Watanabe[21]检测了50株纯培养的鱼腥藻的GC含量,并比较了依据GC含量分类与应用形态学、生理和生化分类的结果,得出相同物种的GC含量是相近的,但是具有相同GC含量的物种却不一定有较近的亲缘关系结果一致。
