两种提取枝角类休眠卵基因组DNA方法的比较*
2014-09-25李思嘉韩小玉韩博平
徐 磊,李思嘉,2,王 晟,韩小玉,韩博平**
(1:暨南大学生态学系,广州510632)
(2:珠江水资源保护科学研究所,广州510611)(3:浙江省发展规划研究院,杭州310012)
休眠是枝角类生活史的重要阶段,枝角类可以通过有性生殖产生休眠卵,使种群进入休眠阶段来应对水体环境的恶化,直到水体环境再度适合生存,休眠阶段打破,种群得以补充和恢复[1-2].类似于高等植物的土壤种子库,大量由卵鞍包被的休眠卵周期性地沉入水体底部,形成一个具有世代周期重叠的休眠卵库.在沉积物保存良好的水体中(深水水体),卵库中枝角类卵鞍种类组成或丰度的变化反映了群落结构的历史动态,可以用来了解水体生态与人类影响的长期变化[3-4].休眠卵的孵化为种群延缓提供种源的同时,不同时期的休眠卵所保存的遗传特征在很大程度上影响着现生种群的遗传多样性.通过对休眠卵遗传信息的研究,可以在时间尺度上了解种群遗传结构的变化,重建种群动态的历史变化过程和水体生态演变,探讨水体环境变化与枝角类适应性.这种基于底泥中的休眠卵库(egg bank)对生态系统中发生的生态和进化过程的分析成为长期生态学和进化生态学研究的重要方法,Kerfoot等[5]称之为重构生态学(reconstruction ecology).
溞属(Daphnia)是淡水枝角类的主要类群,相对其它类群,其个体较大,繁殖速度快,有很强的滤食能力,在生态系统中发挥着极其重要的功能.溞属有性生殖形成的休眠卵被由几丁质外壳构成的卵鞍所包被,可以在水体沉积物中保存较长时间仍然具有萌发活性,这使构建溞属的历史种群遗传信息成为可能.Carvalho和Wolf[6-7]是最早通过孵化沉积物中的休眠卵研究水体浮游动物遗传多样性的学者.Weider等[8]通过等位酶技术研究Constance湖中溞属休眠卵基因频率的变化,发现等位酶PGI的基因频率与水体富营养化有关.然而,他们的研究都基于将休眠卵孵化,培育出大量的克隆体,等位酶技术对实验材料严格的要求使得研究难以深入.因此,直接从休眠卵中获得遗传信息显得尤为重要,目前,国际上许多学者已经通过获取卵库中的遗传信息,发现溞属的遗传结构与富营养化[9-10]、有毒蓝藻水华[11-12]、渔业养殖[13]以及宿主寄生[14]有关.我国也有关于枝角类休眠卵的研究,但主要是有关休眠卵萌发和外形及分布特征的研究[15-17],受休眠卵基因组DNA提取较为困难的限制,尚无对枝角类休眠卵遗传信息的研究结果方面的报道.
目前国际上对于浮游动物DNA的提取主要采用HotSHOT法和相关改良方法[18-19],但此类方法为了避免DNA在抽提过程中的损失,不涉及DNA纯化过程,因此残留的提取液以及动物残体势必会给后期的实验研究带来干扰.本文将尝试使用氯仿异戊醇和玻璃乳法提取单个盔型溞(Daphnia galeata)休眠卵基因组的DNA,并对这2种方法进行比较研究,包括提取效率和随后的PCR扩增检测以及两种方法的准确性验证.
1 材料与方法
1.1 枝角类休眠卵的采集
盔型溞是我国淡水生态系统中广泛分布的大型枝角类.本研究使用的盔型溞休眠卵采集于广东省流溪河水库,该水库(23°45'N,113°46'E)是一座典型的贫中营养的深水水库,正常水位时,水面面积12.25 km2,库容3.25 ×108m3,平均水深26.53 m,最大水深73 m.
2010年7月,使用Uwitec柱状采泥器(奥地利)在位于水库敞水区的中间位置分别采集4~5柱保存较好的沉积物.采集到的沉积物分别切取表层0~10 cm部分以及底层30~40 cm部分(根据水库沉积速率和休眠卵库分布以及孵化的特征,沉积物表层10 cm约为近10年内积累的休眠卵,而沉积物底层30~40 cm代表水库建成后10~20年内积累的休眠卵[4,20-21]),为避免由于采泥器PVC柱子滑动造成不同泥层间的沉积物交叉污染,泥层外围的沉积物切割后需要舍弃[22-23].分层切割后的样品分别用孔径为600、200和35 μm的网筛依次过滤筛选,每个网筛过滤后残留的液体转移至100 ml塑料小白瓶中,保存于4℃冰箱中.分离出含有休眠卵的残余液体在20~50倍放大的解剖镜(Olympus SZX71)和显微镜(Olympus CX21)下进行观察,挑取含有盔型溞休眠卵的卵鞍,并转移至无水乙醇中-20℃保存.
1.2 氯仿异戊醇提取法
将单个卵鞍从无水乙醇中取出,用双蒸水反复清洗3次以上,在解剖镜下将休眠卵从卵鞍中剥离,然后转移至离心管中,加人200 μl体积分数为10%的十二烷基硫酸钠(SDS)消化液和 3 μl蛋白酶 K(20 mg/ml),55℃水浴2 h后加入等体积的氯仿∶异戊醇(24∶1),12000转/min离心10 min,取上清液,加入0.6倍至等体积异丙醇,12000转/min冷冻离心5 min沉淀DNA,用体积分数为70%的乙醇清洗,干燥之后加入30 μl TE缓冲液溶解DNA,-20℃长期保存.
1.3 玻璃乳提取法
休眠卵基因组DNA提取使用Ultra-Sep Gel Extraction Kit凝胶回收试剂盒(美国Omega公司)中的几种试剂,并对标准步骤进行调整,具体方法如下:将单个卵鞍从无水乙醇中取出,用双蒸水反复清洗3次以上,在解剖镜下将休眠卵从卵鞍中剥离,然后转移至离心管中,加人200 μl体积分数为10%的SDS和3 μl蛋白酶K(20 mg/ml),55℃水浴2 h后加入4 μl的玻璃乳和300 μl Binding buffer,12000转/min离心3 min后弃上清液,加入300 μl Washing buffer与沉淀混合,12000转/min离心3 min后弃上清液保留沉淀,放置室温下晾干后,加入30 μl Elution buffer混匀,12000转/min离心2 min,吸取上清液转至新管,-20℃长期保存.
1.4 DNA浓度检测与PCR扩增检测
分别使用两种方法提取表层和底层休眠卵基因组DNA各20个,通过核酸分析仪(Eppendorf Biophotometer Plus)检测浓度和纯度,统计提取效率.为进一步检测DNA提取的成功率,使用线粒体细胞色素氧化酶I(COI)基因引物进行 PCR(BIO-RAD C1000 Thermal Cycler)扩增.COI引物为 LCO1490(GGTCAACAAATCATAAAGATATTGG)和 HCO2198(TAAACTTCAGGGTGACCAAAAAATCA)[24].扩增反应总体积为 30 μl,包括:3 μl 10 × buffer、1.5 mmol/L MgCl2、0.2 mmol/L dNTPs、引物各0.5 μmol/L、0.5 U Taq 酶(TaKaRa)和3 μl模板DNA.扩增程序为94℃预变性4 min,94℃变性1 min,50℃退火1.5 min,72℃延伸1 min,重复40次循环72℃延伸10 min,4℃结束反应.PCR完成后,取2 μl产物用于1%的琼脂糖凝胶检测,并在凝胶成像系统(BIO-RAD Universal Hood II)中观察记录结果.
2 结果

图1 流溪河水库沉积物中盔型溞含休眠卵的卵鞍形态Fig.1 Morphology of ephippium of Daphnia galeata isolated from sediments of Liuxihe Reservoir
从沉积物中获得的盔型溞休眠卵如图1所示,共对40个表层休眠卵进行DNA提取,氯仿异戊醇提取法成功提取8个,玻璃乳提取法成功提取15个,而40个底层休眠卵DNA提取中,氯仿异戊醇提取法仅成功提取3个,玻璃乳提取法成功提取11个.通过核酸分析仪检测浓度和纯度,玻璃乳提取法提取到的休眠卵基因组DNA平均浓度为28.37 ±2.56 ng/μl,要极显著高于氯仿异戊醇提取法(P<0.001,图2a).玻璃乳提取法提取 DNA的A260/A280值范围为 1.67 ~2.02,平均值为 1.85;而氯仿异戊醇提取法提取 DNA的 A260/A280值相对较低,介于1.18 ~1.83之间,平均值为 1.46(图2b).COI扩增产物凝胶电泳结果显示,玻璃乳法提取休眠卵基因组DNA的PCR扩增产物浓度也明显高于氯仿异戊醇法(图3).
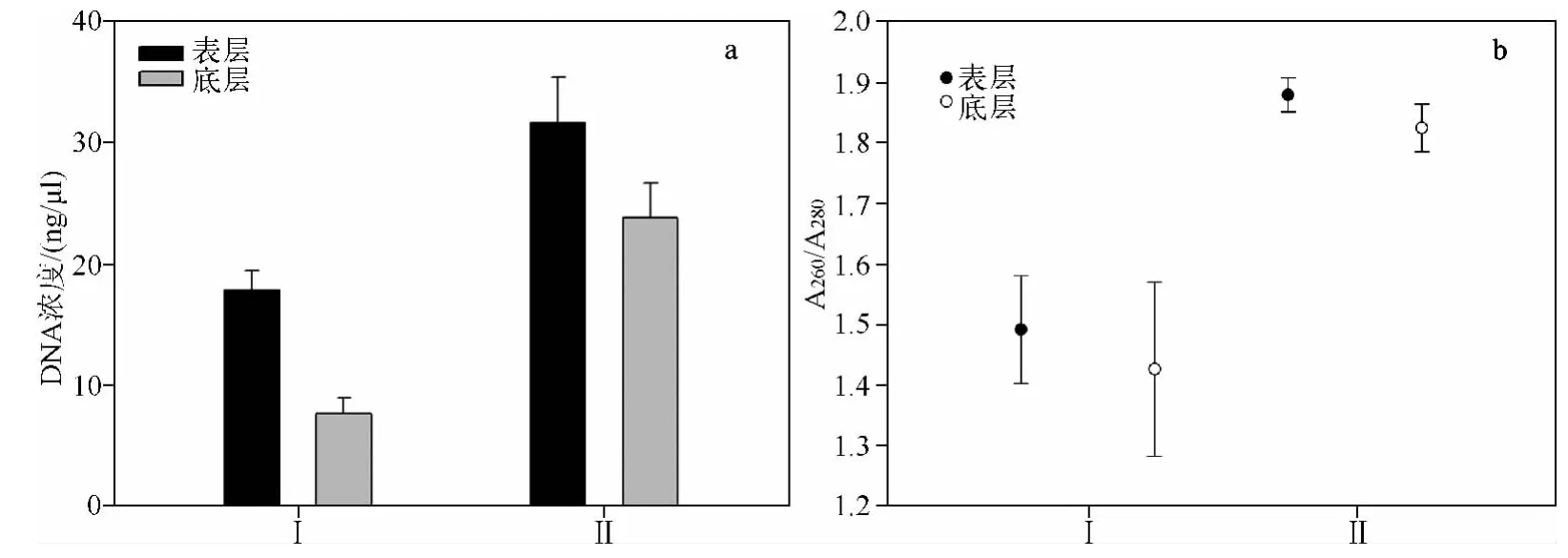
图2 2种方法提取盔型溞休眠卵基因组DNA浓度(a)和纯度(b)比较(Ⅰ代表氯仿异戊醇提取法;Ⅱ代表玻璃乳提取法)Fig.2 Comparison of DNA concentrations(a)and DNA purities(b)of resting eggs by Daphnia galeata using two methods(Ⅰ.Chloroform isoamyl alcohol method;Ⅱ.Ultra-Sep Beads method)
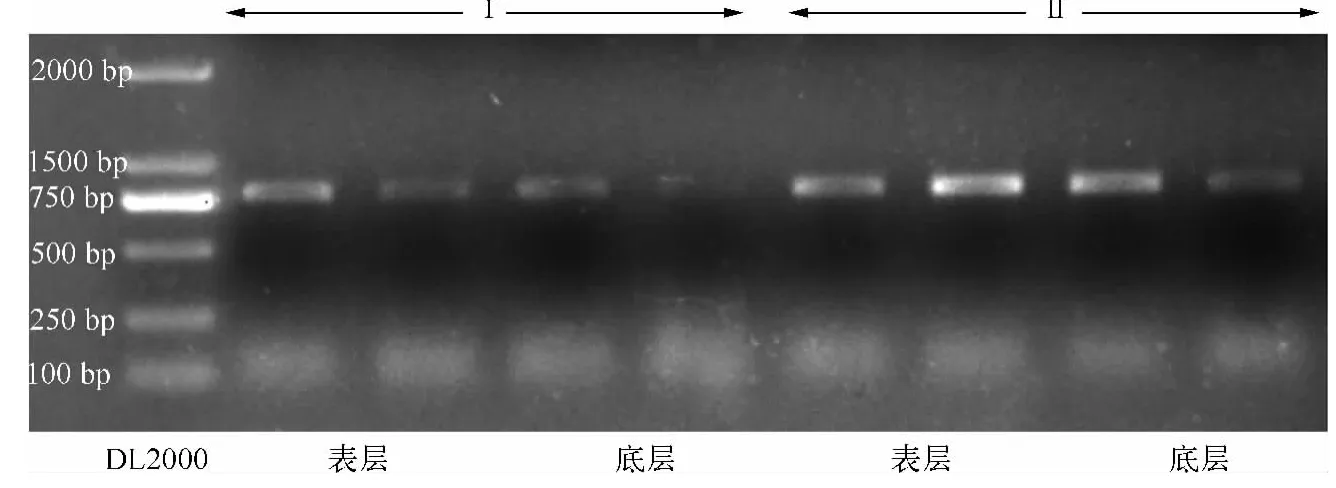
图3 2种方法提取盔型溞休眠卵基因组DNA的COI基因PCR扩增电泳图(Ⅰ代表氯仿异戊醇提取法;Ⅱ代表玻璃乳提取法)Fig.3 PCR products of DNA extractions of resting eggs by Daphnia galeata using two methods(Ⅰ.Chloroform isoamyl alcohol method;Ⅱ.Ultra-Sep Beads method)
3 讨论
溞属作为淡水生态系统的关键种类,其保存在沉积物中休眠卵的DNA信息常被用来研究生态环境变化与遗传结构变化的关系[9-14].因此,获得高质量的DNA是此类研究的基础.通过对比可以看出,2种方法都可用于溞属休眠卵基因组DNA的提取(氯仿异戊醇提取法的成功率为27.5%,玻璃乳提取法的成功率为65.0%),但是从DNA的提取成功率上看,玻璃乳提取法更适合休眠卵基因组DNA的提取,特别是针对底层沉积物保存时间较长的休眠卵,玻璃乳提取法的成功率更高(55.0%),而氯仿异戊醇提取法对底层沉积物中休眠卵DNA提取的成功率仅为15.0%.核酸分析仪检测结果显示,玻璃乳提取法提取的DNA浓度也较氯仿异戊醇法高,对于表层沉积物中的休眠卵,玻璃乳提取法所获得的DNA平均浓度为氯仿异戊醇法的2倍,然而对于在沉积物中保存时间相对较长的底层沉积物休眠卵,玻璃乳提取法所获得的DNA平均浓度却接近氯仿异戊醇法的3倍(图2a).从2种方法的提取过程上来看,氯仿异戊醇法提取步骤繁琐,所用试剂较多,特别是需要氯仿等有毒试剂,增加了对实验操作人员危害的可能.而玻璃乳提取法所用试剂少,步骤简单,整个提取过程所用时间短,吸附在玻璃乳上的DNA可以通过试剂盒中所含的Elution buffer快速洗脱下来,保证DNA的完整性和纯度,可见,玻璃乳提取法提取枝角类休眠卵基因组DNA是一种切实可行的方法.
尽管2种方法都能提取底层沉积物休眠卵的DNA,但提取成功率明显低于表层沉积物休眠卵的DNA提取成功率(氯仿异戊醇法对底层沉积物休眠卵的DNA提取成功率为15.0%,表层沉积物为40.0%;玻璃乳提取法对底层沉积物休眠卵的DNA提取成功率为55.0%,表层沉积物为75.0%),这一差异与休眠卵在沉积物中保存的状态和时间有关.位于沉积物表层2~10 cm中的休眠卵相对于沉积物底层休眠卵,形成时间较晚,容易获得环境条件的直接诱导孵化进入现生种群[4,20,25].因此具有较高的活性,更易从中获得质量较高的DNA.而分布于沉积物底层中的休眠卵为水库新建初期产生的,保存时间较长,含有完整休眠卵的卵鞍数较少,休眠卵萌发活性较低,DNA提取的质量和成功率相对较低,势必会对下游PCR扩增反应产生影响[26].因此,从底层沉积物中尽可能地分离保存较好的休眠卵将直接影响DNA提取的成功率以及后续研究.
[1]Alekseev VR,De Stasio B,Gilbert JJ et al.Diapause in aquatic invertebrates:Theory and human use.Dordrecht:Springer,2007:257.
[2]Cáceres CE.Interspecific variation in the abundance,production,and emergence of Daphnia diapausing eggs.Ecology,1998,79:1699-1710.
[3]Hairston NG,Cáceres CE.Distribution of Crustacean diapause:Micro-and macroevolutionary patterns and processes.Hydrobiologia,1996,320:27-44.
[4]Brendonck L,De Meester L.Egg banks in freshwater zooplankton:evolutionary and ecological archives in the sediment.Hydrobiologia,2003,491:65-84.
[5]Kerfoot WC,Robbins JA,Weider LJ.A new approach to historical reconstruction:combining descriptive and experimental paleolimnology.Limnology and Oceanography,1999,44:1232-1247.
[6]Carvalho GR,Wolf HG.Resting eggs of lake Daphnia I.Distribution,abundance and hatching of eggs collected from various depths in lake sediments.Freshwater Biology,1989,22:459-470.
[7]Wolf HG,Carvalho GR.Resting eggs of lake Daphnia II.In situ observations on the hatching of eggs and their contribution to population and community structure.Freshwater Biology,1989,22:471-478.
[8]Weider LJ,Lampert W,Wessels M et al.Long-term shifts in a microcrustacean egg bank associated with anthropogenic changes in the Lake Constance ecosystem.Proceedings of the Royal Society B:Biological Sciences,1997,264:1613-1618.
[9]Brede N,Sandrock C,Straile D et al.The impact of man-made ecological changes on the genetic architecture of Daphnia species.Proceedings of the National Academy of Sciences of the United States of America,2009,106:4758-4763.
[10]Rellstab C,Keller B,Girardclos S et al.Anthropogenic eutrophication shapes the past and present taxonomic composition of hybridizing Daphnia in unproductive lakes.Limnology and Oceanography,2011,56(1):292-302.
[11]Hairston Jr NG,Perry LJ,Bohonak AJ et al.Population biology of a failed invasion:paleolimnology of Daphnia exilis in upstate New York.Limnology and Oceanography,1999,44:477-486.
[12]Hairston Jr NG,Holtmeier CL,Lampert W et al.Natural selection for grazer resistance to toxic cyanobacteria:Evolution of phaenotypic plasticity?Evolution,2001,55:2203-2214.
[13]Cousyn C,De Meester L,Colbourne JK et al.Rapid,local adaptation of zooplankton behavior to changes in predation pressure in predation pressure in the absence of neutral genetic changes.Proceedings of the National Academy of Sciences of the United States of America,2001,98(11):6256-6260.
[14]Decaestecker E,Gaba S,Raeymaekers JAM et al.Host-parasite‘Red Queen’dynamics archived in pond sediment.Nature,2007,450:870-874.
[15]陆开宏.几种枝角类的卵鞍形态及表面超微结构.动物学报,2001,47(5):565-572.
[16]韩小玉,徐 磊,韩博平等.广东流溪河水库表层沉积物中枝角类休眠卵的多样性及空间分布.生态科学,2011,30(3):272-278.
[17]孟 琼,邓道贵.枝角类休眠卵形成和萌发的研究进展.动物学杂志,2008,43(3):154-160.
[18]Montero-Pau J,Gomez A,Munoz J.Application of an inexpensive and high-throughput genomic DNA extraction method for the molecular ecology of zooplanktonic diapausing eggs.Limnology and Oceanography:Methods,2008,6:218-222.
[19]Ishida S,Otsuki H,Awano T et al.DNA extraction and amplification methods for ephippial cases of Daphnia resting eggs in lake sediments:a novel approach for reconstructing zooplankton population structure from the past.Limnology,2012,13:261-267.
[20]Gyllström M,Hansson LA.Dormancy in freshwater zooplankton:Induction,termination and the importance of benthic-pelagic coupling.Aquatic Sciences,2004,66:274-295.
[21]Shotbolt LA,Thomas AD,Hutchinson SM.The use of reservoir sediments as environmental archives of catchment inputs and atmospheric pollution.Progress in Physical Geography,2005,29:337-361.
[22]Hairston Jr NG,Van Brunt RA,Kearns CM et al.Age and survivorship of diapausing eggs in a sediment egg bank.Ecology,1995,76(6):1706-1711.
[23]Limburg PA,Weider LJ.‘Ancient’DNA in the resting egg bank of a microcrustacean can serve as a palaeolimnological database.Proceedings of the Royal Society B:Biological Sciences,2002,269:281-287.
[24]Folmer O,Black M,Hoeh W et al.DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates.Molecular Marine Biology and Biotechnology,1994,3:294-299.
[25]Hairston Jr NG,Hansen AM,Schaffner WR.The effect of diapause emergence on the seasonal dynamics of a zooplankton assemblage.Freshwater Biology,2000,45:133-145.
[26]Xin Z,Velten JP,Oliver MJ et al.High-throughput DNA extraction method suitable for PCR.Biotechniques,2003,34:820-825.
