白蛋白和石墨烯的结构及其相互作用的分子动力学模拟
2014-09-17丁佳丽王健丹吕晓迎
孙 颖 丁佳丽 宗 皓 王健丹 吕晓迎
(1东南大学生物电子学国家重点实验室,南京 210096)(2东南大学江苏省生物材料与器件重点实验室,南京 210009)(3东南大学软件学院,南京 211189)
白蛋白和石墨烯的结构及其相互作用的分子动力学模拟
孙 颖1,2丁佳丽1,2宗 皓3王健丹1吕晓迎1
(1东南大学生物电子学国家重点实验室,南京 210096)
(2东南大学江苏省生物材料与器件重点实验室,南京 210009)
(3东南大学软件学院,南京 211189)
摘 要:采用分子动力学模拟的方法分析研究了白蛋白与石墨烯表面的相互作用.分子模拟过程采用Gromacs软件进行运算及数据采集分析,并采用VMD软件实现结构的可视化.首先,建立周期边界条件,在边长为9 nm的有限空间中采用恒温恒压系综,对ffgmx力场条件下的白蛋白原始真空构象进行优化,使其在水相中达到平衡态.然后,以水相中得到的稳定的白蛋白作为模型,构建与石墨烯相互作用的总体构象及界面.最后,在周期性边界条件下,经过500 ps的动态过程,完成了白蛋白在石墨烯表面的吸附.对能量、残基轨迹、扭转角等数据进行分析总结,确定了白蛋白在石墨烯表面的吸附位点为白蛋白序列中90~97号肽段90ALA-91GLY-92ILE-93THR-94SER-95ASP-96PHE-97TYR.
关键词:分子动力学模拟;白蛋白;石墨烯;相互作用机制
生物相容性是生物材料研究中的关键科学问题.蛋白质与材料的相互作用是生物材料植入机体后首先发生的重要现象[1],针对其作用规律和机制的研究是生物材料生物相容性研究的重要内容之一.多年来,学者们采用了多种方法和手段(如表面等离激元共振生物传感器法、白光反射干涉生物传感器法、同位素放射性示踪法等)对蛋白质吸附量、蛋白质的构象、蛋白质竞争吸附等进行了研究[2].
鉴于蛋白质结构与功能之间的密切关系,引入固体界面后,其在界面上的吸附过程比普通分子体系复杂得多[3].普通的实验手段无法对蛋白质与材料表面之间相互作用的机理进行深入的研究.如何获得蛋白质的结构信息并从分子水平上研究蛋白质在吸附过程中结构特性的变化,具有重要的理论和应用价值.
分子动力学(MD)模拟是指通过计算分子在一定条件下的运动情况,来模拟分子在真实条件下的状态.它是一种在原子、分子水平上求解多体问题的重要计算机模拟方法,可以预测纳米尺度上的材料动力学特性,为复杂的蛋白质在生物材料表面吸附的理论和实验研究提供有益的补充[4].Kandori等[5]采用分子动力学模拟的方法,研究了牛血清蛋白在表面修饰有羟基磷灰石颗粒的焦磷酸表面的吸附,此吸附是由于两者间羟基等官能团之间产生的氢键相互作用而形成的.
白蛋白是血浆中浓度最高的小分子球状蛋白质,具有维持血管和组织之间的血液胶体渗透压、结合并参与多种小分子物质的运输、代谢产物解毒和再加工、抗氧化及清除自由基等多种重要的生理作用.特别地,白蛋白具有抑制血小板聚集和抗血栓形成等功能,其与生物材料的相互作用具有重要的意义,因此是生物材料血液相容性研究中应用最为广泛的蛋白质之一[1-2,6-7].
随着医学、材料科学和生物技术的发展,关于石墨烯表面与生物大分子吸附行为的研究引起了学者们的广泛关注.作为一种新型二维纳米材料,石墨烯及其衍生物在生物检测、生物成像、药物载体及其肿瘤治疗等方面有着潜在的应用潜力,是一种很有前景的生物医学新材料[8-9].
本文采用分子动力学模拟的方法,对白蛋白在水相中的结构及石墨烯界面上的吸附进行了模拟.于纳秒及皮秒时限内,在原子水平上对白蛋白在石墨烯表面的吸附过程进行分析.基于对能量、残基轨迹、扭转角等数据的分析结果,确定了白蛋白在石墨烯表面的吸附距离和吸附位点,有利于在原子水平上理解白蛋白与石墨烯的作用机理.
1 分子动力学模拟
针对分子系统的分子动力学模拟的具体步骤如下[10]:① 设定模拟所采用的模型;② 给定初始条件;③确定平衡计算过程;④ 计算宏观物理量.进行分子动力学模拟的前提是必须包含结构文件和力场文件,二者缺一不可.结构文件必须是所选软件支持的格式,力场文件则是告诉整个体系里面的每个原子如何运动的文件.在初始数据文件的基础上,将材料和界面有机整合成一个整体后,在力场文件的作用下,使体系运行并完成一个完整的模拟过程.
1.1 模型建立
Material Studio软件中,在单层空间上延拓碳原子的六圆环结构,得到石墨烯的原始文件,通过格式转换得到可用的原始结构图,即为石墨烯的结构图.白蛋白的结构文件来自蛋白质数据库[11].力场是由Gromacs软件中的现有力场ffgmx[12]加入不同键角的碳原子,而后补充白蛋白中含有的原子而得到的.
分子模拟过程采用Gromacs软件进行运算及数据采集分析,并采用VMD软件实现结构的可视化.
1.2 模拟过程
1.2.1 白蛋白在水相中的预平衡
由于从蛋白质数据库中下载的是人血清白蛋白在真空状态下的X射线衍射数据模型,而不是蛋白质在溶液中的状态,因此需要进行预模拟,使白蛋白在水相(即模拟简单的体液状态)中达到一个较好的平衡状态.本文中,将下载得到的白蛋白在真空状态下的X射线衍射数据模型,经过加氢修饰等处理后,放置在水体系中进行1 ns的分子动力学模拟,以便对蛋白质的构型进行优化.其中,水分子采用传统水分子力场模型(SPC模型).
模拟过程中,采取了空间受限模拟.在盒子里装入水溶剂以代替体液,采用恒温恒压(NPT)系综,温度取为310 K,压力取为1个标准大气压,长程静电力采用particle-mesh ewald(PME)处理方法,截断半径设置为1.3 nm,时间步长为2 fs.在力场文件中,手动去除多余的元素后,生成预处理文件;进行能量最小化后,载入白蛋白的结构及SPC模型水分子,进行位置受限模拟,检查能量是否集中;然后,进行没有边界条件下的模拟,观察白蛋白在水相中的折叠状态是否达到稳定,3 ns内可以得到优化的白蛋白模型[13].
1.2.2 白蛋白在石墨烯界面上的吸附
当白蛋白达到水相中平衡的条件后,向体系中载入石墨烯.计算两者的结构大小,确定建造一个边长为9 nm的立方体盒子.将.gro文件作为新的结构文件,导入水分子模型,进行时长为500 ps的模拟.
整个计算过程中体系都处于周期性边界性条件[14]下.体系采用恒温恒压系综,温度设置为310 K,压力为100 kPa.其中,单纯的石墨烯为单层界面,含有5 562个原子.针对石墨烯-水界面体系的模拟中约有10 793个水分子,以模拟人体体液环境,累计共有约74 863个原子.采用各向异性的Berendson控压方法,以确保不引入额外的体系对称性限制.
体系的初始速度设定为Maxwell分布.对于不同的体系,经过约200~500 ps后可使体系的总能量趋于平衡.为确保系统达到热平衡,在平衡模拟过程中检测和分析体系的总能量、温度、压力、体积等数值随时间的变化关系,直到这些值趋于平衡并在一定值附近稳定涨落后,认为体系达到热平衡.
1.2.3 吸附位点的确定
任何界面不可能完全没有间隙,理论上相对距离约0.5 nm[10]时即可认为是相对稳定的吸附.因此,通过计算白蛋白的每个氨基酸与石墨烯界面的相对距离,可以初步将离石墨烯最近的白蛋白的肽段认定为是吸附位点.
2 模拟结果及分析
2.1 水相中白蛋白的预平衡
在蛋白质数据库中下载的是白蛋白在真空状态下的X射线衍射数据模型,经过加氢步骤后得到的白蛋白的构象如图1所示.图1(a)为白蛋白的Cartoon模式,用较厚的光滑条带表示α-螺旋和β-折叠;图1(b)为Surface模式,是由白蛋白进行表面渲染后得到的.
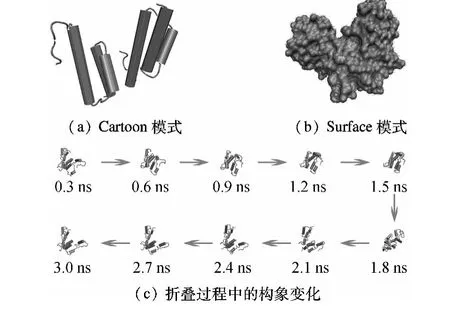
图1 白蛋白构象示意图
若体系总能量和势能收敛且体系趋于动态平衡,则表明预模拟时间充足.此时,残基原子的相对位移曲线表明蛋白质在水相中自我折叠,以达到相对平衡的稳定状态.采用VMD软件对白蛋白在水中的动态折叠过程进行模拟,所得的构象变化示意图见图1(c),每0.3 ns截取1幅图(此处略去水分子,使图像表达更为清晰).可以看到,水相中白蛋白在横轴方向上先被压缩而后恢复,蛋白质的构象较原始状态有了较大的变化,体系能量收敛至稳定,因此它在水溶液中的折叠最终达到了平衡.
2.2 石墨烯界面上白蛋白的吸附
引入石墨烯界面后,白蛋白与石墨烯界面动态结合后的整体构象如图2所示.

图2 白蛋白在石墨烯表面吸附结构示意图
图3为白蛋白在石墨烯表面吸附过程中能量及空间位置的结果,此处采用的是NPT系综.由图3(a)可知,在预定时间t内,相对较高的体系总能量E逐渐收敛并且达到稳定,体系趋于动态平衡,表明预模拟时间充足.均方根(RMS)涨落是MD模拟中反映分子内部运动特征及柔性的一个重要参数.图3(b)为白蛋白整体骨架(模拟系统中作为一个整体进行处理)的位置相对于初始态的位移变化(即RMSF值),通过选择4组蛋白骨架的数据进行最小二乘拟合.所得结果说明,在石墨烯界面存在的条件下,白蛋白中每个氨基酸残基自身构象发生变化,蛋白质在水相中实现自我折叠,继而在石墨烯界面达到动态平衡.从三维空间的3个方向上对蛋白质进行切割,动态变化过程中白蛋白骨架每个截面相对于石墨烯界面的垂直距离Rg如图3(c)所示.Rg体现了计算动态模拟过程中的结构与初始的结构偏差,Rg,x,Rg,y,Rg,z为Rg在x,y,z三个方向上的分量.均方根偏差(RMSD)表示模拟过程中材料维持原有结构的程度,是衡量体系是否稳定的重要依据.由图3(d)可见,在极短的时间(约十几ps)后,作为被吸附界面的石墨烯的构象已经不再变化,说明石墨烯界面在体系中的晶体结构能基本维持不变.白蛋白的存在并没有引起石墨烯构象太大的变化,石墨烯界面仍然保持完整.
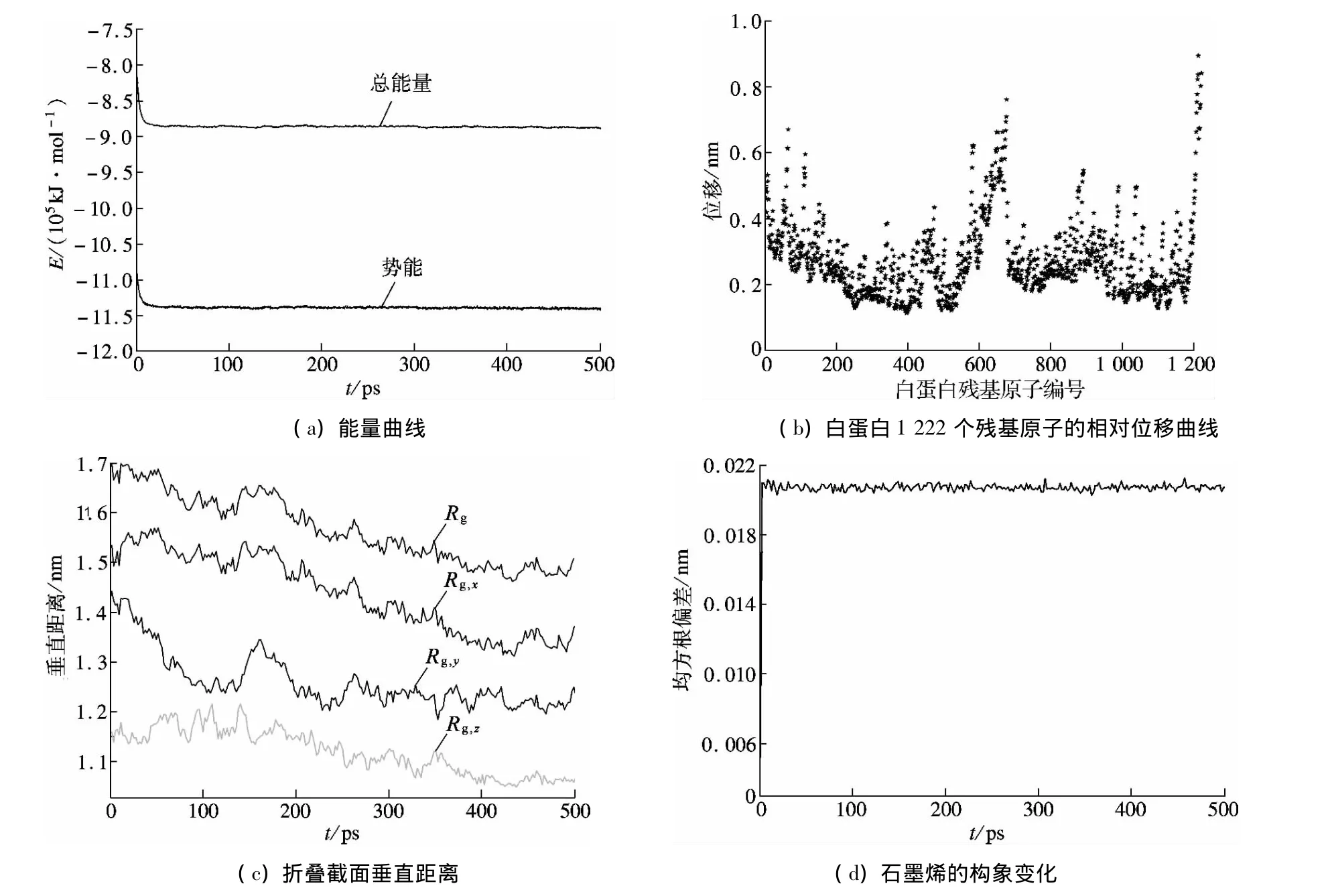
图3 白蛋白吸附到石墨烯界面过程中的能量及空间位置变化
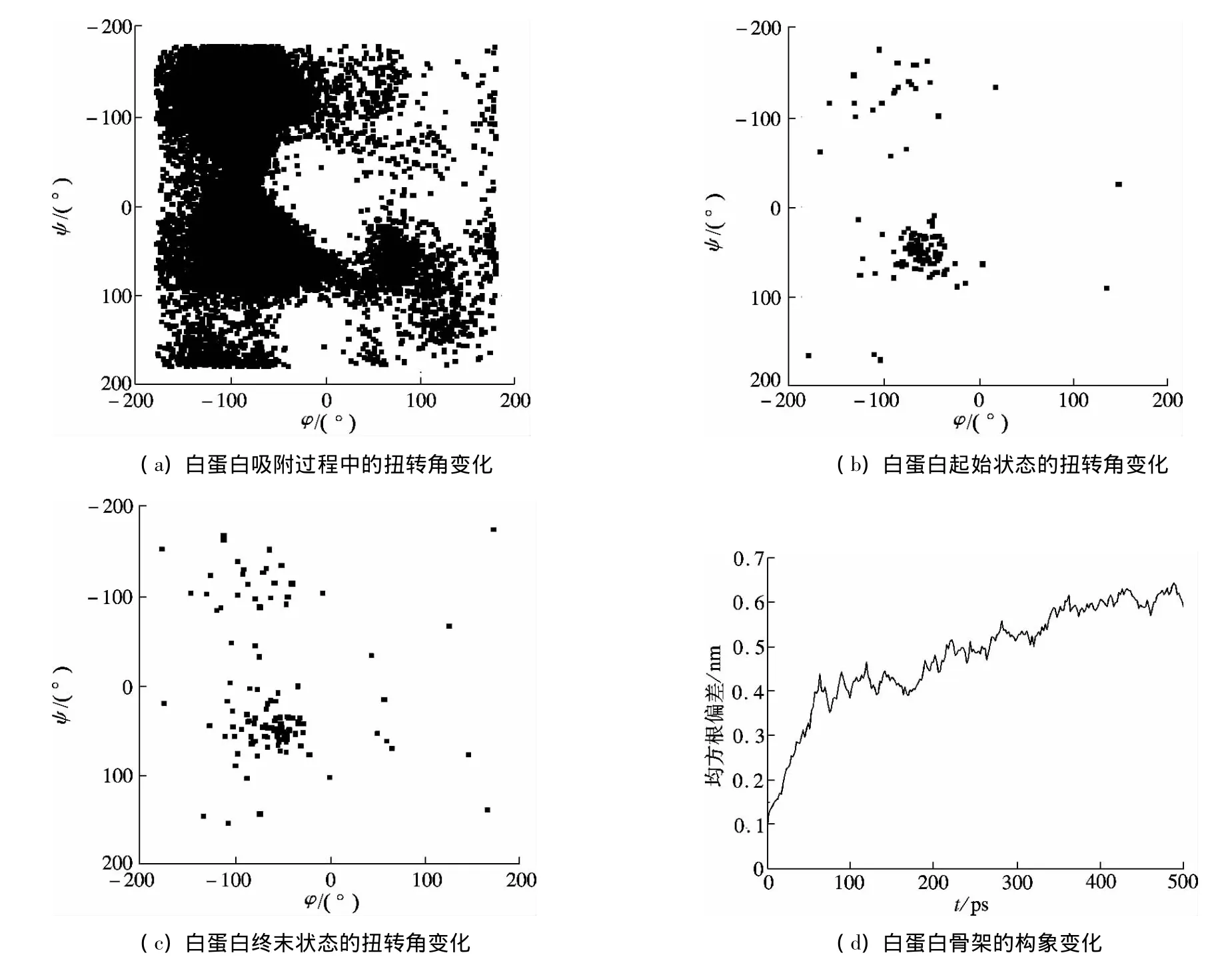
图4 白蛋白吸附到石墨烯表面的过程中的扭转角和骨架构象变化
图4为蛋白质的二面角图(ramachandran plot),表示白蛋白在石墨烯表面吸附过程中具体构象的变化,其中包括扭转角及白蛋白骨架的变化.ψ表示一个肽单位中α碳左边C—N键的旋转角度,φ表示α碳右边C—C键的旋转角度.二面角图以范德华接触距离和肽键键角为基础,横、纵坐标分别为二面角φ和ψ.在与石墨烯界面整个作用过程中,白蛋白的扭转角变化曲线见图4(a).对整个模拟过程进行统计分析后做抽样处理,抽样间隔由模拟时写入轨迹文件的频率决定,不同的抽样间隔对应不同的数据量,但基本可以得到相同的规律.为更清楚地显示蛋白质二面角的变化是否在可允许的范围内,图4(b)和(c)分别给出了起始和终末状态下白蛋白扭转的二面角.可以看到,在起始与终末状态,白蛋白的二面角图像分布已经发生了较大变化,说明成功吸附之后白蛋白的构象因为石墨烯的存在而发生了改变,即β转角增大,α折叠程度降低,左旋化趋势增加.由图4(d)可以看出,在模拟时间内,作为吸附物的白蛋白的构象变化已经稳定,不再发生较大变化,说明白蛋白与石墨烯的相对构象已经趋于固定.此时,水相中由于石墨烯界面的存在,其相互作用使得白蛋白的构象发生了改变.经过500 ps的模拟,整个反应体系已经达到了稳定的动态平衡,石墨烯界面和白蛋白的相对位置固定.
2.3 石墨烯界面白蛋白的吸附位点
图5为白蛋白骨架与石墨烯表面距离的示意图.肽段与石墨烯表面的距离定义为白蛋白骨架与石墨烯的质心距离在石墨烯法向量方向上的投影,或白蛋白骨架与石墨烯的法向的距离d.由图可知,引入石墨烯界面后,随着时间的变化,蛋白迅速接近石墨烯,约100 ps时白蛋白骨架与石墨烯的距离已经接近平台期,400 ps后距离稳定为0.65 nm(见图6).由于任何界面不可能完全没有间隙,故此处可认为白蛋白与石墨烯界面已经稳定在一个吸附状态.

图5 白蛋白骨架与石墨烯的距离示意图
确定白蛋白牢固吸附于石墨烯表面后,通过计算每个氨基酸在整个模拟过程中与石墨烯界面的相对距离,发现与石墨烯界面绝对距离最近(0.4 nm)的氨基酸正好是白蛋白肽段上的连续序列90ALA-91GLY-92ILE-93THR-94SER-95ASP-96PHE-97TYR.图6为模拟过程中此段氨基酸序列与石墨烯表面的距离.随着时间的增加,此距离变小,最终稳定在如图所示的范围之内.

图6 吸附的氨基酸序列与石墨烯的平均距离
对于石墨烯和白蛋白体系,经过500 ps的模拟,体系的总能量趋于平衡,说明模拟时间充足.各项数据显示,在适宜的模拟条件下,经过足够长的模拟时间后,白蛋白已经稳定地吸附在石墨烯界面表面.通过分析白蛋白中各个氨基酸相对于石墨烯界面的距离,发现与石墨烯表面相结合的是白蛋白氨基酸序列中90~97号肽段,此段序列即为吸附位点.
Christian等[15]获得的牛血清白蛋白在疏水石墨表面的分子动力学模拟结果显示,两者的吸附部位是配体连接的subdomainⅢ A部分;这说明蛋白质与材料界面存在一个稳定的作用位点,并且该位点很可能是作用的蛋白材料的一部分肽段.另外,Tillmann等[16]指出,利用传统的分子动力学以及转向分子动力学可以模拟得到骨形成蛋白(BMP-2)在石墨烯表面形成的微弱但位点固定的吸附,并且在纳秒级时间内不会发生变性现象;这说明蛋白与固定材料作用时会以相同的状态吸附在材料表面,且短期内不会导致蛋白变性或者材料损坏.上述结果与本文结论一致.
3 结语
本文采用分子动力学模拟的方法,对白蛋白模型水相中的构象进行了优化,建立了单层石墨烯界面.应用Gromacs软件和VMD软件完成了优化后的白蛋白模型、白蛋白与石墨烯界面的共存和相互作用以及白蛋白牢固吸附过程的模拟.通过对白蛋白在水相中构象的优化及在石墨烯界面上蛋白骨架、残基原子、扭转角等数据的分析,确定了白蛋白在石墨烯界面的吸附距离是0.65 nm,并且准确找出了两者之间相互作用的位点.这对蛋白质与材料相互作用的实验以及机理研究具有参考价值及意义.
[1]Lü X Y,Huang Y,Zheng B Z,et al.Comparing ad-sorptive property of natural and synthesized hydroxyapatite to albumin,fibrinogen and IgG[J].Key Engineering Materials,2007,330/331/332:869-872.
[2]Lü X Y,Cui W,Huang Y,et al.Surface modification on silicon with chitosan and biological research[J].Biomedical Materials,2009,4(4):044103.
[3]李艺,程镕时.蛋白质在固体表面吸附的研究进展[J].高分子通报,2007(3):41-49.
Li Yi,Cheng Rongshi.Development of studying for protein adsorption on the solid surface[J].Polymer Bulletin,2007(3):41-49.(in Chinese)
[4]Raffaini G,Ganazzoli F.Protein adsorption on the hydrophilic surface of a glassy polymer:a computer simulation study[J].Physical Chemistry Chemical Physics,2006,8(23):2765-2772.
[5]Kandori K,Tsuyama S,Tanaka H,et al.Protein adsorption characteristics of calcium hydroxyapatites modified with pyrophosphoric acids[J].Colloids and Surfaces B:Biointerfaces,2007,58(2):98-104.
[6]Huang Y,Lü X Y,Qian W P,et al.Competitive protein adsorption on biomaterial surface studied with reflectometric interference spectroscopy[J].Acta Biomaterialia,2010,6(6):2083-2090.
[7]Svendsen I E,Santos O,Sotres J,et al.Adsorption of HSA,IgG and laminin-1 on model hydroxyapatite surfaces-effects of surface characteristics[J].Biofouling,2012,28(1):87-97.
[8]沈贺,张立明,张智军.石墨烯在生物医学领域中的应用[J].东南大学学报:医学版,2011,30(1):223-228.(in Chinese)
Shen He,Zhang Liming,Zhang Zhijun.Application of graphene in biomedicine area[J].Journal of Southeast University:Medical Science Edition,2011,30(1):223-228.(in Chinese)
[9]姜晖,王雪梅,王晨苏.石墨烯类材料在生物医学领域应用的研究进展[J].药学学报,2012,47(3):291-298.
Jiang Hui,Wang Xuemei,Wang Chensu.Research progress of graphene-based materials in the application to biomedicine[J].Acta Pharmaceutica Sinica,2012,47(3):291-298.(in Chinese)
[10]Zhou H,Wu T,Dong X,et al.Adsorption mechanism of BMP-7 on hydroxyapatite(001)surfaces[J].Biochemical and Biophysical Research Communications,2007,361(1):91-96.
[11]Guo S,Shi X,Yang F,et al.Structural basis of transport of lysophospholipids by human serum albumin[EB/OL].(2009-09-13)[2012-10-18].http://www.pdb.org.
[12]Miroslav K,Ján U,Pavel M.Theoretical study of structure changes caused by applying mechanical strain on peptide L24[J].Journal of Molecular Modeling,2010,16(11):1693-1699.
[13]韩振为,周薇.分子模拟蛋白质在氧化锌界面上的吸附[J].天津大学学报,2007,40(3):366-370.
Han Zhenwei,Zhou Wei.Molecular simulation of adsorption of protein on zinc oxide interface[J].Journal of Tianjin University,2007,40(3):366-370.(in Chinese)
[14]Chen X,Wu T,Wang Q,et al.Shield effect of silicate on adsorption ofproteinsonto silicon-doped hydroxyapatite(100)surface [J].Biomaterials,2008,29(15):2423-2432.
[15]Christian M,Herbert M U.Molecular dynamics simulation of free and forced BSA adsorption on a hydrophobic graphite surface[J].Langmuir,2011,27(21):12938-12943.
[16]Tillmann U,Grazia D,Maria A M.Molecular dynamics simulations of the adsorption of bone morphogenetic protein-2 on surfaces with medical relevance[J].Langmuir,2011,27(21):13144-13153.
Molecular dynamics simulations of albumin and graphene structures and their interaction
Sun Ying1,2Ding Jiali1,2Zong Hao3Wang Jiandan1Lü Xiaoying1
(1Key Laboratory of Molecalar and Biomalecular Electronics of Ministry of Education,Southeast University,Nanjing 210096,China)
(2Jiangsu Key Laboratory for Biomaterials and Devices,Southeast University,Nanjing 210009,China)
(3College of Software Engineering,Southeast University,Nanjing 211189,China)
Abstract:Molecular dynamics simulations were used to study the interaction between albumin and graphene surface.During the simulation,Gromacs software was used to collect and analyze data,and VMD software was used to realize visualization of structures.First,the periodic boundary condition was established.The constant temperature and pressure ensemble was adopted in a limited space with the side length of 9 nm,and the original vacuum conformation of albumin was optimized under the force field ffgmx,so that equilibrium in the aqueous phase was reached.Then,the optimized stable albumin in the aqueous phase was used as a model to build the overall conformation and interaction interface with graphene.Finally,after 500 ps of the dynamic process under periodic boundary conditions,the adsorption of albumin onto the graphene surface was completed.The energy,trajectory of residues,torsion angle and other data were analyzed,and the adsorption sites of albumin on the graphene surface were determined to be 90 to 97 peptide 90ALA-91GLY-92ILE-93THR-94SER-95ASP-96PHE-97TYR.
Key words:molecular dynamics simulation;albumin;graphene;interaction mechanism
中图分类号:R318.08
A
1001-0505(2014)01-0123-06
doi:10.3969/j.issn.1001-0505.2014.01.022
收稿日期:2013-04-30.
孙颖(1988—),女,硕士生;吕晓迎(联系人),女,博士,教授,博士生导师,luxy@seu.edu.cn.
基金项目:国家自然科学基金资助项目(31271012).
孙颖,丁佳丽,宗皓,等.白蛋白和石墨烯的结构及其相互作用的分子动力学模拟[J].东南大学学报:自然科学版,2014,44(1):123-128.[doi:10.3969/j.issn.1001-0505.2014.01.022]
