DNA与CdS-NH2纳米粒子的相互作用研究
2014-09-06叶树虹陈受惠
申 源,叶树虹,汪 莉,陈受惠
(江西师范大学化学化工学院,江西 南昌 330022)
DNA与CdS-NH2纳米粒子的相互作用研究
申 源,叶树虹,汪 莉,陈受惠*
(江西师范大学化学化工学院,江西 南昌 330022)
采用荧光光谱、紫外光谱(UV-vis)、圆二色谱(CD)、透射电镜(TEM)、原子力显微镜(AFM)、琼脂糖凝胶电泳等技术研究CdS-NH2-EcoRI复合物与DNA的相互作用.研究发现: CdS-NH2纳米粒子与pBR322DNA结合后会延迟EcoRI的酶切反应.DNA的曲率和纳米粒子的粒径都是影响结合作用的因素,曲率较大的环状DNA比线性DNA能更好地与纳米粒子结合,小粒径的CdS-NH2纳米粒子则更易结合到DNA上.并研究了DNA与CdS-NH2纳米粒子之间的作用机理.
CdS-NH2;EcoRI;DNA;CdS-NH2-EcoRI复合物;荧光光谱;圆二色谱;原子力显微镜;琼脂糖凝胶电泳
0 引言
发光量子点纳米粒子(如CdSe、ZnS、CdS纳米晶体)具有显著的光催化稳定性和一些优越特性(量子尺寸效应、表面效应等),被广泛用于生物定向研究[1-4],如J.F.Campbell等将CdSe/ZnS纳米粒子与包裹在碳纳米管上的DNA结合,利用量子点的发光特性确定DNA的位置[5].CdS纳米粒子在生物化学、生物传感器等相关领域具有潜在的应用价值,因此其与DNA之间相互作用的研究备受关注[6].
CdS纳米粒子与DNA等生物大分子的结合常被应用于生物试验和生物传感器的研究.通常对其表面进行必要的修饰来提高其生物兼容性,已报道有许多方法能够提高这些纳米材料的水溶性等[7-8],如二氧化硅涂层[9-10]、结合亲水配体[11-12].不同的修饰方法能够提高或改变纳米粒子的相应性能,增强生物兼容性[13-14],从而提高其应用价值.如带有亲水基团的半导体纳米粒子因其能够有效地识别荧光能量的定向转移而在定向研究上具有广阔的应用前景,有望被应用于细胞摄取等[7,15].在众多的报道中,对小粒径的CdS纳米粒子采取改变修饰官能团的方法操作相对简单,具有较大的潜在应用价值.如K. Susumu等将纳米粒子的末端修饰了氨基、羧基等几种不同的官能团,能够更好地与蛋白素结合[16].
本文合成了CdS-NH2纳米粒子,结合UV-vis[17]、CD[18]、AFM[19]、琼脂糖凝胶电泳[20]等技术手段,研究了线性λDNA和环状pBR322DNA与CdS-NH2纳米粒子之间的相互作用.结果显示CdS-NH2纳米粒子与DNA之间同样发生了沟槽结合作用,同时—NH2与DNA分子的磷酸骨架之间还存在着氢键作用,促进了CdS-NH2纳米粒子与DNA的结合.
1 实验
1.1 仪器和实验
λ-DNA(48 502 bp)、pBR322DNA(4 361 bp)、λ-HindIII digest DNA Marker (with bands of 23 130,9 416,6 557,4 361,2 322 and 2 027 bp)、TAE缓冲液(40 mmol·L-1Tris-Acetate,2 mmol·L-1EDTA)、EcoRI (大肠杆菌 RY13)、琼脂糖等购于中国大连TaKaRa生物试剂公司;GelRed Nucleic Acid Gel stain (10000X in water)购于美国Biotium公司;Na2S·9H2O和CdCl2,购于上海阿拉丁;L-半胱氨酸(Cys,97%)购于Sigma-Adlrich.其他试剂均购于中国北京化学试剂公司.所有试剂使用前均没有进一步纯化,溶液均由超纯水(>18 MΩ·cm) 配制.云母(KAl12(AlSi3)O10(OH)2)购于中国长春.
仪器:上海爱建AJ-III型原子力显微镜;FEI公司Technai S-twin透射电子显微镜;伯乐Mini-PROTEAN电泳仪;Perkin-Elmer Lambda 35紫外分光光度仪;Perkin-Elmer LS 55 luminescence荧光分光光度仪;Bio-Logic公司MOS-450/AF-CD圆二色谱仪.
1.2 实验过程
1.2.1 CdS-NH2纳米粒子的制备 CdS-NH2纳米粒子的合成方法参照文献[21-23],合成过程如图1所示.将1.0 mmol·L-1的Cys溶于100 mL超纯水中,在搅拌的条件下通入氮气60 min.用0.5 mol·L-1Tris 调控溶液的pH值在8.5~9.0之间.随后缓慢加入0.5 mmol·L-1的CdCl2溶液,使n(Cys)∶n(Cd2+)=2∶1,搅拌反应30 min后加入10 mL 0.5 mmol·L-1的Na2S溶液,溶液呈无色.然后密封在47 ℃水浴下孵育2 h,这时溶液转为澄清的黄绿色.随后通入N230 min以除去未反应完全的Na2S.所得CdS-NH2溶液在室温下保存.由于合成过程中溶液pH值在8.5~9.0之间,大于等电点5.05,CdS-NH2纳米粒子表面带负电荷.该纳米粒子粒径大小,可通过调节S/Cd2+物质的量之比来控制[23].本工作中分别选取n(S)∶n(Cd2+)为1∶1和1∶2,制备了粒径约1.8 nm和4.2 nm的纳米粒子.

图1 CdS-NH2纳米粒子合成示意图
1.2.2 样品的光谱分析 合成的CdS溶液在60 ℃条件下真空干燥得到CdS粉末,取适量与KBr研磨压片后在干燥恒温的条件下进行傅立叶红外光谱测定.相关参数设定为DTGS检测器,分辨率为4 cm-1,扫描次数为32次.
配制一定浓度的DNA溶液和CdS-NH2溶液,DNA与CdS-NH2的混合溶液均置于37 ℃的恒温水浴中孵育30 min后使用.使用1 cm石英比色皿,测试的波长范围为200 ~ 600 nm.
配制5.0 ng·μL-1λ-DNA、0.1 mmol·L-1CdS溶液、一定浓度的KI溶液.DNA与CdS-NH2的混合溶液均置于37 ℃的恒温水浴中孵育30 min后使用.使用1 cm 石英比色皿,在375 nm的激发波长下测试400~700 nm范围的荧光光谱.
配制20.0 ng·μL-1λ-DNA和0.2 mmol·L-1的CdS-NH2溶液.DNA与CdS-NH2的混合溶液均置于37 ℃的恒温水浴中孵育30 min.使用1 cm 石英比色皿,以200 nm.min-1的速度在200~400 nm的范围内扫描圆二色谱.
1.2.3 样品的TEM表征 用超纯水配制0.5 mmol·L-1CdS-NH2溶液,取约15 μL均匀倾倒于400目的铜网上,室温干燥后在200 kV的加速电压下进行HRTEM检测.
1.2.4 样品的AFM表征 首先剪切大小约为1 cm×1 cm的云母,使用前确保其表面抛光无明显裂纹.然后将1.0 ng·μL-1λ-DNA溶液、1.0 ng·μL-1pBR322DNA溶液分别与1 mmol·L-1CdS-NH2溶液混合,分别加入1 mmol·L-1醋酸钙溶液,在37 ℃条件下恒温孵育30 min.然后,分别取约30 μLλ-DNA-CdS-NH2、pBR322DNA-CdS-NH2溶液滴于新粘制的云母上,吸附约13 min后,依次用1∶1超纯水/无水乙醇、无水乙醇冲洗5次左右,置于干燥器内干燥即可.AFM选用轻敲模式,在室温条件下操作,采用标准的硅探针(力常数0.6~6.0 N·m-1),其共振频率为60~150 kHz.
1.2.5 样品的琼脂糖凝胶电泳表征 使用1%琼脂糖在120 V电压下进行约25 min的电泳测试.测试中使用4 μL GelRed进行染色,使用TAE缓冲液(40 mmol·L-1Tris-Acetate,2 mmol·L-1EDTA).
配制40 ng Marker,40 ng DNA溶液,1 mmol·L-1CdS-NH2溶液,将DNA-CdS-NH2混合液在37 ℃条件下恒温孵育.使用1 U EcoRI和DNA以及复合物于37 ℃恒温孵育不同时间后进行酶切反应.
2 结果与讨论
2.1 CdS-NH2纳米粒子的表征


图2 (A)CdS-NH2纳米粒子的TEM图;(B)CdS-NH2纳米粒子红外光谱图
将合成的2种粒径大小的CdS-NH2纳米粒子进行AFM表征和紫外光谱测试,结果如图3所示.由于量子点尺寸效应,CdS-NH2特征吸收峰位置由515 nm蓝移至380 nm和480 nm,如图3(A)和图3(C)所示[26].所得的CdS-NH2纳米粒子的尺寸可根据Brus公式[27]进行计算:
E=Egbulk+ħ2/8r2(1/mom*e+1/mom*h)-
1.8e2/4πεεor,
(1)
其中Egbulk为体相材料能隙,ħ为普朗克常量,r为粒子半径,m*e为电子有效质量,m*h为空穴有效质量,mo为自由电子有效质量,e为自由电子有效电荷,εo为空穴介电常数,ε为相对介电常数.由此算得CdS-NH2纳米粒子粒径分别为1.76 nm和4.13 nm.
原子力显微镜(AFM)用于表征CdS-NH2纳米粒子的形貌特征,如图3(A)和图3(C)所示,表征了2种不同粒径大小的CdS-NH2纳米粒子,图中CdS-NH2纳米粒子较均匀的分布在云母表面.这2图分别对应于图3(B)和图3(D)中纳米粒子TEM图.从图中能直观地得出CdS-NH2纳米粒子的粒径大小分别为(1.78±0.03) nm和(4.23±0.05) nm.
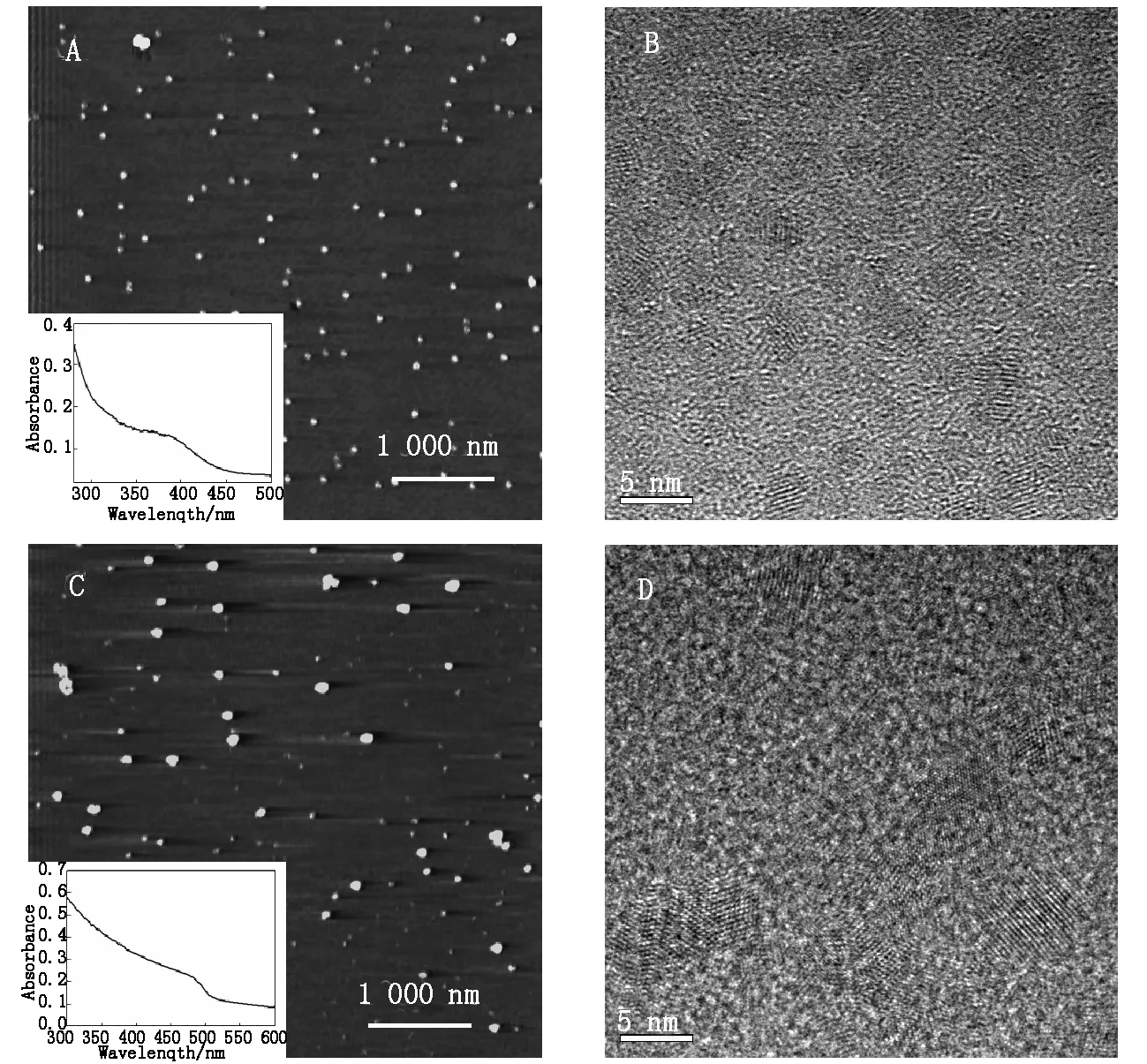
图3 (A)、(B)为小粒径CdS-NH2纳米粒子的AFM表征图、TEM图;(C)、(D)为大粒径CdS-NH2纳米粒子的AFM表征图、TEM图.插图为CdS-NH2纳米粒子紫外光谱图
2.2 CdS-NH2纳米粒子与DNA的相互作用
紫外光谱法是一种判断CdS-NH2纳米粒子与DNA之间相互作用的模式的简单检测方法.如果小分子与DNA之间是嵌插结合,那么加入DNA后小分子的紫外吸收峰会发生较为明显的红移和减色效应[28];若是静电结合或沟槽结合,则紫外吸收峰发生微弱的减色效应,峰位置一般不红移或红移不明显[29].图4(A)和图4(B)分别为1.0 mmol·L-1CdS-NH2与5 ng·μL-1λ-DNA、5 ng·μL-1pBR322DNA相互作用的紫外光谱图.图4中约375 nm处为CdS-NH2纳米粒子的紫外吸收峰位置.随着DNA的加入,CdS-NH2纳米粒子在375 nm处的吸收峰发生了减色效应,这表明CdS-NH2纳米粒子与DNA之间发生了相互作用[28-30].相比线性λ-DNA,同浓度的环状pBR322DNA与CdS-NH2结合后,CdS-NH2吸收峰减色更加明显,这反映了曲率更大的环状DNA与纳米粒子的结合作用比线性DNA分子稍强.根据CdS-NH2纳米粒子的结构特点判断CdS-NH2纳米粒子与DNA的磷酸骨架之间存在着氢键作用.该氢键作用一定程度上增强了CdS-NH2纳米粒子与DNA的结合作用.
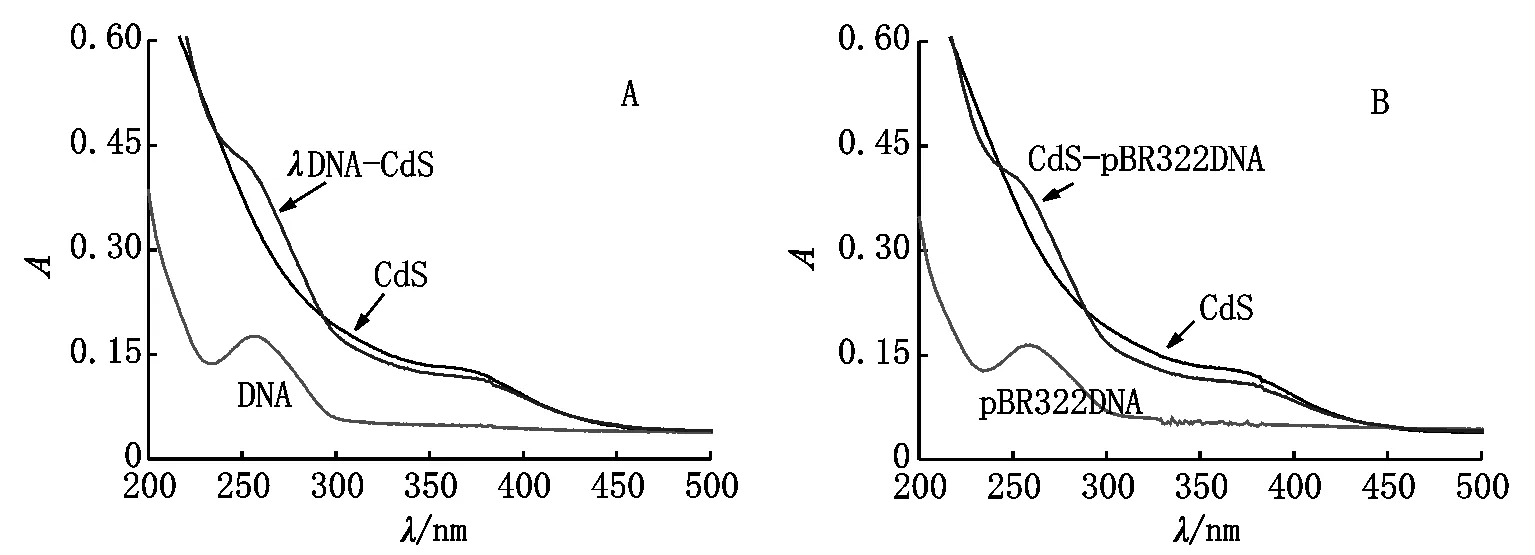
图4 (A) λ-DNA(5 ng·μL-1),CdS-NH2(1.0 mmol·L-1)-λ-DNA(5 ng·μL-1)紫外光谱图.(B) pBR322DNA(5 ng·μL-1),CdS-NH2-pBR322DNA紫外光谱图
本文使用荧光猝灭法来进一步判断CdS-NH2纳米粒子与DNA之间的作用方式.带负电的DNA磷酸骨架能够排斥带负电的猝灭剂.若小分子与DNA是嵌插结合,则小分子嵌插进入DNA分子双链中在一定程度上保护了其荧光不被猝灭,使猝灭程度减弱[31];若是沟槽结合,则小分子的荧光强度很容易被阴离子猝灭剂猝灭[32].猝灭剂KI常用于判断小分子与DNA的结合方式,通过计算比较KI对CdS-NH2纳米粒子、CdS-NH2-DNA体系的荧光猝灭常数可得到相应的结论.图5分别是KI对CdS-NH2纳米粒子、CdS-NH2-DNA体系的荧光猝灭情况.根据Stern-Volmer方程可计算荧光猝灭常数[32]:
劳动最光荣,劳动最崇高,劳动最伟大,劳动最美丽。逐梦新征程,唱响新时代的奋斗者之歌,工会组织要为劳动模范、大国工匠发挥作用搭建平台、提供舞台,为劳模工匠传承技能、传承精神创造条件,培养造就更多劳动模范、大国工匠。要大力弘扬劳模精神、劳动精神、工匠精神,让诚实劳动、勤勉工作蔚然成风,引导广大职工以劳动和创造托起中国梦。
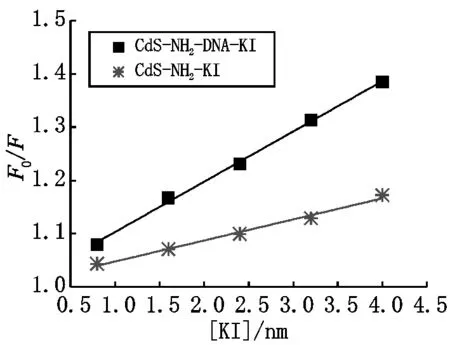
图5 KI对0.1 mmol·L-1 CdS-NH2纳米粒子、CdS-NH2 (0.1 mmol·L-1)-DNA(5 ng·μL-1)体系的猝灭常数线性图.KI的浓度为0.8,1.6,2.4,3.2,4.0 mmol·L-1.
F0/F= 1 +KSV[Q],
(2)
其中F0为KI不存在时的荧光强度,F为加入KI后的荧光强度,KSV为猝灭常数,[Q]为猝灭剂浓度.根据公式(2),由F0/F对[Q]作工作曲线,得到CdS-NH2纳米粒子、CdS-NH2-DNA体系的荧光猝灭常数分别为3.9×105L·mol-1和9.4×105L·mol-1.这说明CdS-NH2纳米粒子与DNA结合后,KI对其的荧光猝灭程度不但没有减弱,反而增强.结果证明CdS-NH2纳米粒子与DNA之间为沟槽结合.
为深入探究CdS-NH2纳米粒子与DNA之间的结合方式,本文研究了单链DNA(ssDNA)和双链DNA(dsDNA)对CdS-NH2纳米粒子的荧光猝灭效果,如图6所示.试验中将dsDNA在沸水中加热10 min之后迅速置于冷水中,即可得到ssDNA.若是静电结合,ssDNA和dsDNA对CdS-NH2纳米粒子具有相同的猝灭效果;若是嵌插结合(小分子嵌插入dsDNA的双螺旋结构中),由于ssDNA没有双螺旋结构,无法实现嵌插结合,ssDNA对CdS-NH2纳米粒子的猝灭效果将很微弱;若是沟槽结合(小分子通过疏水作用力结合到DNA分子的沟槽当中),由于ssDNA的碱基对大量的暴露出来,将增强小分子与DNA碱基对之间的作用力,ssDNA对CdS-NH2纳米粒子的猝灭效果将增强[33-34].从图6可以看出,与dsDNA相比,ssDNA对CdS-NH2纳米粒子的猝灭作用明显更强.这证明了CdS-NH2纳米粒子与DNA的结合方式是沟槽结合.
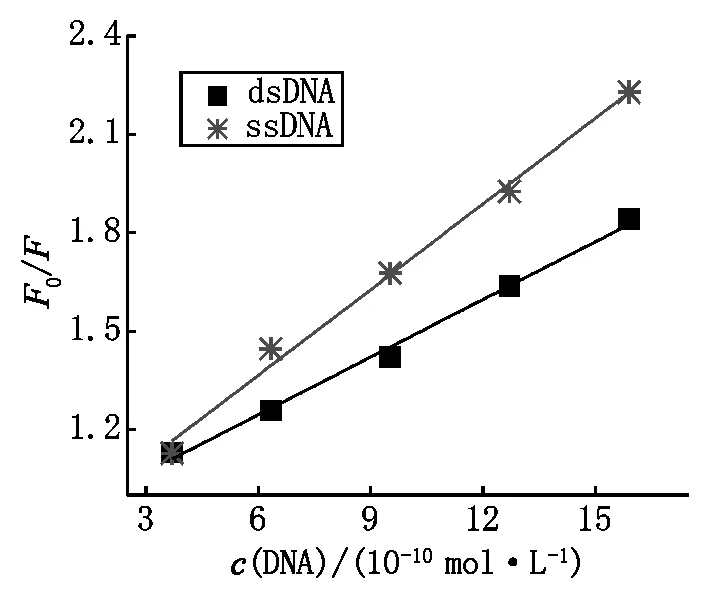
图6 dsDNA和ssDNA对0.1 mmol·L-1 CdS-NH2纳米粒子的Stern-Volmer荧光猝灭图
圆二色谱技术能够反映小分子与DNA发生结合作用后DNA结构的变化情况.B构型DNA在远紫外区(220~320 nm)能够表现出特定的圆二色谱图形[35],结合作用导致的DNA结构的变化能够直接表现为圆二色谱峰形的变化.从图7可以看出,λ-DNA在约276 nm处有一正峰(由于碱基堆积),约246 nm处有一负峰(由于不对称的核苷酸的螺旋结构),这说明λ-DNA是典型的B构型DNA[35].小分子与DNA沟槽结合对DNA的碱基堆积和螺旋结构影响很小或者没有影响,不影响其构型[36].而发生嵌插作用时可削弱DNA碱基之间的π-π堆积作用使双螺旋结构变得松散,因而CD谱中的正负峰有明显的变化.CdS-NH2纳米粒子在远紫外区没有圆二色谱特征峰.由图7可知,λ-DNA与CdS结合后,λ-DNA在276 nm和246 nm处的峰形几乎没有发生变化,这证明CdS-NH2对λ-DNA的构型没有产生影响,与沟槽结合的作用方式相符合.
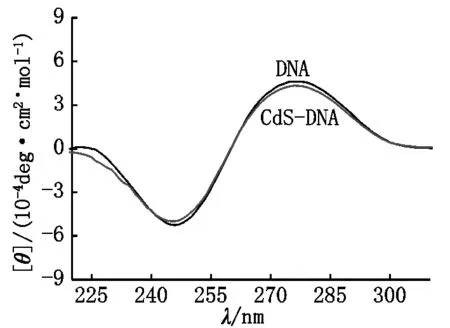
图7 λ-DNA(20 ng·μL-1),CdS-NH2(0.2 mmol·L-1)-λ-DNA(20 ng·μL-1)圆二色光谱图
AFM是用于表征DNA与小分子之间相互作用形貌结构的常用仪器[37-38],它能够清晰准确直观地反映样品表面的形貌,扫描尺度精细,像素清晰.图8为DNA与CdS-NH2纳米粒子结合后的形貌特征.其中(A)、(B)为λ-DNA与不同粒径的CdS-NH2纳米粒子结合的形貌图,(C)为pBR322DNA与小粒径CdS-NH2纳米粒子结合的形貌图.图8(A)中显示与DNA结合的CdS-NH2粒径主要集中在(0.98±0.02) nm左右,结合数量较多;图8(B)中显示与DNA结合的CdS-NH2粒径主要集中在(2.82±0.15) nm左右,结合数量较少.对比图8(A)、图8(B),表明并非所有粒径大小的CdS-NH2纳米粒子都能罗好地与DNA结合,粒径尺寸较大的CdS-NH2纳米粒子与DNA的结合数量较少.结果表明DNA在不同直径大小的CdS-NH2纳米粒子中能够选择其中一种粒径CdS-NH2纳米粒子结合,存在氢键作用并没有改变这一特点.
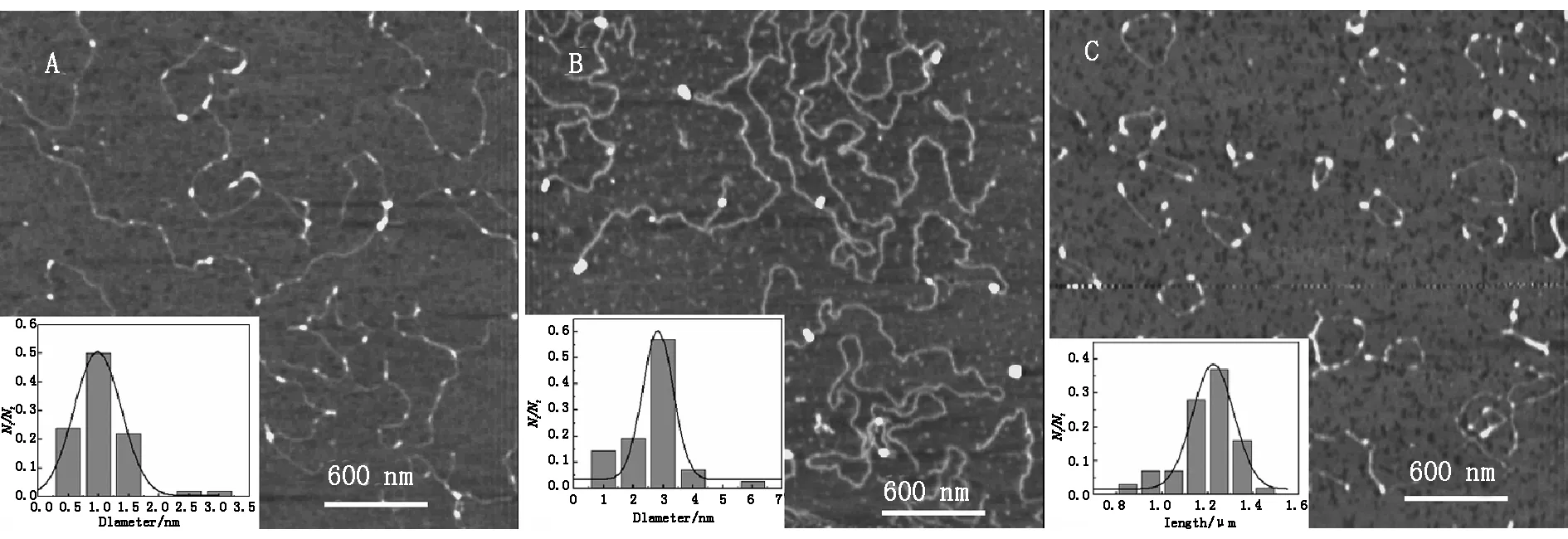
(A)CdS-NH2(1.76 nm)-λ-DNA(1.0 ng·μL-1)形貌图;(B)CdS-NH2(4.13 nm)-λ-DNA(1.0 ng·μL-1)形貌图.插图为结合到λ-DNA上的CdS-NH2粒径分布柱状图;(C) CdS-NH2(1.76 nm)-pBR322DNA(1.0 ng·μL-1)形貌图,插图为pBR322DNA长度柱状统计图.
小分子与DNA若是嵌插结合,则会导致DNA分子链增长,而沟槽结合则不会影响DNA分子的长度.图8(C)中插图为pBR322DNA长度柱状统计图,由图可知,与CdS-NH2纳米粒子结合后pBR322DNA长度约为1.28 μm,与未结合的DNA长度一致,该结果证明它们之间为沟槽结合.采用统计分析的方法,选取粒径约为1 nm的CdS-NH2纳米粒子来统计分析CdS-NH2纳米粒子与DNA的结合数量.图(A)中λ-DNA链上平均每微米约有3个CdS-NH2纳米粒子;图(C)中pBR22DNA分子上平均每微米约有4.5个CdS-NH2纳米粒子.对比发现CdS-NH2纳米粒子与环状的pBR322DNA结合数量较线性的λ-DNA结合数量更大.有文献报道,由于环状质粒DNA比线性DNA分子具有更大的曲率和更小的刚性,接触作用面积更大,与其它分子结合消耗的能量更小,因此能更好地结合[39].同时发现,较小粒径的CdS-NH2纳米粒子也更容易与环状pBR322DNA发生沟槽结合作用
另外,CdS-NH2纳米粒子与DNA结合数量较大,这与—NH2与DNA分子磷酸骨架之间存在氢键促进了结合作用的推断相符合.
2.3 CdS-NH2纳米粒子对EcoRI与DNA酶切作用的影响
EcoRI是一种常用的限制性核酸内切酶,通过识别DNA链上的特定碱基序列位点(GAATTC)对DNA进行酶切作用[40-41].在37 ℃条件下恒温1 h(1×H缓冲液)1 U EcoRI能够剪切1 μgλ-DNA、0.4 μg pBR322DNA.CdS-NH2纳米粒子与DNA通过静电结合后,是否会影响蛋白质(EcoRI)对DNA的剪切作用,从而对EcoRI剪切DNA这一生物活性是否有影响,通过琼脂糖凝胶电泳测试结果,分析不同条带的DNA分子片段(如不同大小DNA分子片段,环状或线性等不同形状)便可得出结论.
图9(B)为CdS-NH2纳米粒子与DNA相互作用凝胶电泳图.作为图9(A)的对照,图9(B)中λ-DNA、pBR322DNA与CdS-NH2纳米粒子结合孵育1.5 h后DNA的电泳条带位置均没有变化,这表明CdS-NH2纳米粒子对DNA没有剪切作用.
如图9(A)所示,泳道1为λ-DNA,泳道2、3分别为恒温37 ℃ 1.0 h、1.5 h后EcoRI对CdS-NH2-λ-DNA酶切反应.泳道3中出现了5条不同位置的条带,这说明泳道3中的λ-DNA被EcoRI剪切成了5条不同分子大小的片段,而泳道2中的λ-DNA没有被剪切.这证明CdS-NH2纳米粒子与λ-DNA结合后对EcoRI的剪切作用产生了一定的影响,阻碍了酶切反应的进行,使在1.0 h内EcoRI对λ-DNA没有发生酶切作用.但随着时间的增加(至1.5 h)EcoRI仍然能够识别λ-DNA上的酶切位点并进行酶切作用.由泳道5、6可以看出,在恒温孵育1.0 h时酶切反应没有发生,直到恒温孵育1.5 h发生了酶切反应,即环状pBR322DNA被EcoRI剪切开环,CdS-NH2纳米粒子与pBR322DNA结合后对EcoRI的生物活性的影响效果比较显著.这可能是由于CdS-NH2与DNA除了静电结合外还存在氢键作用,使得pBR322DNA上结合了更多数量的CdS-NH2纳米粒子,一定程度上阻碍了EcoRI在pBR322DNA上结合位点的识别,从而影响了剪切作用.这一现象表明纳米粒子与DNA分子的这种非特异性结合能够阻碍EcoRI对其在DNA链上的结合位点的特异性识别,而DNA上的这种非特异性结合的纳米粒子数量越多,对EcoRI酶活性的影响越显著,并且对碱基对较少的质粒DNA的酶切作用影响更加明显.

泳道1~6分别为:λ-DNA、EcoRI与λ-DNA-CdS-NH2恒温孵育1.0 h,EcoRI与λ-DNA-CdS-NH2恒温孵育1.5 h、pBR322DNA,EcoRI与pBR322DNA-CdS-NH2恒温孵育1.0 h,EcoRI与pBR322DNA-CdS-NH2恒温孵育1.5 h.(B)CdS-NH2纳米粒子与DNA相互作用凝胶电泳图.泳道1~5分别是λ-DNA、λ-DNA-CdS-NH2恒温孵育1.5 h,CdS-NH2、pBR322DNA、pBR322DNA-CdS-NH2恒温孵育1.5 h.
3 结论
本文使用紫外光谱、荧光光谱、圆二色谱、透射电镜、原子力显微镜、琼脂糖凝胶电泳等技术手段,研究了CdS-NH2纳米粒子与2种DNA(线性λ-DNA和环状pBR322DNA)之间的相互作用.结果表明DNA与CdS-NH2纳米粒子之间为沟槽结合和氢键作用;DNA的曲率和纳米粒子的粒径都是影响结合作用的因素(曲率较大的环状DNA比线性DNA能更好的与纳米粒子结合,粒径为1 nm左右的CdS-NH2纳米粒子更容易结合到DNA上);同时发现CdS-NH2纳米粒子能影响EcoRI对DNA的酶切作用,从而延迟EcoRI对DNA的酶切反应.本文为研究纳米粒子与DNA相互作用的动力学提供参考.
[1]Alivisatos A P.Semiconductorclusters,nanocrystals and quantum dots [J].Science,1996,271:933-937.
[2] Murray C B,Kagan C R,Bawendi M G.Synthesis and characterization of monodisperse nanocrystals and close-packed nanocrystal assemblies [J].Annu Re Mater Sci,2000,30:545-610.
[3] Murray C B,Norris D J,Bawendi M G.Synthesis and characterization of nearly monodisperse CdE (E = sulfur,selenium,tellurium) semiconductor nanocrystallites [J].J Am Chem Soc,1993,115:8706-8715.
[4] 赵孝梨,陈昱瑾,董入源,等.超声辅助磁性纳米四氧化三铁催化过氧化氢降解亚甲蓝染料的研究 [J].西南大学学报:自然科学版,2012,34(5):51-56.
[5] Campbell J F,Tessmer I,Holden Thorp H,et al.Atomic force microscopy studies of DNA-wrapped carbon nanotube structure and binding to quantum dots [J].J Am Chem Soc,2008,130:10648-10655.
[6] Mirkin C A.Programming the assembly of two- and three-dimensional architectures with DNA and nanoscale inorganic building blocks [J].Inorg Chem,2000,39:2258-2272.
[7] Medintz I L,Uyeda H T,Goldman E R,et al.Quantum dot bioconjugates for imaging,labelling and sensing [J].Nat Mater,2005,4:435-446.
[8] Michalet X,Pinaud F F,Bentolila L A,et al.Quantum dots for live cells,in vivo imaging,and diagnostics [J].Science,2005,307:538-544.
[9] Bruchez M Jr,Moronne M,Gin P,et al.Semiconductor nanocrystals as fluorescent biological labels [J].Science,1998,281:2013-2016.
[10] Gerion D,Pinaud F,Williams S C,et al.Synthesis and properties of biocompatible water-soluble silica-coated CdSe/ZnS semiconductor quantum dots [J].J Phys Chem B,2001,105:8861-8871.
[11] Chan W C W,Nie Shuming.Quantum dot bioconjugates for ultrasensitive nonisotopic detection [J].Science,1998,281:2016-2018.
[12] Wang Qiangbin,Xu Yang,Zhao Xiaohang,et al.A facile one-step in situ functionalization of quantum dots with preserved photoluminescence for bioconjugation [J].J Am Chem Soc,2007,129:6380-6381.
[13] Gao Xiaohu,Cui Yuanyuan,Levenson R M,et al.In vivo cancer targeting and imaging with semiconductor quantum dots [J].Nat Biotechnol,2004,22:969-976.
[14] Clapp A R,Medintz I L,Mattoussi H.Förster resonance energy transfer investigations using quantum-dot fluorophores [J].Chem Phys Chem,2006,7:47-57.
[15] Delehanty J B,Medintz I L,Pons T,et al.Self-assembled quantum dot-peptide bioconjugates for selective intracellular delivery [J].Bioconjugate Chem,2006,17:920-927.
[16]Susumu K,Tetsuo Uyeda H,Medintz I L,et al.Enhancing the stability and biological functionalities of quantum dots via compact multifunctional ligands [J].J Am Chem Soc,2007,129:13987-13996.
[17] Chakraborty B,Basu S.Interaction of BSA with proflavin:A spectroscopic approach [J].J Lumin,2009,129:34-39.
[18] Prado-Gotor R,Grueso E A.kinetic study of the interaction of DNA with gold nanoparticles:mechanistic aspects of the interaction [J].Phys Chem Chem Phys,2011,13:1479-1489.
[19] Guo Cunlan,Liu Zhelin,Xu Fugang,et al.Surface-relevant regulable DNA toroids induced by dopamine [J].J Phys Chem B,2009,113:6068-6073.
[20] Costa F O,Neuparth T,Helena Costa M,et al.Detection of DNA strand breakage in a marine amphipod by agarose gel electrophoresis:exposure to X-rays and copper [J].Biomarkers,2002,7(6):451-463.
[21] Kho R,Claudia L,Martinez T,et al.A simple colloidal synthesis for gram-quantity production of water-soluble ZnS nanocrystal powders [J].J Colloid Interf Sci,2000,227:561-566.
[22] Bae W,Mehra R K.Cysteine-capped ZnS nanocrystallites:Preparation and characterization [J].J Inorg Biochem,1998,70:125-135.
[23]Chen Jinlong,Zheng Aifang,Gao Yingchun,et al.Functionalized CdS quantum dots-based luminescence probe for detection of heavy and transition metal ions in aqueous solution [J].Spectrochimica Acta Part A,2008,69:1044-1052.
[24] Brus L E.Electronic wave functions in semiconductor clusters:experiment and theory [J].J Phys Chem,1986,90:2555-2560.
[25] Koneswaran M,Narayanaswamy R.Mercaptoacetic acid capped CdS quantum dots as fluorescence single shot probe for mercury(II) [J].Sensor Actuat B-Chem,2009,139:91-96.
[26] Zhang Z H,Chin W S,Vittal J J.Water-soluble CdS quantum dots prepared from a refluxing single precursor in aqueous solution [J].J Phys Chem B,2004,108:18569-18574.
[27] Steigerwald M L,Brus L.Semiconductor crystallites:a class of large molecules [J].Accounts Chem Res,1990,23:183-188.
[28] Ulrich K H.Molecular aspects of ligand binding to serum albumin [J].Pharmacol Rev,1981,33:17-53.
[29] Deepa S,Mishra A K.Fluorescence spectroscopic study of serum albumin-bromadiolone interaction:fluorimetric determination of bromadiolone [J].J Pharm Biomed Anal,2005,38(3):556-563.
[30] 倪永标,卢春,韩二伟,等.PIF同步荧光猝灭法测定Ga(Ⅲ) [J].江西师范大学学报:自然科学版,2011,35(3):269-271.
[31] Li Xiaoling,Hu Yanjun,Wang Hong,et al.Molecular spectroscopy evidence of berberine binding to DNA:comparative binding and thermodynamic profile of intercalation [J].Biomacromolecules,2012,13:873-880.
[32]Sahoo D,Bhattacharya P,Chakravorti S.Quest for mode of binding of 2-(4-(dimethylamino)styryl)-1-methylpyridinium iodide with calf thymus DNA [J].J Phys Chem B,2010,114:2044-2050.
[33]Cai Changqun,Chen Xiaoming,Ge Fei.Analysis of interaction between tamoxifen and ctDNA in vitro by multi-spectroscopic methods [J].Spectrochimica Acta Part A,2010,76:202-206.
[34] 王瑞玲,徐丽繁,邓承雨,等.光谱法研究PBBHAMF与小牛胸腺DNA的相互作用 [J].江西师范大学学报:自然科学版,2014,38(1):6-10.
[35] Neidle S.Nucleic acid structure and recognition [M].New York:Oxford University Press,2002.
[36] Silvestri A,Barone G,Ruisi G.The interaction of native DNA with Zn(II) and Cu(II) complexes of 5-triethyl ammonium methyl salicylidene ortho-phenylendiimine [J].J Inorg Biochem,2007,101:841-848.
[37] Ye Jingyong,Umemura K,Ishikawa M,et al.Atomic force microscopy of DNA molecules stretched by spin-coating technique [J].Anal Biochem,2000,281:21-25.
[38] Umemura K,Komatsu J,Uchihashi T,et al.Atomic force microscopy of recA-DNA complexes using a carbon nanotube tip [J].Biochem Biophys Res Commun,2001,281:390-395.
[39] Mahtab R,Rogers J P,Singleton C P,et al.Preferential adsorption of a “kinked” DNA to a neutral curved surface:Comparisons to and implications for nonspecific DNA-protein interactions [J].J Am Chem Soc,1996,118:7028-7032.
[40] Alison D P,Kerper P S,Doktycz M J,et al.Direct atomic force microscope imaging of EcoRI endonuclease site specifically bound to plasmid DNA molecules [J].Proc Natl Acad Sci USA,1996,93:8826-8829.
[41] Allison D P,Kerper P S,Doktycz M J,et al.Mapping individual cosmid DNAs by direct AFM imaging [J].Gemomics,1997,41:379-384.
(责任编辑:刘显亮)
TheStudyontheInteractionsbetweenDNAandCdS-NH2Nanoparticles
SHEN Yuan,YE Shu-hong,WANG Li,CHEN Shou-hui*
(College of Chemistry and Chemical Engineering,Jiangxi Normal University,Nanchang Jiangxi 330022,China)
In this work,the interaction between DNA and CdS-NH2-EcoRI composites was investigated by infrared spectroscopy,UV-vis spectroscopy,fluorescence spectroscopy,CD spectroscopy,atomic force microscope,agarose gel electrophoresis,etc.It was shown that the restriction enzyme reaction of EcoRI would be retarded after CdS-NH2nanoparticles combined with pBR322DNA.Both the curvature of DNA and the particle size of nanoparticles are the crucial factors influencing the groove binding.Circular-like DNA with larger curvature is more easily combined with nanoparticles than linear DNA.And CdS-NH2nanoparticles with smaller size were more easily combined with DNA.The interaction mechanism of DNA and CdS-NH2nanoparticles was also studied in this work.
CdS-NH2;EcoRI;DNA;CdS-NH2-EcoRI composites;fluorescence spectroscopy;CD spectroscopy;atomic force microscope;agarose gel electrophoresis
2014-06-20
国家自然科学基金(2105005),江西省教育厅科技课题(GJJ10389)和江西师范大学青年成长基金(5487)资助项目.
陈受惠(1978-),男,江西南昌人,讲师,主要从事光谱分析研究.
1000-5862(2014)06-0624-08
Q 523;TB 383.1
:A
