奥司他韦、扎那米韦和帕拉米韦对人H7N9流感病毒神经氨酸酶的抑制作用
2014-08-31蔚晓晖
蔚晓晖,刘 伟
郑州大学生物工程系生命科学学院 郑州 450001
奥司他韦、扎那米韦和帕拉米韦对人H7N9流感病毒神经氨酸酶的抑制作用
蔚晓晖,刘 伟#
郑州大学生物工程系生命科学学院 郑州 450001
#通讯作者,男,1967年7月生,博士,教授,研究方向:生物信息学与药物设计,E-mail: weiliu@zzu.edu.cn
H7N9流感病毒;神经氨酸酶;分子动力学;分子对接
H7N9流感病毒属于甲型流感病毒,是一种新亚型流感病毒,2013年3月首次在人类中检测出[1-2]。目前尚未有明显证据表明该病毒可以在人与人之间传播[3],但H7N9病毒之间的遗传变化表明其病毒较易传播给人类,并且和SARS一样可导致严重的呼吸道症状及死亡[4]。目前较常用的抗流感病毒药物主要有奥司他韦、扎那米韦和帕拉米韦,它们可以通过抑制神经氨酸酶(neuraminidase,NA)来抑制流感病毒的繁殖。NA是一种流感病毒糖蛋白,能催化水解唾液酸末端的N-乙酰基神经氨酸与邻近寡糖D-半乳糖或是D-氨基半乳糖之间的α(2,6) 或α(2,3) 糖苷键,使成熟的病毒颗粒最终脱离宿主细胞,感染新的上皮细胞,造成流感病毒在患者体内的扩散。虽然甲型流感病毒NA活性中心的结构在流感病毒中是较为保守的,但流感病毒基因易重组突变,NA活性位点上氨基酸残基的改变有可能使病毒对NA抑制剂产生明显抗药性[5-6]。因此,对NA药物抑制特性的研究有助于对人感染H7N9流感病毒的防控工作。作者利用同源建模和分子对接方法,通过对药物分子与H7N9 NA结合能的比较,分析药物分子对NA的抑制作用。
1 材料与方法
1.1序列比对与同源建模从NCBI数据库中获得所有H7N9流感病毒NA的完整序列,包括6条人感染H7N9流感病毒NA序列,利用clustalx进行序列比对。选取人感染H7N9流感病毒A/Hangzhou/1/2013的NA序列(NCBI序列号:AGI60300.1, AGI)和其他来源H7N9流感病毒株A/wild bird/Korea/A3/2011的NA序列(NCBI序列号:AEK84752.2, AEK)进行同源建模。同源建模采用CPHmodels-3.2网络服务器[7],可获得对应序列的三维空间结构。
1.2分子动力学平衡分子动力学模拟采用Amber11,蛋白质力场采用ff99力场[8]。NA模型在进行动力学模拟之前还需要利用Xleap程序进行处理,包括增加氢原子、增加抗衡离子以使体系中性化以及增加8.5 μm厚的基于TIP3P模型的八面体水环境。分子动力学过程优化分为两个阶段:首先约束溶质,对溶剂分子进行优化;再无约束优化整个系统。每个阶段均采用最陡下降法优化2 500步,再采用共轭梯度法优化2 500步。优化之后用60 ps时间使系统缓慢升温到298 K,在298 K下进行500 ps的体系平衡过程,平衡之后再进行1.5 ns分子动力学模拟,最后选取体系能量最低的构像状态作为NA的稳定结构。按上述方法处理,分别获得AGI和AEK的稳定结构。
1.3分子对接模拟从PDB数据库里获得野生型流感H5N1病毒NA与奥司他韦复合物(WT-OTV, PDB Code: 2HU4)的结构作为模板,然后将WT-OTV分别与平衡后的AGI和AEK的稳定结构进行叠加,删除WT-OTV的NA,分别得到了AGI、AEK与奥司他韦复合物(AGI-OTV、AEK-OTV)的结构。同法获得AGI、AEK分别与扎那米韦和帕拉米韦复合物(AGI-ZNV、AEK-ZNV和AGI-PRV、AEK-PRV)的结构。对获得的6种复合结构,分别采用Autodock 4.0 程序[9]进行分子对接。对接选用的格点盒子大小为40点×40点×40点,格点间距0.375埃。运用Lamarckian 遗传算法,对小分子进行100次的独立对接实验,能量极大值设为2.5×106,最大变异代数设为27 000,最后得到这些复合物与药物小分子的结合能。
2 结果与讨论
6条人感染H7N9病毒NA均出自长三角地区,序列比对的结果表明,序列相似度很高,而且在活性位点均未发生变异,由此初步判断这次人感染H7N9病毒NA结构比较保守。除人感染H7N9流感病毒NA序列外,只有AEK序列在第118位活性位点发生了R到T的突变。
在分子动力学模拟过程中,为了判断体系是否平衡,分别计算了动能、势能、总能和NA的主链Cα原子的RMSD值。RMSD是指体系在模拟过程中产生的结构相对于晶体结构随时间变化的均方根偏差,也是衡量体系是否稳定的重要依据。图1表示的是NA主链Cα原子的RMSD值随时间的变化值,其中,横坐标表示的是时间,纵坐标表示的是RMSD值,由图中可以看出时间大于350 ps以后,AGI和AEK的主链Cα原子的RMSD值达到1.5埃左右并保持平衡。同时体系动能、势能和总能的变化迅速达到稳定值,AGI的动能、势能和总能分别保持在62 760、-292 880、-230 120 kJ/mol,AEK分别保持在83 680、-376 560、-292 880 kJ/mol,表明AGI和AEK在500 ps的动力学模拟过程中都达到了平衡,因此,可从模拟的最后阶段选取NA的稳定结构。
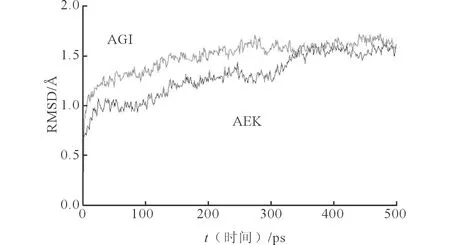
图1 NA的主链Cα原子在平衡阶段的动力学模拟的RMSD值变化
AGI和AEK与奥司他韦、扎那米韦和帕拉米韦的结合能如表1所示。从表1可以看出,奥司他韦、扎那米韦和帕拉米韦对AGI的抑制作用比较明显;而AEK与药物分子的结合能力较AGI略大,第118位活性位点R到T的突变并未减弱药物分子对AEK的抑制作用。3CL2是通过对NA和奥司他韦晶体结构(PDB Code: 3CL2)直接计算获得的数值,该值直接反映了3CL2和奥司他韦之间较强的相互作用[10],其与奥司他韦的结合能与AGI和AEK接近。上述结果说明奥司他韦、扎那米韦和帕拉米韦对人感染H7N9病毒株NA的抑制作用比较明显。
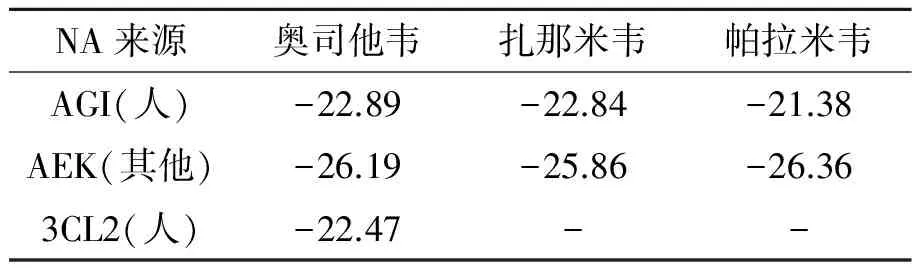
表1 AGI和AEK与奥司他韦、扎那米韦和帕拉米韦的结合能 kJ/mol
综上所述,人流感H7N9病毒株NA序列变化小,结构比较保守,未发生活性位点突变;药物分子对其的抑制作用明显,因此对H7N9流感病毒在体内释放会有较好的抑制效果。但AEK活性位点的突变说明H7N9 NA有可能产生抗药性突变,需引起关注。
[1]Alcorn, T. As H7N9 spread in China, experts watch and wait[J]. Lancet, 2013, 381(9875): 1347
[2]张宝,黄克勇,郭劲松,等.H7N9病毒的来源和重组模式[J].南方医科大学学报,2013,33(7):1017
[3]Horby P.H7N9 is a virus worth worrying about[J].Nature,2013,496(7446):399
[4]From SARS to H7N9: will history repeat itself?[J]. Lancet, 2013, 381(9875):1333
[5]Colman PM, Varghese JN, Laver WG. Structure of the catalytic and antigenic sites in influenza virus neuraminidase[J]. Nature, 1983, 303(5912): 41
[6]McKimm-Breschkin JL. Resistance of influenza viruses to neuraminidase inhibitors:a review[J].Antiviral Res, 2000, 47(1) : 1
[7]Nielsen M, Lundegaard C, Lund O, et al. CPHmodels-3.0-Remote homology modeling using structure guided sequence profiles[J]. Nucleic Acids Research, 2010, 38(Web Serve issue): W576
[8]Duan Y, Wu C, Chowdhury S, et al. A point-charge force field for molecular mechanics simulations of proteins based on condensed-phase quantum mechanical calculations[J]. J Comput Chem, 2003, 24(16): 1999
[9]Morris G, Goodsell D, Halliday R, et al. Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function[J]. J Comput Chem, 1998, 19(14): 1639
[10]Collins PJ,Haire LF, Lin YP,et al. Crystal structures of oseltamivir-resistant influenza virus neuraminidase mutants[J]. Nature, 2008, 453(7199):1258
(2013-08-15收稿 责任编辑李沛寰)
10.13705/j.issn.1671-6825.2014.03.046
