应用微卫星和下颌骨形态分析法对不同昆明小鼠种群进行遗传分析
2014-08-14韩喜彬王立辛苏玉虹巴彩凤
韩喜彬,王立辛,苏玉虹,巴彩凤
(1.辽宁医学院实验动物中心,锦州 121001; 2.辽宁医学院畜牧兽医学院,锦州 121001)
昆明(KM)小鼠中国应用广泛的封闭群实验小鼠,在科研、教学和检定实验中被大量使用,经过多年繁育不同单位的种群携带着或多或少改变的基因位点,这种变化位点的累计最终会导致具体的性状改变,使实验数据的重复性大大降低,所以急需对KM小鼠乃至所有封闭群动物建立遗传学标准,通过遗传监测使其保持遗传稳定性[1]。国内实验动物学界已经意识到昆明小鼠的遗传质量问题,较早的报道证明了不同来源的KM小鼠在体型特征、血液指标和实验反应特性等方面存在差别[2-4];而近年以STR分析KM小鼠遗传特征的报道较多,研究结果发现不同种群之间存在中等或较大程度的遗传背景差异[5-7]。遗传控制的关键在于遗传的稳定性,即群体基因的数量和基因型频率始终保持不变,从而贮藏现存的遗传杂合性,从这一角度考量基因水平的监测必不可少,另外,动物实验是针对个体表型的测量和观察,遗传控制的结果是表型稳定,所以必须测量宏观指标。本研究选择微卫星法和下颌骨形态法对KM小鼠进行遗传分析,将遗传的内因、外因相结合,以期为封闭群动物的遗传学标准建立提供参考。
1 材料和方法
1.1 实验动物
昆明小鼠60只,(20±1)g,雌雄各半,SPF级。30只购自中国医科大学实验动物部8周龄昆明小鼠(SCXK(辽)2008-0005,简称A种群);30只购自大连医科大学实验动物中心(SCXK(辽)2008-0002,简称B种群)。两个昆明小鼠种群都引种自北京国家啮齿类实验动物种子中心,封闭时间8年以上。实验完成于辽宁医学院实验动物中心(SYXK(辽)2008-0004)。
1.2 实验方法
1.2.1 下颌骨形态分析:下颌骨测量位点参照Festing[8]建立的方法,共利用11个变异函数,1~6为高度测量点,7~11为长度测量点,均为右侧下颌骨(支);标本制作过程为煮沸3 min,胰酶37℃消化24 h,清水洗净,拔掉门齿,以黑色背景扫描成图像,再以Photoshop 8.0分析图像获得数据(图1)。
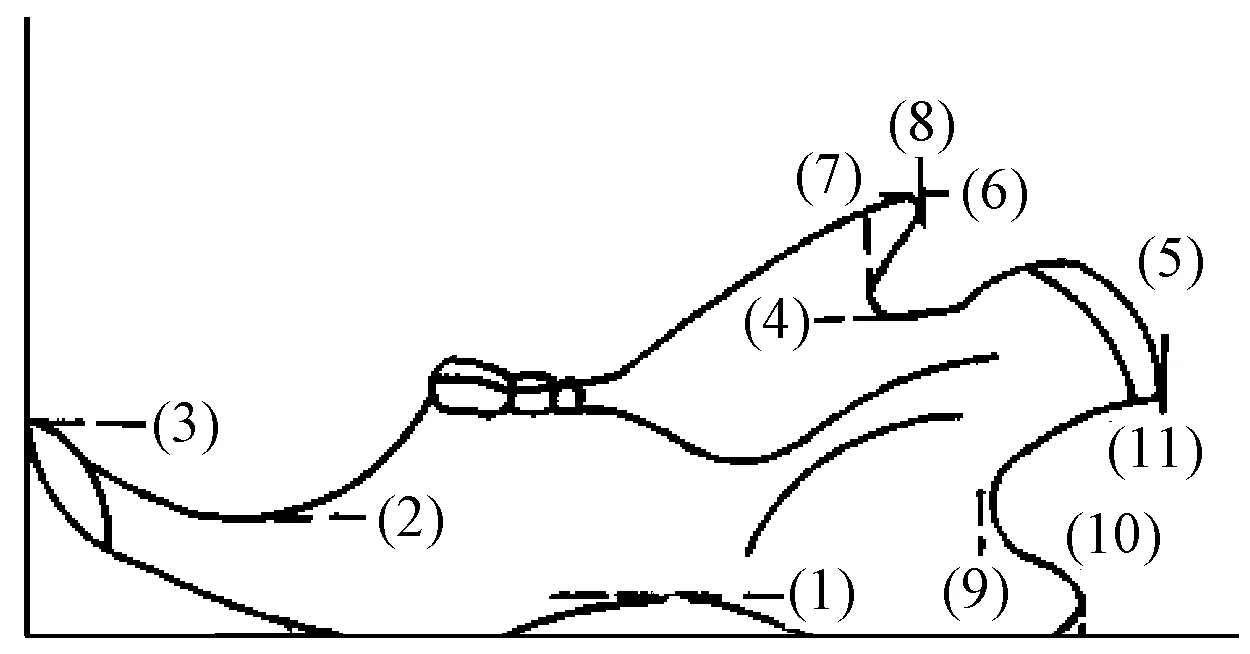
(1~6为高度变量,7~11为长度变量)
1.2.2 微卫星检测:常规酚氯法提取基因组DNA,参考小鼠基因组数据库 (http://www.informatics.jax.org)及本实验室之前的研究结果[9],选取10个分布在不同染色体的微卫星位点,每个位点多态信息丰富,引物序列见表1。优化PCR反应体系,PCR产物进行10%聚丙烯酰胺凝胶电泳,AgNO3染色,根据泳动距离进行结果判读,泳动距离最长的带设为英文字母a,依次为b、c等,泳动距离一致的为单态性(单一条带),若有差异即为多态性(多个条带)。所用试剂均购自大连宝生物公司。平均杂合度(he)、平均多态信息含量(PIC)和遗传距离(DA)分析分别按Nei(1974)、Bosein(1980)和Nei(1983)计算(如下)。
1.2.2.1 平均杂合度、多态信息含量
式中n为特定基因位点上的等位基因数,Pi、Pj分别等于等位基因的频率,he为杂合度。
1.2.2.2 遗传距离
式中:
Xij—X群体中第j个座位上第i个等位基因的频率;
Yij—Y群体中第j个座位上第i个等位基因的频率;

表1 微卫星位点引物序列及反应条件
mj— 第j个座位上的等位基因数;
r— 检测的座位数。
1.2.3 数据处理:采用SPASS 13.0统计软件处理数据。
2 结果
2.1 下颌骨形态分析结果
下颌骨变量测量结果见表2、图2。两单位KM小鼠种群11项参数中有5个参数差别明显,B种群2项高度参数X4、X5,2项长度参数X9、X10都大于A种群,差异极显著 (P﹤0.01),A种群只有高度参数X6大于B种群,达到差异显著(P﹤0.05),说明两个种群小鼠的下颌骨形态存在一定差别。
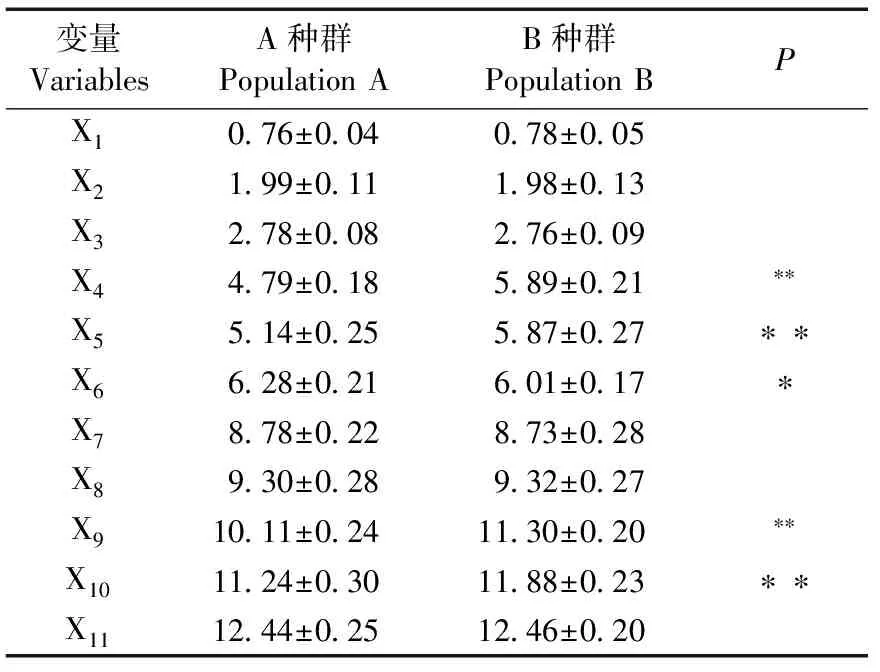
表2 KM小鼠下颌骨参数测量结果

图2 KM小鼠下颌骨扫描图像
2.2 微卫星检测结果
10个微卫星位点检测结果见表3、图3。两家单位共检测出28个等位基因、31个基因型,最多等位基因数4个,最少1个,平均杂合度(he)0.472,平均多态信息含量(PIC)0.382。A种群KM小鼠共检出27个等位基因、28个基因型、平均杂合度0.525、平均多态信息含量0.402;B种群KM小鼠共检出26个等位基因、27个基因型、平均杂合度0.418、平均多态信息含量0.361。两个种群在D2NDS3等5个位点基因型分布不同,A种群的遗传多样性略高于B种群,两个种群遗传距离为0.076。
3 讨论
在KM小鼠等封闭群动物的繁育过程中,遗传污染、基因突变和选择压都能造成遗传差异,这种差异体现在异地种群间,也体现在同一种群相隔较远的代次间,遗传监测的价值在于及时、尽早地发现这些变化,在这些变化固定下来之前对种群进行调整,避免基因分布的改变。本研究以微卫星法和下颌骨分析法获得了两个单位KM小鼠的遗传学数据,两个种群的个别位点和形态参数差别明显,说明两个种群的分化是存在的,根据此结果无法说明种群合格与否,因为至今没有形成KM小鼠封闭群的定性或定量标准,检测结果只能说明其遗传特征。在国内的相关研究报道中,能够对封闭群动物的遗传学指标做出明确说明的不多,其中吴艳花等、李薇等以微卫星技术对国内遗传背景明确的几个小型猪、长爪沙鼠的种群遗传特征进行了分析,并以群体平均杂合度或平衡状态做为群体合格与否的指标[10,11]。其他以微卫星技术检测实验大鼠或小鼠遗传结构的报道并不罕见,检测结果不但能说明群体遗传概貌,也能区分一些品系[12,13];以遗传监测为目的的下颌骨形态学检测方面的报道很少,陈哲等通过对3种白化小鼠下颌骨变量的研究,探讨了其在品系鉴别中的应用价值[14],吴宿慧等探讨了下颌骨测量技术在性别鉴定中的作用[15],这些研究丰富了封闭群实验动物的遗传学数据,虽然未能建立标准,也具备一定参考价值。

表3 KM小鼠10个微卫星位点检测结果

1:ab;2:aa;3:ac;4:ac;5:ac;6:ab
现今世界范围内被承认的通用标准封闭群有美国Charles River Laboratories CD-1种群(ICR小鼠和SD大鼠)以及日本CLEA的Wistar大鼠种群,两个机构基础群的建立方法相同,都取自遗传背景明确的多个种群,力求建立该品系完整基因库,再选择恰当的监测方法,在长期的繁育过程中确定具体的监测指标和操作规程,并以此方法指导选种、留种和迁移等。KM小鼠在我国分布广泛,各地种群遗传差异大,分化明显,更为重要的是遗传背景不明确,所以,目前各个单位有必要对各自种群进行连续的监测,完善基础数据,为KM小鼠遗传学监测标准的建立打下基础,让这个具有中国特色的品系稳定延续,并可促进我国实验动物标准化事业的发展。
4 结论
两个KM小鼠种群存在基因结构和表型特征的差异,群间轻度遗传分化。
参考文献:
[1] 朱玉峰, 王元占, 杨培梁, 等. 封闭群大小鼠国际遗传标准化 [C]. 第五届中南地区实验动物科技交流年会, 2005.
[2] 张周, 徐永平, 夏春梅. 不同来源KM小鼠在药物抑瘤作用重复实验中的结果差异原因初探 [J]. 上海实验动物科学, 2000, 20(4 ):240 -241.
[3] 李洪涛, 杜春燕, 章金涛, 等. 两个昆明小鼠封闭群部分生物学特性的比较[J]. 河南医科大学学报, 2000, 35(4):318-319.
[4] 赵国际, 陈国强, 朱冬琴, 等. 中国6个KM小鼠群体外形形态特征调查 [J]. 上海实验动物科学, 2001, 21(4):239-240.
[5] 班建荣, 胡芹宝, 张新创. 两个封闭群SPF级昆明小鼠遗传背景调查[J]. 微生物学免疫学进展, 2013, 41(5):6-9.
[6] 商海涛, 魏泓, 岳秉飞, 等. 应用微卫星标记对三个昆明小鼠封闭群的遗传学研究[J]. 实验动物科学,2009, 26(2):1-6.
[7] 张建琴, 申士小, 任连生, 等. 微卫星座位对KM封闭群小鼠的遗传分析[J]. 四川动物, 2008, 27(5):791-793.
[8] Festing M, Staats J. Standardized nomenclature for inbred strains of rats. Fourth listing [J]. Transplantation, 1973, 16:221-245.
[9] 王立辛, 韩喜彬, 苏玉虹. KM小鼠和ICR小鼠遗传特征分析[J]. 安徽农业科学, 2012, 28(35):19-24.
[10] 吴艳花, 徐玲玲, 杜小燕, 等. 封闭群实验用小型猪遗传标准的建立[J]. 实验动物科学, 2010, 27(6):33-35.
[11] 李薇, 江其辉, 杜小燕, 等. 封闭群长爪沙鼠遗传标准的建立[J]. 实验动物科学, 2010, 28(2):31-34.
[12] 谢建云, 冯洁, 胡建华. 微卫星标记在近交系及封闭群小鼠遗传质量检测中的初步应用[J]. 上海交通大学学报: 农业科学版, 2008, 26(4):290-295.
[13] 王洪, 岳秉飞, 刘双环, 等. 小鼠11个品系20个微卫星基因位点的遗传分析[J]. 中国比较医学杂志, 2006, 3(16):136-137.
[14] 陈哲, 赵琳, 陈晓, 等. KM、BALB/c和ICR小鼠下颌骨形态变量的初步探讨[J]. 上海实验动物科学,2004, 24(4):221-224.
[15] 吴宿进, 赵晓进, 胡春红, 等. KM、ICR、BALB/c小鼠下颌骨形态变量性差分析[J]. 河南师范大学学报 (自然科学版), 2005, 33(4):126-128.
