2种豆象线粒体COX1和12SrRNA基因序列的克隆分析
2014-07-24吴培福佟友贵潘涌智熊忠平窦报坤
吴培福 佟友贵 王 琳 潘涌智 熊忠平 窦报坤
(1. 西南林业大学生命科学学院,云南 昆明 650224;2. 昆明市东川区森防站,云南 昆明 654100;3.云南省林业职业技术学院,云南 昆明 650224;4.西南林业大学林学院,云南 昆明 650224)
2种豆象线粒体COX1和12SrRNA基因序列的克隆分析
吴培福1佟友贵2王 琳3潘涌智4熊忠平4窦报坤4
(1. 西南林业大学生命科学学院,云南 昆明 650224;2. 昆明市东川区森防站,云南 昆明 654100;3.云南省林业职业技术学院,云南 昆明 650224;4.西南林业大学林学院,云南 昆明 650224)
分别提取银合欢豆象和合欢豆象的基因组,对其COX1和12SrRNA基因进行测序分析。结果表明:2种豆象的COX1和12SrRNA基因A、T含量较丰富;碱基C和T间有最高的转换率;偏向使用以A或T结尾的密码子;银合欢豆象与合欢豆象进化距离较远,与A.oblongoguttatus进化距离最近,合欢豆象与多型豆象属聚在一个大的分支上。
银合欢豆象;合欢豆象;COX1;12SrRNA;测序分析
新银合欢(Leucaenaleucocephala)与滇合欢(Albiziasimeonis)是南方广泛种植的绿化防护树种,具有重要的经济和生态价值,但其种子分别受银合欢豆象(Acanthoscelidesmacrophthalmus(Schaeffer))和合欢豆象(Bruchidiusterrenus(Sharp))的为害,严重制约了2个树种的自然更新和生态功能的发挥[1-3]。因此,对这2种豆象进行科学研究具有重要的实践意义。纵观多年文献资料,关于2种豆象的研究多集中于形态学特征、生活习性、生物学特性及防治等方面[2,4-7]。银合欢豆象属外来入侵害虫,隶属豆象科(Bruchidae)三齿豆象属(Acanthoscelides),体长2.9~3.8 mm,宽1.8~2.0 mm,锯齿状触角呈红褐色,鞘翅多红褐色,后足腿节大齿的长约为后足胫节基部宽的1.5倍[4,8]。银合欢豆象的食性高度专一,仅为害银合欢属植物,其种群数量与银合欢生长的物候密切相关[4]。合欢豆象又称三带豆象,属豆象科,多型豆象属(Bruchidius),体长2.7~4.5 mm,宽1.6~2.1 mm,椭圆形,锯齿状触角呈赤褐色,鞘翅上各有10列刻点列,后足胫节端部有隆脊和刺突[2]。合欢豆象可为害台湾相思(Acaciaconfusa)和合欢的种子,其活动与气温变化有关[2]。
2种豆象的研究虽然取得了一定的成果,但目前仍缺乏系统研究,尤其是分子生物学方面。近年来,线粒体基因不断地应用于昆虫研究,且取得了较好的成果。为了奠定银合欢豆象和合欢豆象的分子生物学研究基础,本文对2种豆象的线粒体COX1和12SrRNA基因序列进行测序分析,期望为豆象的地理种群、系统分类、遗传多样性等研究提供参考。
1 材料与方法
1.1 试虫来源
银合欢豆象,于2013年1月采集于云南昆明市东川区;合欢豆象,于2013年1月采集于四川西昌市。采集2种豆象相应寄主的豆荚,带回实验室置于大烧杯内,饲养收集羽化的成虫试虫。通过形态学特征鉴定羽化的实验样品,而后置于95%乙醇中,于-20℃冰箱中保存备用。
1.2 试验方法
1.2.1 豆象基因组DNA提取与纯化 分别采用单头相应试虫,利用基因组DNA提取试剂盒(Qiagen,德国)按照试剂盒说明书提取试虫的基因组DNA。配置1%琼脂糖凝胶,检测提取基因组DNA的完整性。将DNA模板置于-20℃冰箱中保存备用。
1.2.2 引物设计及PCR扩增产物 引用Simon[9]设计的兼并引物扩增2种豆象的线粒体基因,即:采用引物C1-J2195和C1-N2776扩增试虫线粒体COX1基因序列;采用引物SR-J14197和SR-N14745扩增试虫线粒体12SrRNA基因序列。COX1基因的扩增片段约为660 bp,12SrRNA基因的扩增片段约为580 bp。引物由上海生工生物有限公司合成。PCR反应总体系为50 μL,其中2×Power Taq PCR MasterMix (BioTeke)25 μL,DNA模板2 μL,20 nmol/L上下游引物各1 μL,灭菌水21 μL。PCR扩增条件为:94℃预变性4 min; 94℃变性45 s,50℃退火45 s,68℃延伸45 s,30个循环;最后72℃总延伸6 min。1%琼脂糖凝胶检测PCR扩增产物。利用琼脂糖凝胶DNA回收试剂盒(BioTeke)回收PCR产物。
1.2.3 PCR产物克隆测序 将纯化后的PCR产物连接于PMD18-T载体上,转换至DH5α感受态细胞中,涂平板进行蓝白斑筛选,挑取阳性克隆菌落,在LB液体培养基中摇菌培养,而后送交北京百泰克生物技术有限公司进行测序。
1.2.4 序列分析 将测序结果用DNAMAN V6.0进行比对,结合测序波峰图确定所测序列的质量。如果测序结果不好,需重新测序。利用GenBank中的Blast程序确定所测序列的准确性。利用Clustal W程序比对分析2种豆象的基因序列,采用MEGA V5.0分析序列间的进化距离、转换率和颠换率。利用MEGA V5.0构建NJ树和ML树,Bootstrap值为 1 000 。构建NJ树时,利用Tamura三参数方法。构建ML树时,采用“GTR+G+I”模型。分析序列时,参考的物种有:Acanthoscelidesoblongoguttatus(AB499967,AY945981),Bruchidiusauratopubens(AY625379,AY625282),Bruchidiusrubicundus(AY625394,AY625298),Bruchidiussubmaculatus(AY625397,AY625301)(种名后括号中的代码分别表示COX1和12SrRNA基因的GenBank登录号)。
2 结果与分析
2.1 目的基因的扩增与克隆
PCR扩增产物经1%琼脂凝胶电泳,分别得到约660 bp和580 bp的扩增条带。阳性克隆经菌液PCR扩增也得到了660 bp和580 bp左右的扩增条带,说明已成功扩增并克隆了2种豆象的COX1和12SrRNA基因序列。
2.2 2种豆象线粒体COX1和12SrRNA基因的测序分析
2.2.1COX1和12SrRNA基因的碱基组成特征 测序结果表明,所测银合欢豆象与合欢豆象COX1基因的片段大小均为660 bp,与预期的结果一致。合欢豆象COX1的基因片段中,A占29.4%,T占38.0%,G占16.5%,C占16.1%,A+T含量为67.4%。银合欢豆象COX1基因片段中,A占29.4%,T占33.9%,G占18.0%,C占18.6%,A+T含量为63.3%。2种豆象COX1基因片段的A、T含量与昆虫线粒体基因富含A、T碱基的组成特征基本一致,也与豆象科其他昆虫的碱基组成相一致,如:A.oblongoguttatusCOX1基因的A+T含量为66.7%,B.submaculatusCOX1基因的A+T含量为67.3%。2种豆象COX1基因片段间的相似度为83.5%。
所测银合欢豆象12SrRNA基因片段的大小为579 bp,合欢豆象12SrRNA基因片段的大小为576 bp。银合欢豆象12SrRNA基因片段中A占41.8%,T占36.8%,G占7.6%,C占13.8%,A+T含量为78.6%。合欢豆象12SrRNA基因片段中A占43.1%,T占35.8%,G占8.7%,C占12.5%,A+T含量为78.9%。2种豆象12SrRNA基因片段的A、T含量也与昆虫线粒体基因富含A、T碱基的组成特征一致。2种豆象12SrRNA基因片段间的相似度为88.3%。
2.2.2COX1和12SrRNA基因的进化信号 为了进一步研究2种豆象与其他物种间的进化关系,本研究对COX1和12SrRNA基因的进化距离进行了分析,结果见表1。

表1 COX1和12S rRNA基因的进化距离
注: 1为A.macrophthalmus(银合欢豆象);2为A.oblongoguttatus;3为B.auratopubens;4为B.rubicundus;5为B.submaculatus;6为B.terrenus(合欢豆象)。左下方数值表示Kimura双参数距离,右上方数值表示p距离。
基于COX1基因的进化距离而言,银合欢豆象与合欢豆象间的进化距离最远(0.208,0.161),与A.oblongoguttatus的进化距离最近(0.192,0.152)。就12SrRNA基因而言,银合欢豆象与B.auratopubens和B.Rubicundus的进化距离最远,与A.oblongoguttatus的进化距离最近。由此可见,利用不同单基因来分析进化关系时,会产生矛盾的结论,需要参考多方面因素进行综合考虑。
基于COX1基因的进化距离而言,合欢豆象与银合欢豆象的进化距离最远(0.208,0.161),与B.auratopubens的进化距离最近(0.116,0.100)。就12SrRNA基因而言,合欢豆象与A.oblongoguttatus的进化距离最远(0.114,0.098),与B.auratopubens的进化距离最近(0.060,0.056)。
为了进一步明确2种豆象的进化方式,本研究对COX1和12SrRNA基因的碱基转换和颠换关系进行分析,结果见表2。
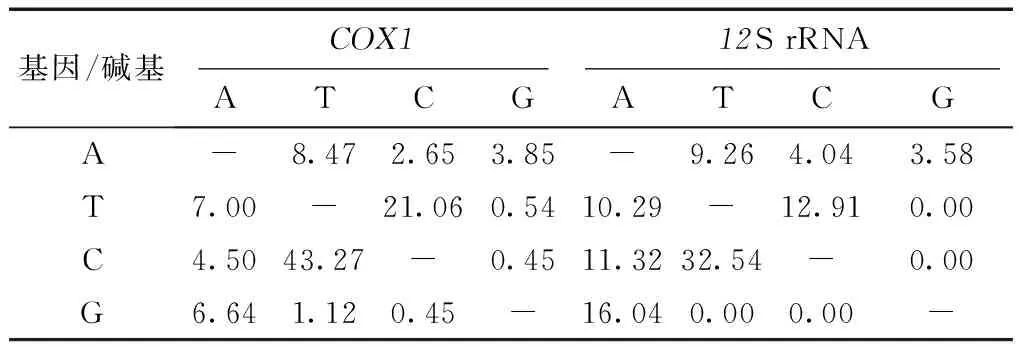
表2 COX1和12S rRNA基因碱基的转换关系
从表2可以看出:无论COX1还是12SrRNA基因,碱基C和T间的转换率均最高,C→T的转换率分别达43.27%和32.54%,T→C的转换率分别达21.06%和12.91%。碱基A和T间的颠换率较高,T→A的颠换率分别达7.00%和10.29%,A→T的颠换率分别为8.47%和9.26%。
2.2.3COX1基因的密码子使用偏向性COX1基因密码子的使用偏向性见表3。

表3 COX1基因中密码子的使用偏向性
注:表中括号前的数据表示银合欢豆象COX1基因中的密码子数量;括号中的数据表示合欢豆象COX1基因中的密码子数量。
从表3可看出:对于银合欢豆象和合欢豆象来说,其COX1基因对密码子的使用具有不同的偏向性,但有共同特征,即四联、六联和双重密码子均偏向于使用以A或T结尾的密码子。银合欢豆象COX1基因中共有29个亮氨酸密码子,其中UUA有14个,CUA有7个,CUU有5个,而以C或G结尾的密码子均为1个。同样,合欢豆象COX1基因中共有30个亮氨酸密码子,其中TTA有21个,CTA有3个,CTT有4个,而以C或G结尾的密码子占的比重很低。对于双重密码子亦有一致的使用偏向性,如苯丙氨酸密码子偏向于使用AUA。
2.3 系统发育树的构建与分析
通过系统发育树(图1)可以看出,基于COX1基因构建的NJ树(图1 A)和ML树(图1 B)以及基于12SrRNA基因构建的NJ树(图1 C)和ML树(图1 D)均有类似的发育树构型。银合欢豆象与A.oblongoguttatus有最近的进化距离,聚在同一大分支上,而合欢豆象与多型豆象属聚在一个大的分支上。该结果与形态学的分类结果完全一致。由于在GenBank中缺乏更多的核苷酸数据,银合欢豆象与合欢豆象更精确的系统发育地位还有待于进一步完善。

3 讨论
3.1 2种豆象COX1和12SrRNA基因的进化特征
以往研究表明,昆虫线粒体基因组具有高A+T含量的特征[10],对线粒体基因的判断具有重要意义。本研究对银合欢豆象与合欢豆象的COX1和12SrRNA基因进行了测序分析,结果显示,2种豆象COX1基因中A+T含量约为67%,12SrRNA基因的A+T含量约为79%。COX1基因的A+T含量低于12SrRNA基因,但明显高于绝大部分核基因的含量。与鞘翅目豆象科的其他成员相比,银合欢豆象和合欢豆象2种基因也具有相似的碱基组成特征,这在一定程度上佐证了昆虫线粒体基因碱基组成的特点[10]。
在系统发育关系方面,进化信号的分析可进一步明确基因进化的方式。本研究通过p距离和Kimura双参数距离的分析表明:基于COX1基因分析时,银合欢豆象与合欢豆象有最远的进化距离;而基于12SrRNA基因分析时,得到了与COX1基因分析不一致的结果。尽管如此,但也有合理的解释。不管是核基因组还是线粒体基因组,其每个基因或一个基因的不同区段可能有不同的进化速率,有不同的进化模式[11]。如在线粒体基因组中,COX1的进化速率最慢,而AT富含区(控制区)的进化速率较快;在12SrRNA基因中,功能区的进化速率较慢,呈中性进化速率,而在其他区域的变异较大[11]。由此可见,采用不同的基因进行进化分析时,因基因进化速率不一,会产生不一致的结果。这需要结合多个基因,或联合核基因与线粒体基因进行分析,才有可能得出更加准确的结果。
研究表明,昆虫线粒体基因偏向使用以A或T结尾的密码子,这与其基因组的高A+T含量有直接关系[10-11]。本研究中,银合欢豆象与合欢豆象的线粒体COX1和12SrRNA基因也偏向使用以A或T结尾的密码子,也具有较高的A+T含量,这与以往报道的其他昆虫结果相一致。该特征还可从另一角度得到反映,即2种基因中C→T的转换率很高。虽然2种基因中有较高的转换率和颠换率,但其相应的氨基酸序列却有几个氨基酸的突变,说明大部分碱基突变属于同义突变。
3.2 系统发育分析
本研究显示,银合欢豆象与A.oblongoguttatus有最近的进化距离,而合欢豆象与多型豆象属聚在一个大的分支上,与形态学的分类结果基本一致[1,2,4-5]。然而,在GenBank中缺乏更多核苷酸数据,有些虫种有部分COX1基因序列,但没有相对应的12SrRNA基因序列,因而,难以达到本文的分析需求(同时具有COX1和12SrRNA基因序列)。银合欢豆象与合欢豆象更精确的系统发育地位还有待于进一步完善。
综上所述,银合欢豆象和合欢豆象的线粒体COX1和12SrRNA基因具有昆虫线粒体基因的组成特征,具有与其他昆虫线粒体基因相类似的进化特征。目前,GenBank数据库中豆象线粒体的核苷酸数据仍较缺乏,这需要对多种豆象的线粒体基因进行测序分析,以便更精确地掌握每个物种的系统发育地位。银合欢豆象和合欢豆象线粒体COX1和12SrRNA基因的测序分析,为2种豆象的地理种群、系统发育、遗传多样性等研究奠定了理论基础。
[1] Wu L H, Wang C P, Wu W J. Description and differentiation of the four larval instars ofAcanthoscelidesmacrophthalmus(Coleoptera: Chrysomelidae: Bruchinae)[J]. Annals of the Entomological Society of America, 2012, 105(2): 259-267.
[2] 孟向东.合欢豆象的生活习性观察[J].昆虫知识,1992,29(5):271-272.
[3] Hizal E, Parlak N N.BruchidiusterrenusandBruchidiussiliquastri(Coleoptera: Chrysomelidae: Bruchinae)-first records for Turkey[J]. Florida Entomologist, 2013, 96(1): 66-70.
[4] 覃新导,周祥,王涛,等.银合欢豆象生物学特性及其防治[J].热带作物学报,2011,32(12):2340-2345.
[5] Sharratt M E J, Olckers T. The biological control agentAcanthoscelidesmacrophthalmus(Chrysomelidae:Bruchinae)inflicts moderate levels of seed damage on its target,the invasive treeLeucaenaleucocephala(Fabaceae),in the KwaZulu-Natal Coastal Region of South Africa[J]. African Entomology, 2012, 20(1): 44-51.
[6] Zhu-Salzman K, Zeng R S. Molecular mechanisms of insect adaptation to plant defense:lessons learned from a Bruchid beetle[J]. Insect Science, 2008, 15(6): 477-481.
[7] Wu L H, Wang C P, Wu W J. Effects of temperature and adult nutrition on the development ofAcanthoscelidesmacrophthalmus,a natural enemy of an invasive tree,Leucaenaleucocephala[J]. Biological Control, 2013, 65(3): 322-329.
[8] 张生芳,覃新导.三齿豆象属几种银合欢豆象的识别[J].植物检疫,2007,21(6):353-355.
[9] Simon C, Buckley T R, Frati F, et al. Incorporating molecular evolution into phylogenetic analysis,and a new compilation of conserved polymerase chain reaction primers for animal mitochondrial DNA[J]. Annual Review of Ecology, Evolution, and Systematics, 2006, 37: 545-579.
[10] Sheffield N C, Song H, Cameron S L, et al. A comparative analysis of mitochondrial genomes in Coleoptera (Arthropoda: Insecta) and genome descriptions of six new beetles[J]. Molecular Biology and Evolution, 2008, 25(11): 2499-2509.
[11] Shao R, Dowton M, Murrell A, et al. Rates of gene rearrangement and nucleotide substitution are correlated in the mitochondrial genomes of insects[J]. Molecular Biology and Evolution, 2003, 20(10): 1612-1619.
(责任编辑 张 坤)
Cloning and Sequence Analysis onCOX1 and 12SrRNAGenes of Two Seed Beetle Species
WU Pei-fu1, TONG You-gui2, WANG Lin3, PAN Yong-zhi4, XIONG Zhong-ping4, DOU Bao-kun4
(1.College of Life Sciences, Southwest Forestry University, Kunming Yunnan 650224, China;2. Forest Disease and Insect Pest Control and Quarantine Station of Dongchuan District, Kunming Yunnan 654100, China;3. Yunnan Vocational College of Forestry , Kunming Yunnan 650224, China;4. College of Forestry, Southwest Forestry University, Kunming Yunnan 650224, China)
The genome DNA was extracted respectively fromAcanthoscelidesmacrophthalmusandBruchidiusterrenus, and theCOX1 and 12SrRNAgenes were sequenced and analyzed to establish the basis for the further molecular study. The results showed thatCOX1 and 12SrRNAof the two beetle species presented higher content of A+T, nucleotide C and T showed higher conversion rate. The two genes had codon usage bias as using codons with the third base A or T. It was showed thatAcanthoscelidesmacrophthalmusshared a longer distance withBruchidiusterrenus, but a shorter distance withAcanthoscelidesoblongoguttatus. While,Bruchidiusterrenuswould be grouped into the same branch of the genusBruchidius. However, the more accurate phylogenetic roles ofAcanthoscelidesmacrophthalmusandBruchidiusterrenusneed to be further studied due to limited nucleotide data in GenBank.
Acanthoscelidesmacrophthalmus;Bruchidiusterrenus;COX1; 12SrRNA; sequencing analysis
2013-09-09
东川区干热河谷主要树种三种害虫发生规律和防治技术研究项目资助;云南省重点学科建设项目(XKZ200905)资助。
潘涌智(1952—),男,硕士,教授。研究方向:种实害虫,森林昆虫。Email:pyz525@126.com。
10.3969/j.issn.2095-1914.2014.02.009
S718.7
A
2095-1914(2014)02-0044-05
第1作者:吴培福(1977—),男,博士,副教授。研究方向:动物进化及疫源疫病。Email:jed-wu2008@126.com。
