膜性肾病差异性表达microRNA的靶基因功能分析
2014-07-03陈文标黄建溶
陈文标, 黄建溶, 戴 勇
(暨南大学附属第二医院,深圳市人民医院,临床医学研究中心,广东 深圳 518020)
膜性肾病(membranous nephropathy,MN)是成年肾病综合征的常见病因之一,国外报道占成年人原发性肾病综合征25% ~40%[1].MN按发病病因可以分为特发性与继发性,特发性膜性肾病病因不明确,多认为是免疫机制有关.继发性膜性肾病常与药物、狼疮及肝炎等有关[2].但是MN的病因尚不清楚,抗体介导的免疫复合物形成启动肾脏组织损伤是主要机制[3].其病理改变是免疫复合物沿肾小球基底膜外侧沉积,形成基底膜“钉突狀”增厚为特征[4].MN是自身免疫性疾病,临床上治疗仍然是采用免疫抑制治疗,但是治疗上缺乏特异性,即便是免疫抑制剂合理使用,常常很难掌握用药度量及缺乏特异性监控标志物.病人在治疗后,有的病情得到缓解,但是带来巨大的副作用.有的病情加深,需要加大剂量[5].为了对MN有更深刻的了解,以期望真正研究MN发病机制,才能更好的指导临床诊断与治疗.本研究从基因层面对MN的差异性表达microRNA及其靶基因功能分析进行探讨,可能为研究MN的病因机制打开新的思路与方法.MicroRNA是一类内生的长度约为20~24nt的小RNA,其在细胞的生长发育,增殖与死亡起着重要的作用[6].最近研究表明每一个microRNA有多个靶基因,多个microRNA可以组合起来精细调控某个基因表达,人类三分之一的基因调控与microRNA有关.所以microRNA在疾病发病起着重要的调控作用.近年来microRNA已经被充分应用到肾脏疾病研究中,且都取得实质性成果[7].例如,MicroRNA-29b的显著性表达可以抑制糖尿病肾病[8];microRNA-223在内皮细胞的表达水平可以作为一种无创的检查用于评估IgA肾病的病情[9];microRNA-443的上调表达常常与肾脏纤维化有着密切联系[10].本研究是在前期的研究基础上,找到MN与正常者之间差异性表达microRNA,运用来源于microRNA的靶基因深入研究MN的生物学功能.靶基因富集的条目与参与的通路可能在一定程度阐明MN的病因.
1 材料与方法
1.1 标本收集
分别抽取MN与健康对照组(NC)静脉全血10 mL,100例MN为2013年深圳市人民医院诊断为特发性膜性肾病患者,经肾脏穿刺检查确定为膜性肾病Ⅲ期,且患者在确定诊断之前没有使用过任何激素与免疫抑制剂.选取100例深圳市人民医院常规体格检查排除肾脏疾病作为健康对照组.100例NC被随机分成10组(NC1~10),每组10人.100例MN被随机分成10组(MN1~10),每组10人.本研究得到深圳市人民医院伦理委员批准,且征得实验参与者的知情同意.
1.2 单个核细胞的分离与总RNA提取
收集到静脉全血10 mL,于采血后1 h内按照淋巴细胞分离液(Ficoll-Paque,PLUS)实验说明手册,使用淋巴细胞分离液(Cedarlane,USA)分离外周血单个核细胞(PBMCs).分离得到的PBMCs按照Trizol(Invitrogen,USA)试剂盒说明书提取总RNA,经Bioanalyer分析仪(Santa clare,CA,USA)检测,满足RNA完整指数(R/N)≥8.0,rRNA比值(28S:18S)≥1,用于小RNA数据库建立.
1.3 小RNA的分离机RNA数据库建立
根据不同样品所测得到的总RNA浓度,每组间各种样品等量混合,即得到20组样品,分别为MN1~10与NC1~10组等质量的总RNA.利用质量分数为15%聚丙烯酰胺凝胶(PAGE)电泳分离总RNA,参照寡核苷酸标志在胶中所处的位置,回收并纯化18~30nt范围的小RNA.运用HiSeq 2000高通量Solexa测序平台(华大基因,深圳)对小RNA进行深度测序.在测序之前,小RNA通过5'接头连接与纯化,3'接头连接与纯化,RT-PCR扩增等步骤制备成CDNA文库.CDNA片段结合在含有接头的芯片上,加入荧光标志Dntp,聚合物与接头引物,经反应扩增,dNTP在参与互补链的系列延伸会释放出荧光,每种碱基具有特定的波才长,荧光信号被测序仪器采集,并用软件转换成测序峰,最终得到各序列的测序信息.
1.4 测序数据的初步分析
HiSeq 2000测序所得到的的为50nt长度序列,通过去接头,去低质量,去污染等过程完成数据处理得到干净序列.统计小RNA的种类及数量,对其进行序列长度分布统计与样品公共序列统计.然后使用SOAP软件(华大基因,深圳)将干净序列RNA与人类基因组序列(NCBI Gene Bank)(http://www.ncbi.nlm.nih.gov/genbank/)进行比对,从而得到已知rRNA,scRNA,snoRNA,snRNA,piRNA,tRNA,mRNA降解片段,重复序列等在所得序列中所占的比例.再将比对后的small RNA序列进一步与microRNA数据库(miRBase 18)(http://www.mirbase.org/)中人的micrRNA序列比对,获取样品中已知micrRNA含量,表达丰度等信息.测序数据分析流程,图1.
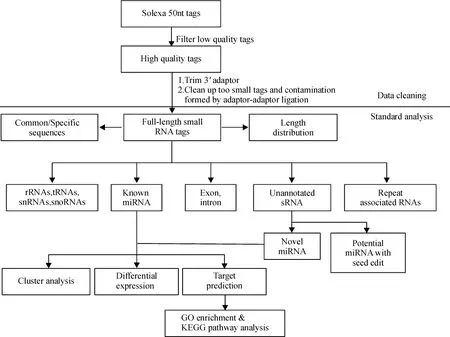
图1 高通量测序分析流程Fig.1 The procedure of analysis by high-throughput sequencing
1.5 样本间micrRNA差异性表达鉴定
为了比较MN1~10与NC1~10组之间差异性表达microRNA,首先应将样本microRNA的表达量归一化到同一数量级,归一化表达量=(某个microRNA的表达量/该样品总表达量)×106.归一化结果中,如果两个样品中某一个表达量为0,则修正为0.01;如果两样品归一化结果都小于1,则这个microRNA不满足数据要求,不能用于差异性表达分析.样品之间microRNA差异性表达用倍比值计算,倍比值=log2(实验组归一化表达量/对照组归一化表达量).当倍比值>0时,表达上调;当倍比值<0时,表达下调.其外,引用P-value值用于分析基因表达谱与避免低表达量microRNA造成数据误差,满足条件P-value≤0.01时,microRNA具有显著差异性表达.
1.6 差异性表达microRNA的靶基因预测及功能分析
借用 TargetScan软件(http://www.targetscan.org/)对差异性表达microRNA的靶基因进行预测,并选取相关靶基因运用DAVID功能注释软件(http://david.abcc.ncifcrf.gov/)向 Gene Ontology 数据库(http://www.geneontology.org/)各个条目映射.计算映射到每一个条目靶基因数目,然后应用超几何检验,找出与整个参考基因背景相比,在靶基因显著富集GO条目.GO富集包括细胞组成(cellular component),分子功能(molecular function)及生物学过程(biological process)三方面内容.KEGG通路分析也是针对靶基因,在生物体内,不同靶基因相互协调行使生物学功能,基于KEGG通路分析有助于更进一步了解基因生物学功能.把靶基因向KEGG数据库(http://www.genome.jp/kegg/)映射,方法与GO富集基本相同.GO富集与KEGG通路检验时,当P-value≤0.05可以确定靶基因显著富集GO功能条目及主要KEGG信号通路.
2 结果
2.1 microRNA的差异性表达
在MN1~10与NC1~10差异性表达microRNA中,有意义的microRNA数量分别为MN1~NC1/440个,MN2~NC2/355个,MN3~NC3/342个,MN4~NC4/351个,MN5~NC5/351个,MN6~NC6/329个,MN7~NC7/329个,MN8~NC8/319个,MN9~NC9/319个,MN10~NC10/337个.分别选取各组最具有差异性表达microRNA前5个上调与下调表达microRNAs,见表 1.其中 has-miR-192-2P作为下调表达microRNA在10个组别出现;has-miR-208b作为上调表达microRNA与has-miR-217,has-miR-23b-5p作为下调表达microRNAs在7个组别出现;hasmiR-95作为下调表达microRNA与has-miR-486-5P作为上调表达microRNA在6个组别出现;has-miR-95作为下调表达microRNA在8个组别出现.

表1 MN与NC1~10差异性表达microRNAsTable1 The differently expressed microRNA between MN and NC1~10 groups

microRNA-name NC(standard expression)MN(standard expression)fold-change(log2 MN/NC) p-value up-regulated/down-regulated hsa-miR-1468 0.01 9.09 9.83 0.00 up hsa-miR-136-3p 0.01 6.12 9.26 0.00 up hsa-miR-449a 0.01 4.14 8.69 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-486-5p 64.24 13 344.79 7.70 0.00 up hsa-miR-215 4 028.37 1.71 -11.20 0.00 down hsa-miR-503 31.16 0.01 -11.61 0.00 down hsa-miR-23b-5p 57.30 0.01 -12.48 0.00 down hsa-miR-217 125.37 0.01 -13.61 0.00 down hsa-miR-320c 216.04 0.01 -14.40 0.00 down MN4~NC4 hsa-miR-208b 0.01 4.86 8.93 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-449c-5p 0.01 1.98 7.63 0.00 up hsa-miR-5683 0.01 1.53 7.26 0.00 up hsa-miR-19a-3p 0.37 51.31 7.10 0.00 up hsa-miR-503 32.30 0.01 -11.66 0.00 down hsa-miR-23b-5p 40.21 0.01 -11.97 0.00 down hsa-miR-4508 41.10 0.01 -12.01 0.00 down hsa-miR-4286 49.08 0.01 -12.26 0.00 down hsa-miR-217 52.96 0.01 -12.37 0.00 down MN5~NC5 hsa-miR-381 0.01 8.19 9.68 0.00 up hsa-miR-509-3p 0.09 40.96 8.78 0.00 up hsa-miR-449a 0.01 4.14 8.69 0.00 up hsa-miR-19a-3p 0.19 51.31 8.10 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-1291 50.13 0.01 -12.29 0.00 down hsa-miR-193a-5p 54.05 0.01 -12.40 0.00 down hsa-miR-483-5p 166.06 0.01 -14.02 0.00 down hsa-miR-129-2-3p 204.70 0.01 -14.32 0.00 down hsa-miR-23b-5p 267.71 0.01 -14.71 0.00 down MN6~NC6 hsa-miR-486-5p 26.56 13 344.79 8.97 0.00 up hsa-miR-208b 0.01 4.86 8.93 0.00 up hsa-miR-133a 0.69 306.05 8.80 0.00 up hsa-miR-449a 0.01 4.14 8.69 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-671-5p 22.19 0.01 -11.12 0.00 down hsa-miR-95 30.00 0.01 -11.55 0.00 down hsa-miR-216b 38.12 0.01 -11.90 0.00 down hsa-miR-216a 66.68 0.01 -12.70 0.00 down hsa-miR-217 191.86 0.01 -14.23 0.00 down MN7~NC7 hsa-miR-486-5p 26.13 13 344.79 9.00 0.00 up hsa-miR-208b 0.01 4.86 8.93 0.00 up

microRNA-name NC(standard expression)MN(standard expression)fold-change(log2 MN/NC) p-value up-regulated/down-regulated hsa-miR-449a 0.01 4.14 8.69 0.00 up hsa-miR-133a 0.81 306.05 8.56 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-503 26.94 0.01 -11.40 0.00 down hsa-miR-95 30.69 0.01 -11.58 0.00 down hsa-miR-216b 34.75 0.01 -11.76 0.00 down hsa-miR-216a 38.56 0.01 -11.91 0.00 down hsa-miR-217 64.38 0.01 -12.65 0.00 down MN8~NC8 hsa-miR-1468 0.01 9.09 9.83 0.00 up hsa-miR-486-5p 26.05 13 344.79 9.00 0.00 up hsa-miR-208b 0.01 4.86 8.93 0.00 up hsa-miR-133a 0.84 306.05 8.51 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-514a-5p 9.95 0.01 -9.96 0.00 down hsa-miR-503 13.57 0.01 -10.41 0.00 down hsa-miR-3653 14.09 0.01 -10.46 0.00 down hsa-miR-95 19.46 0.01 -10.93 0.00 down hsa-miR-1246 36.33 0.01 -11.83 0.00 down MN9~NC9 hsa-miR-1468 0.01 9.09 9.83 0.00 up hsa-miR-486-5p 26.05 13 344.79 9.00 0.00 up hsa-miR-208b 0.01 4.86 8.93 0.00 up hsa-miR-133a 0.84 306.05 8.51 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-514a-5p 9.95 0.01 -9.96 0.00 down hsa-miR-503 13.57 0.01 -10.41 0.00 down hsa-miR-3653 14.09 0.01 -10.46 0.00 down hsa-miR-95 19.46 0.01 -10.93 0.00 down hsa-miR-1246 36.33 0.01 -11.83 0.00 down MN10~NC10 hsa-miR-486-5p 23.02 13 344.79 9.18 0.00 up hsa-miR-133a 0.89 306.05 8.43 0.00 up hsa-miR-195-3p 0.01 2.25 7.81 0.00 up hsa-miR-449c-5p 0.01 1.98 7.63 0.00 up hsa-miR-133b 0.14 9.99 6.20 0.00 up hsa-miR-216b 13.69 0.01 -10.42 0.00 down hsa-miR-95 19.55 0.01 -10.93 0.00 down hsa-miR-216a 34.46 0.01 -11.75 0.00 down hsa-miR-503 36.43 0.01 -11.83 0.00 down hsa-miR-217 104.33 0.01 -13.35 0.00 down
2.2 差异性表达microRNAs靶基因的GO富集分析
GO富集的细胞组成(cellular component)分析中,选取靶基因主要参与的前3个富集条目,见表2.从表2可以看出,差异性表达microRNAs的靶基因主要参与细胞的细胞器与细胞膜的合成.其中细胞膜(membrance)条目在7个组别出现;细胞器官(cell part)条目在6个组别出现.对于差异性表达microRNAs靶基因参与分子功能(molecular func-tion)分析中,表3.靶基因主要参与酶催化,离子结合,分子转导活性.其中离子结合(ion binding)在8个组别出现.而差异性表达microRNAs靶基因参与生物学过程(biological process)分析中,表4.靶基因主要参与细胞代谢过程,生物代谢过程.其中生物代谢过程(biopolymer metabolic process)在5个组别出现;生物调节过程(biological regulation)在3个组别出现.

表2 GO富集的细胞组成分析Table2 Cellular component analysis of GO enrichment

表3 GO富集分子功能分析Table3 Molecular function analysis of GO enrichment
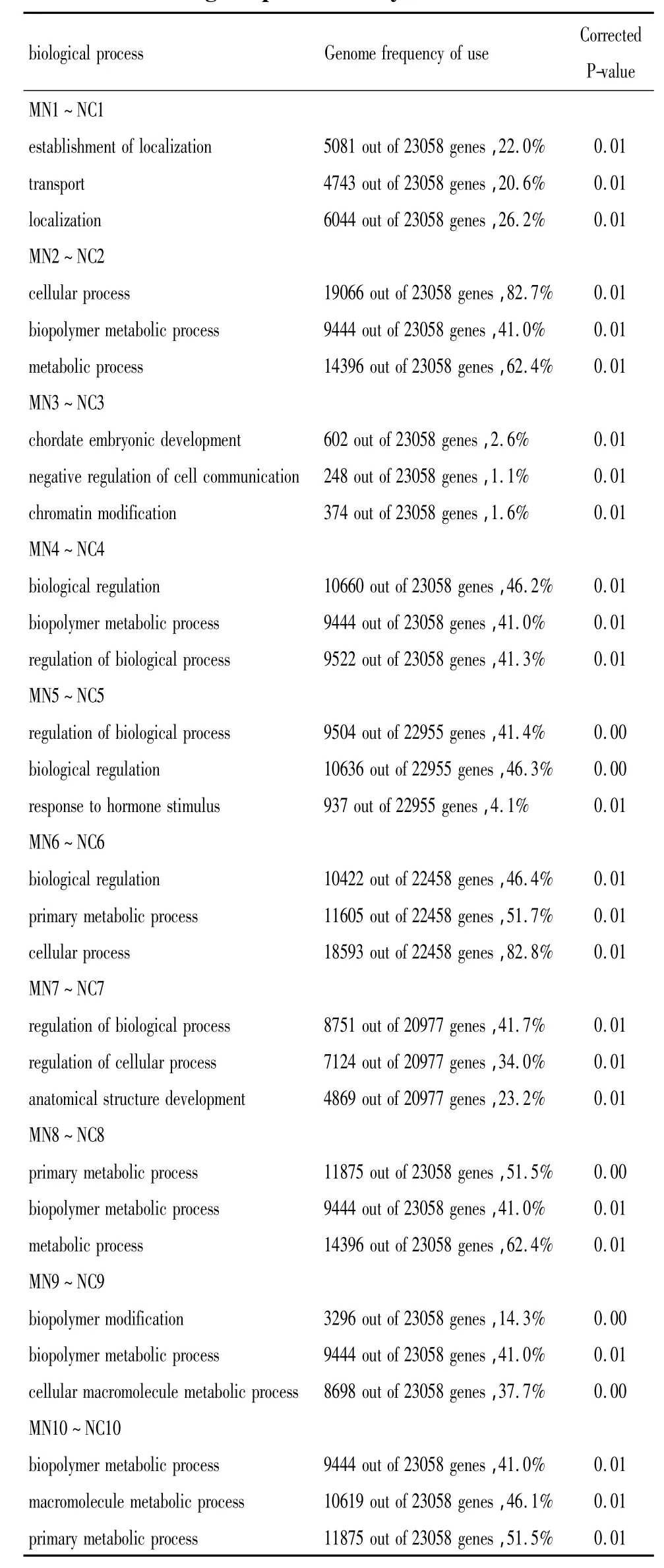
表4 GO富集的生物学过程分析Table4 Biological process analysis of GO enrichment
2.3 差异性表达microRNA靶基因的KEGG分析
分别选取MN1~10与NC1~10差异性表达microRNAs的靶基因主要参与的前3个KEGG通路条目,见表5.从表5可以发现靶基因主要参与代谢通路,癌症通路,MAPK信号通路.其中嘌呤代谢通路(purine metabolism)在6个组别出现;生物合成代谢通路(biosynthesis of secondary)在5个组别出现;癌症通路(pathways in cancer)在6个组别出现;MAPK信号通路(MAPK signaling pathway)在4个组别出现.

表5 差异性表达mciroRNAs靶基因的KEGG通路分析Table5 The differently expressed microRNAs target gene analysis in KEGG pathway
3 讨论
高通量测序平台已经成为基因表达与转录组分析的重要手段,测序平台的高效性与准确性能在短时间产生海量数据,数据可以进行针对性的生物信息分析.高通量测序技术平台为基因组学与其他的交叉学科发展做出了巨大贡献,起着里程碑的意义[11].本实验的研究技术是基于高通量测序平台先进技术,准确找到 MN与NC差异性表达microRNAs.在差异性表达microRNA中,下调表达microRNAs数量要比上调表达microRNAs要多.并且下调表达microRNAs的倍比值要比上调表达的microRNAs要大.但是,microRNAs在许多疾病的研究中发现,上调表达的microRNA的数量要比下调表达要多.许多学者认为microRNA失去监控而过度表达是引起疾病发生的重要原因[12].本课题组在先前的研究发现狼疮肾炎与正常者的mciroRNA比对中,上调表达 mciroRNAs的数量要比下调的多[13].在microRNA作为疾病诊断标志物研究发现,系统性红斑狼疮患者与类风湿关节炎患者相比正常者,microRNA表现明显的上调表达[14].在硬骨化病患者的蛋白质与microRNA关联分析中发现,上调表达的mciroNA有63个,而下调表达的mciroRNA有60个[15].而本研究结果是下调表达microRNAs数量要比上调表达多,并且下调表达microRNAs的倍比值要比上调表达的microRNAs要大.因此,猜测这些差异性表达microRNA在MN的发病机理中起着抑制作用,microRNA的低表达或失表达导致转录水平蛋白质的丢失有关.Jacqueline Ho认为miR-23b,miR-24与miR-26b的大量丢失与肾小球与肾小管损伤有关,它们的存在维持着肾小球率过滤[16].对于本研究发现的大量microRNA的下调表达是否与microRNA的大量丢失有关,需要进一步探讨.
在GO靶基因富集分析中,靶基因在细胞组成当中主要参与细胞膜组成条目中,其中细胞膜(membrance)条目在7个比较组别中,部分膜(membrance part)在5个比较组别中.MN的病理改变是肾小球弥慢性病变,基底膜逐渐增厚,免疫复合物沿肾小球毛细血管壁沉积,而肾小球滤过膜上皮侧有电子致密物质伴有广泛足突融合[17].因此,猜测这些差异性表达microRNAs的靶基因主要参与到细胞的细胞膜组成中,这是否是引起MN基底膜增厚,滤过膜广泛足突融合有关,靶基因是否存在异常的表达而导致基底膜细胞的过度增生.但在本研究中的试验对象没有定位到肾小球的滤过膜,差异性表达的microRNA靶基因参与细胞膜组成也不一定特指虑过膜的内皮细胞,基底膜与脏层上皮细胞.所以这也需要深入探讨.在后期的工作中,以肾脏组织为试验标本也许能得出更准确的实验结果.
在KEGG靶基因通路分析中,靶基因主要参与嘌呤代谢通路,MAPK信号通路.嘌呤代谢紊乱是引起高尿酸血症与痛风的主要原因,而MN的临床表现却少有报道高尿酸与痛风.但是研究报道发现嘌呤代谢紊乱与肾小球纤维化,肾小管率过滤降低有关,长期升高尿酸是肾功能与肾衰竭恶化的一个重要指标[18].假设嘌呤代谢途径是否在将来能成为监控MN病情改变指标,能够在MN出现临床症状之前发现肾小球的微小病理改变.MAPK信号通路是生物体内重要的信号传导系统,在细胞生长,发育,分裂与分化等多种生理病理过程中起着重要作用,特别是在细胞周期运行与基因表达起着重要调控作用[19].MAPK在疾病的炎症,免疫,肿瘤等都参与重要信号传导.据研究,MAPK信号通路活性可以作为肾脏疾病潜在机制,如果MAPK通路得到适当抑制,有利于肾脏疾病的恢复[20].
综上所述,本研究在发现差异性表达microRNAs靶基因,用于分析靶基因的生物信息功能.以期望能更深入研究MN的发病机制原理,为研究MN的病因提供新的思路方法.但是本研究是基于前期大量的试验基础与查阅大量相关文献做出的实验结论,仍有很多不明确地方,这就需要在下一步的研究中继续深入的探讨.
[1] LI L S,LIU Z H.Epidemiologic data of renal diseases from a single unit in China:analysis based on 13,519 renal biopsies[J].Kidney Int,2004,66(3):920-923.
[2] COUSER W G.Membranous nephropathy:a long road but well traveled[J].J Am Soc Nephrol,2005,16(5):1184-1187.
[3] GLASSOCK R J.The pathogenesis of membranous nephropathy:evolution and revolution[J].Curr Opin Nephrol Hypertens,2012,21(3):235-42.
[4] TAGUCHI T,OYAMADA M,HARADA T.[Pathology of membranous nephropathy][J].Nihon Jinzo Gakkai Shi,2011,53(5):684-91.
[5] STAHL R A,HOXHA E,HELMCHEN U.Membranousglomerulonephritis:better therapy with autoantibody monitoring?[J].Dtsch Med Wochenschr,2011,136(34-35):1733-1737.
[6] ZHANG B,WANG Q,PAN X.MicroRNAs and their regulatory roles in animals and plants[J].J Cell Physiol,2007,210(2):279-289.
[7] CHUNG A C,YU X,LAN H Y.MicroRNA and nephropathy:emerging concepts[J].Int J Nephrol Renovasc Dis,2013,6:169-179.
[8] CHEN H Y,ZHONG X,HUANG X R,et al.MicroRNA-29b Inhibits Diabetic Nephropathy in db/db Mice[J].Mol Ther,2014,22(4):842-853.
[9] BAO H,CHEN H,ZHU X,et al.MiR-223 downregulation promotes glomerular endothelial cell activation by upregulating importin alpha4 and alpha5 in IgA nephropathy[J].Kidney Int,2014,85(3):624-635.
[10] KATO M.TGF-beta-induced signaling circuit loops mediated by microRNAs as new therapeutic targets for renal fibrosis?[J].Kidney Int,2013,84(6):1067-1069.
[11] SOON W W,HARIHARAN M,SNYDER M P.Highthroughput sequencing for biology and medicine[J].Mol Syst Biol,2013,9:640.
[12] VASUDEVAN S.Posttranscriptional upregulation by microRNAs[J].Wiley Interdiscip Rev RNA,2012,3(3):311-330.
[13] CHEN W B,TAN K T,HUANG J R,et al.Analysis of microRNAs in patients with systemic lupus erythematosus,using Solexa deep sequencing[J].Connect Tissue Res,2014,55(3):187-196.
[14] WANG H L,PENG W J,OUYANG X,et al.Circulating microRNAs as candidate biomarkers in patients with systemic lupus erythematosus[J].Transl Res,2012,160(3):198-206.
[15] OU M L,ZHANG X,DAI Y,et al.Identification of potential microRNA-target pairs associated with osteopetrosis by deep sequencing,iTRAQ proteomics and bioinformatics[J].Eur J Hum Genet,2014,22(5):625-632.
[16] HO J,NG KH,ROSEN S,et al.Podocyte-specific loss of functional microRNAs leads to rapid glomerular and tubular injury[J].J Am Soc Nephrol,2008,19(11):2069-2075.
[17] KAVOURA E,GAKIOPOULOU H,PARASKEVAKOU H,et al.Immunohistochemical evaluation of podocalyxin expression in glomerulopathies associated with nephrotic syndrome[J].Hum Pathol,2011,42(2):227-235.
[18] KANG D H,NAKAGAWA T.Uric acid and chronic renal disease:possible implication of hyperuricemia on progression of renal disease[J].Semin Nephrol,2005,25(1):43-49.
[19] MARTINEZ-LOPEZ N,SINGH R.ATGs:Scaffolds for MAPK/ERK signaling[J].Autophagy,2014,10(3):535-537.
[20] PENGAL R,GUESS A J,AGRAWAL S et al.Inhibition of the protein kinase MK-2 protects podocytes from nephrotic syndrome-related injury[J].Am J Physiol Renal Physiol,2011,301(3):F509-F519.
