苎麻木质素合成酶CAD基因的cDNA克隆及表达分析
2014-07-02朱伟溢陈建荣彭彦张学文郭清泉赵燕
朱伟溢,陈建荣,彭彦,张学文,郭清泉,赵燕*
(1.湖南农业大学生物科学技术学院,湖南 长沙 410128;2.长沙学院生物工程与环境科学系,湖南 长沙 410003)
苎麻木质素合成酶CAD基因的cDNA克隆及表达分析
朱伟溢1,陈建荣2,彭彦1,张学文1,郭清泉2,赵燕1*
(1.湖南农业大学生物科学技术学院,湖南 长沙 410128;2.长沙学院生物工程与环境科学系,湖南 长沙 410003)
采用PCR结合RACE技术克隆苎麻栽培种湘苎3号CAD基因全长cDNA序列;再运用qRT–PCR技术对木质素含量不同的代表性苎麻品种湘苎1号、湘苎3号、湘潭大叶白和城步青麻的CAD基因在其茎秆韧皮部和木质部的表达进行定量分析;使用1%间苯三酚和25%盐酸对4种苎麻茎横切片进行特异染色,观察品种间木质素染色度。结果表明,获得的苎麻CAD基因cDNA序列全长为1 378 bp(GenBank 登录号,KF758396),编码359个氨基酸,推导为苎麻肉桂醇脱氢酶(BnCAD)。该推导蛋白质与已报道的几种植物木质素合成酶CAD的同源性均达90%以上,其中与蒺藜苜蓿的同源性达97%。运用qRT–PCR方法对BnCAD基因表达的定量分析结果显示,苎麻CAD基因在湘苎1号和湘苎3号的韧皮部表达水平明显高于其在木质部的表达水平,而在城步青麻木质部的表达水平高于其在韧皮部的表达水平,该基因在湘潭大叶白的韧皮部和木质部表达量接近。4种苎麻茎横切片的木质素染色结果表明,品种间木质素染色度与CAD基因在其韧皮部的表达量成正相关。
苎麻;木质素合成酶CAD基因;克隆;表达分析
苎麻(Boehmeria nivea (Linn.) Gaud.)属荨麻科苎麻属草本植物,其纤维是重要的纺织原料,近年来木质纤维被用于生物燃料乙醇发酵[1],是具有较大经济利用价值的纤维植物。低木质素的木质纤维更有利于苎麻作为纺织原料、水土保持植物以及生物燃料等,而研究其木质素合成、组成和代谢的分子生物学规律,可以指导低木质素含量苎麻的选育和对其代谢调控的人工干预。通过对模式植物拟南芥的研究,木质素合成的代谢具体途径已有了较清晰的认识[2],其以苯丙酸为起始,经过一系列生化反应生成木质素化学单体,单体进一步氧化聚合生成相应的木质素。这些木质素通过多种键型连接在一起,随机形成复杂的木质素聚合体,随后该大分子聚合体在植物体内执行相应的生物学功能[3]。
肉桂醇脱氢酶(cinnamyl alcohol dehydrogenase,CAD)是木质素生物合成途径的最后一个关键酶,分离苎麻肉桂醇脱氢酶(BnCAD)是研究木质素生物合成及代谢途径的基础。利用基因工程技术,降低烟草[4]、拟南芥[5]、黑麦草[6]CAD基因的表达量,能够获得木质素含量降低或者单体组分改变的植株。
苎麻木质素生物合成代谢途径中的关键酶CCoAOMT、COMT基因已有报道,陈建荣等[7–8]克隆了CCoAOMT 基因cDNA全长序列,并利用苎麻CCoAOMT基因进行表达干扰导致转基因植株生长势有不同程度的减弱。但关于苎麻中 CAD基因的研究尚未见报道。本研究以苎麻栽培种湘苎3号为材料,拟通过对其转录组测序信息分析,设计引物,采用PCR结合RACE技术克隆其CAD基因全长cDNA分子。对湘苎1号,湘苎3号,湘潭大叶白,城步青麻等4个具有代表性的[9]苎麻品种的CAD基因在韧皮部和木质部的表达进行分析,同时对4种苎麻茎切片进行染色,以进一步分析木质素与CAD基因表达量的关系。
1 材料与方法
1.1 材 料
1.1.1 供试苎麻
供试苎麻品种为苎麻(Boehmeria nivea (Linn.) Gaud.)栽培种湘苎1号、湘苎3号以及野生种湘潭大叶白、城步青麻,均由湖南农业大学苎麻资源圃提供。
1.1.2 菌 株
大肠杆菌DH5α由湖南农业大学细胞生物学实验室提供。
1.2 方 法
1.2.1 苎麻木质素合成酶CAD基因cDNA核心序列的扩增
将苎麻转录组数据(未公开)与已发表的其他植物CAD基因的cDNA进行比对,针对同源性高的核心保守区序列,运用 DNAman软件设计引物CAD–up(5′–CAGGGAAGTTGGGATTGACGATG–3′),CAD–dn(5′–CGTTAGCGGAACAGAAGTCTAG CAT–3′),以扩增CAD基因的cDNA核心序列。
取成熟期的湘苎3号茎秆,用SV Total RNA试剂盒(Promega公司)提取总 RNA,用 Prime-ScriptTM1st strand cDNA Synthesis Kit (TaKaRa)
Ⅱ
进行反转录,得到cDNA作为核心序列扩增的模板。PCR反应体系为:cDNA 模板 1 μL、dNTPs(2.5 mmol/L) 2 μL、10× Buffer 2.5 μL、CAD-up引物1 μL、CAD-dn引物 1 μL、TaqDNA聚合酶(2.5U/μL) 0.5 μL、ddH2O 17 μL。PCR反应条件为:94 ℃预变性5 min;94 ℃变性40 s、56 ℃ 退火40 s、72 ℃延伸1 min,25个循环;72 ℃终末延伸 10 min。PCR产物用胶回收试剂盒(北京康为世纪生物科技有限公司)回收后,连接至pMD18–T载体(TaKaRa),并转化大肠杆菌 DH5α,通过 Amp抗性筛选和X–gal/IPTG 蓝白斑筛选,挑取白色阳性克隆进行菌落PCR检测(检测所用引物是CAD–up和CAD–dn)和酶切检测(酶切所使用的酶是Pst I、Eco RI,均购自 Fermantas)。将检测的阳性单菌落摇菌并提取质粒送上海铂尚生物技术公司测序。
1.2.2 苎麻木质素合成酶CAD基因cDNA末端序列的扩增
以获得的 CAD基因核心序列为模板,设计5′RACE巢式PCR引物CAD 5′–GSP1(5′–ATGGTTG AAGGTGTAAACGG–3′)、CAD 5′–GSP2(5′–CTGCA AGAGTTGACGTAAGTTC–3′)及3′RACE巢式PCR引物CAD 3′–GSP1(5′–CCTACGTGTCCCTGTTGA AGAC–3′)、CAD 3′–GSP2(5′–TGCTAGACTTCTGT TCCGCTAAC–3′)。用5′–Full RACE Kit with TAP (TaKaRa)和3′–Full RACE Core Set with PrimeScript™(TaKaRa)分别进行3′端和5′端的RACE反应。
将上述 RACE产物回收、纯化,分别连接pMD18–T载体并转化大肠杆菌DH5α,通过Amp抗性筛选和 X–gal/IPTG 蓝白斑筛选,挑取白色阳性克隆进行菌落PCR检测(检测引物为5′RACE、3′RACE巢式PCR引物)和酶切检测(用Pst I、Eco RI进行双酶切检测),将检测为阳性的单菌落摇菌并提取质粒送上海铂尚生物技术公司测序。
1.2.3 苎麻木质素合成酶CAD基因拼接序列分析
利用DNAman软件进行序列比对、拼接,用序列处理在线工具包(SMS)进行ORF查找、翻译。在NCBI上进行BLAST和蛋白质序列的CDD搜索。利用MEGA5.1软件构建CAD蛋白分子系统发育树。
1.2.4 苎麻木质素合成酶CAD基因在不同品种的苎麻中的表达
取田间栽种的成熟期苎麻茎杆中部为材料,按以上方法分别提取湘苎1号、湘苎3号、湘潭大叶白、城步青麻4个苎麻品种茎杆的韧皮部和木质部组织的总RNA和cDNA。以苎麻高度保守的内源肌动蛋白Actin1为内参基因,BnCAD为目的基因同时进行荧光定量PCR反应。用于BnCAD 基因扩增的引物为Up(5′–GGCATGAGATTGTTGGAATTG–3′)和Dn(5′– CATGGTTGAAGGTGTAAACGG–3′)。用于苎麻Actin1扩增的引物为Actin1–up(5′–CTCGTCT GCGACAATGGTAC–3′)和Actin1–dn (5′–GACAAT ACCGTGTTCAATGGG–3′)。利用LightCycler 480 System进行荧光定量 PCR反应,具体步骤参照TaKaRa公司的SYBR Premix Ex TaqTM试剂盒进行。
1.2.5 苎麻茎秆切片及显微观察
于8:00–9:00分别取湘苎1号、湘苎3号、湘潭大叶白、城步青麻4种苎麻茎秆的上、中、下部,利用leitz1400型滑走切片机切片,用1%间苯三酚和25%盐酸染色后置于BX51型OLYMPUS研究显微镜下观察。
2 结果与分析
2.1 苎麻CAD基因的克隆
PCR检测结果表明,从湘苎3号茎秆扩增出953 bp的核心片段(图1),与预期目的片段长度相符。根据核心序列使用3′和5′RACE法,扩增到400 bp的3′端片段(图2)和500 bp的5′端片段(图3),均与预期目的片段的长度相符。

图1 BnCAD中间片段扩增结果Fig.1 PCR results for intermediate fragment of BnCAD

图2 BnCAD基因3′ RACE扩增结果Fig.2 Results of BnCAD 3′ RACE amplification
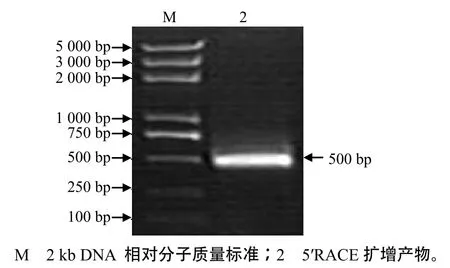
图3 BnCAD基因5′ RACE扩增结果Fig.3 Results of BnCAD 5′ RACE amplification
2.2 苎麻CAD基因拼接结果
用DNAman软件将核心片段和3′端及5′端序列进行拼接,得到长1 378 bp的cDNA序列,其中5′端非翻译区长134 bp、开放阅读框(ORF)长 1 077 bp、3′端非翻译区长167 bp。NCBI比对结果显示,本试验成功克隆了 BnCAD基因序列,并命名为BnCAD1。将该基因登录到 GenBank,登录号为KF758396。
与GenBank已报道的拟南芥、毛白杨、蒺藜苜蓿等多种植物的CAD1蛋白序列进行系统进化关系分析,结果(图4)显示,苎麻肉桂醇脱氢酶CAD与其他植物的CAD1蛋白差异不大,进一步说明该蛋白质属于 CAD1蛋白家族。相对于单子叶植物,BnCAD1与来源于双子叶植物的CAD蛋白家族成员亲缘关系更近,其中与蒺藜苜蓿(Medicago truncatula)的亲缘关系最近,同源性达97%。
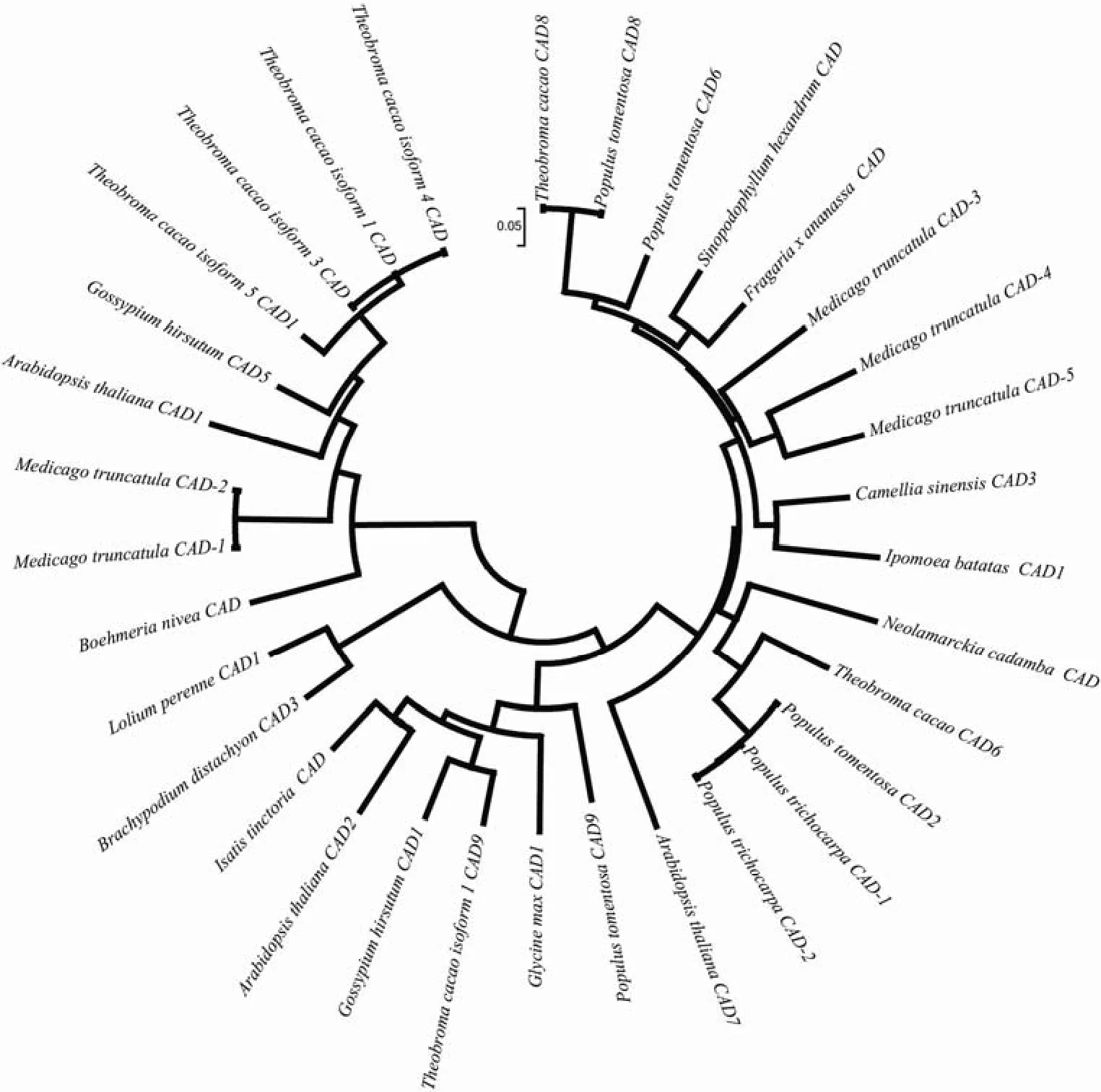
图4 植物中CAD蛋白质氨基酸序列的进化分析结果Fig.4 Phylogenetic analysis of amino acid sequences of CAD proteins from plants
2.3 苎麻CAD基因在4种代表性苎麻品种中的表达

图5 CAD基因在4种苎麻韧皮部和木质部中表达的荧光定量RT–PCR分析结果Fig.5 The fluorescence quantitative RT–PCR analysis of the expression of CAD in the phloem and xylem in the 4 ramie cultivars
选择苎麻Actin1为内参基因,从生长中期的苎麻中提取茎木质部和韧皮部总 RNA,进行qRT–PCR。结果(图5)显示,在4种苎麻的木质部中CAD基因在湘苎1号和湘苎3号的表达量略高于城步青麻和湘潭大叶白,而在韧皮部中湘苎1号的表达量最高,其次是湘苎1号,在城步青麻中的表达量最低,而且湘苎1号、湘苎3号、湘潭大叶白这3种苎麻韧皮部CAD基因的表达量比木质部CAD基因的表达量要高。
2.4 4种代表性苎麻品种茎横切面的染色观察
染色观察结果显示,茎上部的茎横切面染色(图6)结果区别不大,这是由于木质部还处于发育早期,木质素含量均较低,其着色较浅。茎中部横切面染色(图7)差别较大,其中湘苎1号染色最深,其次是湘苎3号、湘潭大叶白,而城步青麻的染色最浅。而茎下部由于木质素在木质部形成沉积,4个苎麻品种的茎横切面染色(图8)均较深且无明显差异。

图6 4种代表性苎麻茎上部的木质部染色Fig. 6 The stain of lignin in top stem of the 4 ramie cultivars
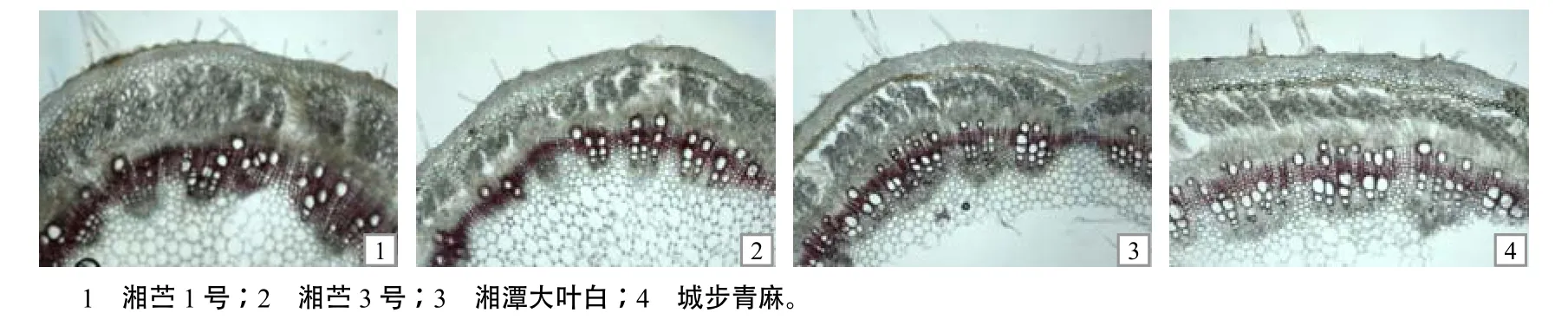
图7 4种代表性苎麻茎中部的木质部染色效果Fig. 7 The stain of lignin in middle stem of the 4 ramie cultivars
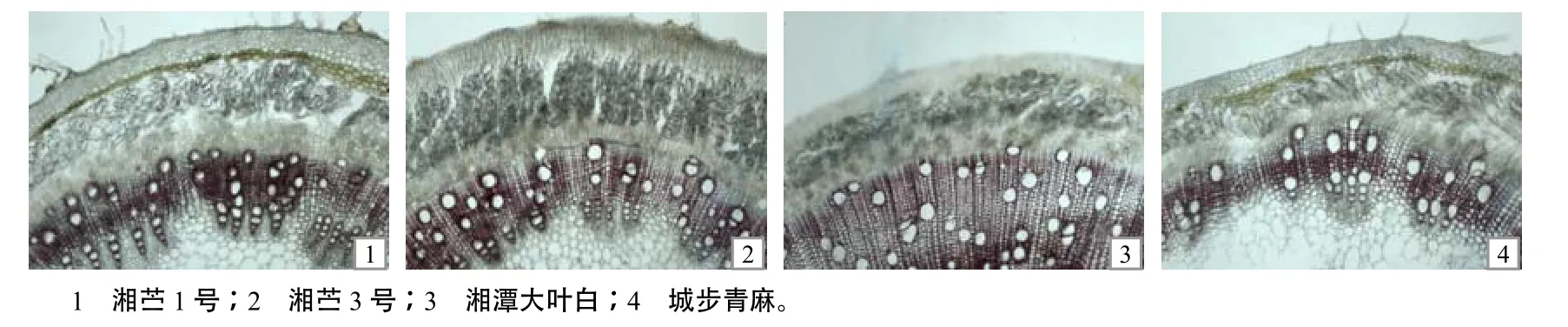
图8 4种代表性苎麻茎下部的木质部染色Fig. 8 The stain of lignin in bottem stem in the 4 ramie cultivars
3 讨 论
苎麻木质素主要由紫丁香基木质素(syringyl lignin,S–木质素)和愈创木基木质素(guaiacyl lignin,G–木质素)2种化学单体聚合而成。目前已经从多种植物中克隆CAD基因,并且通过转基因方法对该基因进行功能研究[10–14]。Halpin等[10]报道,在抑制CAD表达的转基因烟草中,醛醇比的变化大于S/G值,表明CAD 活性降低,抑制松柏醛还原成松柏醇的强度变大,因此,推测CAD 存在不同的CAD 同工酶。反义抑制CAD基因的转基因烟草植株中,木质素含量无明显变化,但其单体组成以及结构都发生较大改变,使工业酸碱提取木质素和木质素作为饲料被消化更容易[4]。Pliate等[15]通过反义抑制杨树 CAD活性,得到与转基因烟草类似结果,即醛醇比的变化大于S/G。除了单基因转化试验,Chabannes等[16]对烟草CCR、CAD进行双抑制,得到的转基因烟草不仅木质素含量大幅度降低,而且其植株表型和木质素性质结构更接近野生型。这些研究都证明了在植物木质素合成中CAD基因对S–木质素和G–木质素合成所起的关键作用。
在对拟南芥CADs基因的研究[17–19]中,发现有9个 CADs基因与木质素合成相关,但只有 AtCAD4 和AtCAD5的表达对木质素合成起关键性作用[18,20],而与BnCAD1同源性较高的 AtCAD1在维管束组织中有明显表达,且在生物代谢途径中的具体功能还未知[20]。另一个与BnCAD1有较高同源性的基因是毛白杨CAD9(PtCAD9),该基因经研究发现在毛白杨的叶片和木质部表达量均较高[20],其功能可能是响应木质素防御途径[21]。经进化分析,与BnCAD1亲缘关系最近的是蒺藜苜蓿(MtCAD1)。Qiao Zhao 等[22]研究发现 MtCAD1基因的缺失能导致细胞壁成分比例发生改变,说明CAD1基因可能通过影响细胞壁的成分比例来改变韧皮部、木质部细胞的结构,从而达到更易降解木质素的目的。
根据前人的研究结果,结合本研究对BnCAD1基因在韧皮部、木质部的表达分析结果说明BnCAD1基因主要在韧皮部表达,且在湘苎1号韧皮部的表达量约是在城步青麻韧皮部表达量的 4倍。另外苎麻茎横切片的木质素间苯三酚特异性染色结果也显示,BnCAD1基因在韧皮部的表达量与苎麻木质部的染色情况呈正相关。可以推测BnCAD1基因在苎麻韧皮部表达后促进木质素单体的合成,而不同类型的单体经一定途径转移到木质部聚集形成木质素,间接验证了 CAD基因在苎麻木质素合成过程中的重要性,而 CAD的表达活性高低是否会影响苎麻木质素含量或单体成分的变化值得进一步的研究。本研究结果还显示BnCAD1在木质部的表达量相差不大。同时,笔者发现4个品种其下部茎秆的木质部染色深浅差别不明显,表明苎麻生长发育后期CAD基因的表达量差异不大,说明 BnCAD1基因有与拟南芥、毛白杨等类似的CADs基因家族,可能还参与其他代谢过程或者具有其他的未知功能,并在长期进化过程中不同的基因之间存在功能分化,而单独降低 CAD基因的表达水平对木质素含量的影响不大,原因则可能是由于基因家族的冗余作用,从而导致其他相关代谢产物发生改变。
[1] 李江,谢天文,刘晓风.木质纤维素生产燃料乙醇的糖化发酵工艺研究进展[J].化工进展,2011,30(2):284–291.
[2] Ruben V,Véronique S,Bartel V.A systems biology view of responses to lignin biosynthesis perturbations in Arabidopsis[J].Plant Cell,2012,24(9):3506–3529.[3] Baucher M,Monties B,Van M M,et al.Biosynthesis and genetic engineering of lignin[J].Crit Rev Plant Sci,1998,17(2):125–197.
[4] Halpin C,Knight M E,Foxon G A.Manipulation of lignin quality by down-regulation of cinnamyl alcohol dehydrogenase[J].Plant,1994,6(3):339–350.
[5] Thévenin J,Pollet B,Letarnec B.The simultaneous repression of CCR and CAD,two enzymes of the lignin biosynthetic pathway,results in sterility and dwarfism in Arabidopsis thaliana[J].Molecular Plant,2011,4(1):70–82.
[6] 胡可,严雪锋,栗丹.沉默CCR和CAD基因培育低木质素含量转基因多年生黑麦草[J].草业学报,2013,22(5):72–83.
[7] 陈建荣,郭清泉,张学文.苎麻CCoAOMT基因全长cDNA克隆与序列分析[J].中国农业科学,2006,39(55):1058–1063.
[8] 邓晶,刘峰,陈建荣,等.苎麻CCoAOMT基因干扰表达载体构建及对烟草的转化[J].湖南农业大学学报:自然科学版,2008,34(2):132–135.
[9] 李建军,郭清泉,陈建荣.21份不同木质素含量的苎麻的 RAPD聚类分析[J].中国麻业,2006,28(3):120–122.
[10] Halpin C.Brown-midrib maize(bm1)–a mutation affecting the cinnamyl alcohol dehydrogenase gene[J]. Plant J,1998,14(5):545–553.
[11] Mackay J J,Booker F L,Sederoff R R.Inheritance,gene expression,and lignin characterization in a mutant pine deficient in cinnamyl alcohol dehydrogenase[J]. Proc Natl Acad Sci,1997,94(15):8255–8260.
[12] Pilonel C,Mulder M M,Boon J J,et al.Involvement of cinnamyl–alcohol dehydrogenase in the control of lignin formation in Sorghum bicolor L[J].Moench Planta,1991,185(4):538–544.
[13] Lapierre C,Pollet B,Toval G,et al.Structural alterations of lignins in transgentic poplars with depressed cinnamyl alcohol dehydrogenase or caffeic acid O–methyltransferase activity have an opposite impact on the efficiency of industrial kraft pulping[J].Plant Physiol,1999,119(1):153–164.
[14] Baucher M,Chabbert B,Besle J M.Down-regulation of cinnamyl alcohol dehydrogenase in transgenic alfalfa (Medicago sativa L.) and the effect on lignin composition and digestibility[J].Plant Molecular Biology,1999,39(3):437–447.
[15] Pilate G,Guiney E,Holt K.Field and pulping performances of transgenic trees with altered lignification[J]. Nature Biotechnology,2002,20:607–612.
[16] Chabannes M,Barakate A,Lapierre C,et al.Strong decrease in lignin content without significant alteration of plant development is induced by simulataneous downregulation of cinnamoyl CoA reductase (CCR) and cinnamyl alcohol dehydrogenase (CAD) in tobacco plants[J].The Plant Journal,2001,28(3):257–270.[17] Sibout R,Eudes A,Pollet B.Expression pattern of two paralogs encoding cinnamyl alcohol dehydrogenases in Arabidopsis.Isolation and characterization of the corresponding mutants[J].Plant Physiol,2003,132(2):848–860.
[18] Kim S J,Kim K W,Cho M H.Expression of cinnamyl alcohol dehydrogenases and their putative homologues during Arabidopsis thaliana growth and development:Lessons for database annotations?[J].Phytochemistry,2007,68(14):1957–1974.
[19] Tronchet M,Balagué C,Kroj T,et al.Cinnamyl alcohol dehydrogenases–C and D,key enzymes in lignin biosynthesis,play an essential role in disease resistance in Arabidopsis[J].Mol Plant Pathol,2010,11(1):83–92.
[20] 张鲁斌,谷会,弓德强,等.植物肉桂醇脱氢酶及其基因研究进展[J].西北植物学报,2011,31(1):0204–0211.
[21] Goffner D,Doorsselaere J V.A novel aromatic alcohol dehydrogenase in higher plants:Molecular cloning and expression[J].Plant Molecular Biology,1998,36(5):755–765.
[22] Somssich I E,Wernert P.Arabidopsis thaliana defenserelated protein ELI3 is an aromatic alcohol:NADP+oxidoreductase[J].Proc Natl Acad Sci USA,1996,93(24):14199–14203.
责任编辑:罗 维
英文编辑:罗 维
cDNA cloning and expression analysis of a lignin synthetase CAD gene in ramie (Boehmeria nivea (L.) Gaud.)
ZHU Wei-yi1, CHEN Jian-rong2, PENG Yan1, ZHANG Xue-wen1, GUO Qing-quan2, ZHAO Yan1*
(1.College of Bioscience and Biotechnology, Hunan Agricultural University, Changsha 410128, China; 2.Department of Biotechnology and Environment Science, Changsha University, Changsha 410003, China)
This study is intended to clone CAD from ramie (Boehmeria nivea L. Gaud. cv Xiangzhu 3), and analyze the expression patterns of CAD in stems from 4 representative ramie cultivar. First, the cDNA sequence of CAD were identified by analyzing the transcriptome data of ramie cultivar Xiangzhu 3 and cloned by RT–PCR combined with RACE method. Secondly, the qRT–PCR was carried out to analyze the expression levels of CAD transcripts in 4 ramie cultivars: Xiangzhu 1, Xiangzhu 3, Xiangtaidayebai and Chengbuqingma. Finally, lignin contents among different cultivars were visually analyzed by histochemical staining of transverse sections of ramie stems using the phloroglucin. The BnCAD cDNA sequence was 1 378 bp in length (GenBank accession number, KF758396) and could be translated into a putative protein with 359 amino acids. Blast and protein structure analysis showed that this cDNA sequence is in high homologous with other plant CAD gene, and with 97% homology compared with Medicago truncatula, which confirmed that this cDNA sequence was the CAD gene. qRT–PCR analysis revealed that the expressions of CAD was much higher in phloem than xylem in Xiangzhu 1 and Xiangzhu 3, while Chengbuqingma the opposite, and no obvious difference was observed in Xiangtandayebai. Histochemical lignin assays showed that the staining degree between different cultivars is directly proportional to the quantitative expression of CAD transcripts, suggesting an important role of CAD during ramie lignin synthesis.
ramie; lignin synthase CAD gene; cloning; expression analysis
S563.101
A
1007−1032(2014)05−0481−06
10.13331/j.cnki.jhau.2014.05.006
投稿网址:http://www.hunau.net/qks
2014–03–21
国家自然科学基金项目(31071457);湖南省科技计划项目(2012NK3062);长沙市科技计划项目(K1309014–31)
朱伟溢(1989—),女,湖南常德人,硕士研究生,主要从事分子细胞生物学研究,258077287@qq.com;*通信作者,zhaoyan 0585@163.com
