检测Sortase A酶催化蛋白质连接效率的新型报道系统
2014-06-15李健王鹏举崔云凤邹培建秦刚1天津科技大学生物工程学院天津300457中国科学院天津工业生物技术研究所工业酶国家工程实验室天津300308
李健,王鹏举,崔云凤,邹培建,秦刚1 天津科技大学生物工程学院,天津 300457 中国科学院天津工业生物技术研究所 工业酶国家工程实验室,天津 300308
检测Sortase A酶催化蛋白质连接效率的新型报道系统
李健1,2,王鹏举1,2,崔云凤2,邹培建2,秦刚2
1 天津科技大学生物工程学院,天津 300457 2 中国科学院天津工业生物技术研究所 工业酶国家工程实验室,天津 300308
利用分子进化技术对Sortase A酶的催化活性进行定向改造已经成为当前的研究热点和重点,为了高效筛选特定催化活性的Sortase A酶突变体,需要建立一种能够快速准确鉴定Sortase A酶活性的方法。为此,设计了由GFP-LPETG蛋白和GGGYK-Biotin组成的新型报道底物系统。利用重组基因工程技术,成功制备GFP-LPETG蛋白,野生型Sortase A酶及近期报道的一个高活性突变型Sortase A酶。以GFP-LPETG及GGGYK-Biotin为报道底物建立连接体系,结果显示,连接反应动力学可直接通过SDS-PAGE凝胶电泳法精确测定。用此连接体系比较野生型与突变型Sortase A酶催化的连接反应效率,证实高活性突变酶具有野生酶无法比拟的高催化活性。此报道系统简单快速灵敏,可应用于系统的筛选,为后续进一步定向优化Sortase A酶奠定了基础。
Sortase A酶,连接反应效率,报道底物,GFP-LPETG蛋白,GGGYK-Biotin,高活性突变体
蛋白质连接技术广泛应用于生物医学工程领域,根据其反应特征可分为化学连接法和酶催化连接法[1–2]。目前,化学连接法在蛋白质连接技术中仍发挥重要作用,但存在制备工艺复杂、对蛋白质活性有损伤、连接产物不均一等问题,相比而言,以Sortase酶为代表的酶催化蛋白质连接法展现出反应条件温和、反应效率高、底物特异性强等明显优势,代表了蛋白质连接技术的发展趋势。
Sortase酶是一组存在于革兰氏阳性细菌中介导表面蛋白与细胞壁共价结合的蛋白酶,负责细胞表面蛋白的修饰和细菌鞭毛的构建[3–7]。当前研究最广泛的是来自金黄色葡萄球菌Staphylococcus aureus的Sortase A酶 (EC 3.4.22.70),其结构、功能及催化原理已有透彻研究[8–14]。Sortase A酶具有一个典型的类似Ⅱ型膜蛋白的N端跨膜区域和C端富集正电荷的尾巴。此酶催化C端含有LPxTG (x代表任一种氨基酸残基) 序列的底物,与N端含有寡聚Gly序列的底物进行连接反应[15–17]。底物识别序列中的Leu和Pro通过疏水作用结合到Sortase A酶活性区域,导致识别序列中Thr与Gly形成的肽键靠近Sortase A酶活性中心的Cys184和Arg197[18],Cys184和His120反向质子化,促成Cys184的巯基攻击Thr与Gly之间酰胺键的羰基,形成一个短暂的四面体氧负离子过渡状态,Arg197的氢键作用有助于此状态稳定;Gly的离开导致形成一个稳定的酰基-酶中间体,此中间体被另一底物的N端氨基亲核攻击,从而打破稳定状态,形成新的连接产物。另外,Sortase A酶存在一个钙离子的结合位点,钙离子的存在会增加酶的催化效率[19–20]。
Sortase A酶催化的连接反应因其便捷性,已被证实可应用于蛋白质与蛋白质、肽、核酸类似物、糖等活性物质的连接,以及蛋白质的固定化[21],展示出良好的应用前景。但天然Sortase A酶催化的连接反应耗时长、产率低,离大规模实际应用尚有一定距离。正因如此,不同实验室开展了一系列对此酶的改造,包括提高酶催化活性及改变底物识别特征等[17,22-26]。最近,Chen等利用酵母展示系统,经过多轮筛选获得了比野生型Sortase A酶活性高140多倍的突变体[25]。与野生型相比,此酶发生4个位点的突变:P94S、D160N、D165A和K196T,根据已知的三维结构,突变位点全部位于底物结合区域附近,推测突变型酶更利于底物进入结合区域,使得连接效率大大提升。而Piotukh等则成功利用噬菌体展示技术筛选获得底物识别序列改变为APxTG的突变型Sortase A酶[26]。目前,检测Sortase A酶突变体活性的方法有HPLC、MS、荧光成像法等,操作过程繁琐且需要特殊设备[25-26]。
本研究旨在开发一种简单灵敏的用于检测连接反应效率的报道底物系统,通过将此系统应用于野生型及高活性突变Sortase A酶催化的连接反应以证实该检测系统的可靠性,为后续定向改造Sortase A酶提供通用的筛选鉴定方法。
1 材料与方法
1.1 材料
1.1.1 菌株及质粒
金黄色葡萄球菌Staphylococcus aureus购于中国普通微生物菌种保藏管理中心;大肠杆菌Escherichia coli DH5α、BL21(DE3) 购于北京全式金生物科技有限公司;表达载体pET-24d(+)-GB1和pET-24d(+)-GFP为本实验室构建。
1.1.2 主要试剂
细菌基因组DNA提取试剂盒、琼脂糖购于天根生化科技 (北京) 有限公司;聚合酶Phusion Hot Start Ⅱ DNA 聚合酶,限制性内切酶NcoⅠ和KpnⅠ,Phusion Site-Directed Mutagenesis Kit,蛋白Marker均购自Thermo Scientific公司;T4 DNA 连接酶购于全式金公司;高纯度质粒小提试剂盒和PCR高纯度片段回收试剂盒购自北京康为世纪生物科技有限公司;Ni Sepharose 6 Fast Flow购于GE公司;人工合成短肽GGGYK-Biotin (分子量为707 Da,纯度99%) 及GGGYK (分子量为481 Da,纯度99%) 由北京中科亚光生物技术有限公司合成;其他试剂均为分析纯或色谱纯。
1.2 方法
1.2.1 GFP-LPETG和野生型Sortase A基因片段的获得
以质粒pET-24d(+)GFP为模板,使用引物GFP For和GFP Rev (表1),PCR扩增GFP-LPETG基因片段。PCR反应程序为:98 ℃预变性30 s;98 ℃变性10 s,59 ℃退火20 s,72 ℃延伸20 s,25个循环;72 ℃延伸5 min。依据金黄色葡萄球菌中srtA基因序列 (GenBank Accession No. AF162687.1),分别设计上下游引物 SrtA For和SrtA Rev (表1),以提取的金黄色葡萄球菌基因组DNA为模板,采用上述方法扩增编码srtA基因61–221位的读码框。
1.2.2 pET-24d(+)-GFP-LPETG、pET-24d(+)-SrtA及pET-24d(+)-SrtAM表达载体的构建
以NcoⅠ和KpnⅠ双酶切处理经琼脂糖凝胶回收的GFP-LPETG的 PCR扩增产物,与经 NcoⅠ和KpnⅠ双酶切处理的pET-24d(+)载体片段连接,转化至感受态E. coli DH5α,挑取单克隆进行酶切鉴定,正确的克隆测序验证,命名为pET-24d(+)-GFP-LPETG。采用同样方法构建pET-24d(+)-SrtA载体。在其基础上,利用Phusion Site-Directed Mutagenesis Kit,依次采用引物P94S For/P94S Rev,D160N/ D165A For和D160N/D165A Rev,K196T For/ K196T Rev,对srtA基因进行定点突变,每轮突变后测序确证,最终获得突变型酶Sortase A酶表达载体pET-24d(+)-SrtAM。
1.2.3 GFP-LPETG、Sortase A及突变型Sortase A酶的诱导表达
分别将表达载体pET-24d(+)-GFP-LPETG、pET-24d(+)-SrtA及pET-24d(+)-SrtAM 转化至E. coli BL21(DE3)感受态细胞,挑取单克隆在含有50 μg/mL卡那霉素的LB液体培养基中培养,加入IPTG 至0.2 mmol/L,于37 ℃诱导3 h。

表1 PCR引物的设计Table 1 Design of PCR primers
1.2.4 GFP-LPETG、Sortase A及突变型Sortase A酶的的亲和纯化
6 000 r/min、4 ℃离心15 min收集菌体。用缓冲液(25 mmol/L Tris-HCl,pH 7.5,250 mmol/L NaCl,20 mmol/L 咪唑)重悬,高压匀浆仪破碎菌体。裂解液中添加0.3% Triton X-100,4 ℃、16 000 r/min离心50 min,上清经0.45 µm滤膜过滤,使用自动蛋白纯化仪AKTA进行Ni-NTA亲和层析。目的蛋白经20–250 mmol/L 咪唑线性梯度洗脱,SDS-PAGE凝胶电泳检测。蛋白样本经4 ℃透析,超滤浓缩并测定浓度,于–80 ℃保存。
1.2.5 连接反应效率检测系统
在连接体系中分别加入1 μmol/L Sortase A酶 (野生型或突变型),2 μmol/L GFP-LPETG蛋白及30 μmol/L 合成短肽GGGYK-Biotin (或合成短肽GGGYK),37 ℃反应,分别于设定时间点取样并终止反应。所有样本经SDS-PAGE凝胶电泳,考马斯亮蓝 (Commassise blue R250)染色,脱色后电泳结果经全自动多色荧光及化学发光凝胶成像系统拍照,利用LANE 1D软件对相应条带进行定量,计算连接效率。
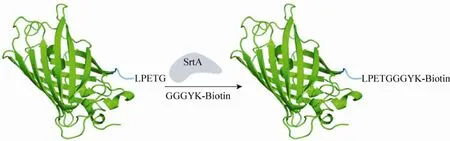
图1 报道底物设计Fig. 1 Design of reporter substrates.
2 结果与分析
2.1 报道底物设计
检测Sortase A酶的催化活性,需要一种简单、快捷、灵敏的连接反应效率分析系统。为实现此目的,根据Sortase A酶介导连接反应的底物特征,将Sortase A酶识别序列LPETG引入GFP蛋白C端;另一底物则选用短肽GGGYK-Biotin (GGGYK作为对照)。预期连接产物为GFP-LPETGGGYK-Biotin,反应过程如图1所示。生物素分子的引入,是希望通过链霉亲和素分子印迹法来实现高灵敏度检测连接产物的生成。
2.2 pET-24d(+)-GFP-LPETG与pET-24d (+)- SrtA载体构建
以本实验室构建的含有GFP编码的质粒pET-24d(+)GFP为模板,PCR扩增完整的GFP编码片段,Sortase A酶识别序列LPETG的核酸编码通过下游引物GFP Rev引入到扩增片段3′端。结果显示,扩增片段大小约750 bp,与预期相符 (图2)。将PCR片段经Nco Ⅰ和Kpn Ⅰ位点插入质粒pET-24d(+),测序完全正确,命名为pET-24d(+)-GFP-LPETG。
先前报道指出,Sortase A酶N端60个氨基酸组成跨膜区域,截掉此跨膜区对酶活没有显著影响,却更利于重组蛋白可溶性制备[27]。据此,以金黄色葡萄球菌基因组DNA为模板,利用PCR成功扩增截短N端60个氨基酸编码的srtA基因片段,扩增片段大小约450 bp,符合预期 (图3) 。PCR片段经Nco Ⅰ和Kpn Ⅰ位点插入质粒pET-24d(+),测序验证与srtA基因序列完全一致,命名为pET-24d(+)-Srt A。
2.3 GFP-LPETG蛋白及野生型Sortase A酶的表达纯化
将pET-24d(+)-GFP-LPETG及pET-24d(+)-SrtA质粒分别转化至BL21(DE3)菌株,诱导表达,重组报道底物GFP-LPETG蛋白与野生型Sortase A酶主要以可溶形式存在。经Ni-NTA柱亲和层析纯化,得到高纯度的目的蛋白,经SDS-PAGE,报道底物GFP-LPETG大小为36 kDa (图4),野生型Sortase A大小为26 kDa (图5)。

图2 GFP基因的PCR扩增Fig. 2 PCR amplification of GFP gene. M: DNA molecular weight markers; 1: PCR product of GFP gene.
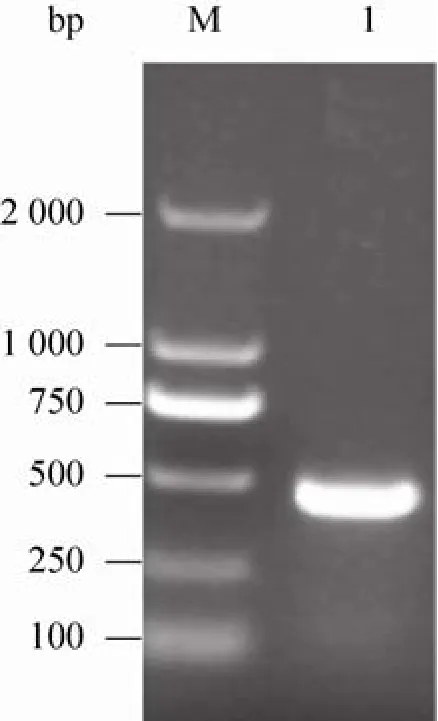
图3 金黄色葡萄球菌srtA基因的PCR扩增Fig. 3 PCR amplification of Staphylococcus aureus srtA gene. M: DNA molecular weight markers; 1: PCR product of srtA gene.

图4 Ni-NTA柱亲和纯化GFP-LPETG的SDS-PAGE分析Fig. 4 SDS-PAGE analysis of purified GFP-LPETG via Ni-NTA affinity chromatography. M: protein marker; 1: soluble fraction of whole cell lysate; 2: flowthrough; 3–4: wash; 5: elution of purified GFP protein.

图5 Ni-NTA柱亲和纯化野生型Sortase A酶的SDS-PAGE分析Fig. 5 SDS-PAGE analysis of purified wildtype Sortase A protein via Ni-NTA affinity chromatography. M: protein marker; 1: soluble fraction of whole cell lysate; 2: flowthrough; 3: purified Sortase A protein.
2.4 Sortase A酶介导的连接反应效率分析系统的建立
分别将合成的底物短肽GGGYK及GGGYKBiotin,与GFP-LPETG蛋白组成连接反应底物对,经野生型Sortase A酶催化,SDS-PAGE凝胶电泳追踪连接产物GFP-LPETGGGYK及GFP-LPETGGGYK-Biotin的形成。结果显示,使用底物GGGYK进行反应,连接产物GFPLPETGGGYK与未反应底物GFP-LPETG条带过于接近,因而无法准确判断连接反应进程 (图6,泳道8–13)。与此对照,使用底物GGGYK-Biotin进行反应,连接产物GFP-LPETGGGYK-Biotin与未反应底物GFP-LPETG的SDS-PAGE电泳迁移率存在明显差异,随反应时间延长连接产物逐渐增多,并伴随底物GFP-LPETG逐渐减少 (图6,泳道1–6)。这表明,以GGGYK-Biotin与GFP-LPETG为报道底物的连接反应体系,仅需SDS-PAGE凝胶电泳经考马斯亮蓝染色就可以快速、灵敏地追踪Sortase A酶催化的连接反应进程,最初设想的引入链霉亲和素分子印迹步骤已无实施必要。

图6 SDS-PAGE分析野生型Sortase A催化蛋白质连接效率Fig. 6 SDS-PAGE assay monitoring ligation efficiency mediated by wildtype Sortase A. 1–6: ligation between GFP-LPETG and GGGYK-Biotin, with 0, 1, 2, 3, 4, 12 hours of reaction, respectively; 7: GFP-LPETG; 8–13: ligation between GFP-LPETG and GGGYK, with 0, 1, 2, 3, 4, 12 hours of reaction, respectively (: substrate GFP-LPETG; : ligation product GFP-LPETGGGYK-Biotin;: wildtype Sortase A).
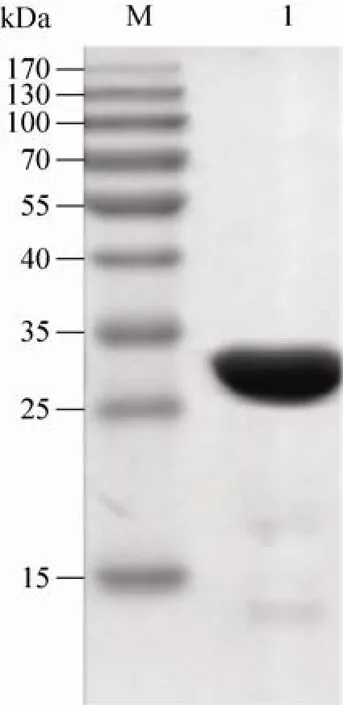
图7 Ni-NTA柱亲和纯化高活力突变型Sortase A酶的SDS-PAGE分析Fig. 7 SDS-PAGE analysis of purified high activity mutant Sortase A protein via Ni-NTA affinity chromatography. M: protein marker; 1: purified high activity Sortase A mutant protein.
2.5 pET-24d(+)-SrtAM载体的构建及突变型Sortase A酶的表达纯化
GFP-LPETG与GGGYK-Biotin为报道底物的连接反应体系中,野生型Sortase A酶仅显示出较低的催化活性,连接反应经过12 h仅完成约30% (图6,泳道6),这与实际应用仍有较大距离。为此,参照Chen等近期报道的高活性突变Sortase A酶序列[25],以pET-24d(+)-SrtA为模板,依次对P94、D160、D165、K196位氨基酸进行定点突变,测序确认定点突变成功,命名为pET-24d(+)-Srt AM。
将pET-24d(+)-SrtAM质粒转化至BL21 (DE3)菌株,诱导表达。与野生型Sortase A酶类似,重组突变型酶主要以可溶形式存在,采用Ni-NTA柱亲和层析纯化,得到高度纯化产物:突变型Sortase A酶大小为26 kDa (图7),符合预期。
2.6 野生型及突变型Sortase A酶连接效率比较
利用已建立的GGGYK-Biotin与GFPLPETG报道底物连接体系 (见2.3),在相同的反应条件下测定并比较野生型与突变型Sortase A酶催化连接产物形成的速率。结果显示,突变型酶具有高催化活性,连接体系中连接产物迅速积累:底物与酶一经混合立即取样便可检出连接产物 (图8,泳道7),至30 min时反应已超过60%,至120 min反应接近完成 (图8,泳道9,12;图9)。与之相比,野生型酶的活性则低得多,反应至60 min,连接产物尚不明显,至120 min反应仅完成20%左右 (图8,泳道1–6;图9)。以上实验结果,确证Chen等报道的突变型Sortase A酶的催化反应动力学较野生型酶大幅提升,可使连接反应在1–2 h接近完成。
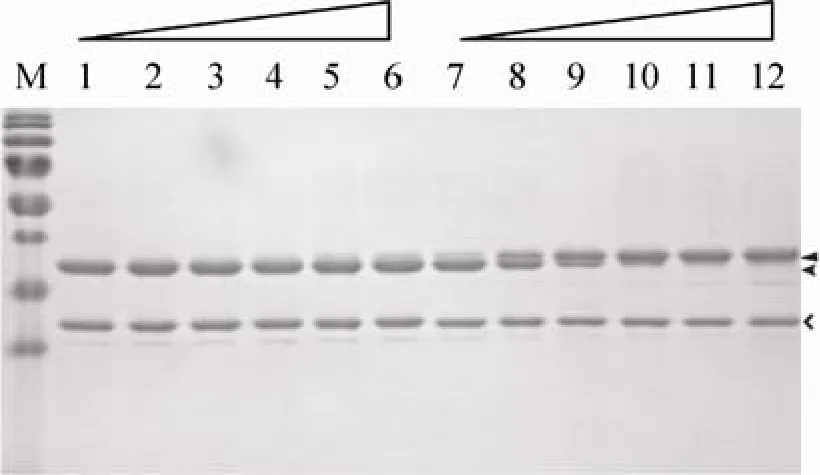
图8 SDS-PAGE比较野生型与突变型Sortase A催化的蛋白质连接效率Fig. 8 SDS-PAGE assay comparing ligation efficiency mediated by wildtype and mutant Sortase A. 1–6: wildtype Sortase A mediated ligation between GFP-LPETG and GGGYK-Biotin, with 0, 10, 30, 60, 90, 120 minutes of reaction respectively; 7–12: mutant Sortase A mediated ligation between GFP-LPETG and GGGYK-Biotin, with 0, 10, 30, 60, 90, 120 minutes of reaction respectively (: substrate GFP-LPETG;: ligation product GFP-LPETGGGYK-Biotin;: wildtype or mutant Sortase A).
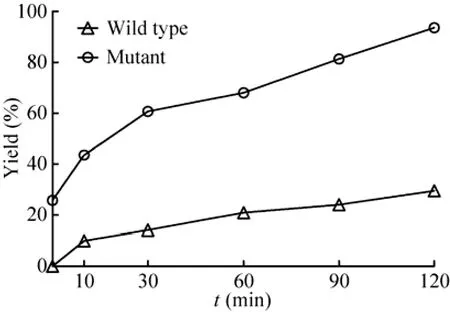
图9 野生型与突变型Sortase A的连接效率比较Fig. 9 Comparison of ligation efficiency mediated by wild type and mutant Sortase A.
3 讨论
Sortase A酶催化的蛋白质连接反应,因其反应条件温和、特异性强,已在生物医药工程领域展现出巨大的应用潜力[21],但天然Sortase A酶催化的连接反应效率较低,影响了它的广泛应用。正因如此,基于Sortase A酶催化特性的定向分子改造已经成为当前的研究热点和重点,较成功的例子有,近期David Liu和Dirk Schwarzer实验室分别在提高酶催化活性及改变底物识别特征等方面取得进展[25–26],有趣的是,筛选得到的关键突变都发生在底物结合区域附近。这些报道显示出Sortase A酶具有广阔的改造潜力,同时也启示,对Sortase A酶的改造重点应聚焦于底物结合区域附近的位点上,通过饱和突变方法辅以系统筛选是比较可行的方案。目前用于检验Sortase A酶突变体催化活性的分析技术,过程复杂且需特殊设备,成为对突变体活性进行系统鉴定分选的主要制约因素,因此迫切需要建立一种简单灵敏的用于监测连接反应进程的方法。
为此,本研究设计了由GFP-LPETG蛋白和GGGYK-Biotin短肽组成的新型报道底物,并利用重组基因工程技术,成功制备GFP-LPETG蛋白,野生型Sortase A酶及Irwin Chen等报道的高活性突变酶。以GFP-LPETG及GGGYKBiotin为底物建立连接体系,并应用于野生型及高活性突变型Sortase A酶催化的连接反应,以证实该检测系统的可靠性。最初在底物中引入生物素分子,是希望通过链霉亲和素分子印迹法来实现高灵敏度检测连接产物的生成;出乎意料的是,GGGYK-Biotin虽与GGGYK仅相差一个生物素分子(226 Da),但连接产物的电泳迁移率相比底物GFP-LPETG却产生明显的降低,使得连接反应进程可直接通过SDS-PAGE凝胶电泳法精确测定。对此现象的一种解释是,连接产物中的Biotin分子侧链具有刚性构象,造成连接产物在SDS-PAGE电泳过程中受阻,致其电泳迁移率与分子量不再呈线性对应关系。连接反应分析步骤的大大简化,将显著促进后续突变体筛选规模的扩大。在实际筛选过程中,尚需对SDS-PAGE凝胶电泳法初步筛选出的阳性样本作进一步验证,预期初筛之后的阳性样本数目将大大减少,可以采用最初设计的链霉亲和素分子印迹法独立确证连接产物的生成效率。
在本研究建立的连接体系下,分析比较野生型与突变型酶的催化效率,证实Irwin Chen等报道的高活性突变酶具有野生酶无法比拟的高催化效率,连接反应可以在2 h左右趋近完成,这在以往的连接反应中是不可能实现的。考虑到很多蛋白质在较高温度 (如37 ℃) 下不稳定,尽可能缩短反应时间对很多活性物质至关重要。对比传统化学连接反应制备过程繁琐、产物不均一以及反应耗时长等缺陷,此突变型酶催化的连接反应展现了明显优势。在本研究基础上,高活性突变酶已被成功应用于蛋白质的固定化及大分子量蛋白质的NMR核磁结构测定过程中 (待发表)。
综上所述,本研究成功开发一种新型的连接反应报道系统,具有简单快速灵敏的特点,可应用于大规模筛选,为后续进一步定向优化Sortase A酶提供了通用简易的筛选鉴定方法。同时,成功制备了一种已报道的高活性突变型Sortase A酶,可广泛应用于各种高效蛋白质连接过程。
REFERENCES
[1] Hackenberger CP, Schwarzer D. Chemoselective ligation and modification strategies for peptides and proteins. Angew Chem Int Ed Engl, 2008, 47(52): 10030–10074.
[2] Song LP, Huang H. Protein splicing and its application. Chin J Bitotech, 2003, 19(2): 249–254 (in Chinese).宋利萍, 黄华. 蛋白质剪接及其应用. 生物工程学报, 2003, 19(2): 249–254.
[3] Luo LX, Shi ZM. Sortase: new target enzyme in Gram-positive pathogens. Prog Biochem Biophys, 2006, 33(9): 828–833 (in Chinese).罗立新, 施周铭. 一种抗革兰氏阳性致病菌新型靶酶—分选酶. 生物化学与生物物理进展, 2006, 33(9): 828–833.
[4] Mazmanian SK, Liu G, Ton-That H, et al. Staphylococcus aureus sortase, an enzyme that anchors surface proteins to the cell wall. Science, 1999, 285(5428): 760–763.
[5] Necchi F, Nardi-Dei V, Biagini M, et al. Sortase A substrate specificity in GBS pilus 2a cell wall anchoring. PLoS ONE, 2011, 6(10): e25300.
[6] Kang HJ, Baker EN. Structure and assembly of Gram-positive bacterial pili: unique covalent polymers. Curr Opin Struct Biol, 2012, 22(2): 200–207.
[7] Spirig T, Weiner EM, Clubb RT. Sortase enzymes in Gram-positive bacteria. Mol Microbiol, 2011, 82(5): 1044–1059.
[8] Mazmanian SK, Ton-That H, Schneewind O. Sortase-catalysed anchoring of surface proteins to the cell wall of Staphylococcus aureus. Mol Microbiol, 2001, 40(5): 1049–1057.
[9] Ton-That H, Liu G, Mazmanian SK, et al. Purification and characterization of sortase, the transpeptidase that cleaves surface proteins of Staphylococcus aureus at the LPXTG motif. Proc Natl Acad Sci USA, 1999, 96(22): 12424–12429.
[10] Huang X, Aulabaugh A, Ding W, et al. Kinetic mechanism of Staphylococcus aureus sortase SrtA.Biochemistry, 2003, 42(38): 11307–11315.
[11] Zong Y, Bice TW, Ton-That H, et al. Crystal structures of Staphylococcus aureus sortase A and its substrate complex. J Biol Chem, 2004, 279(30): 31383–31389.
[12] Race PR, Bentley ML, Melvin JA, et al. Crystal structure of Streptococcus pyogenes sortase A: implications for sortase mechanism. J Biol Chem, 2009, 284(11): 6924–6933.
[13] Ilangovan U, Ton-That H, Iwahara J, et al. Structure of sortase, the transpeptidase that anchors proteins to the cell wall of Staphylococcus aureus. Proc Natl Acad Sci USA, 2001, 98(11): 6056–6061.
[14] Frankel BA, Kruger RG, Robinson DE, et al. Staphylococcus aureus sortase transpeptidase SrtA: insight into the kinetic mechanism and evidence for a reverse protonation catalytic mechanism. Biochemistry, 2005, 44(33): 11188–11200.
[15] Suree N, Liew CK, Villareal VA, et al. The structure of the Staphylococcus aureus sortase-substrate complex reveals how the universally conserved LPXTG sorting signal is recognized. J Biol Chem, 2009, 284(36): 24465–24477.
[16] Kruger RG, Otvos B, Frankel BA, et al. Analysis of the substrate specificity of the Staphylococcus aureus sortase transpeptidase SrtA. Biochemistry, 2004, 43(6): 1541–1551.
[17] Bentley ML, Lamb EC, McCafferty DG. Mutagenesis studies of substrate recognition and catalysis in the sortase A transpeptidase from Staphylococcus aureus. J Biol Chem, 2008, 283(21): 14762–14771.
[18] Tian BX, Eriksson LA. Catalytic mechanism and roles of Arg197 and Thr183 in the Staphylococcus aureus sortase A enzyme. J Phys Chem B, 2011, 115(44): 13003–13011.
[19] Naik MT, Suree N, Ilangovan U, et al. Staphylococcus aureus Sortase A transpeptidase. Calcium promotes sorting signal binding by altering the mobility and structure of an active site loop. J Biol Chem, 2006, 281(3): 1817–1826.
[20] Hirakawa H, Ishikawa S, Nagamune T. Design of Ca2+-independent Staphylococcus aureus sortase A mutants. Biotechnol Bioeng, 2012, 109(12): 2955–2961.
[21] Dasgupta S, Samantaray S, Sahal D, et al. Isopeptide ligation catalyzed by quintessential sortase A: mechanistic cues from cyclic and branched oligomers of indolicidin. J Biol Chem, 2011, 286(27): 23996–24006.
[22] Liew CK, Smith BT, Pilpa R, et al. Localization and mutagenesis of the sorting signal binding site on sortase A from Staphylococcus aureus. FEBS Lett, 2004, 571(1/3): 221–226.
[23] Frankel BA, Tong Y, Bentley ML, et al. Mutational analysis of active site residues in the Staphylococcus aureus transpeptidase SrtA. Biochemistry, 2007, 46(24): 7269–7278.
[24] Nobbs AH, Vajna RM, Johnson JR, et al. Consequences of a sortase A mutation in Streptococcus gordonii. Microbiology, 2007, 153(Pt 12): 4088–4097.
[25] Chen I, Dorr BM, Liu DR. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci USA, 2011, 108(28): 11399–11404.
[26] Piotukh K, Geltinger B, Heinrich N, et al. Directed evolution of sortase A mutants with altered substrate selectivity profiles. J Am Chem Soc, 2011, 133(44): 17536–17539.
[27] Ton-That H, Mazmanian SK, Faul KF, et al. Anchoring of surface proteins to the cell wall of Staphylococcus aureus. Sortase catalyzed in vitro transpeptidation reaction using LPXTG peptide and NH(2)-Gly(3) substrates. J Biol Chem, 2000, 275(13): 9876–9881.
(本文责编 郝丽芳)
A novel reporter system monitoring Sortase A catalyzed protein ligation efficiency
Jian Li1,2, Pengju Wang1,2, Yunfeng Cui2, Peijian Zou2, and Gang Qin2
1 College of Bioengineering, Tianjin University of Science and Technology, Tianjin 300457, China 2 National Engineering Laboratory for Industrial Enzymes, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
Efforts on directed evolution of sortase A to optimize its catalytic properties have been undertaken and shown the promise. To facilitate screening of sortase A mutants with expected catalytic properties, a novel ligation efficiency monitoring system, including reporter substrates GFP-LPETG and GGGYK-Biotin, was developed. GFP-LPETG, wild type sortase A, and a recently reported high activity sortase A mutant were prepared recombinantly from Escherichia coli BL21 (DE3). Taking advantage of the newly designed reporter system, the ligation efficiency catalyzed by wild type and mutant form of sortase A could be sensitively monitored via SDS-PAGE directly. Consistent with previous report, the mutant sortase A displayed much higher catalytic activity compared to wild type enzyme, indicating the new reporter system is easily and fast handled and sensitive. The application of this reporter system into systemic screening will facilitate future directed optimization of sortase A.
sortase A, protein ligation efficency, reporter substrates, GFP-LPETG, GGGYK-Biotin, high activity mutant
April 21, 2013; Accepted: July 29, 2013
Peijian Zou. Tel/Fax: +86-22-24828722; E-mail: zou_pj@tib.cas.cn Gang Qin. Tel/Fax: +86-22-24828722; E-mail: qin_g@tib.cas.cn
李健, 王鹏举, 崔云凤, 等. 检测Sortase A酶催化蛋白质连接效率的新型报道系统. 生物工程学报, 2014, 30(2): 284-293.
Li J, Wang PJ, Cui YF, et al. A novel reporter system monitoring Sortase A catalyzed protein ligation efficiency. Chin J Biotech, 2014, 30(2): 284-293.
Supported by: Program of “One Hundred Talented People” of the Chinese Academy of Sciences (No. KSCW2-YW-BR-4), Tianjin Municipal Science & Technology Project (No. 11ZCZDSY08100).
中国科学院百人计划项目 (No. KSCW2-YW-BR-4), 天津科技支撑计划项目 (No. 11ZCZDSY08100) 资助。
