灰霉病菌抗药位点及其分子检测方法研究进展
2014-06-13马雪梅
张 鑫, 马雪梅
北京工业大学生命科学与生物工程学院,北京100124
灰霉病(grey mould)是一种重要的世界性植物真菌病害,由灰葡萄孢(Botrytis cinerea Pers.)侵染所致。B.cinerea属半知菌亚门,有性态为富氏葡萄孢盘菌(Botryotinia fuckeliana(de Bary)Whetz.),该病菌寄主范围广泛,危害多种蔬菜、经济作物、粮食作物以及观赏性植物。据统计,灰葡萄孢已被列为世界十大真菌病害之一[1]。目前对灰霉病主要以化学防治为主,包括苯并咪唑类(benzimidazoles)、N-苯氨基甲酸酯类(phellylcarbaliates)、二甲酰亚胺类(dicarboximides)、甾醇生物合成抑制剂类(sterol biosynthesis inhibitors,SBIs)、醌氧化抑制剂类(quinone oxidation inhibitors,QoIs)、琥珀酸脱氢酶抑制剂类(succinate dehydrogenase inhibitors,SDHIs)、苯氨基嘧啶类(anilinopyrimidines)和吡咯类(pyrroles)等,但自从使用杀菌剂以来,关于灰葡萄孢对不同作用机制的杀菌剂产生抗药性的报道不断出现,且具有新作用机制的杀菌剂开发缓慢,使化学类杀菌剂的药效面临挑战,导致无法有效控制灰霉病的发生。因此,利用分子生物学方法及早发现并检测抗药菌株的出现,对于灰霉病的防治工作具有重要意义,本文就近年来已研究报道的灰霉病菌抗药位点进行了总结,并对其分子检测方法进行简要综述。
1 抗药位点概述
灰霉病菌(B.cinerea)被杀菌剂抗性行动委员会(Fungicide Resistance Action Committee,FRAC)认定为具有高风险的病原真菌[2],其抗药菌株的出现频率也很高,近年来已报道的与灰葡萄孢杀菌剂抗性相关的主要突变位点如表1所示。

表1 灰葡萄孢抗药位点Table 1 Resistant mutations to fungicides in B.cinerea.
2 分子检测方法
传统真菌杀菌剂抗药性检测方法(菌丝生长法和孢子萌发法)检测周期长,耗时耗力,检出率低,对于抗性频率小于1%的抗药菌很难检出,且对一些离体培养困难的病原菌检测很难。此时,分子检测方法就显示出了很大的优越性,其简单、迅速、准确、灵敏的优点越来越受到广大科研工作者的青睐,如下是目前灰霉病菌抗药位点的主要分子检测方法。
2.1 测序法
随着DNA测序的自动化及成本的不断降低,测序法越来越多的被应用于杀菌剂抗药突变位点的研究[13~16],是确定抗药位点及类型的金标准。该方法主要是通过对不同个体菌株的同一个基因或者基因片段进行PCR扩增,测序后比对基因序列,从而找出变异的碱基。灰霉病菌对SBIs类杀菌剂环酰菌胺(fenhexamid)、SDHIs类杀菌剂啶酰菌胺(boscalid)[17]、二甲酰亚胺类杀菌剂异菌脲(iprodione)[18,19]等的抗药分子机制的研究中均采用过测序法。测序法直观准确,可检测未知的突变位点并做到精确定位,国内外学者经常采用直接测序法或者结合其他方法检测基因变异。但该方法工作量大,耗时耗力,对于大批量抗药菌株的研究则不太适合。
2.2 切割扩增多态性标记法(CAPS)
切割扩增多态性(cleaved amplified polymorphic sequence,CAPS)又称限制性片段长度多态性聚合酶链式反应(polymerase chain reaction-restriction fragment length polymorphism,PCR-RFLP),是检测点突变的常用方法[20,21]。CAPS的检测原理是基于单碱基突变导致的抗药性菌株获得或丧失某些限制性酶切位点,与敏感菌株相比,用限制性内切酶酶切得到的目的片段大小不一,以此来区分敏感性和抗药性菌株。
灰霉病菌线粒体细胞色素b基因(cytochrome b,cytb)G143A位点的突变是病原菌对QoIs类杀菌剂产生抗药性的根本原因[17]。2012年,De Miccolis等[2]在对抗 QoIs类杀菌剂肟菌酯(trifloxystrobin)的分子机制的研究中,采用CAPS方法对细胞色素b基因(cytb)进行分子检测。由于cytb第143位密码子GGT到GCT的改变导致核酸序列产生了一个AluI(5'-AG↓CT-3')的酶切位点,设计特异性引物扩增包含变异位点在内cytb基因片段,然后选用限制性内切酶AluI对PCR产物进行酶切,经琼脂糖凝胶电泳即可将39株灰霉病菌野生型、69株抗药突变体完全区分开。另外,在柑橘和草莓灰霉病菌对QoIs类杀菌剂醚菌酯(kresoxim-methyl)和嘧菌酯(azoxystrobin)的抗药性分子机制的研究中也采用了CAPS的方法[22]。
该方法的优点是操作简单,无需昂贵仪器,但只能做到定性检测,无法定量,且并不是所有突变位点处都有限制酶酶切位点的变化,因此该方法具有一定的局限性。如果酶切反应不完全会影响结果的判断,故经常与测序法同时使用。
2.3 扩增阻滞突变系统(ARMS)
扩增阻滞突变系统(amplification refractory mutation system,ARMS)别名很多,又称 AS-PCR(allele specific PCR)[23,24]、ASA(allele-specific amplification)、PASA(PCR amplifications of specific alleles)和MAMA(mismatch amplification mutation assay)。其基本原理为:Taq DNA聚合酶由于缺少3'→5'外切酶活性,引物3'末端碱基错配会导致PCR产物急剧减少,以此针对野生型和突变型基因设计3条引物,其中2条引物的3'末端分别与正常碱基或者突变碱基匹配,第3条引物为另一侧通用引物,含正常碱基的引物对只能扩增获得野生型菌株的基因片段,而抗药菌株的基因片段无法获得,同理,含突变碱基的引物对只能对抗药菌株进行扩增,最后通过琼脂糖凝胶电泳技术即可区分突变型与野生型基因。
灰霉病菌琥珀酸脱氢酶铁硫蛋白亚基(succinate dehydrogenase iron-sulfurprotein subunit,SdhB)H272Y或H272R处突变是抗SDHIs类杀菌剂的主要原因。2011年,Yin等[25]在针对抗SDHIs类杀菌剂啶酰菌胺(boscalid)的苹果灰霉病菌SdhB基因的H272Y和H272R突变位点进行检测时,设计了一条通用上游引物272Y/R-F,两条等位基因特异性下游引物。针对H272Y突变类型,第272位密码子由CAC(BosS)变为TAC(BosR),其中一条下游引物272Y-R的3'末端碱基为A,与突变碱基T互补;同理,针对H272R突变类型(CAC→CGC),另一条等位基因特异引物272R-R的3'末端碱基为C,与突变碱基G互补。采用272Y/R-F+272Y-R,272Y/R-F+272R-R引物对分别检测66株boscalid抗性菌株(BosR)和106株boscalid敏感菌株(BosS),其中65株抗药菌扩增得到了232 bp的目的片段,而106株敏感菌株和1株抗药菌株未获得相应条带,后经测序表明这1株抗药菌株属于另外一种突变类型(P225F)。由此可见,采用AS-PCR的方法可准确检测包含H272Y或者H272R突变位点的灰霉病菌抗药菌株。此外,作者在1个PCR反应中加入了3条引物(272Y/R-F,272Y-R和272R-R),此多重AS-PCR的方法可同时检测H272Y型和H272R型突变。
采用 AS-PCR 的方法,Bardas等[24]以及 Yin等[26]在对抗 QoIs类杀菌剂唑菌胺酯(pyraclostrobin)的奇异果灰霉病菌、苹果灰霉病菌cyt b基因进行分子检测时,成功检测出G143A型突变。该方法的缺点是容易出现非特异性扩增导致假阳性或假阴性结果。
2.4 四引物扩增阻滞突变系统(Tetra primer ARMS-PCR )
四引物扩增阻滞突变系统是在ARMS-PCR方法的基础上衍生而来。其特点是在突变碱基两侧设计两条外引物及两条等位基因特异性内引物,将4条引物同时加入一个反应体系中,PCR结束后可得到不同长度的特异性扩增片段,经凝胶电泳分析片段有无及大小即可直接区分野生型及突变型[27]。外引物兼具反应阳性对照及与两条内引物分别配对选择性扩增突变型与野生型的作用。2009年,Munoz等[28]采用该方法对抗苯并咪唑类灰葡萄孢E198A位点进行检测。
Tetra primer ARMS-PCR对单核苷酸突变检测具有简便、快速、价格低廉等优点,但需要对引物浓度、内外引物比例、退火温度、Taq酶浓度等各种PCR反应条件进行优化,才能保证该方法的准确性和灵敏性,优化步骤费时费力。
2.5 Real-time AS-PCR
Real-time PCR是在常规PCR基础上加入荧光标记探针或相应的荧光染料,不用进行PCR后处理或电泳检测,可通过荧光强度变化直接进行定性和定量分析。具有操作简便、快速、高效、高敏感性等特点。Real-time PCR已广泛应用于点突变检测[29]。
Real-time AS-PCR是在AS-PCR的基础上在PCR体系中加入SYBR Green荧光染料,利用荧光信号积累实时监测整个PCR反应的进程。在对QoIs类杀菌剂肟菌酯的抗性分子机制的研究中,Real-time AS-PCR表现出很高的特异性,能够在少至0.1 ng的DNA中检测到G143A突变,且检测结果和CAPS及测序法得到的结果一致[2]。但该方法的一个限制因素是可能会出现非特异性扩增,虽然这种限制因素不会影响多态性的检测,但会影响定量的准确性,尤其是对一些低丰度突变基因的检测。
2.6 等位基因特异性探针及引物扩增
甾醇是生物细胞膜的重要组分,SBIs类杀菌剂环酰菌胺抑制甾醇生物合成过程中的3-酮基还原酶的活性,从而影响细胞膜的功能,干扰细胞正常新陈代谢,影响菌体生长,甚至导致细胞死亡[30]。2012 年,Billard 等[31]采用了一种精确、高效且可准确定量的等位基因特异性探针及引物扩增(allele specific probe and primer amplification assay,ASPPAA)方法检测灰霉病菌3-酮基还原酶基因 (3-keto reductase,erg27)F412S、F412V 和F412I三种类型的突变,并对突变型与野生型混合基因组中erg27基因的不同DNA多态性进行了成功检测,灵敏度达1%。
ASPPAA PCR的原理是:利用Taq酶5'→3'外切酶活性,将探针酶切降解,使5'报告基团(FAM)和3'淬灭基团分离,从而荧光监测系统可接收到荧光信号。其特点是针对突变位点设计一条与DNA一条链结合的等位基因特异性引物以及与另外一条DNA链结合的等位基因特异性TaqMan-MGB探针,TaqMan-MGB探针的5'端连接Report基团 FAM,3'端连接 Quencher基团MGB-NFQ(minor groove binder-nonfluorescent quencher)。为减少非特异性杂交,引物和探针之间最多只能有3个核苷酸的重叠(见图1)。为了区别erg27四种类型的等位基因,研究人员共设计了4对引物和探针,每一对组合特异针对一种类型的基因。为了提高特异性,分别在特异性引物的3'末端的3或5个碱基处人为引入一个突变,并以干扰极限值(interference limit,IL)对此方法的非特异性扩增情况进行了分析,结果表明该方法的非特异扩增现象基本可以忽略不计。
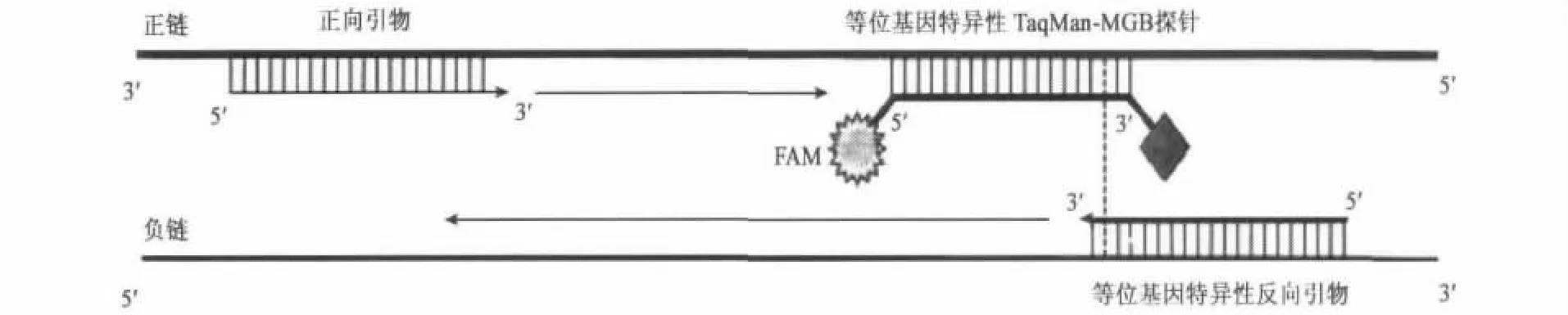
图1 ASPPAA PCR原理示意图[31]Fig.1 Principle schematic diagram of the ASPPAA PCR[31].
相对于常用的AS real-time PCR方法而言,ASPPAA PCR由于包括引物和探针两种特异筛选,其非特异扩增的几率降低至少4倍,这种新的方法可用来鉴定混合DNA中复杂的SNP位点,且错误率小于1%。另一优点是MGB的存在可提高探针Tm值,使高Tm值的探针的长度缩短,便于设计,尤其对富含AT序列的探针设计有很大帮助,且TaqMan-MGB探针3'端的 Quencher基团为非荧光淬灭基团(NFQ),可大大降低荧光本底。此外,TaqMan-MGB探针可以增大配对与非配对模板间的Tm值差异,故TaqMan-MGB探针杂交的稳定性大大提高,实验结果更精确,分辨率更高。但此方法前期需要进行引物及探针的优化,耗费时间较长,成本较高。
2.7 双杂交探针法(又称LightCycler技术)
灰霉病菌抗苯并咪唑类杀菌剂的主要分子机制是由于198位或200位氨基酸发生改变。2008年,Banno等[3]通过荧光杂交探针和熔解曲线分析,建立了一种快速检测抗药突变位点的Realtime PCR法。针对苯并咪唑类抗药菌的突变位点E198A、E198K、E198V和F200Y设计了一对引物TUB-HPF1和TUB-HPR1,其224 bp的扩增产物中包含198位及200位密码子。另外设计两个探针,其中sensor probe(TUB-U27198-Sn)可以和包含突变位点的区域杂交,其3'末端标记有供体荧光基团荧光黄(fluorescein),另一个 anchor probe(TUB-U27198-An)的5'末端标记有受体荧光基团LC-Red 640,当两个探针和PCR产物中的目的序列杂交时,两个基团相互靠近发生荧光共振能量传递,在LightCycler仪器上进行实时PCR和熔解曲线分析,熔解峰对应的温度与 sensor probe和PCR产物间的同源性相关,且不同位置的碱基突变或同一位置的不同碱基的变化都会影响杂交的稳定性。结果显示,携带有 E198A(GAG→GCG)、野生型、E198K(GAG→AAG)、F200Y(TTC→TAC)的BenA基因的不同变化,导致 sensor probe 分别在 68.7℃、62.0℃、59.4℃和57.2℃产生可辨的熔解峰,由此即可判断突变类型。在BenA基因的检测过程中,在63.4℃出现了一个未知的熔解峰,经测序分析,发现了一个新的突变类型E198V(GAG→GTG);编码组氨酸激酶的BcOS1基因突变导致灰葡萄孢对二甲酰亚胺类杀菌剂产生抗药性[6,32]。Banno 等[3]采用该方法对BcOS1基因的三种突变类型也进行了成功检测,包括I365S(ATC→AGC)、V368F(GTC→TTC)+Q369H(CAG→CAC)和 Q369P(CAG→CCG)。
这些数据显示,基于Real-time PCR的杂交探针技术,只要探针设计合适,仅利用一对探针和一对引物即可对苯并咪唑类杀菌剂的多种突变位点进行检测,且同样适用于对二甲酰亚胺类杀菌剂抗药位点的研究。另外,在研究抗QoIs类杀菌剂灰葡萄孢菌株的G143A位点中同样采用了该方法[33]。
相比PCR-RFLP和AS-PCR,双杂交探针法可在一个反应内对多种突变类型进行检测,无需后续任何操作,如限制酶酶切和凝胶电泳,且可检测未知的突变类型。但该方法需要昂贵的仪器及两个探针的合成,成本较高,且如果探针设计不合适,对检测结果的影响会非常明显,Banno设计的sensor probe与BenA E198A基因完全匹配,产生了可辨的熔解峰,而采用与野生型序列完全匹配的sensor probe则未得到可辨的熔解曲线[3],由此可见,探针设计至关重要,直接影响结果的判读,由此也为试验设计带来较大困难。
3 展望
灰霉病菌针对六大类杀菌剂的抗药性分子机制已被报道,包括:苯并咪唑类、N-苯氨基甲酸酯类、二甲酰亚胺类、SBIs、SDHIs和 QoIs,本文总结了与抗药性相关的主要突变位点,共涉及5个基因。随着将来研究的深入,并不排除与抗药性有关的其他突变位点的存在。
目前,有多种分子检测方法应用于灰霉病菌抗药位点检测的研究中,每个方法各有利弊。对于已知靶基因中的未知突变类型的检测只有测序法及双杂交探针法。除了Real-time AS PCR、ASPPAA以及双杂交探针法外,其他方法只能做到定性研究。并且目前的方法只能对每个突变位点进行分别检测或者对同一靶基因不同变异碱基进行检测,不能同时分析对多个靶基因的不同变异位点。
当前迫切需要一种快速、简便、高通量的检测方法,如果能找到一种同时检测多种类型杀菌剂抗药菌株方法,对于灰霉病菌抗药性的早期监测将具有重大意义。
[1]Dean R,Van Kan J A,Pretorius Z A,et al..The Top 10 fungal pathogens in molecular plant pathology[J].Mol.Plant Pathol.,2012,13(4):414 -430.
[2]De Miccolis A R,Rotolo C,Masiello M,et al..Genetic analysis and molecular characterisation of laboratory and field mutants of Botryotinia fuckeliana(Botrytis cinerea)resistant to QoI fungicides[J].Pest Manag.Sci.,2012,68(9):1231-1240.
[3]Banno S,Fukumori F,Ichiishi A,et al..Genotyping of benzimidazole-resistant and dicarboximide-resistant mutations in Botrytis cinerea using real-time polymerase chain reaction assays[J].Phytopathology,2008,98(4):397-404.
[4]Faretra F, Pollastro S. Genetic basis of resistance to benzimidazole and dicarboximide fungicides in Botryotinia fuckeliana(Botrytis cinerea)[J].Mycol.Res.,1991,95(8):943-951.
[5]Malandrakis A, Markoglou A, Ziogas B. Molecular characterization of benzimidazole-resistant B.cinerea field isolates with reduced or enhanced sensitivity to zoxamide and diethofencarb[J].Pestic.Biochem.Physiol.,2011,99(1):118-124.
[6]Ma Z,Yan L,Luo Y,et al..Sequence variation in the twocomponent histidine kinase gene of Botrytis cinerea associated with resistance to dicarboximide fungicides[J]. Pestic.Biochem.Physiol.,2007,88(3):300 -306.
[7]Cui W,Beever R E,Parkes S L,et al..Evolution of an osmosensing histidine kinase in field strains of Botryotinia fuckeliana(Botrytis cinerea)in response to dicarboximide fungicide usage[J].Phytopathology,2004,94(10):1129-1135.
[8]Billard A,Fillinger S,Leroux P,et al..Strong resistance to the fungicide fenhexamid entails a fitness cost in Botrytis cinerea,as shown by comparisons of isogenic strains[J].Pest Manag.Sci.,2012,68(5):684 -691.
[9]De Miccolis Angelini R, Habib W, Rotolo C, et al..Selection,characterization and genetic analysis of laboratory mutants of Botryotinia fuckeliana(Botrytis cinerea)resistant to the fungicide boscalid[J].Eur.J.Plant Pathol.,2010,128(2):185-199.
[10]Laleve A, GametS, WalkerA S, et al.. Site-directed mutagenesis of the P225,N230 and H272 residues of succinate dehydrogenase subunitB from Botrytiscinerea highlights different roles in enzyme activity and inhibitor binding[J].Environ.Microbiol.,2013,doi:10.1111/1462 -2920,12282.
[11]Veloukas T,Kalogeropoulou P,Markoglou A N,et al..Fitness and competitive ability of Botrytis cinerea field isolates with dual resistance to SDHI and QoI fungicides,associated with several sdhB and the cytb G143A mutations[J].Phytopathology,2014,104(4):347-356.
[12]Samuel S,Papayiannis L C,Leroch M,et al..Evaluation of the incidence of the G143A mutation and cytb intron presence in the cytochrome bc-1 gene conferring QoI resistance in Botrytis cinerea populations from several hosts[J].Pest Manag.Sci.,2011,67(8):1029-1036.
[13]Carter H E,Cools H J,West J S,et al..Detection and molecular characterisation of Pyrenopeziza brassicae isolates resistant to methyl benzimidazole carbamates[J].Pest Manag.Sci.,2013,69(9):1040 -1048.
[14]Blum M,Waldner M,Gisi U.A single point mutation in the novel PvCesA3 gene confers resistance to the carboxylic acid amide fungicide mandipropamid in Plasmopara viticola[J].Fungal Genet.Biol.,2010,47(6):499 -510.
[15]Hwang T J,Kim N,Kim H B,et al..Change in antibiotic resistance of Helicobacter pylori strains and the effect of A2143G point mutation of 23S rRNA on the eradication of H.pylori in a single center of Korea[J].J.Clin.Gastroenterol.,2010,44(8):536-543.
[16]Mallik I, ArabiatS, Pasche J S, et al.. Molecular characterization and detection of mutations associated with resistance to succinate dehydrogenase-inhibiting fungicides in Alternaria solani[J].Phytopathology.2014,104(1):40 -49.
[17]Leroux P,Gredt M,Leroch M,et al..Exploring mechanisms of resistance to respiratory inhibitors in field strains of Botrytis cinerea,the causal agent of gray mold[J].Appl.Environ.Microbiol.,2010,76(19):6615 -6630.
[18]Oshima M,Fujimura M,Banno S,et al..A Point mutation in the two-componenthistidinekinaseBcOS-1 gene confers dicarboximide resistance in field isolates of Botrytis cinerea[J].Phytopathology,2002,92(1):75-80.
[19]Fillinger S,Ajouz S,Nicot P C, et al..Functional and structural comparison of pyrrolnitrin-and iprodione-induced modifications in the class III histidine-kinase Bos1 of Botrytis cinerea[J].PLoS One.,2012,7(8):e42520.
[20]Klesiewicz K,Nowak P,Karczewska E,et al..PCR-RFLP detection of point mutations A2143G and A2142G in 23S rRNA gene conferring resistance to clarithromycin in Helicobacter pylori strains[J].Acta Biochim.Pol.,2014,61(2):311-315.
[21]Van der Heyden H,Dutilleul P,Brodeur L,et al..Spatial distribution ofsingle-nucleotide polymorphismsrelated to fungicide resistance and implicationsforsampling[J].Phytopathology,2014,104(6):604-613.
[22]Ishii H,Fountaine J,Chung W H,et al..Characterisation of QoI-resistant field isolates of Botrytis cinerea from citrus and strawberry[J].Pest Manag.Sci.,2009,65(8):916 -922.
[23]De Miccolis A R,Masiello M,Rotolo C,et al..Molecular characterisation and detection ofresistance to succinate dehydrogenase inhibitor fungicides in Botryotinia fuckeliana(Botrytis cinerea)[J].Pest Manag Sci.,2014,doi:10.1002/ps.3748.
[24]Bardas G A,Veloukas T,Koutita O,et al..Multiple resistance of Botrytis cinerea from kiwifruit to SDHIs,QoIs and fungicides of other chemical groups[J].Pest Manag.Sci.,2010,66(9):967-973.
[25]Yin Y N,Kim Y K,Xiao C L.Molecular characterization of boscalid resistance in field isolates of Botrytis cinerea from apple[J].Phytopathology,2011,101(8):986 -995.
[26]Yin Y N,Kim Y K,Xiao C L.Molecular characterization of pyraclostrobin resistance and structural diversity of the cytochrome b genein Botrytiscinerea from apple[J].Phytopathology,2012,102(3):315-322.
[27]Medrano R F,de Oliveira C A.Guidelines for the tetra-primer ARMS-PCR technique development[J].Mol.Biotechnol.,2014,56(7):599 -608.
[28]Munoz C,Gomez T S,Volpe M L.Tetra primer ARMS-PCR for identification of SNP in beta-tubulin of Botrytis cinerea,responsible of resistance to benzimidazole[J].J.Microbiol.Methods,2009,78(2):245 -246.
[29]Oleastro M,Menard A,Santos A,et al..Real-time PCR assay for rapid and accurate detection of point mutations conferring resistance to clarithromycin in Helicobacter pylori[J].J.Clin.Microbiol.,2003,41(1):397 -402.
[30]Debieu D,Bach J,Montesinos E,et al..Role of sterol 3-ketoreductase sensitivity in susceptibilitytothefungicide fenhexamid in Botrytis cinerea and other phytopathogenic fungi[J].Pest Manag.Sci.,2013,69(5):642 -651.
[31]Billard A,Laval V,Fillinger S,et al..The allele-specific probe and primer amplification assay,a new real-time PCR method for fine quantification of single-nucleotide polymorphismsin pooled DNA [J]. Appl. Environ.Microbiol.,2012,78(4):1063 -1068.
[32]Leroux P,Fritz R,Debieu D,et al..Mechanisms of resistance to fungicides in field strains of Botrytis cinerea[J].Pest Manag.Sci.,2002,58(9):876 -888.
[33]Banno S,Yamashita K,Fukumori F,et al..Characterization of QoI resistance in Botrytis cinerea and identification of two types of mitochondrial cytochrome b gene[J].Plant Pathol.,2009,58(1):120-129.
