水稻中一个油菜素内酯不敏感突变体的图位克隆
2014-06-13祝步拓路铁刚
祝步拓, 张 芊, 路铁刚
中国农业科学院生物技术研究所,北京100081
油菜素内酯(brassinosteroids,BRs)是植物体内广泛存在的一类甾醇类激素,在植物根茎伸长、维管束分化、光形态建成、种子发芽、生殖发育和向性建成等生长发育过程中发挥着重要作用,此外油菜素内酯还能提高植物的抗逆性和抗病性[1,2]。对于油菜素内酯作用及信号转导的研究已成为植物科学研究的热点。
基于拟南芥突变体的研究,BRs信号转导途径网络调控机制已基本清晰。通过对BRs不敏感突变体bri1(brassinosteriod-insensitive 1)的研究,克隆到了BRs的受体基因BRI1[3]。此突变体表现为矮小、叶片卷曲、叶色深绿、育性下降且不能被外源施加BRs所恢复。此后通过对BRI互作蛋白的研究进一步发现了 BAK1[4,5]。研究表明,BRs在细胞表面与受体BRI1结合,诱导BRI1和激酶抑制蛋白BKI1解离,同时介导BRI1与另一个跨膜受体激酶BAK1结合,BRI1和BAK1蛋白相互磷酸化,介导BR信号的转导。BIN2和BSU1是BRs信号转导过程中的重要组分,当BRs和BRI1蛋白结合时激活BSU1同时使BIN2失活,从而导致下游蛋白BZR1和BES1去磷酸化而激活,BZR1和BES1在核内积累,直接调控BRs调节相关基因的表达;当BRs和BRI1解离时,BIN2被激活,进而 BIN2磷酸化 BZR1和 BES1,从而导致BZR1和BES1的失活[6]。
水稻是全世界最主要的粮食作物之一,油菜素内酯可直接影响水稻的株型,进而影响水稻的生物量和产量[7]。随着水稻基因组学的迅速发展和一系列水稻BRs相关突变体的发现,越来越多的水稻BRs合成和信号转导的关键元件被克隆和印证。水稻BR合成突变体(d2,dl1,brd1和brd2),表现为矮小、去黄化、卷叶和叶直立等表型[8~11]。这些突变体的表型均是由于BRs的合成途径被阻断导致内源BRs不足造成,可被外源施加BRs所恢复。在水稻中第一个被克隆的BRs信号转导相关基因是OsBRI1。OsBRI1是水稻中可能的BRs受体,与拟南芥BRI1无论在结构还是序列上都有着高度的相似性。他的突变体d61表现为矮化、叶片直立、对BRs的敏感性下降[12]。通过对水稻基因组序列的比对搜索,分离到了OsBRI1的两个同源基因OsBRL1和OsBRL3。它们在水稻根中大量表达,并部分参与BRs的信号传递,与 OsBRI1存在着一定的功能冗余性[13]。通过反向遗传学的方法相继克隆到OsBZR1和OsBAK1,并 进 行 了 功 能 研 究[14,15]。OsGSK1 是BIN2在水稻中的同源基因,并在水稻逆境响应中发挥重要作用[16]。目前研究表明,BRs合成和信号转导关键元件在单、双子叶中的作用非常保守但也存在不同之处,并且还有很多BRs相关调控组分仍不清楚。本实验室在水稻突变体库中筛选得到了一个叶夹角减小突变体,其叶型直立、植株高度略矮并且对外源BRs表现不敏感。通过图位克隆将其定位在了水稻第5号染色的2 004 032 bp至2 704 032 bp区间范围内,物理距离为700 kb。此区间尚无BRs相关基因报道。
1 材料与方法
1.1 水稻材料
本实验中水稻野生型材料为粳稻品种日本晴(Oryza sativa L.spp.Japonica cv.Nipponbare)和籼稻品种Dular。水稻BRs不敏感突变体来源于本实验室构建的 T-DNA插入突变体库[17],遗传背景为粳稻品种日本晴,经自交获得表型稳定纯合突变体el植株。
1.2 表型观察
将日本晴和Dular两种亲本与el突变体种植于田间,各种植10行,每行10株。成熟后分别对植株叶夹角进行测量统计。
1.3 遗传分析及基因定位群体的构建
2011年5月在河北廊坊试验基地种植各亲本材料并套袋自交,同时配置Dular/el和日本晴/el杂交种。收取各系自交亲本和F1代(Dular/el;日本晴/el)杂交种子。2012年春在海南三亚种植各亲本和F1代杂交种子,收取亲本和F2代种子。调查统计各个亲本、F1代和F2代群体的植株形态。对F2代群体进行遗传分离规律分析,确定突变体表型的显隐性关系以及是否为单基因控制。
1.4 基因组DNA提取
采用改进的CTAB法[18]提取植株叶片DNA。对F2代植株,按5株/组,选取2组野生型表型单株,每组剪取等量叶片混合,构建野生表型基因池。
1.5 Indel分子标记分析
通过比较粳稻亚种日本晴和籼稻亚种9311的全基因组序列,利用本实验室曹建等开发的多态性引物对Dular/el F2代隐性表型混池进行初定位[19],再用与该混池连锁的初定位标记对隐性F2代全部单株进行连锁分析,确定引物与F2代野生型表型群体的连锁关系。本研究所用的多态性InDel标记见表1。
PCR 扩增体系(10 μL)为:ddH2O 6.1 μL,10 × PCR Buffer 1 μL,MgCl2(25mmol/L)0.6 μL,dNTP Mix(1mmol)0.2 μL,引物(2 pmoL,正向 + 反向)1 μL,Takara rTaq(5 U/μL)0.1 μL,DNA 模板(20 ng/μL)1 μL。PCR 扩增程序为:94℃预变性5min;94℃变性30 s,56℃退火30 s,72℃延伸30 s,5个循环;第二步,94℃变性30 s,58℃退火 30 s,72℃ 延伸 30 s,33 个循环;最后72℃下延伸7min。反应产物在8%聚丙烯酰胺凝胶上电泳,电泳电压200 V,时间2 h 10min。电泳完成后用银染显色,在胶片观察灯下观察结果并照相。将电泳图谱进行数值转换:与隐性亲本带型一致的记为1型带,与显性亲本带型一致的记为2型带,同时具有双亲本带型的记为3型带。

表1 本研究所用的多态性InDel标记Table 1 Polymorphic InDel markers used in this study.
1.6 遗传作图与基因定位分析
利用Mapmaker软件[20]对转换数据进行分析,并进行遗传作图,对突变基因进行定位分析。
1.7 BR敏感性实验
1.7.1 叶夹角敏感性实验 野生型和突变体水稻种子去皮后用75%乙醇灭菌1min,50%次氯酸钠灭菌20min,更换次氯酸钠一次继续灭菌20min,之后在超净台中用灭菌超纯水洗4次,浸泡30min,再次清洗4次,黑暗处萌发48 h。挑取萌发一致的种子接至1/2 MS培养基中,继续黑暗培养8 d。于黑暗处剪取第二叶夹角上下各1cm的位置,分别用 0 nmol/L、10 nmol/L、100 nmol/L、1 000 nmol/L和10 000 nmol/L 24-eBL(BRs类似物)避光处理48 h,随后进行照相。用ImagineJ测量并统计野生型和突变体的叶夹角。
1.7.2 根敏感性实验 野生型和突变体水稻种子同1.7.1灭菌处理、萌发后挑取萌发一致的种子接至含有 0 nmol/L、10 nmol/L、100 nmol/L、1 000 nmol/L、10 000 nmol/L 24-eBL 的 1/2 MS培养基中,光照培养5 d后进行照相、测量。用ImagineJ对野生型和突变体根的长度进行统计。
2 结果与分析
2.1 el的表型鉴定
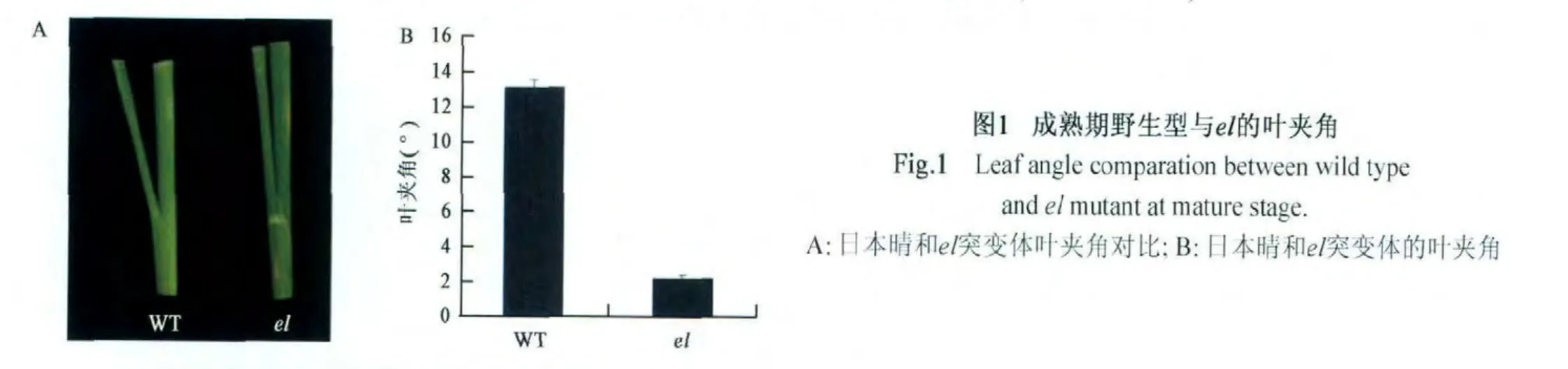
突变体el较野生日本晴植株叶夹角明显减小(见图1A,彩图见封三图版)。对成熟时期植株进行田间测定,日本晴植株叶夹角为13.1±0.2°,而 el突变体为2.3 ±0.1°(见图1B)。

图1 成熟期野生型与el的叶夹角Fig.1 Leaf angle comparation between wild type and el mutant at mature stage.
2.2 el突变体的遗传分析
将el突变体与日本晴进行杂交,获得F1代杂交种子。种植F1代植株全部为叶夹角减小表型。由此表明,叶夹角减小的突变性状受显性基因控制,el为显性突变体。经F1代自交获得的F2代分离群体中,野生型表型96株,突变体表型304株;野生型与突变体的比例符合1∶3的孟德尔遗传理论分离比例(卡方检验P>0.05,无显著性差异)(见表2)。由此表明,叶夹角减小的突变表型由显性单基因控制。
将el突变体与Dular杂交获得F1代,而后F1代自交获得F2代分离群体,对突变位点进行图位克隆。

表2 F2代分离群体的统计分析Table 2 Statistical analysis of the phenotype of F2 segregating population.
2.3 el突变体突变位点的初步定位
用600对Indel引物对亲本Dular和日本晴进行多态性分析,得到151对可以用于Dular和日本晴多态性分析的引物[19]。由于此突变表型为显性突变,选择野生型表型植株进行图位克隆,野生型表型的带型与Dular带型一致。如图2A所示,用上述151对引物对el/Dular F2代群体的2个野生型表型混池(5株/混池)进行分析,发现2个DNA混池与5-2分子标记完全连锁。取20株野生型单株进一步验证了5-2位点的连锁。在5-2上下游合成InDel引物,最终用300棵野生型表型单株将突变位点定位在5号染色体InDel3和InDel4之间700 kb范围内,在Indel3处交换单株为4个,在Indel4处交换单株为6个,与Indel3-1标记共分离(见图2B、2C)。
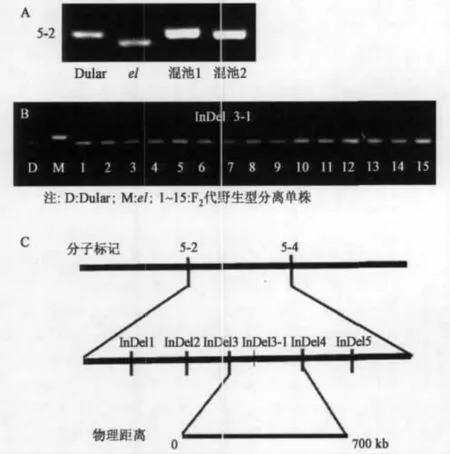
图2 el突变位点粗定位Fig.2 Linkage analysis the mutant locus of el.
2.4 el突变体对BRs不敏感
在水稻中外加BRs通常会直接影响叶片的叶夹角。对el纯合突变体进行叶夹角敏感性实验,结果如图3所示(彩图见封三图版)。在0~10 000 nmol/L不同浓度的24-eBL处理下,野生型的叶夹角分别为 85.2°、155°、157.5°、161.8°和 171.8°;而突变体植株的叶夹角分别为 7.4°、8.4°、11.31°、12.9°和 14.2°。不同浓度的处理下,突变体叶夹角均明显小于野生型,且基本不张开,即对外源BRs没有响应。由此表明el为BRs不敏感突变体。
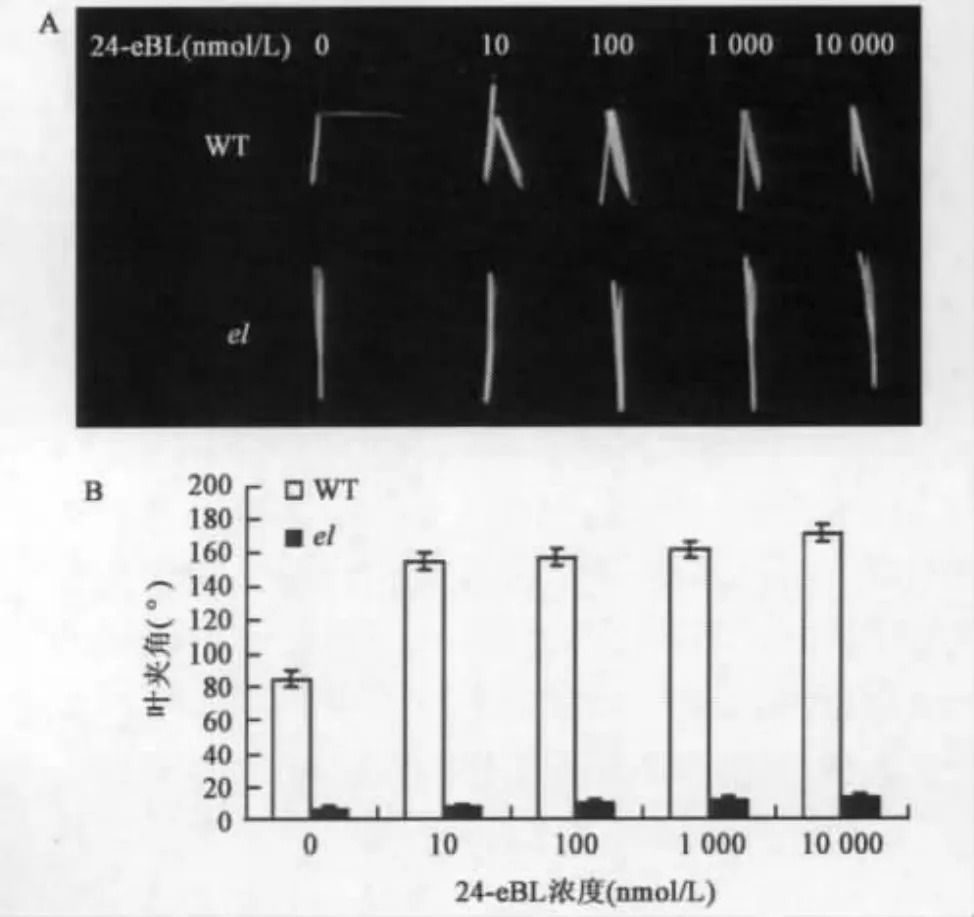
图3 不同浓度外源BRs对el第二叶夹角的影响Fig.3 Lamina joint assay of the el mutant in the presence of BRs in different concentrations.
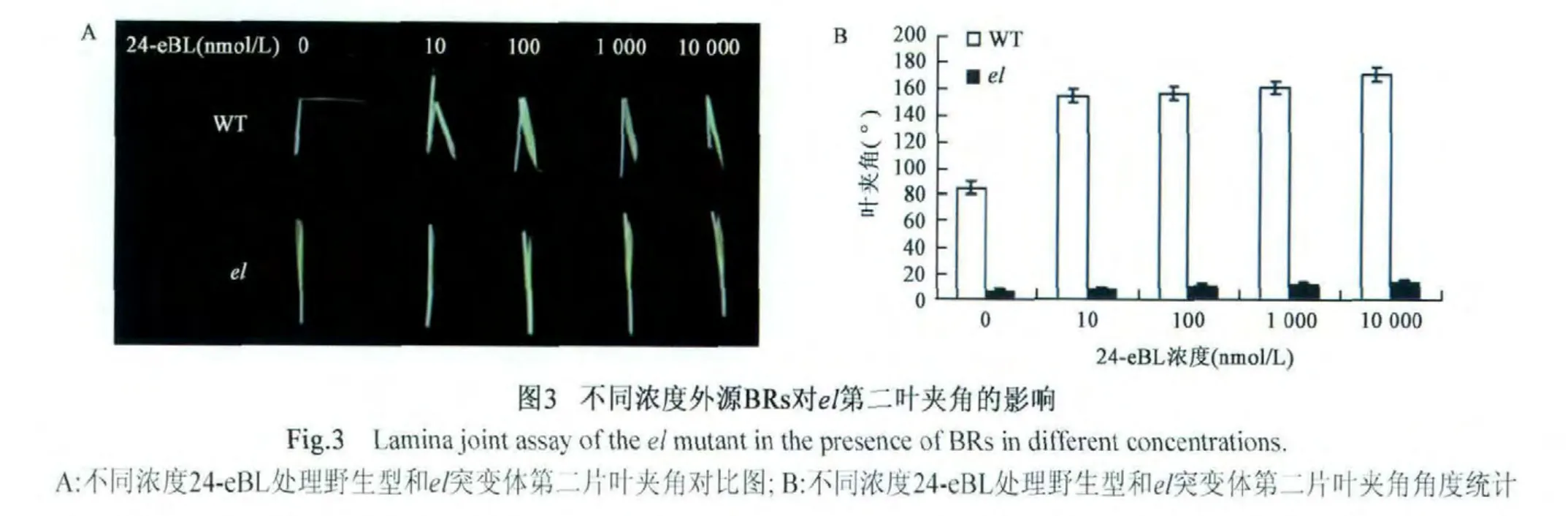
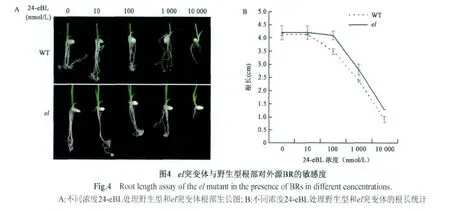
进一步实验检测了植株根生长对外源BR的敏感性,结果如图4所示(彩图见封三图版)。24-eBL可以抑制水稻根的生长,尤其在100 nmol/L以上浓度。突变体根的生长也受到抑制,然而在100 nmol/L以上浓度,24-eBL对突变体根长的抑制程度明显低于野生型。由此可以进一步明确el为BRs不敏感突变体。

图4 el突变体与野生型根部对外源BR的敏感度Fig.4 Root length assay of the el mutant in the presence of BRs in different concentrations.
3 讨论
BRs是一类重要的植物激素,在植物的生长发育以及对外界环境条件的响应中发挥重要作用。BRs的信号转导机制在双子叶植物拟南芥中已经取得重大进展,但在单子叶植物中研究尚不全面。此外,BRs对于提高水稻抗逆性和产量有着密切的关系。因此关于水稻BRs信号转导的研究,有利于进一步明确BRs如何调控水稻的生长发育,为创制水稻理想株型提供理论依据。
BRs不敏感突变体是BRs相关研究的重要材料。目前利用BR不敏感突变体已克隆多个BRs信号转导通路关键基因。水稻对BR最明显的反应就是叶夹角的变化,外源施加微量的BRs就会引起叶夹角增大;而BRs缺失或不敏感都会导致叶片直立。基于这一生理特性,以直立叶夹角为指标筛选水稻突变体,已找到一些水稻BRs合成和信号转导的突变体如 d2、brd1、brd2、d11和d61。本研究从本实验室创制的水稻突变体库中发现了一个叶片直立突变体,经遗传学分析表明此表型为显性单基因控制。且此纯合突变体均表现对外源BRs不敏感:突变体的叶夹角在不同浓度BRs和不同时间处理下均明显小于野生型;此外不同浓度BRs对el的根长抑制程度显著低于野生型(见图3,4)。由此表明此突变体的BRs信号转导通路被抑制,使得水稻无法对BRs信号进行响应。由此可以推断,其叶片直立的表型可能是由于突变体对内源BRs不敏感所致。图位克隆将其突变位点定位在第5号2 004 032 bp至2 713 034 bp区间内。该区间尚无BRs相关基因功能报道。本研究所获得的el为BRs不敏感显性突变体。此研究为克隆水稻BRs信号转导通路关键基因奠定基础。
[1]Fàbregas N,Cano-Delgado A I.Turning on the microscope turret:a new view for the study of brassinosteroid signaling in plant development[J].Physiol.Plantar.,2014,doi:10.1111/ppl.12130.
[2]Krishna P.Brassinosteroid-mediated stress responses[J].J.Plant Growth Regul.,2003,22(4):289 -297.
[3]Clouse S D,Langford M,McMorris T C.A brassinosteroidinsensitive mutant in Arabidopsis thaliana exhibits multiple defects in growth and development[J].Plant Physiol.,1996,111(3):671-678.
[4]Nam K H,Li J.BRI1/BAK1,a receptor kinase pair mediating brassinosteroid signaling[J].Cell,2002,110(2):203 -212.
[5]Li J,Wen J,Lease K A,et al..BAK1,an Arabidopsis LRR receptor-like protein kinase,interacts with BRI1 and modulates brassinosteroid signaling[J].Cell,2002,110(2):213 -222.
[6]Kim T W,Wang Z Y.Brassinosteroid signal transduction from receptor kinases to transcription factors[J].Annu.Rev .Plant Biol.,2010,61:681 -704.
[7]Choudhary S P,Yu J Q,Yamaguchi-Shinozaki K,et al..Benefits of brassinosteroid crosstalk [J].Trends Plant Sci.,2012,17(10):594-605.
[8]Hong Z,Ueguchi-Tanaka M,Umemura K,et al..A rice brassinosteroid-deficient mutant,ebisu dwarf(d2),is caused by a loss of function of a new member of cytochrome P450[J].Plant Cell,2003,15:2900 -2910.
[9]Hong Z,Ueguchi-Tanaka M,Fujioka S,et al..The rice brassinosteroid-deficient dwarf2 mutant,defective in the rice homolog of Arabidopsis DIMIN UTO/DWARF1,is rescued by the endogenously accumulated alternative bioactive brassinosteroid,dolichosterone[J].Plant Cell,2005,17(8):2243 -2254.
[10]Hong Z,Ueguchi-Tanaka M,Shimizu-Sato S,et al..Loss of function of a rice brassinosteroid biosynthetic enzyme,C-6 oxidase,prevents the organized arrangement and polar elongation of cells in the leaves and stem[J].Plant J.,2002,32(4):495-508.
[11]Tanabe S,Ashikari M,Fujioka S,et al..A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant,dwarf11,with reduced seed length[J].Plant Cell,2005,17(3):776 -790.
[12]Yamamuro C,Ihara Y,Wu X,et al..Loss of function of a rice brassinosteroid insensitive 1 homolog prevents internode elongation and bending of the lamina joint[J].Plant Cell,2000,12(9):1591-1606.
[13]Nakamura A,Fujioka S,Sunohara H,et al..The role of OsBRI1 and its homologous genes,OsBRL1 and OsBRL3,in rice[J].Plant Physiol.,2006,140(2):580 -590.
[14]Bai M Y,Zhang L Y,Gampala S S,et al..Functions of OsBZR1 and 14-3-3 proteins in brassinosteroid signaling in rice[J].Proc.Natl.Acad.Sci.USA,2007,104(34):13839 -13844.
[15]Nam K H,Li J.BRI1/BAK1,a receptor kinase pair mediating brassinosteroid signaling[J].Cell,110(2):203 -212.
[16]Koh S,Lee S C,Kim M K,et al..T-DNA tagged knockout mutation of rice OsGSK1,an orthologue of Arabidopsis BIN2,with enhanced tolerance to various abiotic stresses[J].Plant Mol.Biol.,2007,65(4):453 -466.
[17]Wan S Y,Wu J X,Zhang Z G,et al..Activation tagging,an efficient tool for functional analysis of the rice genome[J].Plant Mol.Biol.,2009,69(1 -2):69 -80.
[18]Rogers S O,BendichA J.Extraction of DNA from milligram amounts of fresh,herbarium and mummified plant tissues[J].Plant Mol.Biol.,1985,5(2):69 -76.
[19]曹 建,孙学辉,路铁刚.一份水稻矮杆小粒突变体的形态特征和基因定位[J].生物技术进展,2014,4(3):186-191.
[20]Lander E S,Green P,Abrahamson J,et al..Mapmaker:an interactive computer package for constructing primary genetic linkage maps of experimental and natural populations[J].Genomics,1987,1(2):174 -181.
