海洋生物DNA条形码研究现状与展望
2014-06-01林森杰王路郑连明董云伟柳淑芳丁少雄叶乃好曹文清庄志猛
林森杰,王路,郑连明,董云伟,柳淑芳,丁少雄,叶乃好,曹文清,庄志猛
(1.厦门大学海洋生物多样性与全球变化研究中心,福建厦门 361005;2.中国水产科学院黄海水产研究所,山东青岛 266000)
海洋生物DNA条形码研究现状与展望
林森杰1,王路1,郑连明1,董云伟1,柳淑芳2,丁少雄1,叶乃好2,曹文清1,庄志猛2
(1.厦门大学海洋生物多样性与全球变化研究中心,福建厦门 361005;2.中国水产科学院黄海水产研究所,山东青岛 266000)
海洋生物种类多样,分布广泛,具有复杂性、多样性和趋同性等特点,为了对物种进行更快速、准确地鉴定,急需在传统形态分类学基础上,建立并结合便捷准确的分子鉴定手段。DNA条形码提供了可信息化的分类标准和有效的分类学手段,已成为近年来分类学与生物多样性研究中重要的技术依托。本文概述了DNA条形码当前的发展现状与趋势,并介绍了DNA条形码技术在主要海洋浮游植物(红藻、褐藻、绿藻、硅藻、甲藻)、无脊椎动物(海绵动物、刺胞动物、甲壳动物和软体动物等)和鱼类中的研究进展,以及不同条形码基因针对于不同生物类群的有效性和适用性,指出了目前条形码技术在各海洋类群中存在的主要问题,并对未来的相关工作做了展望,希望为今后我国的海洋生物DNA条形码研究提供理论基础。
DNA条形码;海洋生物;形态分类;分子分类进化;条形码基因
1 引言
面对未来食品能源短缺和环境恶化的危机,广袤的海洋正在成为我们赖以生存与发展的“第二疆土”。在海洋资源中,海洋生物是现今和未来人类所依赖的最主要、最直接的资源,其不仅能够维持近海生态系统结构与功能的稳定性,同时也是重要的食物资源、药源化合物库和能源库,对于维护和谐的生态环境及促进人类发展具有重要意义。物种的快速、准确鉴定是进行海洋生物科学研究和资源保护及可持续性利用的基础。海洋生物门类繁多、分布广泛,有的门类因个体小而外部形态特征不明显(如浮游植物),有的门类在不同环境诱导下存在表型可塑性和趋同进化(converging evolution)现象,从而导致海洋生物隐存种属的普遍存在;而对生物体残片(如动物食道中部分消化的饵料种)、鱼卵仔鱼等也缺乏足够的可供鉴定的形态信息,使得传统形态学分类方法在解决这些问题方面存在着诸多局限性。此外,传统形态分类不仅耗时费力,而且过分依赖研究者的个人经验和观察手段,且随着传统形态分类学家队伍的日益缩减,基于形态学特征的传统生物分类研究已难以对众多的海洋生物进行准确分类。目前急需在传统形态分类学基础上,建立便捷准确的分子鉴定手段,形成适合于海洋生物类群的物种鉴定标准和技术规范。
DNA条形码技术是利用生物共有的、种间差异明显的一段DNA序列来鉴定物种[1],它提供了可信息化的分类学标准和规范的鉴别技术,成为发展最快的前沿学科之一。所用的DNA片段要求长度较短、易于扩增、变异率适度,种间遗传距离明显大于种内遗传距离,以形成相应的条型码间隔(图1)。目标片段还需要具有足够的数据库信息,以便在获得DNA序列后,进行序列比对和人工矫正,构建简易的进化树(邻接法Neighbor-joining)并计算遗传距离来判断未知样品与数据库中已知种类的关系[2]。这一技术使物种鉴定过程实现信息化和标准化,突破了传统形态分类对鉴定者个人能力和经验的过度依赖,并可利用有机体的残片和组织进行快速有效的鉴定,能够在较短时间内建立易于利用的分类系统。概而言之,通过DNA条形码,能够准确地鉴定形态相似种、隐存种以及处于不同生活史阶段的物种,为研究海洋物种提供了海量的、稳定的和可比较的数据,有助于海洋生物在系统进化、遗传变异、群落动态及相互关系等方面的研究[3—5]。
近10年来,国内外很多文章综述了海洋生物条形码的研究概况[4,6-8],但大多聚焦于某一类的海洋生物。为方便参考和比较,本文将整合最近的研究成果,综述DNA条形码技术在海洋浮游植物、无脊椎动物和鱼类中的研究现状,并对DNA条形码技术在未来海洋生物学研究中的应用前景做出展望。
2 DNA条形码技术的起源及发展现状
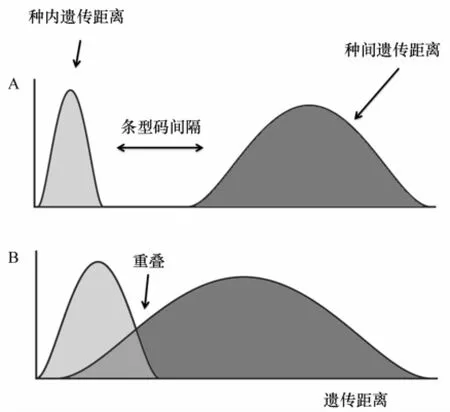
图1 示意DNA片段作为条型码的条件Fig.1 The qualification of a gene/fragment as the DNA barcode marker
由于动物界线粒体(mt)DNA进化速率快于细胞核DNA[9],包括细胞色素B(COB)和细胞色素C氧化酶第一亚基(COⅠ)基因在内的mtDNA早已被用于系统发育和系统地理学研究[10],然而DNA条形码这一概念2003年才由Hebert等首次提出[1]。Hebert等对动物界中除刺胞动物门以外的所有动物门,共11个门13 320个物种的COⅠ基因序列进行分析后发现,其序列间的差异能够很好的区分所有研究物种,并因此认为COⅠ基因适合作为动物DNA条形码基因[11]。此后,越来越多的研究都表明此技术可以进行准确的物种鉴定。目前,利用COⅠ基因片段为标记,已经实现了部分动物类群的条形码鉴定,如蝴蝶[12—15]、鸟类[16—19]和鱼类[20—23]。然而在两栖爬行动物的某些类群中COⅠ基因种内歧化程度高,致使种内与种间的遗传距离发生部分重叠现象,给此基因作为条型码标记带来挑战[24]。在陆生植物、原生生物(单细胞生物)中细胞器基因的遗传变异较缓慢[9],COⅠ种间差异较小,不适合作为DNA条型码,推动了寻找合适的条型码标记的研究。陆生植物的条形码标记DNA探索时间较长[25—27],于2009年的第三次国际DNA条形码学术大会上才确立了以叶绿体rbcL(1,5-二磷酸核酮糖羧化酶/加氧酶大亚基)和matK(叶绿体成熟酶K基因)两个基因片段作为核心的植物DNA标准条形码,用叶绿体trnH-psbA(组氨酸tRNA基因到光合系统Ⅱ反应中心D1蛋白基因之间)的间隔区和核内核糖体RNA基因间隔区(ITS;通常包括ITS1、5.8S、ITS2)为植物DNA条形码的补充条码,并推动了植物条形码的相关项目和组织,如Grass-BOL、Tree-BOL、CFTS Plot和基于植物区系的条形码研究。
随着条形码技术的逐步推进,各类物种传统分类信息和DNA条形码数据相继在网络数据库上发布。截至2013年2月,BOLD(Barcode of Life Data Sys-tems)数据库(http://www.barcodinglife.org/)已收录的DNA条形码序列约250万条,其中具有完整DNA条形码序列(包含准确的物种信息和条形码序列信息)的物种有19万个[28]。很多国际组织建立了自己的DNA条形码数据库,如ABBI(All Birds Barcoding Initiative,http://www.barcodingbirds.org/),All Leps(http://www.lepbarcoding.org/),TBI(Tephritid Barcode Initiative),MBI(Mosquito Barcode Initiative)等。
海洋生物多样性高,形态复杂多变,DNA条型码技术至关重要但其研究相对落后。截至2014年6月,已确认的世界海洋物种数为213 611(WoRMS;http://www.marineSpecies.org),而已被描述形态学特征的物种仅23 712个,远远低于曾经预测的1 000 000个物种[29]。随着DNA条形码技术的广泛应用,海洋生物相关的条形码序列在近几年迅速增多,与此同时海洋中的隐存种和新物种也在被不断地发现和定义。通过十年时间的调查研究,“国际海洋生物普查组织”(the Census of Marine Life,CoML)发现了约1 200个新海洋物种,另外有超过5 000个物种还在被鉴定和描述中(Co ML website,June 2014)。在BOLD与Co ML共同协作下建立的“海洋生物条形码组织”(marine barcode of life,MarBOL),已收录6 199个海洋物种的37 182个条形码信息(MarBOL website,June 2014)。目前该组织正在迅速地壮大发展,努力实现收录5 451个不同海洋物种条形码信息的目标。另外,还有一些针对不同生物群体、生态系统和地理位置的诸多条形码组织如FISH-BOL,Sponge Barcoding Project,SharkBOL,ReefBOL,Polar Barcode of Life等,也在近年来不断的发展壮大[5]。而海洋藻类(浮游植物和大型藻)、浮游动物和底栖动物的DNA条型码研究起步较晚,还未找到国际普遍认可的统一的条形码基因,属于目前海洋生物分类学的研究热点之一。
3 海洋生物研究领域常见DNA条形码基因
细胞核编码的条形码基因主要包括核糖体RNA的18S(the small-subunit r RNA)、28S(the large-subunit rRNA)和ITS(the internal transcribed spacer)。由于核糖体RNA编码基因相对保守,不同区域进化速率各异,适宜于不同分类阶元,核基因一直是分类鉴定和系统演化关系分析的主要依据,其在数据库中的序列信息也较为完善,例如18S的V4和V9区经常被用于新一代高通量测序。而近年来随着ITS在真菌、藻类、陆生植物和某些动物类群中的广泛应用,其在数据库中的序列信息也在迅速扩增,被认为是最具潜力的条形码之一。
相对于植物线粒体基因组,动物线粒体DNA上的基因排列相对紧凑,两个rRNA(12S rRNA和16S r RNA)相邻且没有内含子,22个tRNA位于rRNA和13个蛋白编码基因(细胞色素C氧化酶COⅠ、COⅡ、COⅢ;ATP酶亚基ATPase6、8;COB;以及NADH的7个基因ND1-ND6,ND4L)之间。基于长度和进化速率的考虑,线粒体的13个编码基因中只有COⅠ、COB符合要求,其中COⅠ因其5’端(COⅠ-5P)区域更容易设计较保守的通用性引物,且很少出现内含子和插入缺失现象,被认为是许多鸟类、昆虫和鱼类等的理想DNA条形码。目前海洋动物常用的标准条形码基因也以COⅠ为主,且该基因在一些海洋藻类(红藻、褐藻)中也得到了尝试和应用,但在珊瑚、海葵、海绵等特殊群体中由于COⅠ的进化速率过慢,仍然需要寻找和评估其他的条形码标记。
除陆生植物外,叶绿体水平上的条形码研究主要集中在藻类,常见基因包括rbcL、UPA(universal primer-based amplicon或通用引物扩增产物,是基于叶绿体的核糖体大亚基的RNA基因)、tufA(叶绿体延伸因子)、rbcSp(Plastid Rubisco operon spacer)等。其中rbcL长约1 400 bp,在红藻和硅藻中都有研究;UPA片段是叶绿体23S rRNA基因的V结构域,长约370 bp,由于其具有较高的通用性和分辨力,被认为适用于红藻的条形码研究;而tufA的高扩增率和无内含子被认为适合大型绿藻的鉴定分析。
4 海洋藻类DNA条形码研究进展
海洋藻类的DNA条形码研究主要集中在硅藻、红藻和褐藻上,目前核基因ITS和28S rDNA的D1-D3区,叶绿体基因rbcL和UPA[30],线粒体基因COⅠ和COB等已在硅藻[31—33]、甲藻[34—36]等单细胞藻类、以及大型藻[33]中先后被应用,这些基因在不同的物种中各有优势,很难找到像动物的COⅠ序列那样可以在物种鉴定中较通用的条形码,目前尚未发现适合藻类的统一条型码基因,现阶段的研究以两个或两个以上的条形码基因共同使用为主。
4.1 红藻及褐藻DNA条形码的研究进展
红藻门是大型藻中种类最丰富的一个门类,其形态在种内和种间具有高度可变性,且易受环境影响,生活史和生殖方式都很复杂,使得形态学分类非常困难[37-40]。大型红藻已研究的DNA条形码包括COI基因、UPA基因、28S rDNA、rbcL基因等,其中COI和rbcL被认为较适合作为红藻门的条形码基因[33,41],而UPA的种间差异度理想,也在很多类群中得到认可[31]。Saunders等[37—38,42]利用COI基因成功区分了形态上难以分类的三大混合种,并发现了加拿大地区的新物种和外来入侵种,认为COⅠ适合作为此类群的条形码基因。Tan等对同一群体的不同区段进行比较,也认为COⅠ和rbcL更适合作为整个红藻门的条形码基因[43]。但也有数据表明rbcL并不适合红藻门的分类鉴定,Hind和Saunders对珊瑚藻属Chiharaea中的3个种和2个新变种进行研究,发现rbcL序列的种间差异性和遗传变异速率与COⅠ-5P和ITS不同,可能有杂交或基因渗入现象发生[44]。
褐藻的形态差异很大,Clayton将褐藻纲Phaeophyceae分为15个目,从小型的丝状体到全长可达60 m的巨藻[45—46]。目前褐藻的分子系统学研究主要集中在r RNA基因、COⅠ、rbcL和rbcS(二磷酸核酮糖羧化酶/加氧酶的小亚基)序列分析上。对于期待值较高的COⅠ基因,已有的研究结果显示此基因能够准确地区分墨角藻属Fucus[47]和昆布科Laminariaceae[48]中的不同物种;Mattio和Payri在针对马尾藻属不同条形码基因的分析时,也暗示了线粒体基因mtsp(mitochondrial targeting signal peptide)、COⅠ和COⅢ作为褐藻条形码基因的可能性[49]。但是在翅藻属Alaria中,Lane等发现单一的COⅠ基因在此类群的种间差异不足,而COⅠ-5P、ITS及叶绿体基因rbcSp 3个基因片段的组合则能更有效地区分大部分种类,但作者也认为在特殊种群中的欠缺,可能不足以影响COⅠ作为褐藻条形码基因的普遍适用性[50]。Poong等比较了几个褐藻属的rbcL和COⅠ基因,发现虽然COⅠ相对于rbcL无法解决不同类群的系统进化关系,但仍能用于该类群的物种鉴定,所以两个基因相结合才能准确的鉴定该类群并反应它们的系统进化关系,相对于红藻的DNA条形码技术,褐藻相关方面的研究尚显薄弱,需要进一步的发展[51]。
4.2 绿藻DNA条形码的研究进展
目前已有的藻类基因序列数据库中,绿藻的序列信息仅占6%左右,已尝试过的条形码包括COⅠ、rbcL、tufA、ITS等。其中tufA在鉴别海洋大型绿藻时扩增效率高(除刚毛藻属)、种间差异度大、很少存在内含子,被认为更适合用于绿藻的分类鉴定,而rbcL在部分种群中明显存在的内含子严重阻碍了此基因作为理想条形码的可能性[52—53]。尽管在某些群体(Pseudomuriella)中COⅠ表现出明显的种间差异性,却很难找到合适的通用引物[53]。另外ITS区域也在绿藻群体中表现出一定潜力,例如5.8S+ITS2区域能明确地区分小球藻属Chlorella的已知种并藉此发现新种[54],并成功地鉴定了日本沿海本土和引入的典型绿藻物种[55]。刚毛藻科作为绿藻中的特殊群体,除ITS有一定(较低)的成功率外,其他基因都无法区分其不同物种[52],最近研究也发现ITS和18S r DNA序列种间差异明显大于种内差异,似乎可以用于作为刚毛藻科物种分类的辅助基因[56]。
4.3 硅藻DNA条形码的研究进展
硅藻是海洋浮游植物中生物量最高的类群之一,承载着大约20%的光合固碳量,在海洋和淡水中分布广泛,然而对其多样性和地理分布情况我们还知之甚少,急需利用简单快捷的条形码技术对该群体做更深入的研究。
目前尚未找到合适的、统一的硅藻DNA条形码基因。尽管早先的研究有认为COⅠ的种间差异明显,适合作为部分硅藻如鞍型藻属Sellaphora的条形码标签[57]。但越来越多的研究表明rbcL、28S和ITS基因更具作为硅藻条形码基因的潜力[58],而COⅠ尽管种间差异明显,但在某些群体中很难获取和测序[59]。目前研究认为rbcL-3′端(3P)扩增测序效率极高,出现内含子的概率较小,适合作为硅藻的首选条形码[60],而对于双基因(double-gene)条形码技术所需要的次选条形码,较合适的两个基因包括ITS区和28S区。例如,MacGillivary和Kaczmarska在对比ITS、18S和rbcL-3P后建议将rbcL-3P与变异率较高的5.8S+ITS2区域搭配使用[61];Momiz等发现ITS区域能快速准确地区分硅藻纲Mediophyceae与Bacillariophyceae等类群的不同种类[61-62]。而Hamsher等在Sellaphora群体中尝试不同基因后认为28S更适合作为次选条形码基因[63],且在中心藻纲[64]和骨条藻属[65]的研究也发现28S似乎效果最佳。另外,Zimmermann等对18S的V4区(约400 bp)进行了评估,发现此区域能有效区分不同种,包括区分鞍形藻属Sellaphora中的混合种,反映了高通量测序中利用18S的V4区研究硅藻生物多样性的可靠性[66]。
4.4 甲藻DNA条形码的研究进展
甲藻具有真核生物中最大的核基因组,而因为大部分的基因水平转移到细胞核内的缘故其线粒体与叶绿素体中却包含了最少的基因。核基因中存在大量的复制现象,而不同拷贝之间由于长期进化与复制的原因一般具有微小的差异[67—69],由此产生的基因组内多态可能影响条形码技术在此类群中的应用。针对甲藻的条形码研究,起步较晚且相对复杂,我们不但要筛选合适的条形码基因,还要避免某些条形码基因多态性对测序分析、引物设计及多样性评估所带来的干扰。
目前已被尝试过的条形码基因包括ITS、28S(D1 -D3)、COⅠ和COB等,其中ITS和28S似乎更适合作为此类群的条形码基因,例如Stern等利用ITS对甲藻中的78个物种进行了研究,认为它可以有效地区分甲藻中的绝大多数物种,但研究中普遍存在的基因组内多态会影响这一标记基因在现场样品中的应用,需要同其他条形码基因辅助使用[36]。此前Litaker等利用ITS区分析了甲藻14属不同物种的种内种间遗传距离,发现当两条ITS序列的遗传距离(p-value)大于等于一个临界值时(p≥0.04)即可认为是不同的种,证明了ITS作为甲藻条形码基因的巨大潜力[34]。Gu等则利用28S结合形态学特征在中国沿海发现并定义了新种Takayama xiamenensissp. nov.[70],同时利用28S和ITS成功地分析了黄海海域Gymnodinium中3个种的进化关系[71]。
近年对COⅠ和COB的分析结果也证明了核基因ITS作为条形码基因的优越性。Lin等研究认为线粒体COB比COⅠ更适用些,其应用发现美国长岛湾的甲藻具有很高的多样性,甚至包括以前只在极地发现的极冰甲藻Polarella glacialis类群,但COB的种类分辨率尚不理想,且存在引物通用性不强、数据库序列稀缺等问题[35]。Stern等利用COⅠ鉴定了甲藻15个属中的不同物种,但仍无法解决一些分类中常见的问题属,如Alexandrium,Symbiodinium和Protoceratium等[71]。Raho等尝试利用COⅠ和COB作为区分鳍藻属(Dinophysis)的条形码基因,发现相对于分辨力较差的核基因和COB而言,COⅠ表现出了更强的种间差异性,但也无法有效区分鳍藻属中的所有物种[72]。
利用不同基因尝试解决形态上较难区分的种属,近年来在一些重要甲藻类群中取得了一定成效,例如利用28S、18S和ITS将包含有3个形态种的亚历山大藻塔玛复合群(Atama species complex)重新划分成了遗传差异显著的5个新种[2,73—74],并提出ITS相对于18S和28S能更明确地区分此类群,且普遍存在的多态型并不会影响不同种类的准确鉴定[2]。在珊瑚白化过程中生态意义巨大的虫黄藻Symbiodinium也是甲藻分类中典型的问题属,Pochon等发现除ITS区外还有3个基因(COⅠ,rad24,actin)也适合作为虫黄藻属的辅助条形码基因[75];LaJeunesse等也利用ITS、COB、23S r RNA和单拷贝微卫星Sym15基因结合形态生理学等数据定名了Symbiodinium minutumsp.nov.及S.psygmophilumsp.nov.两个新种[76]。
4.5 藻类DNA条形码技术应用的初步框架与展望
目前对于除上述之外的其他门类的微藻研究尚少,但也有一定进展[77]。Saunders和McDevit提出了针对于海洋大型藻类和硅藻的不同条形码基因和标准操作流程,并建议用双条形码方法对藻类进行准确鉴别,即在28S D2/D3作为通用辅助条形码的前提下,COⅠ-5P作为红藻与褐藻基本条形码,rbcL-3P和tufA分别作为硅藻和绿藻的基本条形码[33]。另外,根据已有研究[2,35,72]和未发表数据我们建议用ITS作为甲藻的首选条形码,Saunders等建议的藻类通用辅助条形码28S D2/D3也适用于此群体。
虽然现在涉及藻类的DNA条形码的研究组织还较少,且初步的应用框架并不适用于所有藻类,但相信随着物种的发现和条形码研究的深入,会有越来越多的藻类的DNA条形码信息被录入数据库供研究者参考使用,针对藻类的标准操作流程和应用体系也会被逐步完善。藻类DNA条形码的研究也将会成为藻类物种分类鉴定和系统分化研究中的一种发展趋势,为藻类的分类鉴定和新物种的发现、生物多样性的评价等研究提供准确、快速的信息,还可以用于对外来入侵物种的评估及外来物种的识别等研究[78—80]。
5 海洋无脊椎动物类群的条形码研究进展
5.1 海绵动物DNA条形码研究进展
海绵动物被认为是最原始的后生动物类群,在海洋生态系统和生物化工制药中具有重要地位[81]。目前已被描述的物种有8 000多个,而初步预测全世界至少有15 000个种[82]。由于形态结构(如骨骼、骨针的排列方式)的高度相似性,分类学家很难进行物种鉴定,因此它也是唯一建立全球性条形码计划(Sponge Barcoding Project,http://www.spongebarcoding.org)的无脊椎动物类群。然而已有的研究显示,在较高等的后生动物类群中普遍适用的COⅠ-5P区在海绵动物中保守性较高,很难区分同一属内的不同种,并不适合作为此类群的条形码标记,相反COⅠ下游的I3-M11区[83]和ITS区[84—85]被证明更适合作为海绵动物的条形码片段。
5.2 刺胞动物DNA条形码研究进展
刺胞动物门包括珊瑚纲、水螅纲、钵水母纲、十字水母纲和立方水母纲五大类群。由于珊瑚纲生物(海葵、珊瑚和海鳃等)的线粒体DNA进化速率较低,COⅠ被证明不适合作为物种鉴定的分子标记,使得目前刺胞动物门DNA条形码标记尚未统一[5,86]。Moura等应用16S片段分析了北大西洋6科68种水螅,证实了其在水螅物种鉴定中的适用性,同时还发现16S片段在种以下分类阶元及属、科水平上均具有较好的分辨力[87]。然而最近也有研究结果显示,利用COⅠ可以成功地对浮游水螅类进行分类[88]。另外,以COI[89-91]和16S[92]为标准基因进行水母类的DNA条形码研究已获得广泛应用,并被认可为此类群的标准条形码基因。
5.3 甲壳动物DNA条形码的研究进展
甲壳动物是海洋浮游动物中最为丰富的类群,其条形码研究相对完善,COⅠ片段被认为可作为理想的标准基因。Radulovici等扩增了加拿大湾端足、十足、磷虾、等足和糠虾等5目39科60属87种507个个体的COⅠ序列,发现95%的序列可对应到已鉴定种类,种间遗传距离超过种内遗传距离的25倍,另有4个种类其种内遗传距离较高(3.78%~13.6%),显示出隐种存在的可能[93]。在小型甲壳动物桡足类中,COⅠ也被证实适合作为此类群的标准条形码基因[88,94—96],但也有研究在比较了COⅠ和12S srRNA后推测12S可能更适合作为隆剑水蚤科Oncaeidae的条形码基因[97]。
磷虾目约包含90多个种,分隶于2科11属,均为海洋浮游种类,其鉴定所依据的形态特征比较细微,尤其是幼虫和幼体的鉴定给多样性的准确评估带来困难。COⅠ在此类群的种间差异明显,通过遗传距离的统计可以有效地研究近缘种的系统关系,地理遗传差异及隐存种的预测[95,98]。
5.4 软体动物DNA条形码的研究进展
软体动物种类较多,现存约8万多种,为动物界第二大门类,DNA条形码技术目前已经在腹足纲[99—102]、双壳纲[103—105]、头足纲[106—108]等的分类中得
到广泛应用,但在单板纲、多板纲、无板纲及掘足纲等中研究较少。目前在软体动物研究中广泛使用的DNA条形码是COⅠ基因,但线粒体基因自身还存在单亲遗传、异质性、基因渗入等问题,还需要结合16S和ITS等作为辅助基因。因此,DNA条形码在此类群的进一步研究还需要和形态学、生态学等其他特征结合使用[109—110]。Liu等利用COⅠ和ITS基因对中国沿海的16个及日本沿海的一个栉江珧群体进行分析,探讨隐存种的存在以及个体之间是否发生杂交,通过COⅠ系统进化分析发现了6个明显分化的支系,而通过ITS系统发生分析发现了5个明显分化的支系,此外还在同一个体中发现多种ITS序列,表明支系间曾发生过杂交,此结果也显示了DNA条形码在历史演化研究中的重要作用[111]。此外借助DNA条形码还可以发现一些具有表型差异,但不具有遗传差异的现象。如Nakano发现在原有分类方式中命名为N.helmsi(E.A.Smith,1894)及N.virescens(Oliver,1926)的两个种均属于N.elongata(Quoy&Gaimard,1834)[101];Teske等研究发现非洲东南部潮间带4种同时分布的帽贝Siphonariidae为不同表型的同一种帽贝[112]。
5.5 原生动物DNA条形码的研究进展
原生动物虽然结构简单,比较原始,但种类多、数量大、分布广,在海洋生态系统中占有相当重要的位置。当前原生动物的分类学研究多集中于应用18S和28S rDNA等片段分析其系统关系[113—116]。
目前的DNA条形码研究还仅限于某些类群,例如,COI能准确区分纤毛虫属Tetrahymena中的不同群体,适合作为纤毛虫类标准条形码基因[117]。Santoferrara等则比较了18S、28S及18S高变异区(V4和V9)等4个片段,结果显示28S有效鉴定出了86%的形态类型,可作为沙壳纤毛虫目Tintinnida的有效条形码标记[118]。结合形态特征和基于18S等基因的系统学分析,已鉴定出了多个新属和新种[119—120],同时ITS1-5.8S-ITS2片段也成功地用于分析盾纤虫属Aspidisca的系统关系[121]。
5.6 栉水母DNA条形码的研究进展
栉水母动物分布广,较为常见,目前已发现160余种,其中瓣水母作为入侵种,在地中海海域曾大规模聚集,在海洋浮游动物中也占有相当重要的位置。然而,由于遗传差异较大,该类群尚未有DNA条形码的相关研究。Ortman分析了20种栉水母的COⅠ、ITS1和28S序列,发现COⅠ、28S片段进化速率较低,而ITS1也缺乏足够的种间遗传距离。因此,该类群的条形码研究工作有待寻找更为适合的条形码DNA片段[122]。
5.7 毛颚动物DNA条形码的研究进展
毛颚动物是海洋动物中结构特殊、分类位置尚待确定的一个类群,共鉴定超过100种,其COⅡ和COⅠ片段均被证明可作为条形码研究的标准片段,但后者更被广泛采纳。Peijnenburg等利用COⅡ序列将来自大西洋、地中海和黑海的86个毛箭虫Sagitta setosa个体归类成4个种群,并推测可能存在1个隐种[123]。另外,COⅠ也被成功的用于毛颚动物的种类鉴定、隐种的发现、种群结构和遗传多样性等方面的分析[124—126]。
5.8 其他类群无脊椎动物DNA条形码的研究进展
轮虫的种类鉴定常依赖于实验室培养条件下的显微观察,而对其表型与生殖隔离、环境变化的关系尚不得而知,因此,利用DNA条形码技术对该类群的隐存种及种群分布进行研究具有重大意义。Birky以COⅠ片段分析了多个轮虫类群,指出该片段在轮虫中有着理想的进化速率,可作为该类群条形码的标准基因[127]。
多毛类大多营底栖生活,其幼体须经过浮游阶段,主要分布于沿岸低盐水域,也有少数类群终生浮游。近年来研究发现,过去认为的许多广布种类实际上可分为诸多近缘种,Carr等应用COⅠ分析了加拿大周边太平洋、大西洋和北冰洋三大海域的多毛类多样性,发现属间类群的遗传距离超过属内的40倍,指出该片段是有效的物种鉴定工具,有助于物种的准确识别[128]。
作为阶段性浮游生物,浮游幼虫有着较强的扩散能力,是一种重要的资源补充途径。对于缺乏必要形态分类信息的浮游幼虫,DNA条形码的应用使得该类群生物的物种多样性评估水平显著提高。由于海洋浮游幼虫涵盖了多毛类、甲壳动物、棘皮动物、软体动物和纽形动物等诸多类群,一般使用两个或两个以上条形码基因能更准确进行物种鉴定。例如,结合COⅠ和16S片段成功鉴定出了新西兰海域24种浮游幼虫[129]和香港海域的网采浮游虾蛄幼体[130];Heimeier等结合16S和18S序列共鉴定出53个分子操作分类单元(molecular operation taxonomic Unit,MOTU,即最低可能分类学单元),极大地提高了浮游动物幼虫的鉴定水平[131];Webb等比较了COⅠ、16S和18S后发现COⅠ的扩增成功率最高,比较适合作为首选片段,以促进浮游幼虫多样性的评估[132]。
另外,随着全球贸易的进一步发展,压舱水携带入侵生物排放的问题日益加剧。对许多海洋生物类群,休眠卵的存在使其得以渡过不良环境,保持种群生存,同时也赋予它们跟随压舱水扩散至陌生水域的能力。Briski等应用16S和COⅠ片段分析了五大湖区压舱水带入的休眠卵,其中约64%的个体鉴定到种,其余36%的个体到属或者科级别,证实了使用DNA条形码可以快速检测出压舱水中入侵浮游动物的休眠卵[133]。
5.9 海洋无脊椎动物DNA条形码研究存在的不足
总的来说,海洋无脊椎动物的DNA条形码研究工作已经在物种鉴定、隐种和亚种甄别、种群系统地理学等方向逐步展开。但是,值得注意的是,当前海洋无脊椎动物各主要类群的条形码研究进展极不平衡,在研究较为热门的类群,比如甲壳动物、软体动物和毛颚动物,其标准基因基本确定,相应的应用工作也广泛展开;在原生动物中,基于18S和28S基因的系统学分析和新种鉴定已获得广泛共识,其相关的条形码工作已具备坚实的基础;而在海绵、珊瑚、栉水母、浮游幼虫等类群,由于形态鉴定难度大和线粒体基因进化速率的异常,使得研究者们一直在努力寻求更适合的条形码基因。另外,作为脊索动物的某些类群(文昌鱼纲、海樽纲等)序列信息的过于稀少,也严重影响了此类群条形码研究进程[6]。
6 海洋鱼类DNA条形码研究进展
鱼类是脊椎动物中最具多样性的类群,几乎占了脊椎动物总数的一半。截至2013年10月,FishBase记录的鱼类多达32 700种(http://www.fishbase. org/)。由于鱼类早期发育过程复杂多样且“同种异形”和“异种同形”现象普遍存在,加大了传统形态学分类鉴定的难度,另有许多鱼类的早期发育过程非常类似,其卵子、幼体形态特征种间差异不显著,从而导致了鱼卵仔稚鱼分类鉴定难度较大,DNA条形码的研究则为解决这些难题提供了重要保障。近年来人们甚至尝试用水体中的DNA(掉入水体中的体表细胞或分泌物)来检测鱼类的种群分布和活动范围[134],使条形码技术成为鱼类迁徙、洄游等相关研究的便利工具。
6.1 国际数据库平台收录鱼类DNA条形码进展
在短短10年时间内,鱼类DNA条形码研究取得了长足进展和巨大成就。截至2013年10月,BOLD数据库收录了近17.75万条辐鳍鱼纲(Actinopterygii)鱼类的DNA条形码序列信息,隶属于46个目1.48万种(见表1)。其中DNA条形码序列数最多的类群为鱼类中最大目——鲈形目(Perciformes),占辐鳍鱼纲鱼类DNA条形码总数的44%。位居第二和第三的分别是鲤形目与鲇形目,分别占辐鳍鱼纲鱼类DNA条形码总数的10%和6%。BOLD数据库中的鱼类DNA条形码采自全球95个国家和地区,其中贡献最大的国家是美国,提交了1.3万余条序列;而中国提交的鱼类DNA条形码序列数仅为1 354条,居第16位(表2)。

表1 2013年10月为止BOLD数据库收录的46个目鱼类DNA条形码序列数量Tab.1 DNA barcoding sequences of 46 orders of fish in the BOLD database as of October 2013

表2 2013年10月止为BOLD数据库提交鱼类DNA条形码序列排名前20位的国家和地区Tab.2 The top 20 countries and regians that provided most DNA barcoding sequences of fish in the BOLD database as of October 2013
由BOLD发起的鱼类DNA条形码计划(Fish Barcode of Life,FISH-BOL),目标是获取全球3万多种鱼类的DNA条形码信息并建立鱼类DNA条形码信息网(http://www.fishbol.org/)。当前FISHBOL已收录了9万多条鱼类DNA条形码,隶属于6个亚纲的1万种鱼类(表3),完成了三分之一的工作目标。BOLD数据库与FISH-BOL数据库收录的鱼类DNA条形码信息将极大地方便科学家系统而准确地掌握鱼类在分类地位、遗传结构、分布地区和进化历史等方面的信息,为开展鱼类种质资源研究和生物多样性保护工作奠定了坚实的基础。

表3 2013年10月止FISH-BOL数据库收录的鱼类DNA条形码序列数量Tab.3 The number of DNA barcoding sequences of fish documented in the FISJH-BOL database as of October 2013
6.2 鱼类DNA条形码的应用
COⅠ基因是鱼类DNA条形码研究最常用的标记,被广泛应用于物种鉴别和多样性分析。目前BOLD数据库收录的鱼类DNA条形码信息基本上都来自于此基因。Ward等利用COⅠ基因成功区分了澳大利亚、北大西洋陆架等地区的不同种类[20,135],并通过重建系统发生关系进一步验证了COⅠ基因序列作为海洋鱼类DNA条形码标准序列的可行性。在我国,COⅠ在南海硬骨鱼类、鲌属鱼类、石首鱼科Sciaenidae、鲽形目Pleuronectiformes等类群的分类鉴定中也取得了较好的成效[136—138]。另外,COⅠ序列作为条形码还被广泛用于鉴别市场上出售的食用鱼片的真伪[139]。
为了估测鱼类群落的营养结构以及每种鱼类在群落中的营养水平,进一步研究生态系统中食物链和食物网上的物质循环和能流格局,现代鱼类生态学中采用胃含物分析法开展鱼类的食性研究。近年来,DNA条形码技术已在胃含物分析研究中发挥了一定的作用。Dunn等利用该技术分析6个物种的深海鲨鱼的胃含物组成,能够区分上述种类在捕食对象上存在的显著差异[140]。DNA条形码技术能够更快速准确的鉴定海洋生物幼体物种。分布于北极和亚北极的鲑科鱼类,尤其是其仔稚鱼,难以依据形态学特征加以区分辨别,但Schlei等对其3个属的49个体进行了COⅠ基因序列分析,发现其中48个体都能够被正确鉴定[141]。Webb等的研究结果证明DNA条形码技术可以用于鉴定南极洲物种的幼体,但是其分辨力受到DNA数据库中可用于序列比对的成体序列数量的局限,因此建立一个海洋生物DNA条形码数据库十分必要[132]。
然而,目前部分研究表明COⅠ基因并非适用于所有的鱼类种类鉴别,Kruck等发现仅凭外部形态特征和COⅠ基因很难鉴别鱚属的两个近缘种S.analis和S.ciliata,只有利用多基因条形码(multi-gene barcoding)技术,集合线粒体ND2、ATP酶及核基因RAG2序列,才能对S.analis和S.ciliata进行准确鉴别,并发现所检测的样品中存在着杂交个体[23]。Page等评估了COⅠ、COB、ATPase及控制区等4个线粒体基因片段作为条形码基因在检测澳大利亚昆斯兰东南区域淡水鱼类种类鉴别的适用性,发现前3个基因都能准确鉴别各科属鱼类,在进化速率上ATPase稍快于COⅠ和COB,而控制区则不适合进行种间鉴别[142]。此外,某些特定类群的鱼类容易发生种间杂交现象,而仅凭线粒体DNA很难对杂交个体进行准确鉴别,如石斑鱼科鱼类,已被广泛证实易于发生种间杂交,而利用多基因条形码技术,使用核基因RYR3结合线粒体基因序列,则可以鉴别出杂交个体及其双亲种类(未发表资料)。因此我们认为,对鱼类某些类群来说,采用多基因条形码技术可能是一个更合理的方案。
7 研究总结与展望
目前我国已发现的海洋生物约占全球已知海洋生物物种的10%[4],还有众多物种未被发现或还属于隐存种。DNA条形码技术使物种鉴定过程能够实现信息化和标准化,在较短时间内建立易于利用的分类系统有助于我们去发现这些新物种及隐存种,让人们对海洋生物多样性有全新的认识,并显著地提升人类监测、理解、保护以及利用海洋生物资源的能力。
从DNA条型码概念提出迄今已超过10年,其在海洋生物中的应用得到较快发展。虽然COI在动物界应用较广,但并不适用所有生物。已被尝试的基因较多,因生物类别而异;除COI及其他线粒体DNA片段(如16S),主要有28SD2/D3、ITS、rbcL、tufA、UPA等(表4)。已有的研究表明,海洋生物的许多大类群很难寻求到像动物界一样较为统一的条形码基因,我们需要根据不同的海洋生物类群,去确立具有针对性的,适合于该类群的,且通过长期研究验证并被国际所认可的标准条形码基因。某些复杂的类群如藻类,还要筛选出较为通用且合适的辅助条形码片段(如28S的D1-D3区)。针对不同类群确立2~3个有效的条型码基因可能是确保条型码技术分类、鉴定准确性的必要手段。另外,使用不同条形码基因对同一类群进行鉴定时可能会得到不同的结果,其原因可能是该类群复杂的生殖方式(如线粒体的母性或父性遗传),基因内部的插入缺失变异或某些基因在数据库中信息量的不足等,这就需要我们一方面选择更多的不同遗传水平的条形码基因进行更大规模的分析,一方面对形态特征进行更加细微的观察,从而对问题类群进行全面整体的评测。
随着生物多样性保护、药物开发、食品安全及资源调查等方面的社会需求日益增长,选择DNA条形码还必须有一定的针对性和兼顾性,在节约高效的前提下考虑到不同领域的特殊需求,努力使DNA条形码的选择满足于不同的科学研究和实际应用。因此充分利用物种的遗传(大部分物种没有基因组)信息,在动物、植物和微生物中筛选适宜的DNA条形码基因,仍然是今后一段时间内国际DNA条形码研究的主要任务。当然,DNA条形码技术是传统形态分类学的有力而且必要的补充,物种标准DNA条形码的确定必须以形态分类学研究为佐证;DNA条形码数据库不仅仅收录物种的DNA条形码,还包括物种形态分类学的信息,每一个物种的鉴定须依靠分子证据和形态分类学结果的相互参照及验证。传统形态分类学和现代分子分类学的有机结合是DNA条形码研究的发展的正确方向。
DNA条形码技术不仅有助于对物种多样性的认识,还对物种间群落关系多样性(如食物网结构复杂性)的研究提供了有力工具。近年来利用分子标记分析甲壳动物等的食性及食物组成的研究正在兴起[143—146],相信随着DNA条型码技术的不断成熟完善,我们对海洋食物网结构多元性的认识将显著提高。
当然,任何技术都有其相应的局限性和缺点,此技术在应用于某些特殊群体(杂交种,近期分化种,混合种,进化速率较低的物种)时还存在诸多问题,另外,假基因的存在[147]、基因差异性拷贝造成的多态、通用引物的难以获取[36]、基因的渗入现象[148]、易于污染的操作流程、遗传差异计算的不统一性等,都阻碍了DNA条形码技术快速的前进步伐。我们还需要经过长期的研究与实践,具有针对性的解决这些问题,为条形码技术的稳步发展奠定基础。

表4 在主要海洋生物类别尝试过的条形码基因Tab.4 The genes that have been attempted as barcodes in major groups of marine organisms

续表4
致谢:本文在厦门大学筹建海洋生物多样性与全球变化研究中心过程中,以及在中心召开的“郑重生物多样性论坛”2013年(首届)学术研讨会蕴酿而成期间得到许多同行特别是国家海洋局第三海洋研究所的林茂研究员,厦门大学的李少菁、许振祖、黄加祺教授,刘敏、王德祥、李凌等副教授及孟珊珊的大力支持,特此感谢。
[1] Hebert P D,Ratnasingham S,de Waard J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society B,2003,270(S1):96-99.
[2] Wang L,Zhuang Y,Zhang H,et al.DNA barcoding species in Alexandrium tamarense complex using ITS and proposing designation of five species[J].Harmful Algae,2014,31:100-113.
[3] Schander C,Willassen E.What can biological barcoding do for marine biology?[J].Marine Biology Research,2005,1(1):79-83.
[4] 李琪,邹山梅,郑小东,等.DNA条形码及其在海洋生物中的应用[J].中国海洋大学学报,2010,40(8):43-47.
[5] Radulovici A E,Archambault P,Dufresne F.DNA barcodes for marine biodiversity:Moving fast forward?[J].Diversity,2010,2(4):450-472.
[6] Bucklin A,Steinke D,Blanco-Bercial L.DNA barcoding of marine metazoa[J].Annual Review of Marine Science,2011,3(1):471-508.
[7] Weigt L A,Driskell A C,Baldwin C C,et al.DNA barcoding fishes[J].Methods Mol Biol,2012,858:109-126.
[8] Velmurugan S,Prasannakumar C,Manokaran S,et al.DNA barcodes for marine fungal identification and discovery[J].Fungal Ecology,2013,6(5):408-418.
[9] Graur D,Li W H.Fundamentals of Molecular Evolution[M].Sunderland:Sinauer Associates,1999.
[10] Avise J,Ellis D.Mitochondrial DNA and the evolutionary genetics of higher animals[J].Philosophical Transactions of the Royal Society of London.B,Biological Sciences,1986,312(1154):325-342.
[11] Hebert P D,Cywinska A,Ball S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society of London B,2003,270(1512):313-321.
[12] Hebert P D,Penton E H,Burns J M,et al.Ten species in one:DNAbarcoding reveals cryptic speciesin the neotropical skipper butterfly astraptes fulgerator[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(41):14812-14817.
[13] Elias M,Hill R I,Willmott K R,et al.Limited performance of DNA barcoding in a diverse community of tropical butterflies[J].Proceedings of the Royal Society B:Biological Sciences,2007,274(1627):2881-2889.
[14] Hausmann A,Haszprunar G,Segerer A H,et al.Now DNA-barcoded:the butterflies and larger moths of Germany[J].Spixiana,2011,34(1):47-58.
[15] Wilson J J,Sing K W,Sofian A M.Building a DNA barcode referencelibrary for the true butterflies(Lepidoptera)of peninsula Malaysia:What about the subspecies?[J].PloS ONE,2013,8(11):e79969.
[16] Johnsen A,Rindal E,Ericson P G,et al.DNA barcoding of Scandinavian birds reveals divergent lineages in trans-Atlantic species[J].Journal of Ornithology,2010,151(3):565-578.
[17] Park H Y,Yoo H S,Jung G,et al.New DNA barcodes for identification of Korean birds[J].Genes&Genomics,2011,33(2):91-95.
[18] Lijtmaer D A,Kerr K C,Stoeckle M Y,et al.DNA barcoding birds:From field collection to data analysis[J].Methods Mol Biol,2012,858:127 -152.
[19] Aliabadian M,Beentjes K K,Roselaar C S,et al.DNA barcoding of Dutch birds[J].Zookeys,2013,365:25-48.
[20] Ward R D,Zemlak T S,Innes B H,et al.DNA barcoding Australia's fish species[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2005,360(1462):1847-1857.
[21] Zhang J B.Species identification of marine fishes in China with DNA barcoding[J].Evidence-Based Complementary and Alternative Medicine,2011,2011:10.
[22] Becker S,Hanner R,Steinke D.Five years of FISH-BOL:brief status report[J].Mitochondrial DNA,2011,22(S1):3-9.
[23] Krück N C,Tibbetts I R,Ward RD,et al.Multi-gene barcoding to discriminate sibling species within a morphologically difficult fish genus(Sillago)[J].Fisheries Research,2013,143:39-46.
[24] Vences M,Thomas M,Bonett R M,et al.Deciphering amphibian diversity through DNA barcoding:chances and challenges[J].Philosophical Transactions of the Royal Society B,2005,360(1462):1859-1868.
[25] Kress W J,Erickson D L.A two-locus global DNA barcode for land plants:the codingrbcL gene complements the non-coding trn H-psb A spacer region[J].PloS ONE,2007,2(6):e508.
[26] Lahaye R,van der Bank M,Bogarin D,et al.DNA barcoding the floras of biodiversity hotspots[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(8):2923-2928.
[27] Hollingsworth P M,Forrest L L,Spouge J L,et al.A DNA barcode for land plants[J].Proceedings of the National Academy of Sciences of the U-nited States of America,2009,106(31):12794-12797.
[28] Stoeckle M Y,Coffran C.TreeParser-aided Klee diagrams display taxonomic clusters in DNA barcode and nuclear gene datasets[J].Scitific Reports,2013,3:2635.
[29] Grassle J F,Maciolek N J.Deep-sea species richness-regional and local diversity estimates from quantitative bottom samples[J].American Naturalist,1992,139(2):313-341.
[30] Sherwood A R,Presting G G.Universal primers amplify a 23s rDNA plastid marker in eukaryotic algae and cyanobacteria[J].JPhycol,2007,43(3):605-608.
[31] Alverson A J.Molecular systematics and the diatom species[J].Protist,2008,159(3):339-353.
[32] Ellegaard M,Godhe A,Härnström K,et al.The species concept in marine diatom:LSU rDNA-based phylogenetic differentiation inSkeletonema marionoi/dohrnii(Bacillariophyceae)is not reflected in morphology[J].Phycologia,2008,47(2):156-167.
[33] Saunders G W,McDevit D C.Methods for DNA barcoding photosynthetic protists emphasizing the macroalgae and diatoms[J].DNA Barcodes-Methods in Molecular Biology,2012,858:207-222.
[34] Litaker W R,Vandersea M W,Kibler S R,et al.Recognizing dinoflagellate species using ITS r DNA sequences[J].Journal of Phycology,2007,43(2):344-355.
[35] Lin S,Zhang H,Hou Y,et al.High-level diversity of dinoflagellates in the natural environment,revealed by assessment of mitochondrialcox1 andcobgenes for dinoflagellate DNA barcoding[J].Applied and Environmental Microbiology,2009,75(5):1279-1290.
[36] Stern R F,Andersen R A,Jameson I,et al.Evaluating the Ribosomal Internal Transcribed Spacer(ITS)as a candidate dinoflagellate barcode marker[J].PloS ONE,2012,7(8):e42780.
[37] Saunders G W.Applying DNA barcoding to red macroalgae:a preliminary appraisal holds promise for future applications[J].Philosophical Transactions of the Royal Society B,2005,360(1462):1879-1888.
[38] Saunders G W.Routine DNA barcoding of Canadian Gracilariales(Rhodophyta)reveals the invasive species Gracilaria vermiculophylla in British Columbia[J].Molecular Ecology Resources,2009,9(S1):140-150.
[39] 崔翠菊,张立楠,王娜,等.藻类DNA条形码研究进展[J].中国生物工程杂志,2012,32(9):113-117.
[40] 吴菲菲,杜国英,茅云翔.红藻DNA条形码鉴定技术研究[Z].中国科技论文在线,2013.
[41] Kucera H,Saunders G W.A survey of Eangiales(Rhodophyta)based on multiple molecular markers reveals cryptic diversity[J].Journal of Phycology,2012,48(4):869-882.
[42] Saunders G W.A DNA barcode examination of the red algal family Dumontiaceae in Canadian waters reveals substantial cryptic species diversity. 1.The folioseDilsea-Neodilseacomplex and weeksia[J].Botany,2008,86(7):773-789.
[43] Tan J,Lim P E,Phang S M,et al.Assessment of four molecular markers as potential DNA barcodes for red algaeKappaphycus Doty andEucheumaJ.Agardh(Solieriaceae,Rhodophyta)[J].PloSONE,2012,7(12):e52905.
[44] Hind K R,Saunders G W.Molecular markers from three organellar genomes unravel complex taxonomic relationships within the coralline algal genusChiharaea(Corallinales,Rhodophyta)[J].Molecular Phylogenetics and Evolution,2013,67(2):529-540.
[45] 徐涤,秦松.海洋褐藻分子系统学研究进展[J].海洋科学,2002,26(2):19-22.
[46] McDevit D C,Saunders G W.A DNA barcode examination of the Laminariaceae(Phaeophyceae)in Canada reveals novel biogeographical and evolutionary insights[J].Phycologia,2010,49(3):235-248.
[47] Kucera H,Saunders G W.Assigning morphological variants ofFucus(Fucales,Phaeophyceae)in Canadian waters to recognized species using DNA barcoding[J].Botany,2008,86(9):1065-1079.
[48] McDevit DC,Saunders GW.On the utility of DNA barcoding for species differentiation among brown macroalgae(Phaeophyceae)including a novel extraction protocol[J].Phycological Research,2009,57(2):131-141.
[49] Mattio L,Payri C.Assessment of five markers as potential barcodes for identifyingSargassumsubgenusSargassumspecies(Phaeophyceae,Fucales)[J].Cryptogamie Algologie,2010,31(4):467-485.
[50] Lane C E,Lindstrom S C,Saunders G W.A molecular assessment of northeast PacificAlariaspecies(Laminariales,Phaeophyceae)with reference to the utility of DNA barcoding[J].Molecular Phylogenetics and Evolution,2007,44(2):634-648.
[51] Poong S W,Lim P E,Phang S M,et al.A molecular-assisted floristic survey of crustose brown algae(Phaeophyceae)from Malaysia and Lombok Island,Indonesia based onrbcL and partialcox1 genes[J].Journal of Applied Phycology,2014,26(2):1231-1242.
[52] Saunders G W,Kucera H.An evaluation ofrbcL,tufA,UPA,LSU and ITSas DNA barcode markers for the marine green macroalgae Cryptogamie[J].Algologie,2010,31(4):487-528.
[53] Fucikova K,Rada J C,Lukesova A,et al.Cryptic diversity within the genusPseudomuriella Hanagata(Chlorophyta,Chlorophyceae,Sphaeropleales)assessed using four Barcode markers.Nova Hedwigia,2011,93(1/2):29-46.
[54] Bock C,Krienitz L,Proeschold T,et al.Taxonomic reassessment of the genusChlorella(Trebouxiophyceae)using molecular signatures(barcodes),including description of seven new species[J].Fottea,2011,11(2):293-312.
[55] Kawashima Y,Akasaki T,Matsumoto Y,et al.Species identification of imported and Japanese commercial green algal products based on phylogenetic analyses using the nrITS2 and 5S r DNA spacer regions[J].Fisheries Science,2013,79(3):521-529.
[56] 丁兰平,马元元,黄冰心.DNA条形码技术在大型海藻学研究中的应用及前景[J].海洋科学,2012,36(11):103-109.
[57] Evans K M,Wortley A H,Mann D G.An assessment of potential diatom“barcode”genes(cox1,rbcL,18Sand ITS rDNA)and their effectiveness in determining relationships inSellaphora(Bacillariophyta)[J].Protist,2007,158(3):349-364.
[58] Hamsher S E,Saunders G W.A floristic survey of marine tube-forming diatoms reveals unexpected diversity and extensive co-habitation among genetic lines of the Berkeleya rutilans complex(Bacillariophyceae)[J].European Journal of Phycology,2014,49(1):47-59.
[59] Trobajo R,Mann D G,Clavero E,et al.The use of partialcox1,rbcL and LSU rDNA sequences for phylogenetics and species identification within the Nitzschia palea species complex(Bacillariophyceae)[J].European Journal of Phycology,2010,45(4):413-425.
[60] Moniz M B,Kaczmarska I.Barcoding diatoms:Is there a good marker?[J].Molecular Ecology Resources,2009,9:65-74.
[61] MacGillivary M L,Kaczmarska I.Survey of the efficacy of a short fragment of the rbcL gene as a supplemental DNA barcode for diatoms[J]. Journal of Eukaryotic Microbiology,2011,58(6):529-536.
[62] Moniz M B,Kaczmarska I.Barcoding of diatoms:nuclear encoded ITS revisited[J].Protist,2010,161(1):7-34.
[63] Hamsher S E,Evans K M,Mann D G,et al.Barcoding diatoms:exploring alternatives to COI-5P[J].Protist,2011,162(3):405-422.
[64] Lee M A,Faria D G,Han M S,et al.Evaluation of nuclear ribosomal RNA and chloroplast gene markers for the DNA taxonomy of centric diatoms[J].Biochemical Systematics and Ecology,2013,50:163-174.
[65] Kooistra W H,Sarno D,Baizano S,et al.Global diversity and biogeography ofSkeletonemaspecies(Bacillariophyta)[J].Protist,2008,159(2):177-193.
[66] Zimmermann J,Jahn R,Gemeinholzer B.Barcoding diatoms:evaluation of the V4 subregion on the 18S rRNA gene,including new primers and protocols[J].Organisms Diversity and Evolution,2011,11(3):173-192.
[67] Zhang H,Lin S.Complex gene structure of the form II RUBISCOin the dinoflagellateProrocentrum minimum(Dinophyceae)[J].JPhycol,2003,39(6):1160-1171.
[68] Thornhill DJ,Lajeunesse T C,Santos SR.Measuring rDNA diversity in eukaryotic microbial systems:how intragenomic variation,pseudogenes,and PCR artifacts confound biodiversity estimates[J].Molecular Ecology,2007,16(24):5326-5340.
[69] Gu H F,Luo Z H,Zhang X D,et al.Morphology,ultrastructure and phylogeny ofTakayama xiamenensissp.nov.(Gymnodiniales,Dinophyceae)from the East China Sea[J].Phycologia,2013,52(3):256-265.
[70] Gu HF,Liu T T,Vale P,et al.Morphology,phylogeny and toxin profiles ofGymnodinium inusitatumsp.nov.,Gymnodinium catenatumandGymnodinium microreticulatum(Dinophyceae)from the Yellow Sea,China[J].Harmful Algae,2013,28:97-107.
[71] Stern R F,Horak A,Andrew R L,et al.Environmental barcoding reveals massive dinoflagellate diversity in marine environments[J].PloS ONE,2010,5(11):e13991.
[72] Raho N,Rodríguez F,Reguera B,et al.Are the mitochondrialcox1 andcobgenes suitable markers for species ofDinophysisEhrenberg?[J]. Harmful Algae,2013,28:64-70.
[73] Lilly E L,Halanych K M,Anderson D M.Species boundaries and global biogeography of theAlexandriumtamarensecomplex(Dinophyceae)[J]. Journal of Phycology,2007,43(6):1329-1338.
[74] Miranda L N,Zhang H,Zhuang Y,et al.Phylogenetic analysis guided by intragenomic SSU rDNA polymorphism refines classification of“Alexandrium tamarense”species complex[J].Harmful Algae,2012,16:35-48.
[75] Pochon X,Putnam H M,Burki F,et al.Identifying and characterizing alternative molecular markers for the symbiotic and Free-Living dinoflagellate genus symbiodinium[J].PloSONE,2012,7(1):e29816.
[76] LaJeunesse T C,Parkinson J E,Reimer J D.A genetics-based description ofSymbiodinium minutumsp.nov.andS.psygmophilumsp.nov.(Dinophyceae),two dinoflagellates symbiotic with cnidaria[J].Journal of Phycology,2012,48(6):1380-1391.
[77] Hoef-Emden K.Pitfalls of establishing DNA barcoding systems in protists:The cryptophyceae as a test case[J].PloSONE,2012,7(8):e43652.
[78] Armstrong K F,Ball S L.DNA barcodes for biosecurity:invasive species identification[J].Philosophical Transactions of Lthe Royal Society B,2005,360(1462):1813-1823.
[79] Clarkston B E,Saunders G W.A comparison of two DNA barcode markers for species discrimination in the red algal family Kallymeniaceae(Gigartinales,Florideophyceae),with a description ofEuthora timburtoniisp.nov[J].Botany,2010,88(2):119-131.
[80] Cross H B,Lowe A J,Gurgel CF D.DNA barcoding of invasive species[M]//Fifty Years of Invasion Ecology:The Legacy of Charles Elton.Oxford:Wiley-Blackwell,2011:289-299.
[81] Wörheide G,Erpenbeck D.DNA taxonomy of sponges-progress and perspectives[J].Journal of the Marine Biological Association of the United Kingdom,2007,87(6):1629-1633.
[82] Hooper J N A,Van Soest R W M.Systema Porifera.Guide to the supraspecific classification of sponges and spongiomorphs(Porifera)[M].New York:Plenum,2002.
[83] Erpenbeck D,Hooper J N A,Worheide G.COI phylogenies in diploblasts and the‘Barcoding of Life’-Are we sequencing a suboptimal partition?[J].Molecular Ecology Notes,2006,6(2):550-553.
[84] Park M H,Sim C J,Baek J,et al.Identification of genes suitable for DNA barcoding of morphologically indistinguishable Korean Halichondriidae sponges[J].Molecules and Cells,2007,23(2):220-227.
[85] Pöppe J,Sutcliffe P,Hooper J N,et al.COI barcoding reveals new clades and radiation patterns of Indo-Pacific sponges of the family Irciniidae(Demospongiae:Dictyoceratida)[J].PloS One,2010,5:e9950.
[86] Shearer T L,Coffroth M A.Barcoding corals limited by interspecific divergence,not intraspecific variation[J].Molecular Ecology Resources,2008,8(2):247-255.
[87] Moura C J,Harris D J,Cunha M R,et al.DNA barcoding reveals cryptic diversity in marine hydroids(Cnidaria,Hydrozoa)from coastal and deepsea environments[J].Zoological Scripta,2008,37(1):93-108.
[88] Bucklin A,Hopcroft R R,Kosobokova K N,et al.DNA barcoding of Arctic Ocean holozooplankton for species identification and recognition[J]. Deep-Sea Research PartⅡ,2010,57(1/2):40-48.
[89] Ortman B D,Bucklin A,Pagès F,et al.DNA Barcoding the Medusozoa using mtCOI[J].Deep-Sea Research PartⅡ,2010,57(24/26):2148-2156.
[90] 程方平,王敏晓,王彦涛,等.中国北方习见水母类的DNA条形码分析[J].海洋与湖沼,2012,43(3):451-459.
[91] Laakmann S,Holst S.Emphasizing the diversity of North Sea hydromedusae by combined morphological and molecular method[J].Journal of Plankton Research,2014,36(1):64-76.
[92] Zheng L M,He J R,Lin Y S,et al.16S r RNA is a better choice than COI for DNA barcoding hydrozoans in the coastal waters of China[J].Acta Oceanologica Sinica,2014,33(4):55-76.
[93] Radulovici A E,Sainte-Marie B,Dufresne F.DNA barcoding of marine crustaceans from the Estuary and Gulf of St Lawrence:a regional-scale approach[J].Molecular Ecology Resources,2009,9:181-187.
[94] Bucklin A,Ortman B D,Jenning R M,et al.A“Rosetta Stone”for metazoan zooplankton:DNA barcode analysis of species diversity of the Sargasso Sea(Northwest Atlantic Ocean)[J].Deep-Sea Research PartⅡ,2010,57(24/26):2234-2247.
[95] 王敏晓,程方平,李超伦,等.基于线粒体cox1片段序列的胶州湾浮游动物DNA条形码分析[J].海洋与湖沼,2011,42(5):702-710.
[96] 王仁诚,王敏晓,程方平,等.基于线粒体cox1片段序列胶州湾浮游动物物种组成分析[J].海洋与湖沼,2011,42(5):711-721.
[97] Böttger-Schnack R,Machida R J.Comparison of morphological and molecular traits for species identification and taxonomic grouping of oncaeid copepods[J].Hydrobiologia,2011,666(1):111-125.
[98] Bucklin A,Wiebe P H,Smolenack SB,et al.DNA barcodes for species identification of euphausiids(Euphausiacea,Crustacea)[J].Journal ofPlankton Research,2007,29(6):483-493.
[99] Krug PJ,Ellingson R A,Burton R,et al.A new Poecilogonous species of sea slug(Opisthobranchia:Sacoglossa)from California:comparison with the planktotrophic congenerAlderia modesta[J].Journal of Molluscan Studies,2007,73(1):29-38.
[100] Johnson S B,Warén A,Vrijenhoek R C.DNA Barcoding ofLepetodriluslimpets reveals cryptic species[J].Journal of Shellfish Research,2008,27(1):43-51.
[101] Nakano T,Marshall B A,Kennedy M,et al.The phylogeny and taxonomy of New Zealand Notoacmea and Patelloida species(Mollusca:Patellogastropoda:Lottiidae)inferred from DNA sequences[J].Molluscan Research,2009,29(1):33-59.
[102] Proschwitz T V,Schander C,Jueg U,et al.Morphology,ecology and DNA barcoding distinguishPupilla pratensis(Clessin,1871)fromPupilla muscorum(Linnaeus,1758)(Pulmonata:Pupillidae)[J].Journal of Molluscan Studies,2009,75(4):315-322.
[103] Feng Y,Li Q,Kong L,et al.COⅠ-based DNA barcoding of Arcoida species(Bivalvia:Pteriomorphia)along the coast of China[J].Molecular E-cology Resources,2011,11(3):435-441.
[104] Liu J,Li Q,Kong L F,et al.Identifying the true oysters(Bivalvia:Ostreidae)with mitochondrial phylogeny and distance-based DNA barcoding[J].Molecular Ecology Resources,2011,11(5):820-830.
[105] Sekino M,Yamashita H.Mitochondrial DNA barcoding for Okinawan oysters:a cryptic population of the Portuguese oysterCrassostrea angulatain Japanese waters[J].Fisheries Science,2013,79(1):61-76.
[106] Allcock A L,Barratt I,Eléaume M,et al.Cryptic speciation and the circumpolarity debate:a case study on endemic Southern Ocean octopuses using the COI barcode of life[J].Deep-Sea Research PartⅡ,2010,58(1/2):242-249.
[107] Williams R C,Newman SJ,Sinclair W.DNA barcoding inNautilus pompilius(Mollusca:Cephalopoda):evolutionary divergence of an ancient species in modern times[J].Invertebrate Systematics,2012,26(6):548-560.
[108] Dai L N,Zheng X D,Kong L F,et al.DNA barcoding analysis of Coleoidea(Mollusca:Cephalopoda)from Chinese waters[J].Molecular Ecology Resources,2012,12(3):437-447.
[109] Hebert P D,Stoeckle M Y,Zemlak T S,et al.Identification of birds through DNA barcodes[J].PLoS Biology,2004,2(10):e312.
[110] Dayrat B.Towards integrative taxonomy[J].Biological Journal of the Linnean Society,2005,85(3):407-415.
[111] Liu J,Li Q,Kong L F,et al.Cryptic diversity in the pen shellAtrina pectinata(Bivalvia∶Pinnidae):high divergence and hybridization revealed by molecular and morphological data[J].Molecular Ecology,2011,20(20):4332-4345.
[112] Teske P R,Barker N P,Mc Quaid C D.Lack of genetic differentiation among four sympatric southeast African intertidal limpets(Siphonariidae):phenotypic plasticity in a single species?[J].Journal of Molluscan Studies,2007,73(3):223-228.
[113] Huang J,Dunthorn M,Song W.Expanding character sampling for the molecular phylogeny of euplotid ciliates(Protozoa,Ciliophora)using three markers,with a focus on the family Uronychiidae[J].Molecular Phylogenetics and Evolution,2012,63(3):598-605.
[114] Ishitani Y,UjiiéY,de Vargas C,et al.Phylogenetic relationships and evolutionary patterns of the order collodaria(Radiolaria)[J].PLoS ONE,2012,7(5):e35775.
[115] Santoferrara L F,McManus G B,Alder V A.Phylogeny of the order Tintinnida(Ciliophora,Spirotrichea)inferred from small-and large-subunit r RNA genes[J].The Journal of Eukaryotic Microbiology,2012,59(4):423-426.
[116] Chen L Y,Liu W W,Liu A,et al.Morphology and molecular phylogeny of a new marine hypotrichous ciliate,Hypotrichidium paraconicumn. sp.(Ciliophora,Hypotrichia)[J].Journal of Eukaryotic Microbiology,2013,60(6):588-600.
[117] Chantangsi C,Lynn D H,Brandl M T,et al.Barcoding ciliates:a comprehensive study of 75 isolates of the genusTetrahymena[J].International Journal of Systematic and Evolutionary Microbiology,2007,57(10):2412-2425.
[118] Santoferrara L F,McManus G B,Alder V A.Utility of genetic markers and morphology for species discrimination within the order Tintinnida(Ciliophora,Spirotrichea)[J].Protist,2013,164(1):24-36.
[119] Jiang J,Xing Y,Miao M,et al.Two new marine ciliates,Caryotrichararisetan.sp.andDiscocephalus pararotatoriusn.sp.(Ciliophora,Spirotrichea),with phylogenetic analyses inferred from the small subunit r RNA gene sequences[J].The Journal of Eukaryotic Microbiology,2013,60(4):388-398.
[120] Pan H,Gao F,Lin X,et al.Three newLoxophyllumspecies(Ciliophora:Pleurostomatida)from China with a brief review of the marine and brackishLoxophyllumspecies[J].The Journal of Eukaryotic Microbiology,2013,60(1):44-56.
[121] Huang J,Yi Z Z,Miao M,et al.Phylogenetic relationships within the genusAspidisca(Protozoa,Ciliophora,Euplotida)revealed by ITS1-5.8S -ITS2 region sequences[J].Chinese Journal of Oceanology and Limnology,2011,29(2):277-283.
[122] Ortman B D.DNA Barcoding the Medusozoa and Ctenophora[D].USA:University of Connecticut,2008.
[123] Peijnenburg K T,Breeuwer J A,Pierrot-Bults A C,et al.Phylogeography of the planktonic chaetognath Sagitta setosa reveals isolation in European seas[J].Evolution,2004,58(7):1472-1487.
[124] Jennings R M,Bucklin A,Pierrot-Bults A.Barcoding of arrow worms(phylum Chaetognatha)from three oceans genetic diversity and evolution within an enigmatic phylum[J].PLoSONE,2010,5(4):e9949.
[125] Miyamoto H,Machida R J,Nishida S.Genetic diversity and cryptic speciation of the deep sea chaetognathCaecosagitta macrocephala(Fowler,1904)[J].Deep-Sea Res PartⅡ,2010,57(24/26):2211-2219.
[126] Miyamoto H,Machida R J,Nishida S.Global phylogeography of the deep-sea pelagic chaetognathEukrohnia hamata[J].Progress in Oceanography,2012,104:99-109.
[127] Birky C W.Workshop on barcoded DNA:application to rotifer phylogeny,evolution,and systematics[J].Hydrobiologia,2007,593(1):175-183.
[128] Carr C M,Hardy S M,Brown T M,et al.A trioceanic perspective:DNA barcoding reveals geographic structure and cryptic diversity in Canadian polychaetes[J].PLoS ONE,2011,6(7):e22232.
[129] Sewell M A,Lavery S,Baker C S.Whose larva is that?Molecular identification of planktonic larvae of the Ross Sea[J].New Zealand Aquatic Environment and Biodiversity Report,2006(3):1-46.
[130] Tang R W,Yau C,Ng W C.Identification of stomatopod larvae(Crustacea:Stomatopoda)from Hong Kong waters using DNA barcodes[J]. Molecular Ecology Resources,2010,10(3):439-448.
[131] Heimeier D,Lavery S,Sewell M A.Using DNA barcoding and phylogenetics to identify Antarctic invertebrate larvae:Lessons from a large scale study[J].Marine Genomics,2010,3(3/4):165-177.
[132] Webb K E,Barnes D K A,Clark M S,et al.DNA barcoding:a molecular tool to identify Antarctic marine larvae[J].Deep Sea Research Part II:Topical Studies in Oceanography,2006,53(8/10):1053-1060.
[133] Briski E,Cristescu M E,Bailey S A,et al.Use of DNA barcoding to detect invertebrate invasive species from diapausing eggs[J].Biological Invasions,2011,13(6):1325-1340.
[134] Thomsen P F,Kielgast J,Iversen L L,et al.Detection of a diverse marine fish fauna using environmental DNA from seawater samples[J].PLoS One,2012,7(8):e41732.
[135] Knebelsberger T,Landi M,Neumann H,et al.A reliable DNA barcode reference library for the identification of the North European shelf fish fauna[J].Molecular Ecology Resources,2014,14(5):1060-1071.
[136] 柳淑芳,陈亮亮,戴芳群,等.基于线粒体COI基因的DNA条形码在石首鱼科鱼类系统分类中的应用[J].海洋与湖沼,2010,41(2):221-232.
[137] 李鹏飞,朱文斌,贺舟挺,等.东海带鱼DNA条形码的建立及COI序列变异分析[J].浙江海洋学院学报,2013,32(1):6-9.
[138] 杨凡.中国鲽形目鱼类的DNA分子条形码及褐牙鲆的遗传多样性研究[D].广州:暨南大学,2010.
[139] Di Pinto A,Di Pinto P,Terio V,et al.DNA barcoding for detecting market substitution in salted cod fillets and battered cod chunks[J].Food Chemistry,2013,141(3):1757-1762.
[140] Dunn M R,Szabo A,Mc Veagh M S,et al.The diet of deepwater sharks and the benefits of using DNA identification of prey[J].Deep Sea Research PartⅠ:Oceanographic Research Papers,2010,57(7):923-930.
[141] Schlei O L,Crête-Lafreniere A,Whiteley A R,et al.DNA barcoding of eight North American coregonine species[J].Molecular Ecology Resources,2008,8(6):1212-1218.
[142] Page T,Hughes J.Comparing the performance of multiple mitochondrial genes in the analysis of Australian freshwater fishes[J].Journal of Fish Biology,2010,77(9):2093-2122.
[143] Blankenship L E,Yayanos A A.Universal primers and PCR of gut contents to study marine invertebrate diets[J].Molecular Ecology,2005,14(3):891-899.
[144] Martin D L,Ross R M,Quetin L B,et al.Molecular approach(PCR-DGGE)to diet analysis in young Antarctic krillEuphausia superba[J]. Marine Ecology Progress Series,2006,319:155-165.
[145] Troedsson C,Simonelli P,Nägele V,et al.Quantification of copepod gut content by differential length amplification quantitative PCR(dla-qPCR)[J].Mar Biol,2009,156(3):253-259.
[146] Hu S M,Guo Z L,Li T,et al.Detecting in situ copepod diet diversity using molecular technique:Development of a copepod/symbiotic ciliate-excluding eukaryote-inclusive PCR protocol[J].PLoS ONE,2014,9(7):e103528.
[147] Song H,Buhay J E,Whiting M F,et al.Many species in one:DNA barcoding overestimates the number of species when nuclear mitochondrial pseudogenes are coamplified[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(36):13486-13491.
[148] Kemppainen P,Panova M,Hollander J,et al.Complete lack of mitochondrial divergence between two species of NE Atlantic marine intertidal gastropods[J].Journal of Evolutionary Biology,2009,22(10):2000-2011.
Current status and future prospect of DNA barcoding in marine biology
Lin Senjie1,Wang Lu1,Zheng Lianming1,Dong Yunwei1,Liu Shufang2,Ding Shaoxiong1,
Ye Naihao2,Cao Wenqing1,Zhuang Zhimeng2
(1.Marine Biodiversity and Global Change Research Center,Xiamen University,Xiamen 361005,China;2.Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao 266071,China)
Marine organisms are highly diverse,widely distributed,with high complexity and homoplasy.To enable fast and accurate identification of species,it is imperative to establish molecular techniques,to complement the traditional morphological metbodology.DNA barcoding provides digitalized criteria and effective means for species identification,and is becoming an important technical tool in the research on taxonomy and biodiversity.In this review,we summarize the major recent progress and current trend in DNA barcoding,particularly as it applies to the fields of marine phytoplankton(Rhodophyte,Phaeophyta,Chlorophyta,Bacillariophyta and Dinophyta),invertebrates(Spongia,Cnidaria,Custacea,Mollusca,etc.)and fish.We provide an overview of the deffectiveness and suitability of different barcoding markers in different groups of marine organisms.We also discuss current challenges and future prospects of marine DNA barcoding in hope to provide a framework for future marine DNA barcoding research in China.
DNA barcoding;marine biology;morphological taxonomy;molecular evolution;barcoding marker
S917.4
A
0253-4193(2014)12-0001-17
林森杰,王路,郑连明,等.海洋生物DNA条形码研究现状与展望[J].海洋学报,2014,36(12):1—17,
10.3969/j.issn.0253-4193.2014.12.001
Lin Senjie,Wang Lu,Zheng Lianming,et al.Current status and future prospect of DNA barcoding in marine biology[J].Acta Oceanologica Sinica(in Chinese),2014,36(12):1—17,doi:10.3969/j.issn.0253-4193.2014.12.001
2014-06-26;
2014-08-15。
国家自然科学基金“通过生态基因组学分析探索东海原甲藻的生态适应机制”重点项目(41330959);国家海洋可再生能源”海洋微藻生物柴油规模化制备关键技术与装置的优化、耦联及应用研究”专项资金项(ME2011SW03);外高层次人才引进计划;科技基础性工作专项“我国重要渔业生物DNA条形码信息采集及其数据库构建”(2013FY110700)。
林森杰(1964—),男,教授,主要从事海洋浮游生物多样性及生态基因组学研究。E-mail:senjie.lin@xmu.edu.cn
