鳍足类线粒体基因的选择压力比较及进化关系探讨
2014-06-01申欣田美孟学平程汉良阎斌伦
申欣,田美,孟学平,程汉良,阎斌伦
(1.淮海工学院海洋学院江苏省海洋生物技术重点实验室,江苏连云港 222005;2.中国科学院北京生命科学研究院,北京 100101)
鳍足类线粒体基因的选择压力比较及进化关系探讨
申欣1,2,田美1,孟学平1,程汉良1,阎斌伦1
(1.淮海工学院海洋学院江苏省海洋生物技术重点实验室,江苏连云港 222005;2.中国科学院北京生命科学研究院,北京 100101)
鳍足类Pinnipeds是海洋哺乳动物的重要类群,现存种类包括海象科Odobenidae、海狮科Otariidae和海豹科Phocidae的所有物种。在海豹属Phoca中,atp8基因的Ka/Ks最高,仅为0.084 7(远小于1),因而海豹属所有线粒体蛋白质编码基因的选择压力均非常高。对于群海豹属Pusa,cox1、cox2和nad3基因的Ka/Ks均为0,表明在这3个基因中,所有的核苷酸的替换均为同义替换,没有出现氨基酸的改变。科内、属内基因变异位点的分析表明,nad5、nad4和nad2基因可以作为cox1和cob基因辅助的分子标记。线粒体基因组的系统发育结果,强烈支持食肉目所有12个科均为单系群,并且鳍足类(海狮科、海象科和海豹科)为食肉目Carnivora内部的一个单系群(BPM=100,BPP=100)。在鳍足类内部,海狮科与海象科亲缘关系最近,聚类为海狮总科Otarioidea,然后与海豹科聚类:(海狮科+海象科)+海豹科。在海豹科内部,线粒体基因组的证据支持将海豹科划分为2个亚科,即僧海豹亚科Monachinae和海豹亚科Phocinae。线粒体基因组的证据,强烈支持鼬科Mustelidae与浣熊科Procyonidae关系最近,臭鼬科Mephitidae与熊猫科Ailuridae近缘,同时,鳍足类与[(鼬科+浣熊科)+(臭鼬科+熊猫科)]为姊妹群,而与熊科Ursidae较远。犬科Canidae在犬型亚目Caniformia中分化较早,位于犬型亚目的基部。
海狮科;海豹科;海象科;鳍足类;系统发育;线粒体基因组
1 引言
鳍足类Pinnipeds是海洋哺乳动物的重要类群,现存种类包括海象科Odobenidae、海狮科Otariidae和海豹科Phocidae的所有物种[1—2]。鳍足类身体呈纺锤或流线形,四肢被肥厚的蹼膜连成鳍状。鳍足类的毛皮和肉都具有很高的价值,从而遭受了人类几个世纪的狂捕滥杀,导致不少种类自然种群的数量不断减少甚至灭绝。以加勒比僧海豹Monachus tropicalis为例,数量最多时曾超过25万只,然而由于人类的大量猎杀和栖息地的破坏,最后一次的野外纪录是在1952年,目前已经完全灭绝,与此同时,还有些鳍足类物种处于灭绝的边缘[3—4]。因此,对鳍足类自然种群的保护及其遗传背景的认识刻不容缓。通常认为鳍足类动物起源于一类(或多类)陆生食肉目动物,在约2 000万年前逐渐进入并适应海洋生活,从而开始了由陆地环境到海洋环境的进化历程[5]。事实上,鳍足类本身是否为单一起源(单系群)一直存在激烈争议,另外,鳍足类现存的3个科与陆生食肉目现存类群之间的关系仍然具有诸多分歧[6—7]。后生动物的基因组除了核基因组外,还包括线粒体基因组。线粒体基因组具有基因排列紧凑、成本较低及易于完成等优点,目前被广泛应用到后生动物分子系统进化、群体遗传及生物地理的研究[7—11]。本文全面的分析比较现有的24个鳍足类物种线粒体基因组,系统分析鳍足类线粒体基因组的选择压力和分子标记;同时,结合陆生食肉目9个科的线粒体基因组,全面、系统地分析了鳍足目的起源及其内部的进化关系。
2 材料和方法
2.1 鳍足类线粒体基因组
目前共有24个鳍足类线粒体基因组,分别为海象科Odobenidae的唯一现存种:海象Odobenus rosmarus rosmarus,海狮科Otariidae的纽西兰海狗Arctocephalus forsteri、南非海狗Arctocephalus pusillus、瓜达卢佩海狗Arctocephalus townsendi、北海狗Callorhinus ursinus、北海狮Eumetopias jubatus、澳洲海狮Neophoca cinerea、新西兰海狮Phocarctos hoo犽eri、加州海狮Zalophus californianus,以及海豹科Phocidae的冠海豹Cystophora cristata、髯海豹Erignathus barbatus、灰海豹Halichoerus grypus、豹斑海豹Hydrurga leptonyx、威德尔氏海豹Leptonychotes weddellii、食蟹海豹Lobodon carcinophaga、南象海豹Mirounga leonina、夏威夷僧海豹Monachus schauinslandi、环海豹Phoca fasciata、琴海豹Phoca groenlandica、斑海豹Phoca largha、港海豹Phoca vitulina、里海海豹Pusa caspica、环斑海豹Pusa hispida和贝加尔海豹Pusa sibirica[12—16]。
2.2 线粒体基因组选择压力分析
在检测鳍足类线粒体基因组的选择压力时,以海狮科3个南非海狗属物种(纽西兰海狗、南非海狗与瓜达卢佩海狗)、海豹科4个海豹属物种(环海豹、琴海豹、斑海豹与港海豹)以及3个群海豹属物种(里海海豹、环斑海豹与贝加尔海豹)为数据群。然后,分别借助于Clustal X 2.1软件[17],对线粒体13个蛋白质编码基因(atp6、atp8、cox1-3、cob、nad1-4、nad4L、nad5和nad6)进行多重序列比对(默认参数)。同义替换率(Ks)和非同义替换率(Ka)的计算则是通过DnaSP 5.10.1软件[18]完成。
2.3 基因差异性分析
以8种海狮科与15种海豹科物种线粒体基因组分别作为数据群,统计了2个科内部的基因变异位点,同时,以3个南非海狗属物种、4个海豹属物种和3个群海豹属物种分别作为数据群,统计了3个属内的基因变异位点。借助于Clustal X 2.1软件[17]对15个主编码基因分别进行多重序列比对。随后,线粒体单基因的变异位点,则是通过DnaSP 5.10.1软件[18]来完成。
2.4 进化关系分析
除了24个鳍足类线粒体基因组外,17个陆生食肉目的线粒体基因组(小熊猫Ailurus fulgens、川西小熊猫Ailurus fulgens styani、灰狼Canis lupus、豹Cuon alpinus、臭鼬Mephitis mephitis、东部斑臭鼬Spilogale putorius、海獭Enhydra lutris、水獭Lutra lutra、南美浣熊Nasua nasua、北美浣熊Procyon lotor、大熊猫Ailuropoda melanoleuca、北极熊Ursus maritimus、猎豹Acinonyx jubatus、华南虎Panthera tigris amoyensis、红颊獴Herpestes javanicus、鬣狗Crocuta crocuta和缟鬣狗Hyaena hyaena)也加入到系统发育分析中。首先提取41个物种(共代表食肉目12个科)的13个蛋白质编码基因的核苷酸序列,然后首尾相连,进而通过Clustal X 2.1软件[17]做多重序列比对。对于多重序列比对结果,借助于j Model Test 2.1.3软件[19]进行替换模型的计算比较,当最适替换模型(GTR+I+V)确定后,分别采取最大似然法与贝叶斯法进行系统发育关系重建,使用的软件分别为Phy ML 3.0[20]和MrBayes 3.2[21]。在最大似然树构建时,使用自展值检验法(Bootstrap=1 000),来评估系统发育树中每个分支的可靠性。在贝叶斯分析中,运算1 000 000代,以保证足够的时间达到收敛。
3 结果和讨论
3.1 鳍足类线粒体基因组的主要特征
海狮科8个物种线粒体基因组长度相差较大,介于15 413 bp(纽西兰海狗)和16 736 bp(澳洲海狮)之间,差距达到2 323 bp。与此相比,海豹科15个物种线粒体基因组的长度较为保守,琴海豹的线粒体基因组最短(16 551 bp),南象海豹的线粒体基因组最长(16 970 bp)。海象线粒体基因组的长度与此相当(16 565 bp)(表1)。
现有鳍足类线粒体基因组的基因组成和基因排列非常保守,均共享脊椎动物典型的基因排列。8种海狮线粒体主编码链的A+T含量相差较少,介于58.75%(新西兰海狮)和59.69%(北海狗)之间。与此相比,海豹科线粒体基因组A+T含量差异较大,贝加尔海豹的A+T含量最低(58.12%),南象海豹的A+T含量最高(60.76%)。海象线粒体基因组主编码链的A+T含量为59.41%(表1)。
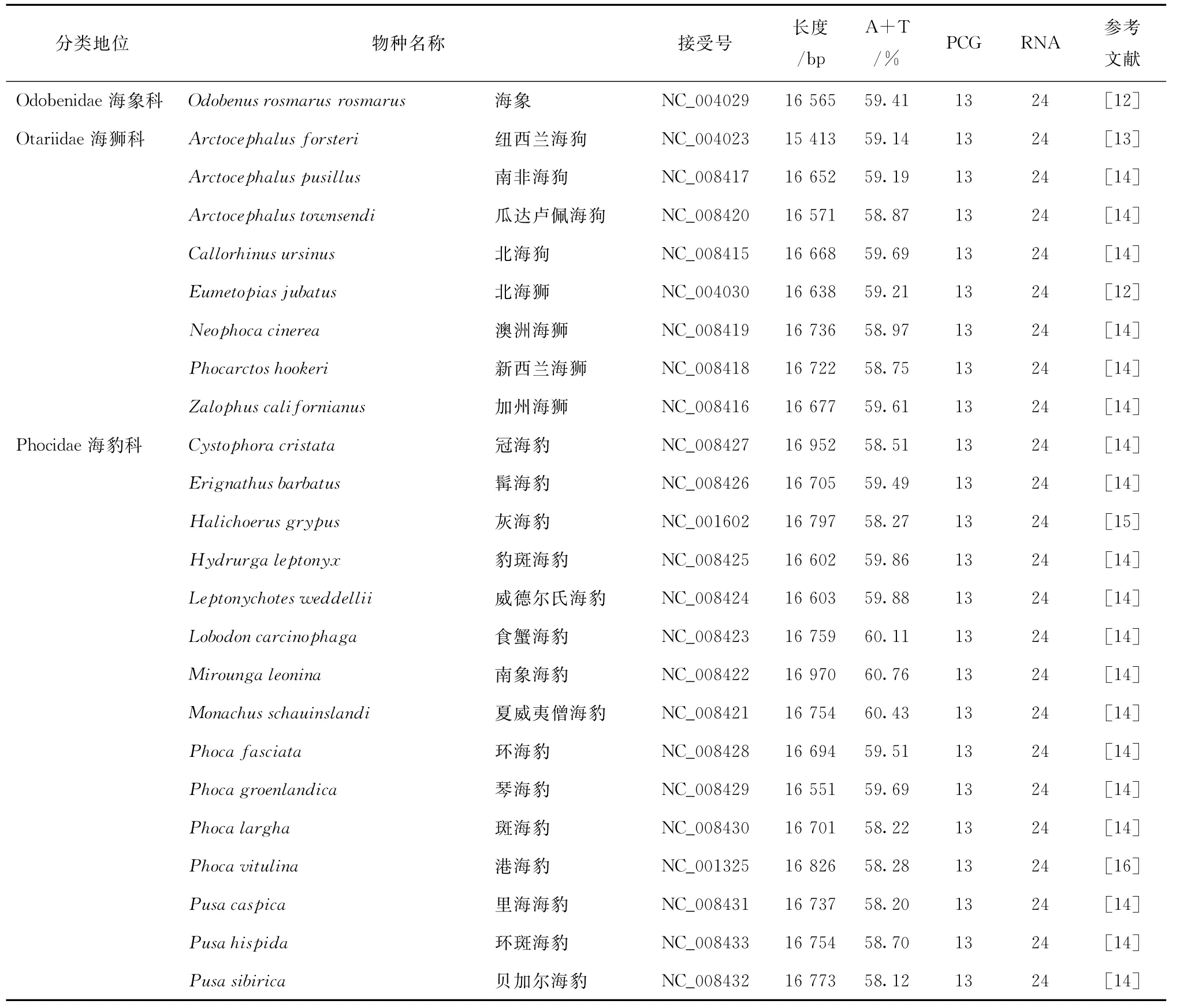
表1 鳍足类线粒体基因组的基本特征
3.2 鳍足类线粒体基因的选择压力分析
在南非海狗属中,cox1基因的Ka/Ks最低,为0.007 7,其次为cox2、nad6和nad3基因(分别为0.014 1、0.015 1和0.019 0),说明在南非海狗线粒体基因组中,这些基因的选择压力较高;而atp8基因的Ka/Ks最高,为0.181 6,其次为nad2、nad5和nad4基因(分别为0.087 2、0.085 7和0.058 6),从而说明它们的选择压力较小。对于海豹属,cox1基因的Ka/Ks最低,为0.005 5,其次为nad3、cox2和nad4L基因(分别为0.006 5、0.017 4和0.018 5);而atp8基因的Ka/Ks最高,仅为0.084 7,因此海豹属线粒体基因组中,所有蛋白质编码基因的选择压力均非常高。在群海豹属,cox1、cox2和nad3基因的Ka/Ks均为0,说明在这3个基因中,所有核苷酸的替换均为同义替换,没有出现氨基酸的改变;而nad4L基因的Ka/Ks最高,为0.2603,其次为atp6、atp8和nad2基因(分别为0.110 9、0.085 9和0.071 6),从而说明这些基因的选择压力较小(图1)。
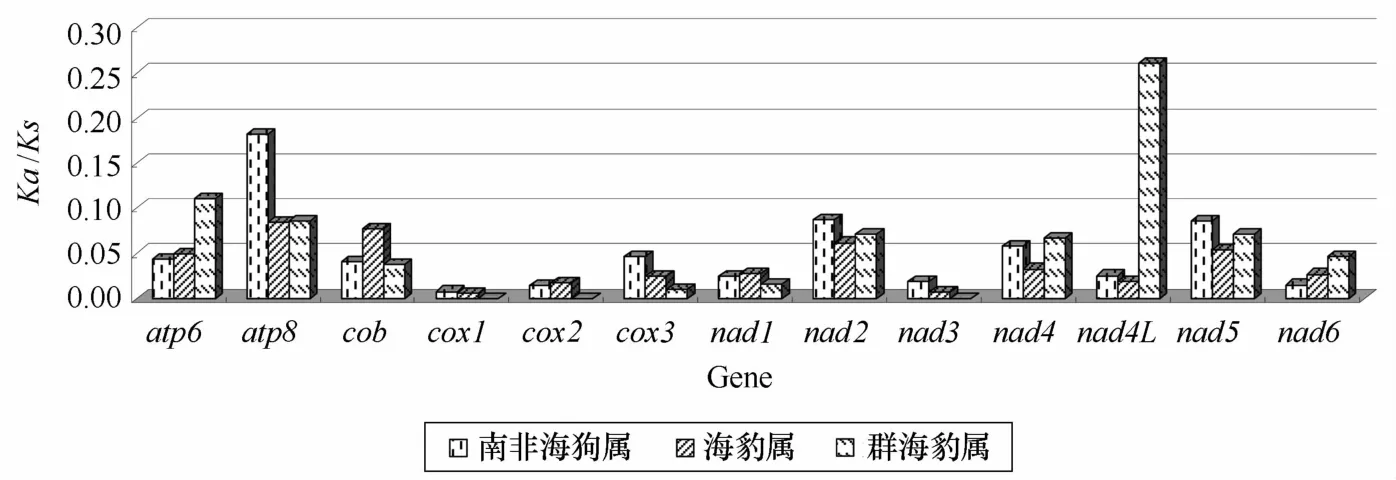
图1 南非海狗属、海豹属和群海豹属线粒体基因选择压力分析
3.3 鳍足类线粒体基因的分子标记分析
南非海狗属内部线粒体基因的变异位点结果表明,nad5基因的变异位点数最多(222个),其次为nad4、cox1、cob和nad2基因,变异位点的数目分别为148、147、127和125个。海豹属和群海豹属线粒体变异位点数的分析结果与南非海狗属类似,nad5、nad4、cox1、cob和nad2基因的变异位点数位于前列。在海狮科线粒体基因组中,nad5基因的变异位点数最多(524个),其次为nad4、cox1、cob和nad2基因,变异位点的数目分别为384、347、274和265个。在海豹科15个线粒体基因组单基因的变异位点分析中,nad5基因的变异位点数最多(635个),其次为nad4、cox1、cob和nad2基因,变异位点的数目分别为475、469、360和351个。综合属内与科内基因变异位点的分析结果,均支持nad5、nad4和nad2基因作为cox1 和cob基因辅助的分子标记,为鳍足类群体遗传及分子生态的研究提供有价值的遗传信息。
3.4 进化关系分析
与其他食肉目动物相比,鳍足类在形态和生活方式上差异巨大,鳍足类动物到底作为一个目与食肉目并列,还是作为食肉目下的一个类群,长期以来一直存在争议[22—24]。目前数据库中共有24个鳍足类线粒体基因组全序列;除了鳍足类线粒体基因组外,17个陆生食肉目的线粒体基因组也包含在系统发育分析中(共41个线粒体基因组,代表12个科),以利于从线粒体基因组层次全面系统的了解食肉目的进化历史。
以线粒体所有蛋白质编码基因为数据群,采用2种建树方法(最大似然法和贝叶斯法),所构建系统发育树的拓扑结构完全一致。线粒体基因组的数据支持鳍足类作为食肉目内部的一个类群(见图2)。同时,根据化石的证据来看,最早的鳍足类动物的化石是中新世时期,而哺乳纲其他各目最晚出现在始新世,这也支持鳍足类不能单列为一个目[5]。食肉目下分为2个分支:猫型亚目和犬型亚目(BPM=100,BPP=100)。
鳍足类本身是否为单系群,以及鳍足类3个科之间的关系存在较多争议[6—7,25]。线粒体基因组的系统发育结果,强烈支持食肉目所有12个科均为单系群,并且鳍足类(海狮科、海象科和海豹科)为单系群(BPM=100,BPP=100)。在鳍足类内部,海狮科与海象科亲缘关系最近,聚类为海狮总科Otarioidea,然后与海豹科聚类:(海狮科+海象科)+海豹科(见图2)。在海豹科内部,线粒体基因组的证据支持将海豹科划分为2个亚科,即僧海豹亚科和海豹亚科。僧海豹属Monachus、象海豹属Mirounga、食蟹海豹属Lobodon、豹海豹属Hydrurga和威德尔海豹属Leptonychotes聚在一支,组成僧海豹亚科(BPM=100,BPP=100)。同时,髯海豹属Erignathus、冠海豹属Cystophora、海豹属Phoca、灰海豹属Halichoerus和群体海豹属Pusa聚在另外一支,组成海豹亚科(BPM =100,BPP=100)。
除了鳍足类是否为单系群的问题外,鳍足类3个科与陆生食肉目各科之间的亲缘关系一直以来存在激烈争议[7,25]。线粒体基因组的证据,强烈支持鼬科与浣熊科关系最近(BPM=100,BPP=100),臭鼬科与熊猫科近缘(BPM=65,BPP=100),(鼬科+浣熊科)与(臭鼬科+熊猫科)互为姊妹群(BPM=99,BPP =100)。同时,在陆生食肉目中,鳍足类与[(鼬科+浣熊科)+(臭鼬科+熊猫科)]亲缘关系最近(BPM=78,BPP=100),而与熊科较远。犬科在犬型亚目中分化较早,位于犬型亚目的基部(见图2)。
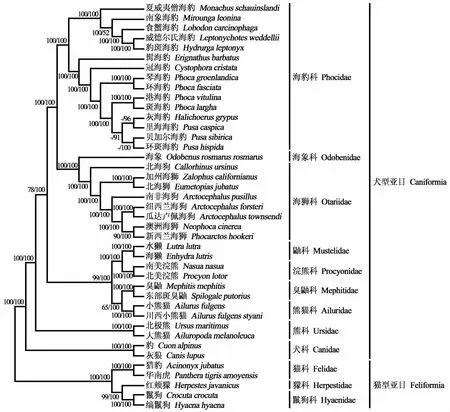
图2 基于线粒体蛋白质编码基因构建的进化关系树
4 结论
现有鳍足类线粒体基因组的基因组成和基因排列非常保守,均共享脊椎动物典型的基因排列。在南非海狗属中,cox1基因的Ka/Ks最低,其次为cox2、nad6和nad3基因,说明在南非海狗线粒体基因组中,这些基因的选择压力较高;而atp8基因的Ka/Ks最高,其次为nad2、nad5和nad4基因,从而说明这些基因存在较小的选择压力和功能束缚。对于海豹属,cox1基因的Ka/Ks最低,其次为nad3、cox2和nad4L基因;而atp8基因的Ka/Ks最高且远小于1,因此海豹属线粒体基因组中,所有蛋白质编码基因的选择压力均非常高。在群海豹属,cox1、cox2和nad3基因的Ka/Ks均为0,说明在这3个基因中,所有核苷酸的替换均为同义替换,没有出现氨基酸的改变。综合属内与科内基因变异位点的分析结果,均支持nad5、nad4和nad2基因作为cox1和cob基因辅助的分子标记。线粒体基因组的系统发育结果,强烈支持鳍足类作为食肉目内部的一个类群。同时,强烈支持食肉目所有12个科均为单系群,并且鳍足类(海狮科、海象科和海豹科)本身为单系群。在鳍足类内部,海狮科与海象科亲缘关系最近,聚类为海狮总科,然后与海豹科聚类:(海狮科+海象科)+海豹科。在海豹科内部,线粒体基因组的证据支持将海豹科划分为2个亚科,即僧海豹亚科和海豹亚科。线粒体基因组的证据,强烈支持鼬科与浣熊科关系最近,臭鼬科与熊猫科近缘,同时,鳍足类与[(鼬科+浣熊科)+(臭鼬科+熊猫科)]为姊妹群,而与熊科较远。
[1] Reeves R R,Stewart B S,Clapham P J,et al.Guide to marine mammals of the world[M].New York:Knopf,2002.
[2] Berta A,Sumich J L,KovacsKM.Marine mammals:evolutionary biology[M].Boston,London:Elsevier/Academic Press,2006.
[3] McClenachan L,Cooper A B.Extinction rate,historical population structure and ecological role of the Caribbean monk seal[J].Proc Biol Sci,2008,275(1641):1351—1358.
[4] Debrot A O.A review of records of the extinct west indian monk seal,Monachus tropicalis(carnivora:phocidae),for the netherlands antilles[J]. Marine Mammal Science,2000,16(4):834—837.
[5] Rybczynski N,Dawson M R,Tedford R H.A semi-aquatic Arctic mammalian carnivore from the Miocene epoch and origin of Pinnipedia[J].Nature,2009,458(7241):1021—1024.
[6] Delisle I,Strobeck C.A phylogeny of the Caniformia(order Carnivora)based on 12 complete protein-coding mitochondrial genes[J].Mol Phylog enet Evol,2005,37(1):192—201.
[7] Arnason U,Gullberg A,Janke A,et al.Mitogenomic analyses of caniform relationships[J].Mol Phylogenet Evol,2007,45(3):863—874.
[8] Yu L,Peng D,Liu J,et al.On the phylogeny of Mustelidae subfamilies:analysis of seventeen nuclear non-coding loci and mitochondrial complete genomes[J].BMC Evol Biol,2011,11:92—107.
[9] Boore J L,Macey J R,Medina M.Sequencing and comparing whole mitochondrial genomes of animals[J].Methods Enzymol,2005,395:311—348.
[10] Curole J P,Kocher T D.Mitogenomics:digging deeper with complete mitochondrial genomes[J].Trends Ecol Evol,1999,14(10):394—398.
[11] Boore J L.Animal mitochondrial genomes[J].Nucleic Acids Res,1999,27(8):1767—1780.
[12] Arnason U,Adegoke J A,BodinK,et al.Mammalian mitogenomic relationships and the root of the eutherian tree[J].Proc Natl Acad Sci U S A,2002,99(12):8151—8156.
[13] Lin Y H,McLenachan P A,Gore A R,et al.Four new mitochondrial genomes and the increased stability of evolutionary trees of mammals from improved taxon sampling[J].Mol Biol Evol,2002,19(12):2060—2070.
[14] Arnason U,Gullberg A,Janke A,et al.Pinniped phylogeny and a new hypothesis for their origin and dispersal[J].Mol Phylogenet Evol,2006,41 (2):345—354.
[15] Arnason U,Gullberg A.Comparison between the complete mtDNA sequences of the blue and the fin whale,two species that can hybridize in nature[J].J Mol Evol,1993,37(4):312—322.
[16] Arnason U,Johnsson E.The complete mitochondrial DNA sequence of the harbor seal,Phoca vitulina[J].J Mol Evol,1992,34(6):493—505.
[17] Larkin M A,Blackshields G,Brown N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947—2948.
[18] Rozas J,Sanchez-DelBarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496—2497.
[19] Darriba D,Taboada G L,Doallo R,et al.j Model Test 2:more models,new heuristics and parallel computing[J].Nat Methods,2012,9(8):772.
[20] Guindon S,Dufayard J F,Lefort V,et al.New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of Phy ML 3.0[J].Syst Biol,2010,59(3):307—321.
[21] Ronquist F,Teslenko M,van der Mark P,et al.MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space [J].Syst Biol,2012,61(3):539—542.
[22] Yonezawa T,Kohno N,Hasegawa M.The monophyletic origin of sea lions and fur seals(Carnivora;Otariidae)in the Southern Hemisphere[J]. Gene,2009,441(1/2):89—99.
[23] Higdon JW,Bininda-Emonds O R,Beck R M,et al.Phylogeny and divergence of the pinnipeds(Carnivora:Mammalia)assessed using a multigene dataset[J].BMC Evol Biol,2007,7:216—234.
[24] Flynn JJ,Finarelli J A,Zehr S,et al.Molecular phylogeny of the carnivora(mammalia):assessing the impact of increased sampling on resolving enigmatic relationships[J].Syst Biol,2005,54(2):317—337.
[25] Yu L,Luan P T,Jin W,et al.Phylogenetic utility of nuclear introns in interfamilial relationships of Caniformia(order Carnivora)[J].Syst Biol,2011,60(2):175—187.
Selection pressure comparison and evolutionary relationships exploration of pinnipeds mitochondrial genes
Shen Xin1,2,Tian Mei1,Meng Xueping1,Cheng Hanliang1,Yan Binlun1
(1.JiangsuKey Laboratory of Marine Biotechnology,College of Marine Science,HuaihaiInstitute of Technology,Lianyungang 222005,China;2.BeijingInstitutes of Life Science,Chinese Academy of Sciences,Beijing 100101,China)
Current pinnipeds is one important group of marine mammals,which including all species in three families (Odobenidae,Otariidae and Phocidae).In the genus Phoca,theKa/Ks rate of atp8 gene is highest and only 0.084 7(much less than 1),so all Phoca mitochondrial protein-coding genes bear very high selection pressure.In Pusa,Ka/Ks rates of three genes(cox1,cox2 and nad3)are equal to 0,indicating that replacement of all nucleotides in these three genes are synonymous substitutions and none amino acid change.Analyses of genetic variations within family and genus indicted that nad5,nad4,and nad2 genes can be used as molecular markers assisted to cox1 and cob gene.Phylogenetic results based on mitochondrial genomes strongly support that all 12 families from Carnivora are monophyletic group,and pinnipeds(Otariidae,Odobenidae and Phocidae)are one monophyletic group within Carnivora(BPM=100,BPP=100).Within the pinnipeds,the relationship between Otariidae and Odobenidae is close(Otarioidea),then cluster with Phocidae:(Otariidae+Odobenidae)+Phocidae.Evidences of mitochondrial genomes support that Phocidae are divided into two subfamilies(Monachinae and Phocinae).Mitochondrial genomic evidence strongly supports close relationship between Mustelidae and Procyonidae.Meanwhile,Mephitidae clustering with Ailuridae is supported powerfully.Pinnipeds as a sister group to[(Mustelidae+Procyonidae)+(Mephitidae+Ailuridae)],then cluster with the family Ursidae.Canidae is differentiation earliest and is located in base position of the suborder Caniformia.
Otariidae;Phocidae;Odobenidae;Pinnipeds;phylogeny;mitochondrial genome
Q915.874
A
0253-4193(2014)06-0087-07
2013-07-01;
2013-09-02。
国家自然科学基金(40906067);香江学者计划(XJ2012056);中国博士后科学基金(2012M510054,2012T50218);中央财政支持地方高校发展专项资金(CXTD04);江苏省“青蓝工程”人才基金资助项目(苏教师[2010]27号);江苏高校优势学科建设工程资助项目。
申欣(1981—),男,山东省成武县人,副教授,博士,主要从事基因组学和分子系统学研究。E-mail:shenthin@163.com
申欣,田美,孟学平,等.鳍足类线粒体基因的选择压力比较及进化关系探讨[J].海洋学报,2014,36(6):87—93,
10.3969/j. issn.0253-4193.2014.06.011
Shen Xin,Tian Mei,Meng Xueping,et al.Selection pressure comparison and evolutionary relationships exploration of pinnipeds mitochondrial genes[J].Acta Oceanologica Sinica(in Chinese),2014,36(6):87—93,doi:10.3969/j.issn.0253-4193.2014.06.011
