雷竹林土壤氨氧化微生物对不同肥料的响应
2014-05-29徐秋芳沈振明李松昊李永春
郭 帅 ,徐秋芳,沈振明,李松昊,秦 华,李永春
(1.浙江农林大学 环境与资源学院,浙江 临安 311300;2.浙江农林大学 浙江省森林生态系统碳循环与固碳减排重点实验室,浙江 临安 311300;3.浙江临安市林业科技推广总站,浙江 临安 311300)
20世纪后期,用土壤微生物数量、土壤酶等生物指标来表征土壤品质及土壤微生物多样性曾受到广泛关注[1-2]。土壤微生物多样性代表着土壤微生物群落结构的稳定性,同时也反映土壤胁迫和土壤生态机制对群落的影响,因此,土壤微生物是土壤可持续利用的重要指标[3]。硝化作用的微生物代谢包括氨氧化作用和亚硝化作用2个过程,分别由氨氧化细菌(ammonia oxidizing bacteria,AOB),氨氧化古菌(ammonia oxidizing archaea,AOA)和亚硝酸盐氧化菌催化进行,其中由铵盐氧化成亚硝酸盐的第1步过程是硝化作用的限速步骤,与土壤氮素循环密切相关,硝化细菌将氨或者铵盐氧化为硝酸盐,增加了土壤中氮元素移动性,对喜硝酸盐植物有利,但同时也增加了土壤中氮素淋失、反硝化作用还原成氮气,从而增加了土壤中氮素损失的风险,对农业生产不利[4-6]。有研究认为,基于氨氧化细菌和氨氧化古菌生态位的不同和它们在土壤生态系统大尺度范围内对环境变化的敏感性,氨氧化细菌和氨氧化古菌可以作为土壤检测有效的生物学指标[7]。研究发现,氨氧化微生物的群落结构和多样性受多种环境因子的影响,主要包括氨态氮[8]和氧气[9]、环境 pH 值[10-11]、 温度[12-13]、 底物浓度等。因此,研究不同施肥制度下土壤氨氧化微生物的多样性,可为土壤环境质量的变化提供重要依据[14]。Zhang等[15]研究了长期施肥对太湖流域水稻土细菌群落结构的影响。Chu等[16]研究施肥对砂质壤土中氨氧化细菌多样性的影响发现,施用氮肥土壤amoA基因的多样性高于不施氮肥的土壤。而Colloff等[17]的研究显示,施用尿素的土壤中amoA基因的多样性未见增长。贺纪正等[18]应用实时荧光定量核酸扩增检测系统(qPCR),克隆和测序多方法综合研究发现,红壤旱地长期氮磷钾复合肥、以及氮磷钾与有机肥配合施肥能显著提高氨氧化古菌和细菌的amoA功能基因数量,而单施氮肥则反而降低了功能基因的数量,不同施肥土壤氨氧化细菌群落结构没有显明差异,但对氨氧化古菌的群落结构组成产生明显的影响。雷竹Phyllostachys violascens林由于具有产量高、成林快、出笋早、笋味鲜的特点,目前,在长江以南各省均有栽培,特别是20世纪80年代推广以冬季地表增温覆盖和施肥为核心的反季节集约栽培技术以来,因为经济效益高,集约栽培面积不断扩大,竹农对雷竹经营强度不断加大,超过一般农作物。集约经营雷竹林土壤有机质急剧增加[19],为土壤微生物的活动提供大量能源和碳源。雷竹林大量施用化学氮肥的经营方式对土壤氮循环相关微生物的影响则更突出,氮循环相关微生物的变化反过来影响土壤氮素循环和氮素的利用率。为了探究不同肥料施用后短期内对土壤氨氧化微生物的影响,在施肥后1周采集土壤样品并分析土壤氨氧化古菌和氨氧化细菌群落结构,揭示大剂量施肥后土壤氨氧化微生物的快速响应。
1 研究地区与分析方法
1.1 研究地概况
研究区位于浙江省临安市,30°14′N,119°42′E,位于浙江省西北部,属中亚热带季风气候区,全年降水量为1628.6 mm,全年平均气温16.4℃,全年日照时数1847.3 h。雷竹产区有粗放经营和集约经营2种管理模式,前者施肥量小,且冬季不覆盖,全年总用量为2250 kg·hm-2硝态氮复合肥[m(氮)∶m(五氧化二磷)∶m(氧化钾)=16∶16∶16,折合纯氮为360 kg],分别在每年的5月和9月份施肥,2次比例为60%和40%,每年4月时采收自然笋。而集约雷竹林冬季覆盖大量的有机物料以提高土温度,全年施肥量是粗放经营的2倍,施肥3次·a-1,分别在5月上旬、9月中下旬和11月底覆盖以前,3次施肥量比例控制在35%,30%和35%。本试验选择成林时间为15 a的长期粗放经营雷竹林作为试验地,2010年12月进行首次冬季覆盖经营,6月将覆盖物翻入土壤。
1.2 试验设计
在临安市板桥镇雷竹林地,按照雷竹集约经营要求于2010年12月7日进行冬季(第1次)施肥,翌日覆盖竹叶;2011年6月20日,揭去部分覆盖物后将剩余的竹叶翻入土壤,10 d后(6月30日)进行春季(第2次)施肥,2次的施肥方案相同(表1),为了比较覆盖与否对土壤氨氧化微生物的影响,增设了前期不覆盖的对照1与施肥相同但覆盖的处理1的比对试验(第1个试验);覆盖后不同肥料处理对土壤氨氧化微生物的影响的试验为4个处理(第2个试验),分别为对照2(不施肥),处理1,处理2和处理3,施肥品种及用量见表1,其中处理1为2个试验共用。各处理以含氮量360 kg·hm-2为标准折算成相应的肥料用量,重复3个·处理-1,随机区组排列,共计15个小区,小区面积5 m×5 m。按表1中试验设计要求将肥料均匀地散施于地表再翻入10 cm土层。

表1 各处理化肥品种、用量及田间排列顺序Table1 Design of the trial with different fertilizers and the order of treatments in field
1.3 土壤样品采集
2011年7月8日(施肥后7 d),在每个小区用土钻多点随机采集表层0~20 cm土壤样品,混匀装入无菌密封袋,放入事先准备好的冰盒带回实验室。新鲜土壤充分混匀后,去除石块和动植物残体,过2 mm钢筛。样品存放于-70℃冰箱内,待冷冻干燥后用于土壤细菌总DNA的提取,供土壤氨氧化细菌和氨氧化古菌(AOA)群落结构及定量分析,剩余的样品存放于-20℃冰箱中保存以备后续分析。
1.4 主要试剂与引物
Power Soil TM Total DNA Isolation Kit试剂盒购于美国Mo Bio公司,Taq DNA聚合酶购于大连TaKaRa公司,定量聚合酶链式反应(PCR)试剂采用Takara公司的SYBR Premix Ex TaqTM(Perfect Real Time)(TaKaRa Code∶DRR041),荧光染料SYBR greenⅠ购于美国Invitrogen公司,引物由生工生物工程(上海)有限公司合成。
1.5 分析方法
1.5.1 土壤总DNA的提取及纯化 采用Power Soil TM Total DNA Isolation K试剂盒提取土壤总DNA,按试剂盒说明书操作,通过10 g·kg-1琼脂糖电泳对所提取DNA效果进行鉴定,提取的DNA保存于-40℃冰箱中。
1.5.2 土壤DNA的PCR扩增 以土壤总DNA为模板扩增氨氧化古菌氨单加氧酶基因amoA片段采用引物对 GC-Arch-amoAR 和 Arch-amoAF,Arc-amoAF:5′-STA ATGGTCTGGCTTAGACG-3′;GC-Arc-amoAR:5′-GCGGCCATCCATCTGATTGT-3′,扩增AOB氨单加氧酶基因amoA片段采用引物对GC-amoA-1F和amoA-2R,GC-amoA-1F:5′-GGGGTTTCTACTGGTGGT-3′;amoA-2R:5′-CCCCTCGGGAAAGCCTTCTTC-3′。使用Bio-Rad公司的S1000TM Thermal Cycler PCR仪对土壤DNA进行扩增。20.0 μL反应体系如下:预混合液(premix) 10.0 μL,引物(50 μmol·kg-1)各 0.2 μL,牛血清白蛋白(BSA)0.2 μL,模板 DNA 0.2 μL,用无菌双蒸水补足至20.0 μL。PCR反应参数:94℃预变性5 min,94℃变性45 s,55℃退火1 min,72℃延伸1 min,35个循环,最后72℃延伸10 min。取3.0 μL PCR反应产物用10.0 g·kg-1的琼脂糖凝胶进行电泳,检测产物及其长度。
1.5.3 变性梯度凝胶电泳(DGGE)使用DcodeTM Universal Mutation Detection System (Bio-Rad),AOB amoA基因PCR产物检测是在在变性梯度为40%~65%的60.0 g·kg-1聚丙烯酰胺凝胶上进行变性梯度凝胶电泳;氨氧化古菌 amoA基因PCR产物检测是在变性梯度为22.5%~42.5%的60 g·kg-1聚丙烯酰胺凝胶上进行变性梯度凝胶电泳。电泳前对PCR产物定量、校正,使上样量一致。在60℃,80 V条件下电泳13.5 h后,用Gel red染料染色30 min,染色结束后用GelDocTM EQ(Bio-Rad)凝胶成像系统成像,使用Quantity One 4.4软件(Bio-Rad)进行图像分析。
1.5.4 实时荧光定量PCR(Real-time PCR)使用CFX96TM Real-Time System(Bio-Rad)仪器进行氨氧化古菌和氨氧化细菌的功能基因的实时定量分析,进而确定样品的原始拷贝数。氨氧化细菌和氨氧化古菌功能基因定量引物序列分别为:amoA-1F:5′-ggggTTTCTACTggTggT-3′amoA-2R:5′-CCCCTCgggAAAgCCTTCTTC-3′Arch-amoAF:5′-STAATGGTCTGGCTTAGACG-3′Arch-amoAR:5′-GCGGCCATCCATCTGTAT GT-3′。20 μL 反应体系如下:SYBR Premix Ex TaqTM 10.0 μL,引物(50 μmol·L-1)各 0.2 μL,模板DNA1.0 μL,无菌双蒸水补足至20.0 μL。氨氧化古菌反应程序:95℃,3.0 min;35×(95℃,30 s;55℃,30 s;72℃,30 s终点读板);溶解曲线65.0~95.0℃,增量0.5℃,0:05+终点读板。氨氧化细菌反应程序:95℃,3.0 min;38×(95℃,30 s;55℃,30 s;72℃,30 s终点读板);溶解曲线65.0~95.0℃,增量0.5℃,0:05+终点读板。样品DNA浓度与拷贝数之间的转化公式:摩尔数M=DNA质量浓度(μg·L-1)/(重组质粒片段长度×324.5×2),Nc=6.02×1023×M。
1.5.5 数据分析 采用SPSS 18.0统计软件进行数据处理,Duncan单因素方差分析比较各处理之间的差异显著性(P<0.05)。采用Bio-Rad公司的Quantity One 4.4进行DGGE指纹图谱分析。用Quantity One软件进行Shannon多样性指数(H)和Pielou均匀度指数(EH)的计算和聚类分析,其中聚类分析用非加权组平均法(UPGMA)。计算公式如下:H=-Σ(ni/N)ln(ni/N),EH=H/lnS。其中∶ni为单一条带的强度,N为所有条带的总强度;S为每一泳道总的条带数。
2 结果与分析
2.1 雷竹林土壤氨氧化细菌群落结构及多样性分析
2.1.1 土壤氨氧化细菌微生物群落变性梯度凝胶电泳(DGGE)图谱分析 应用变性梯度凝胶电泳(DGGE)技术对PCR产物进行分离,通过指纹图谱可以清楚看到分离为若干条带(图1A),不同处理的土壤样品显示出不同的条带类型,对DGGE图谱进行统计发现:不同处理土壤在DGGE图谱中电泳条带的数目、亮度、迁移的位置均存在一定程度的差异。在DGGE图谱上,处于不同位置的DNA条带及其相对浓度(亮度)可能代表微生物群落中某一特定微生物种及其在群落中的相对丰度[20]。通过分析DGGE图谱发现所有的土壤样品共有31条位置不同的条带,比本地阔叶林、杉木Cunninghamia lanceolata 林[21-22]等土壤少,但比旱地农田[18]和水稻土壤[23]则多。不同土壤样品间共同条带 17 条,占条带总类型的54.8%,但这些共性条带的亮度不同;此外,还有不少条带(6,10,12,13,23,24,26,30)是大多数样品的共有条带,说明不同施肥处理土壤有较高比例的共性氨氧化细菌种类,其中半数以上氨氧化细菌的物种完全相同,只是丰度不同。与总体较高比例共性条带形成显明对照的是,虽然同一处理不同重复之间条带位置基本一致,但亮度上有明显差异,特别是处理1和处理3。可能的原因有二:其一是因为田间试验时的肥料以固体撒施的方法进行,无法达到完全均匀;其二是用于DNA提取的土壤样品量太少(0.3 g),样品的代表性受到影响,虽然是多点的混合样品,但新鲜土壤不能像风干土那样混合充分。不同处理间的差异情况,平均条带由高到低的顺序依次是:对照2(29条)>处理2(28条)>处理1(25条)>对照1(23条)>处理3(22条)。对照1的条带模式与其他处理的差异更明显,其中22号条带亮度特别强,30号条带亮度是对照1的特色条带,而其他条带则不如其他处理强,说明前期覆盖以及后期竹叶翻入土壤的大量有机物质对氨氧化细菌的影响。对照1和处理1施用同样的含有硝态氮的复合肥,条带模式明显不同。前期覆盖的处理1条带数多于未覆盖的对照1,说明前期覆盖以及覆盖物翻入土壤对土壤氨氧化细菌有影响,但发现两者有1条(条带29)共性条带比其他施肥处理的亮,可能是复合肥的作用结果。处理3的条带数比其他处理少,可能与氯化钾中氯离子对微生物的抑制作用所致。Shannon指数反映了群落中物种的变化度或差异度,受样本总数和均匀度的影响,一般来说,物种丰富度高且分布较均匀的群落的 Shannon指数较高[24]。Shannon多样性指数由高到低的顺序依次是:对照2>处理2>处理1>对照1>处理3(表2),不同处理间氨氧化细菌多样性指数差异性显著(P<0.05),而施用复合肥的圴匀度指数(EH)也明显高于尿素处理(表2)。

图1 土壤样品氨氧化细菌amoA基因扩增片段的DGGE分析结果(A)以及DGGE条带图谱的聚类分析(B)Figure1 DGGE analytical result and cluster analysis of amoA fragment of ammonia-oxidizing bacterial communities from the soil samples

表2 不同施肥土壤氨氧化细菌和氨氧化古菌的多样性Table2 Diversity properties of soil AOB and AOA calculated from DGGE band pattern data under different fertilization treatments
2.1.2 土壤氨氧化细菌的DGGE图谱聚类分析 聚类分析可了解不同处理对氨氧化细菌群落结构的定量影响。利用聚类分析非加权组平均法(UPGMA)对不同施肥处理下的氨氧化细菌群落结构DGGE图谱进行分析得图1B。土壤样品明显分为两大组:对照1单独为一组,其他处理为另一组,但相似度值略大于临界值0.6,说明前期覆盖对土壤氨氧化细菌的影响大于施肥处理,但结构没有发生显著变化。所有前期覆盖处理的土壤样品,除了异常的处理1,处理2和处理3外又分为2组,其中处理3(尿素+氯化钾)为一组,进一步证明处理3施入氯化钾中的氯元素对土壤的氨氧化细菌群落结构有一定影响。以上结果分析表明,覆盖和不同肥料的集约经营措施在短期内能降低氨氧化细菌的多样性,改变土壤氨氧化细菌的群落结构,但变化幅度尚未超出相似度临界值0.6,如长期采用覆盖措施并施用同样的肥料,则土壤氨氧化细菌的群落结构可能发生明显改变;施用复合肥(处理1)的土壤氨氧化细菌丰富度显著大于(P<0.05)尿素与氯化钾混合施肥(处理3),施用复合肥对维持土壤氨氧化细菌的群落多样性有一定好处。
2.2 雷竹林土壤氨氧化古菌群落结构及多样性分析
2.2.1 长期施肥氨氧化古菌微生物群落DGGE图谱分析 应用DGGE技术对雷竹林土壤氨氧化古菌能基因amoA的PCR产物进行分离。所有的土壤样品共有26条位置不同的条带(图2A),比相同土壤样品的氨氧化细菌条带数少,而宋亚娜等[23]在水稻土中检测到的氨氧化古菌种类明显多于氨氧化细菌种类。所有处理DGGE条带的位置基本相同,但不同处理条带的亮度有差异,处理3的3个重复有很好的一致性,且条带亮度大,其中条带1明显比其他处理亮、而条带20则明显弱于其他处理外,其他位置的条带没有显示处理间的规律性差异,相反重复间差异大于处理间。用于氨氧化古菌与氨氧化细菌扩增的为同一DNA提取液,不同重复间氨氧化古菌的差异明显大于氨氧化细菌,说明土壤氨氧化古菌对高浓度肥料比氨氧化细菌敏感,然而尿素+氯化钾对氨氧化古菌则没有不利影响。其他研究报道:氨氧化古菌比氨氧化细菌受施肥、植被及土壤pH值等环境影响较小[21,25]。可能的解释是,氨氧化古菌比氨氧化细菌在一定范围内受环境影响小于氨氧化细菌,但大量施肥这种强度干扰对氨氧化古菌的影响反而大于氨氧化细菌,当然这只是推测,确切的结果必须通过进一步的培养实验来证实。不同处理之间氨氧化古菌物种的差异不大,但Shannon多样性指数(Shannon index)从平均值看,对照1(前期未覆盖+复合肥)大于处理1(前期覆盖+复合肥),说明前期覆盖及大量的竹叶翻入土壤可能会降低雷竹林地氨氧化古菌的物种多样性。雷竹林去掉覆盖物后不同施肥处理间Shannon多样性指数由高到低的顺序依次是:处理3>处理2>对照2>处理1,由此可以看出,施加尿素态氮肥能够增加土壤中氨氧化古菌的种群丰富度,但是处理1的复合肥是含硝态氮的复合肥,而处理2和处理3所含的是铵态氮的氮肥。均匀度指数(EH)对照1最高,处理2最低,其他处理之间则没差异(表2)。土壤氨氧化古菌对覆盖和不同肥料的反应正好与氨氧化细菌相反。
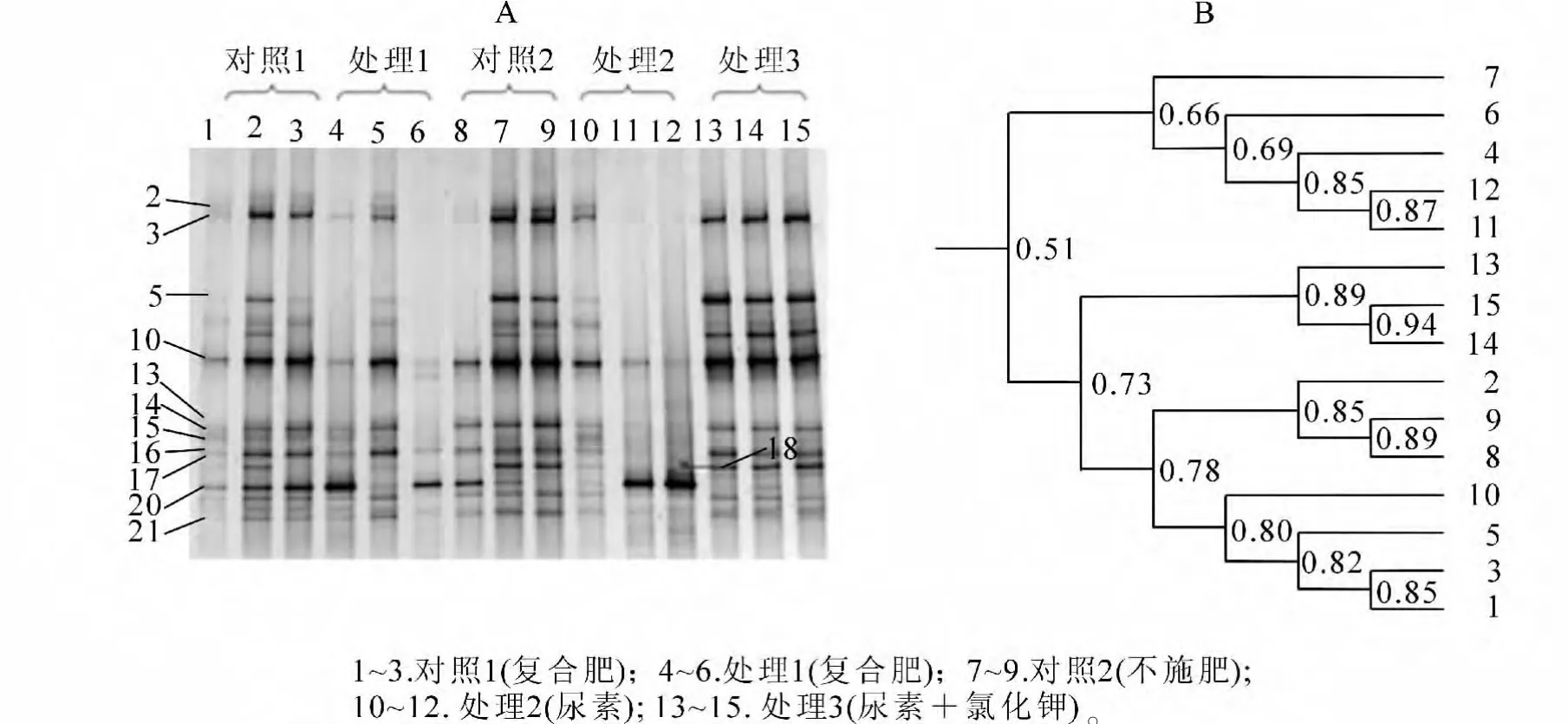
图2 土壤样品氨氧化古菌amoA基因扩增片段的DGGE分析结果(A)以及DGGE条带图谱的聚类分析(B)Figure2 DGGE analytical result and cluster analysis of amoA fragment of ammonia-oxidizing archaea communities from the soil samples
2.2.2 土壤氨氧化古菌的DGGE图谱聚类分析 为进一步了解施肥处理对氨氧化古菌群落结构的影响,利用聚类分析非加权组平均法(UPGMA)对不同施肥处理下的氨氧化古菌群落结构DGGE图谱进行分析。观察结果(图2B)得出,所有样品分为2组,上面一组为不同处理中出现异常低的样品(4,6,7,11,12),其余样品聚为一类的相似值为0.73,说明不同处理间结构相似度较高,不同肥料对氨氧化古菌群落结构组成影响较氨氧化细菌小。以上结果分析表明,不同施肥处理土壤氨氧化古菌的群落结构变化也不大,大部分样品群落的相似度为0.73,不同处理间氨氧化古菌物种没有差异,但丰度差异较大,其中处理3土壤氨氧化古菌结构多样性显著高于处理1(P<0.05)。
2.3 雷竹林土壤氨氧化细菌和氨氧化古菌amoA基因定量分析
通过amoA基因的拷贝数可以分别估算氨氧化细菌和氨氧化古菌的丰富度,应用Real-Time PCR技术获得各样品起始拷贝数换算出每克干土的氨氧化古菌和氨氧化细菌的amoA基因拷贝数(Nc,图3),氨氧化细菌的amoA基因拷贝数最多的是处理3(干土拷贝数4.47×107·g-1),其他依次为对照2,处理2和处理1,对照1最少,其中处理3土壤氨氧化细菌的amoA基因拷贝数要显著高于(P<0.05)对照1,是对照1的2.50倍,而与其他的处理之间没有显著性差异。不同处理土壤的氨氧化古菌除处理3外没有显著差异。不同样品的定量分析结果与DGGE图中条带数量与强度基本一致,条带多亮度大则基因拷贝数多,然而也有例外的情况,氨氧化细菌的处理3条带数明显少于其他处理,但拷贝数反而最高,处理2的氨氧化古菌的条带数明显比其他样品少,但基因拷贝数的差异则没有条带明显,以上情况说明,有些条带因为浓度低不能在DGGE上显示出来,PCR-DGGE通常只能检测到微生物群落中数量上大于1%的优势种群[26],但定量分析则可以检测到所有基因片段,因此定量分析能更准确地反应基因的丰度。处理3的氨氧化古菌和氨氧化细菌基因拷贝均为最高,而且重复间的差异也最小,说明施肥后虽然部分种类的氨氧化细菌被弱化,不能在DGGE图上显示,但从功能基因数量角度看尿素+氯化钾施用后没有产生不良影响。
不同处理的土氨氧化古菌与氨氧化细菌 amoA基因拷贝数的比值(NcAOA/NcAOB)范围为 1.18~2.70。前期覆盖的4个处理中处理3最高,与贺纪正等[18]对酸性红壤长期施肥定位试验地不同处理的氨氧化细菌和氨氧化古菌研究一致,NcAOA/NcAOB比值最高的是处理3(7.47),其他处理均低于对照,即使是施用有机肥,说明施肥降低了NcAOA/NcAOB的比值,改变了氨氧化细菌和氨氧化古菌的组成和结构。土壤氨氧化细菌和氨氧化古菌的组成受土壤pH值、施肥和土地利用、土壤类型[27]等因素的影响,碱性土壤NcAOA/NcAOB比值大于酸性土壤[25]。在不施肥的自然森林土壤中,许多因素如土壤pH值、有效磷、微生物量碳、硝酸盐和总氮可能影响氨氧化微生物的组成,养分丰富、pH值中性则amoA基因拷贝数多[28]。而对于大量施肥的土壤,养分水平总体很高,小幅变化则不会引起土壤氨氧化微生物结构的改变。本试验中,2次大量施肥使土壤养分水平处于过高状态,因此影响功能基因丰度的因素是肥料品种及组合方式而非养分水平。结合比较不同处理氨氧化细菌和氨氧化古菌的DGGE条带数和定量分析数据发现,对强度经营的雷竹林土壤要维持较好的土壤硝化功能和氮素循环,建议采用处理3的肥料搭配。

图3 不同处理下氨氧化古菌amoA基因和氨氧化细菌amoA基因的丰富度Figure3 Abundance of ammonia oxidizing bacterial amoA genes and ammonia oxidizing archaeal amoA genes under different treatments
3 结论
不同施肥处理土壤有较高比例的共性氨氧化细菌种类,群落结构尚未发生明显改变;前期未覆盖的对照1与其他前期曾覆盖的所有处理的氨氧化细菌群落结构差异较明显,同为前期覆盖的处理3与其他处理又稍有不同;处理1土壤的氨氧化细菌多样性显著高于处理3(P<0.05),而基因的丰度则处理3最高。不同施肥处理土壤氨氧化古菌的群落结构变化不大,除去处理1,对照2和处理2中的异常样品后,群落的相似度高达0.73;处理3与其他处理有2个条带亮度存在明显差异,其余处理间条带则没有差异,但丰度的差异较大。处理3土壤氨氧化古菌结构多样性显著高于处理1(P<0.05),基因的丰度也是处理3最高。综合比较不同处理氨氧化细菌和氨氧化古菌的DGGE条带的多样性以及定量分析得出结论,处理3的氨氧化微生物多样性和功能基因最丰富,处理2最差。建议生产上采用尿素与氯化钾搭配施用,有利于土壤氨氧化微生物的活动和氮素循环利用。
[1]LOVELL R D,JARVIS S C,BARDGETT R D.Soil microbial biomass and activity in long-term grassland∶effects of management changes [J].Soil Biol Biochem,1995,27:969-975.
[2]BÖHME L,LANGER U,BÖHME F.Microbial biomass,enzyme activities and microbial community structure in two European long-term field experiments [J].Agric Ecosyst Environ,2005,109:141-152.
[3]BRUSSAARD L,DE RUITER P C,BROWN G G.Soil biodiversity for agricultural sustainability[J].Agric,Ecosyst Environ,2007,121(3)∶233-244.
[4]LEININGER S,URICH T,SCHLOTER M,et al.Archaea predominate among ammonia oxidizing prokaryotes in soils [J].Nature,2006,442∶806-809.
[5]PROSSER J I,NICOL G W.Relative contributions of archaea and bacteria to aerobic ammonia oxidation in the environment[J].Environ Microbiol,2008,10∶2931-2941.
[6]KOWALCHUK G A,STEPHEN J R.Ammonia-oxidizing bacteria∶a model for molecular microbial ecology [J].Ann Rev Microbiol,2001,55(1)∶485-529.
[7]WESSÉN E,HALLIN S.Abundance of archaeal and bacterial ammonia oxidizers-possible bioindicator for soil monitoring [J].Ecol Indic,2011,11∶1696-1698.
[8]OKANO Y,HRISTOVA K R.Application of real-time PCR to study effects of ammonium on population size of ammonia-oxidizing bacteria in soil[J].Appl Environ Microbiol,2004,70∶1008-1016.
[9]JENSEN K,SLOTH N P.Estimation of nitrification and denitrification from microp rofiles of oxygen and nitrate in model sediment systems [J].Appl Environ Microbiol,1994,60∶2094-2100.
[10]FRIJLINK M J,ABEE T,LAANBROCK H J.The bioenergetics of ammonia and hydroxylamine oxidation in nitrosomonas europaca by altering pH to affect NH3availability [J].Appl Environ Microbiol,1997,63∶4588-4592.
[11]BURTON S,PROSSER J I.Autotrophic ammonia oxidation at low pH through urea hydrolysis [J].Soil Biochem,1998,30∶509-515.
[12]FIERER N,CARNEY K M,HORNER-DEVINE M C,et al.The biogeography of ammonia-oxidizing bacterial communities in soil[J].Microbial Ecol,2009,58∶435-445.
[13]SUNDBERG C,STENDAHL J S K,TONDERSKI K.Over land flow systems for treatment of landfill leachates∶Potential nitrification and structure of the ammonia-oxidising bacterial community during a growing season[J].Soil Biol Biochem,2007,39∶127-138.
[14]贺纪正,张丽梅.氨氧化微生物生态学与氮循环研究进展[J].生态学报,2009,29(1)∶406-415.HE Jizheng,ZHANG Limei.Advances in ammonia-oxidizing microorganisms and global nitrogen cycle [J].Acta Ecol Sin,2009,29(1):406-415.
[15]ZHANG Pingjiu,ZHENG Jufeng,PAN Genxing et al.Changes in microbial community structure and function with in particle size fractions of a paddy soil under different long-term fertilization treatments from the Tai Lake Region,China [J].Coll Surf B Bioint,2007,58∶264-270.
[16]CHU H Y,FUJII T,MORIMOTO S,et al.Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil [J].Appl Environ Microbiol,2007,73(2)∶485-491.
[17]COLLOFF M J,WAKELIN S A,GOMEZ D,et al.Detection of nitrogen cycle genes in soils for measuring the effects of changes in land use and management[J].Soil Biol Biochem,2008,40(7)∶1637-1645.
[18]HE Jizheng,SHEN Jupei,ZHANG Limei,et al.Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long term fertilization practices [J].Environ Microbiol,2007,9∶2364-2374.
[19]姜培坤,周国模,徐秋芳.雷竹高效栽培措施对土壤碳库的影响[J].林业科学,2002,38(6)∶6-11.JIANG Peikun,ZHOU Guomo,XU Qiufang.Effect of intensive cultivation on the carbon pool of soil in Phyllostachys praecox stands [J].Sci Silv Sin,2002,38(6)∶6-11.
[20]KUNTAL M H,ANAND S,MISHRA B,et al.Impact of long-term application of fertilizer,manure and lime under intensive cropping on physical properties and organic carbon content of an Alfisol [J].Geoderma,2008,148∶173-179.
[21]刘卜榕.亚热带4种主要森林土壤氨氧化微生物群落结构及多样性研究[D].临安:浙江农林大学,2012.LIU Burong.Community and Diversity of Ammonia-oxidizing Microbes in Four Major Types of Forest in Subtropics ofChina [D].Lin’an∶Zhejiang A&F University,2012.
[22]田甜.毛竹林土壤氨氧化微生物功能基因多样性[D].临安:浙江农林大学,2011.TIAN Tian.Functional Gene Diversity of Ammonia-oxidizing Microbes of Soil in Phyllostachys pubescens [D].Lin’an∶Zhejiang A&F University,2011.
[23]宋亚娜,林智敏,林捷.不同品种水稻土壤氨氧化细菌和氨氧化古菌群落结构组成[J].中国生态农业学报,2009,17(6)∶1211 - 1215.SONG Yana,LIN Zhimin,LIN Jie.Composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea communities in paddy soils of different rice cultivars [J].Chin J Eco-Agric,2009,17(6)∶1211 - 1215.
[24]MAGURRAN A E.Ecological Diversity and Its Measurement[M].Princeton∶Princeton University Press,1988.
[25]SHEN Jupei,ZHANG Limei,ZHU Yongguan,et al.Abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea communities of an alkaline sandy loam [J].Environ Microbiol,2008,10(6)∶1601-1611.
[26]VALLAEYS T,TOPP E,MUYZER G,et al.Evaluation of denaturing gradient gel electrophoresis in the detection of 16S rDNA sequence variation in rhizobia and methanotrophs [J].FEMS Microbiol Ecol,1997,24(3)∶279 - 285.
[27]GIRVAN M S,BULLIMORE J,PRETTY J N,et al.Soil type is the primary determinant of the composition of the total and active bacterial communities in arable soils [J].Appl Environ Microbiol,2003,69∶1800 - 1809.
[28]HAYDEN H L,DRAKE J,IMHOF M,et al.The abundance of nitrogen cycle genes amoA and nifH depends on land-uses and soil types in South-Eastern Australia [J].Soil Biol Biochem,2010,42∶1774 - 1783.
