含有CRISPR序列的噬菌体耐受菌的筛选
2014-05-18滑玉会张志毅安小平米志强尹秀云陈建魁童贻刚
滑玉会,黄 勇,张志毅,安小平,米志强,尹秀云,陈建魁,童贻刚
规律成簇的间隔短回文重复(clustered regularly interspaced short palindromic repeats,CRISPR)是细菌内存在的专门针对噬菌体及质粒等外源基因的获得性免疫系统[1],这种抗性作用的发挥类似于真核生物中的RNA干扰作用。在噬菌体治疗过程中,细菌往往会对噬菌体产生耐受,导致耐受噬菌体的菌株出现。关于噬菌体耐受菌株的筛选,国内外已有较多的研究[1-2],如通过筛选嗜热链球菌抗噬菌体突变株来提高发酵酸奶的生产性能,以及通过筛选噬菌体耐受菌来研究细菌耐受噬菌体的机制。该研究通过PCR方法筛选含有CRISPR系统的大肠埃希菌,以含有CRISPR系统的大肠埃希菌为指示菌分离噬菌体,进而在人为条件下筛选出耐受噬菌体的大肠埃希菌,拟利用这些细菌分析噬菌体和CRSIPR系统之间的关系,为进一步了解CRISPR的作用特点和提高噬菌体治疗效果提供依据。
1 材料与方法
1.1 材料 临床分离的70株致病性大肠埃希菌来自于军事医学科学院附属307医院检验科,用于分离噬菌体以及筛选噬菌体耐受株;PCR试剂盒购自北京全式金生物技术有限公司;胶回收试剂盒购自天津宝瑞生物技术有限公司;LB培养基:胰蛋白胨10 g,酵母提取物5 g,氯化钠10 g,用蒸馏水定容至1 L,调至 pH 7.0,121 ℃ 下高压灭菌 20 min。固体LB培养基需添加1.5%琼脂粉,半固体LB培养基需添加0.75%琼脂粉。SM缓冲液:氯化钠5.8 g,硫酸镁2.0 g,pH 7.5,1 mol/L Tris-HCl 50 ml,2%明胶5 ml,加水至1 L;化学试剂均为国产分析纯;2720型PCR仪(ABI公司);DYY-6C电泳仪(北京六一仪器厂);BIO-RAD Gel Doc XR凝胶成像系统。
1.2 含有CRISPR系统大肠埃希菌的筛选 根据CRISPR database(http://crispr.u-psud.fr/crispr/)公布的E.coli菌株的CRISPR序列特征,利用网站中FlankAlign选项的 Flanking sequence Alignement Tool,分别选取200 bp长度的大肠埃希菌CRISPR序列right flanking sequence和left flanking sequence,按照同源性比对结果将其分为15个组,将每组对应的序列导入到CLC软件里,分别得到一个consensus序列,利用NCBI网站上的primer-blast功能,设置各种参数,经过引物特异性检测,只有4对引物符合要求。P10(F:TACCGTTGGTGAAGGAGCTG,R:TTCCGGTGGATTTGGATGGG)、P13(F:TTGAGTTTCAGA CCTGGGGC,R:TGCTACCGTTGGTGAAGGAG)、P14(F:CTGGGAGTTCTACCGCAGAG,R:CCCCGGAGTATTTGGATGGT)、P15(F:GGAAAATGGGAGCTGGGTGT,R:ACCTGGGGAGAAAACAGACG)。采用PCR法对供试菌株进行CRISPR序列扩增,反应体系参照PCR试剂盒使用说明,回收PCR产物并测序。
1.3 相关噬菌体的分离 以含有CRISPR的菌株E.coli 147-30为指示菌,于军事医学科学院附属307医院污水处理处收集未经消毒处理的污水,12 209 r/min离心2次,每次10 min以除去固体杂质,收集上清液;用0.22 μm滤器过滤除菌,取4 ml滤液加入2 ml经高压灭菌的3×LB培养基中,接种500 μl过夜培养的宿主菌E.coli 147-30,混合均匀后置于摇床内,37℃过夜培养。12 209 r/min离心10 min收集上清液,0.22 μm滤器过滤,滤液即为含有噬菌体的原液。
将噬菌体原液10倍梯度稀释,分别取各梯度噬菌体稀释液100 μl和500 μl对数期的宿主菌混合均匀后,室温放置10 min以使其充分吸附后加入45℃上层半固体培养基5 ml,混合均匀,然后倾倒入固体培养基平板上,凝固后置于37℃培养,观察噬菌斑的出现。用无菌枪头从噬菌斑分布均匀的平板上挑取单个噬菌斑,加入到对数期的宿主菌中,置于37℃摇床中培养6 h,12 209 r/min离心10 min,0.22 μm滤器过滤,滤液置于4℃保存。重复以上步骤3~4次,以得到大小和形态保持一致的噬菌斑。
噬菌体形态的电镜观察:将保存在SM缓冲液中的噬菌体滴在覆有碳膜的铜网上,室温晾干后用滤纸吸去未经吸附的溶液;滴一滴磷钨酸(PTA)染色2 min,用滤纸吸去多余的液体;室温干燥后用HITACHI HT7700型透射电子显微镜进行观察,记录噬菌体颗粒的形态和大小。
1.4 大肠埃希菌噬菌体耐受株的筛选 采用PEG沉淀的方法[3]浓缩噬菌体IME-EC1以使其达到一个较高的滴度(约1010PFU/ml),大肠埃希菌噬菌体耐受株的筛选参照文献[4-5]报道,具体方法简述如下:将过夜培养的宿主菌进行10倍梯度稀释,然后分别取各梯度稀释液100 μl分别与100 μl噬菌体悬液(1010PFU/ml)混合均匀,吸附10 min后加入到45℃上层半固体培养基中,混合均匀,然后倾倒入固体培养基平板上,凝固后置于37℃培养24 h至单克隆出现。
随机挑取平板上产生的单克隆,加入到5 ml经高压灭菌的LB液体培养基中,置于37℃中过夜培养,采用点滴法[6]来鉴定单克隆对噬菌体是否产生了真正的耐受以及耐受能否稳定的保持到下一代细菌,具体步骤为:将培养至对数期的敏感菌和耐受菌分别加入到融化的半固体LB培养基中,待凝固后,取2 μl的噬菌体原液滴在双层平板上,待平板晾干后倒置于37℃培养观察噬菌斑的有无。敏感菌的平板上能够产生比较透亮的噬菌斑,而耐受菌的平板上,由于耐受菌对噬菌体不再敏感,则不会产生噬菌斑。
2 结果
2.1 含有CRISPR系统大肠埃希菌的筛选 采用菌落 PCR的方法,分别采用 4对人工设计的CRISPR特异性引物对70株大肠埃希菌的CRISPR序列进行扩增,结果从70株供试菌株中检测到42株大肠埃希菌中含有CRISPR序列,见表1。选择其中1株大肠埃希菌(E.coli 147-30)用于后续相关噬菌体的分离以及噬菌体耐受菌筛选实验。见图1。设计的4对特异性引物中,有3对引物(10、13、15)扩增出CRISPR序列。将E.coli 147-30的PCR产物直接进行序列分析,结果显示该3对引物扩增的序列互相重叠,可以拼接成一个较长的contig序列,用 CRISPR database 网站(http://crispr.u-psud.fr/crispr/)中CRISPR Finder功能分析该CRISPR序列结构特征,显示该位点共有18个重复单元,其中重复序列长为29 bp,序列为CGGTTTATCCCCGCTGGCGCGGGGAACAC,间隔序列长为32 bp,见表2。

图1 E.coli 147-30的CRISPR序列扩增结果
2.2 相关噬菌体的分离 以含有CRISPR的菌株E.coli 147-30为指示菌,于军事医学科学院附属医院未经消毒处理的污水分离到1株噬菌体,命名为IME-EC1,噬菌斑的大小不均一,如针孔状并且透明,见图2。纯化后的噬菌体IME-EC1经负染后在透射电镜下的形态见图3。该噬菌体呈蝌蚪形,具有典型的二十面体结构,头部宽约70 nm,长约100 nm,尾部长约115 nm,尾部底端具有数根尾丝,属于肌尾噬菌体科。
2.3 大肠埃希菌噬菌体耐受株的筛选 采用PEG沉淀的方法浓缩噬菌体 IME-EC1,滴度约为1010PFU/ml。用浓缩得到的高滴度噬菌体筛选得到了大肠埃希菌噬菌体耐受菌,采用点滴法鉴定筛选得到的耐受菌,见图4。由于147-30R1对噬菌体产生了耐受性,所以平板上没有发现透亮的噬菌斑。选用其中1株噬菌体耐受菌(命名为147-30R1)用于后续实验的研究。147-30R1在连续培养数代以后能够继续稳定保持对噬菌体IME-EC1的耐受。

表1 CRISPR序列在大肠埃希菌中的分布情况

表2 E.coli 147-30的CRISPR序列相关信息
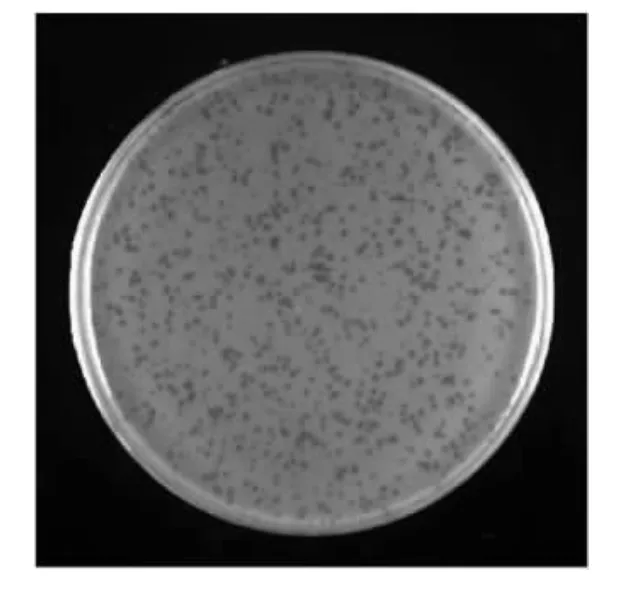
图2 噬菌斑的形态

图3 噬菌体在电镜下的形态 ×40 000

图4 点滴法鉴定噬菌体耐受菌
3 讨论
在细菌和古细菌中广泛存在CRISPR-Cas获得性免系统是新发现的一个机制,该机制能够限制相应噬菌体、质粒等外源DNA的入侵。研究[1]表明CRISPR系统能够为Streptococcus thermophiles提供抵抗噬菌体感染的获得性免疫,显示了间隔序列和噬菌体基因组序列之间存在同源性。在噬菌体、质粒等外源性DNA入侵的过程中,新的重复序列(repeats,R)-间区序列(spacers,S)片段被获得并被插入到前导序列和原来第一个重复序列之间,从而使宿主获得抵抗相应噬菌体、质粒等再次入侵的获得性免疫能力。目前,CRISPR系统作为一种能够抵御噬菌体感染的防御机制,对其作用的其他分子作用机制并不十分了解。
为了更清楚的研究细菌耐受噬菌体现象和CRISPR系统之间的相互关系,根据CRISPR database数据库中已经公布的大肠埃希菌的CRISPR序列的信息,依据其Flanking sequence的同源性比对结果设计了4对特异性的引物,对4对引物筛选出的CRISPR序列进行分析发现,4对引物所扩增的CRISPR序列其实是一个CRISPR位点,但是由于菌株自身的差异导致其与每条引物结合的特异性不同,所以才会出现有些引物呈阳性,有些引物为阴性的结果,这与后期序列分析结果吻合。首先从本实验室保存的细菌库中成功筛选到42株含有CRISPR序列的大肠埃希菌,然后又以含有CRISPR序列的大肠埃希菌为指示菌分离得到了相应的噬菌体IME-EC1,成功筛选了含有CRISPR序列的噬菌体耐受菌,为后续研究CRISPR系统与细菌耐受噬菌体现象之间的相互关系打下了基础。在噬菌体IMEEC1的培养过程中,发现在平板上产生的噬菌斑的大小常不均一,这可能与培养基pH、琼脂浓度、宿主菌的浓度、培养温度、培养时间有关,即使是在同一培养条件下、同一平板上的噬菌斑大小也常不一致。另外,已经完成了对噬菌体敏感菌E.coli 147-30和耐受菌E.coli 147-30R1的全基因组测序,以分析噬菌体耐受菌中CRISPR系统的序列特征,期望从分子机制上解释噬菌体耐受菌出现的原因,相关的生物信息学分析工作正在进行。
总之,成功建立了筛选含有CRISPR序列的大肠埃希菌的方法,从70株大肠埃希菌中筛选到42株含有CRISPR序列的细菌,筛选率达到60%。序列分析显示筛选的E.coli 147-30大肠埃希菌中所含有的CRISPR序列在数据库中是独特的,这也丰富了大肠埃希菌中CRISPR序列的信息资源。同时筛选到含有CRISPR序列的噬菌体耐受菌,敏感菌和耐受菌中所含有的巨大的序列信息对以后更加深入的研究细菌耐受噬菌体的现象提供了基础数据。
[1]Barrangou R,Fremaux C,Deveau H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709 -12.
[2]冷一非,赵述淼,朱 辉,等.嗜热链球菌抗噬菌体菌株的选育[J].中国酿造,2010,7(220):118-20.
[3]萨姆布鲁克J,拉塞尔D W.分子克隆实验指南[M].黄培堂,译.北京:科学出版社,2002:186-7.
[4]Johnson K,Lory S.Characterization of Pseudomonas aeruginosa mutants with altered piliation[J].J Bacteriol,1987,169(12):5663-7.
[5]Guerrero-Ferreira R C,Viollier P H,Ely B,et al.Alternative mechanism for bacteriophage adsorption to the motile bacterium Caulobacter crescentus[J].Proc Nati Acad Sci U S A,2011,108(24):9963-8.
[6]Kim M,Ryu S.Characterization of a T5-like coliphage,SPC35,and differential development of resistance to SPC35 in Salmonella enterica serovar Typhimurium and Escherichia coli[J].Appl Environ Microbiol,2011,77(6):2042 - 50.
