急诊重症监护病房感染患者耐氨基苷鲍曼不动杆菌的耐药机制
2014-05-13赵雪于沛涛徐芝君顾清裘力锋王弋
赵雪,于沛涛,徐芝君,顾清,裘力锋,王弋
(1.杭州市第一人民医院急诊科,杭州 310003;2.北京大学基础医学院,北京 100191)
急诊重症监护病房感染患者耐氨基苷鲍曼不动杆菌的耐药机制
赵雪1,于沛涛2,徐芝君1,顾清1,裘力锋1,王弋1
(1.杭州市第一人民医院急诊科,杭州 310003;2.北京大学基础医学院,北京 100191)
目的 研究急诊重症监护病房(EICU)感染患者鲍曼不动杆菌16S rRNA甲基化酶armA、氨基苷核苷转移酶ANT(3′′)-Ia、氨基苷磷酸转移酶APH(3′)-I、氨基苷乙酰转移酶AAC(6′)-Ib检出率,分析耐氨基苷鲍曼不动杆菌的耐药机制。方法收集48株鲍曼不动杆菌均经VITEK系统鉴定,并采用琼脂对倍稀释法测定最低抑菌浓度(MIC),采用聚合酶链反应(PCR)法检测酶基因。结果48株鲍曼不动杆菌中氨基苷类抗菌药物高耐药菌株28株,低耐药菌株20株。高耐药菌16S rRNA armA和低耐药菌16S rRNA armA检出率分别为71.43%和0.00%(P<0.01),高耐药菌APH (3′)-I和低耐药菌APH(3′)-I检出率分别为60.71%和5.00%(P<0.01),高耐药菌ANT(3′′)-Ia和低耐药菌ANT(3′′)-Ia检出率分别为82.14%和5.00%(P<0.01),高耐药菌AAC(6′)-Ib和低耐药菌AAC(6′)-Ib检出率分别为53.57%和5.00%(P<0.01)。结论氨基苷钝化酶和16S rRNA甲基化酶在氨基苷高耐药鲍曼不动杆菌中检出率高,与氨基苷类抗菌药物高耐药密切相关。
鲍曼不动杆菌;氨基苷钝化酶基因;16S rRNA甲基化酶基因;重症监护病房,急诊
鲍曼不动杆菌近年来已成为常见医院感染致病菌,因其高检出率和高耐药率而受到高度关注。急诊重症监护病房(emergency intensive care unit,EICU)患者分离鲍曼不动杆菌亦逐年增多,鲍曼不动杆菌已成为EICU患者感染主要致病菌之一[1]。随着抗菌药物广泛应用,鲍曼不动杆菌对氨基苷类、喹诺酮类、β-内酰胺类抗菌药物均呈现药耐性,16S rRNA甲基化酶可使所有氨基苷类抗菌药物失活[2]。编码16S rRNA甲基化酶的基因主要包括rmtA、rm tB、rm tC、rm tD、rmtE、armA和npmA等7种,其中主要检出的是armA亚型[3-4]。氨基苷类抗菌药物耐药主要机制为细菌产生钝化酶[5],钝化酶主要包括氨基苷磷酸转移酶(aminoglycoside phosphotransferases,APH)、氨基苷核苷转移酶(aminoglycoside nucleotidyltransferases, ANT)、氨基苷乙酰转移酶(aminoglycoside acetyltransferases,AAC)三类,共三十余种[6]。为了分析EICU耐氨基苷鲍曼不动杆菌耐药相关基因状况,笔者检测了从EICU感染患者分离鲍曼不动杆菌具有代表性的基因:16S rRNA甲基化酶armA、氨基苷核苷转移酶ANT(3′′)-Ia、氨基苷磷酸转移酶APH(3′)-I、氨基苷乙酰转移酶AAC(6′)-Ib,报道如下。
1 资料与方法
1.1 一般资料 2010年1月~2012年12月,EICU感染患者分离鲍曼不动杆菌48株,感染部位主要是下呼吸道和尿道,分别占57.6%和22.4%。收集1周内或住院期间反复培养出现的同部位相同菌株,不重复统计。
1.2 培养和鉴定 按《全国临床检验操作规程》(2005年版)临床微生物常规鉴定程序操作培养,用法国生物梅里埃公司VITEK系统鉴定。
1.3 最低抑菌浓度(minimal inhibitory concentration, MIC)测定 采用琼脂对倍稀释法测定菌株对抗菌药物的MIC,药敏结果判断按2010年美国临床和实验室标准协会所制定的标准执行。
1.4 质控菌株 采用铜绿假单胞菌ATCC27853、大肠埃希菌ATCC25922、鲍曼不动杆菌ATC19606。
1.5 仪器与试剂 试剂盒均购自北京天根生化科技有限公司。聚合酶链反应(polymerase chain reaction, PCR)基因扩增仪(型号:TProfessional Trio,德国Whatman Biometra公司)
1.6 方法 以阿米卡星MIC≥256μg·m L-1为标准[7],高于该值为高耐药菌,低于该值为低耐药菌。氨基苷钝化酶基因PCR扩增[8],以GenBank中基因序列设计引物,引物序列见表1,退火温度均为55℃。上下游引物各1μL,模板液2μL,PCR预混液12.5μL,余量超轻水补足至25μL。经1%琼脂糖凝胶电泳检测产物,用凝胶成像系统观察结果。
1.7 统计学方法 应用SPSS19.0版软件进行Χ2检验,以P<0.05为差异有统计学意义。
2 结果
2.1 耐药菌 高耐药菌和低耐药菌分别为28和20株。
2.2 氨基苷钝化酶基因测序 经ABI自动DNA测序仪测序,与GenBank已登录的基因序列比对,同源性为99%,16S rRNA armA、APH(3′)-I、ANT(3′′)-Ia、 AAC(6′)-Ib基因序列登录号分别为EU140571、ACYR02000001、GU304661、EF690695。

表1 氨基苷钝化酶基因PCR扩增引物序列Tab.1 PCR amplification primers of aminoglycoside-modifying enzymes gene
2.3 16S rRNA甲基化酶和氨基苷钝化酶基因在高耐药菌和低耐药菌中检出率比较 高耐药菌ANT(3′′)-Ia检出率最高,其次是16S rRNA armA,低耐药菌未检测到16S rRNA armA,见表2。
2.4 16S rRNA甲基化酶和氨基苷钝化酶基因检出情况 高耐药菌16S rRNA armA、APH(3′)-I、ANT(3′′)-Ia、AAC(6′)-Ib 4种酶基因都检测到的菌株数最多,共9株,见表3。低耐药菌1株检出APH(3′)-I、ANT(3′′)-Ia、AAC(6′)-Ib 3种钝化酶基因,其他均未检出。
3 讨论
氨基苷类抗菌药物耐药鲍曼不动杆菌已经成为院内感染的主要病原菌之一,且多为高水平耐药,其主要耐药机制是产生钝化酶[3],以及新近发现的产生16S rRNA甲基化酶[2]等。不同的钝化酶有相对应的特异性作用底物,通常介导一种或几种相似结构的氨基苷类抗菌药物产生耐药性[9],16S rRNA甲基化酶由质粒介导。
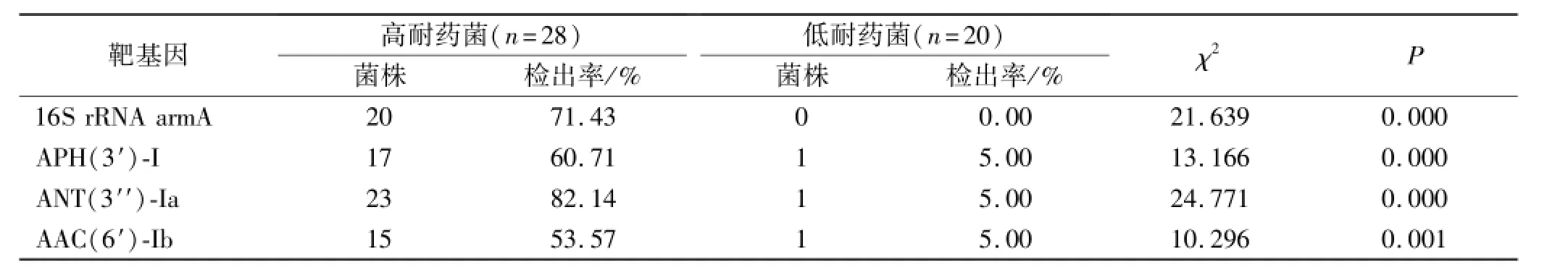
表2 16S rRNA甲基化酶和氨基苷钝化酶基因在高耐药菌和低耐药菌中检出率Tab.2 Detection rate of am inoglycoside-mod ifying enzym es gene and 16S rRNA methylase gene in high resistant and low resistant Acinetobacter baumannii
氨基苷类抗菌药物MIC为256μg·mL-1时具有阳性预测意义[7]。本研究以阿米卡星MIC≥256μg·mL-1的菌株为高耐药菌株,MIC<256μg·mL-1的菌株为低耐药菌株。分离的48株鲍曼不动杆菌中, 28株为高耐药菌株,20株为低耐药菌株。
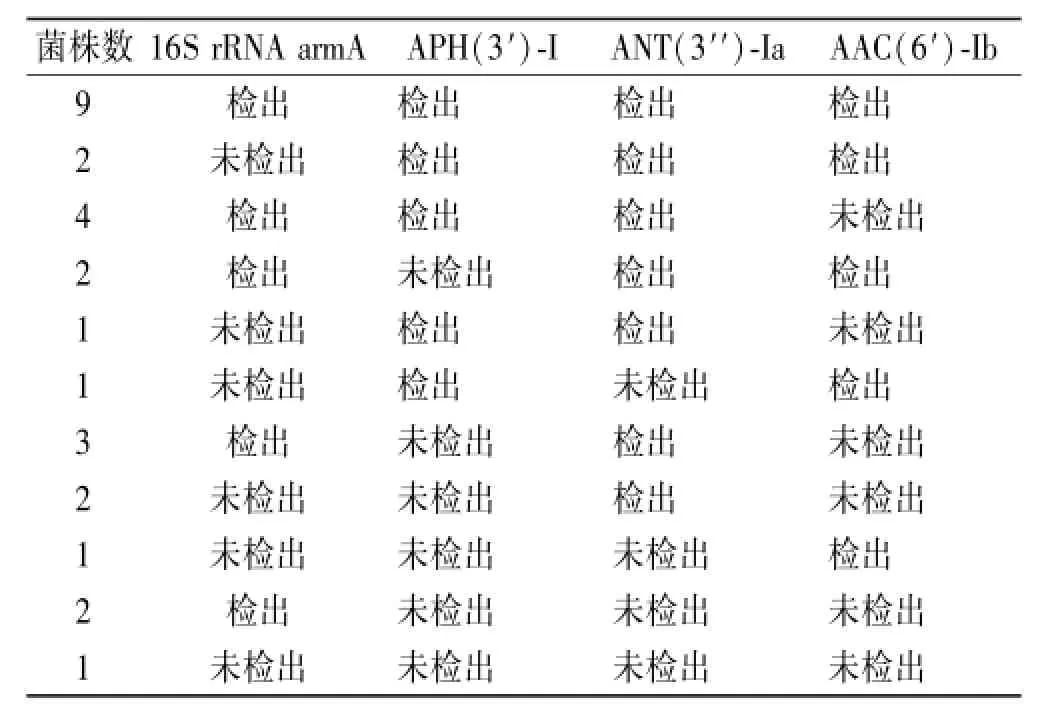
表3 高耐药菌中16S rRNA arm A、APH(3′)-I、ANT (3′′)-Ia、AAC(6′)-Ib 4种酶基因检出情况Tab.3 Detection on 16S rRNA arm A、APH(3′)-I、ANT (3′′)-Ia、AAC(6′)-Ib gene in highly resistan t Acinetobacter baumannii
本实验结果显示,EICU患者中分离的鲍曼不动杆菌高耐药株携带16S rRNA armA基因阳性率较高,氨基苷钝化酶基因核苷转移酶ANT(3′′)-Ia、磷酸转移酶APH(3′)-I、乙酰转移酶AAC(6′)-Ib阳性率依次降低。不同地区钝化酶差异较大,美国以ANT(2′′)最常见,日本以AAC(6′)常见,可能与不同地区抗菌药物应用差异较大有关。
同时检出3种钝化酶基因的菌株,对氨基苷类抗菌药物均呈现高度耐药,原因应为3种基因表达产物作用底物覆盖了所有氨基苷类抗菌药物;而仅检出一种或两种钝化酶基因的菌株,对不同氨基苷类抗菌药物中有些品种存在低耐药或敏感菌株,其机制应与只介导一种或几种相似结构的氨基苷类抗菌药物有关[9],如APH(3′)-I对卡那霉素、核糖霉素、新霉素等产生耐药性,ANT(3′′)-I对链霉素、大观霉素等产生耐药性,AAC(6′)-I对阿米卡星、奈替米星、妥布霉素等产生耐药性。16S rRNA甲基化酶的出现给原有氨基苷类抗菌药物针对拮抗钝化酶进行结构改造的研发思路带来很大的障碍。
本研究显示,氨基苷类抗菌药物高耐药鲍曼不动杆菌耐药与氨基苷钝化酶和16S rRNA甲基化酶基因表达有关,与DOI等[2]报道一致。1株未检出氨基苷钝化酶基因菌株呈现高耐药,1株检出3种钝化酶基因菌株呈现低耐药,说明鲍曼不动杆菌耐药机制复杂,存在其他耐药机制,如:核糖体结合位点改变、外排泵过度表达、外膜孔蛋白表达缺失等,具体耐药机制有待进一步研究。
总之,及时发现耐药菌株及流行趋势,可有效延缓细菌产生耐药性,控制耐药菌传播,为临床合理选用抗菌药物提供依据。
[1] 赵雪,于沛涛,王弋,等.急诊重症监护病房病原学调查及临床分析[J].北京医学,2013,35(3):178-181.
[2] DOIY,ADAMS JM,YAMANE K,et al.Identification of16S rRNA methylase producingAcinetobacter baumanniiclinical strains in North America[J].Antimicrob AgentsChem other, 2007,51(11):4209-4210.
[3] ZHOU Y,YU H,GUO Q,et al.Distribution of 16S rRNA methylases among different species of Gram-negative bacilli with high-level resistance to am inoglycosides[J].Eur JClin Microbio Infect Dis,2010,29(11):1349-1353.
[4] DAVISM A,BAKER K N,ORFE L H,et al.Discovery of A gene conferring multiple-aminoglycoside resistance inEscherichia coli[J].Antimicrob Agents Chemother,2010,54 (6):2666-2669.
[5] MINGEOT-LECLERCQ M P,GLUPCZYNSKIY,TULKENS PM,et al.Am inoglycosides:activity and resistance[J]. Antimicrob Agents Chemother,1999,43(4):727-737.
[6] NEMEC A,DOLZANI L,BRISSE S,et al.Diveristy of aminoglycoside-resistance genes and their association with class I integrons among strains of pan-EuropeanAcinetobacter baumanniiclones[J].JMed Microbiol,2004,53(Pt12): 1233-1240.
[7] LEE H,YONG D,YUM J H,et al.Dissemination of 16S rRNA methylase-mediated highly amikacin-resistant isolates ofKlebsiella pneumoniaeandAcinetobacter baumanniiin Korea[J].Diagn Microbiol Infect Dis,2006,56(3):305-312.
[8] NOPPE-LECLERCQ I,WALLET F,HAENTJENS S,et al. PCR detection of aminoglycoside resistance genes:a rapid molecular typing method forAcinetobacter baumannii[J]. Res Microbiol,1999,150(5):317-322.
[9] YOKOYAMA K,DOIY,YAMANE K,et al.Acquisition of 16S rRNA methylase gene inPseudononas aeruginosa[J]. Lancet,2003,362(9399):1888-1893.
DOI 10.3870/yydb.2014.05.008
Drug Resistance Mechanism of Patients Infected with Aminoglycoside-resistant Acinetobacter Baumannii in Emergency Intensive Care Unit
ZHAO Xue1,YU Pei-tao2,XU Zhi-jun1,GU Qing1,QIU Li-feng1,WANG Yi1
(1.Department of Emergency,the First Poeple's Hospital of Hangzhou,Hangzhou 310003,China;2.School of Basic Medicine,Peking University, Beijing 100191,China)
ObjectiveTo investigate drug resistance mechanism of am inoglycoside-resistant Acinetobacter baumannii by detecting 16S rRNA methylase gene and three common genes of aminoglycoside-modifying enzymes in Acinetobacter baumannii infected patients at EICU.MethodsThe 48 Acinetobacter baumannii strains were collected,and antimicrobial susceptibility tests were performed by VITEK automicroscan.The MIC was detected by 2-fold agar dilutionmethod,and genes were analyzed by polymerase chain reaction(PCR).ResultsAmong48 strains,28 werehighly resistant to aminoglycosides and 20 showed lower resistances.The 16S rRNA armA,APH(3')-I,ANT(3'')-Ia,AAC(6')-Ib geneswere detected in 71.43%,60.71%,82.14%, and 53.57%of the 28 highly resistant strains,but only present in 0.00%,5.00%,5.00%,and 5.00%of the low-resistant isolates(P<0.01).ConclusionThe aminoglycoside-modifying enzymes and 16S rRNA methylase were frequently found in Acinetobacter baumannii clinical isolates,which is closely related to the high-level resistance to aminoglycoside antibiotics.
Acinetobacter baumannii;Aminoglycoside-passivating enzymes genes;16S rRNA methylase gene; Intensive care unit,emergency
R978.12;R969.3
A
1004-0781(2014)05-0579-03
2013-07-05
2013-09-06
赵雪(1978-),男,浙江杭州人,主治医师,在读硕士,从事急诊医学研究。电话:(0)13750888506,E-mail:15130726@qq.com。
于沛涛(1969-),男,北京人,副教授,学士,从事基础医学研究。电话:(0)13701096687,E-mail:yupeitao@126.com。
