地下水中病原菌及其快速检测技术研究进展
2014-04-26郜志云朱岗辉
文 一,郜志云,白 辉,朱岗辉
环境保护部环境规划院,北京100012
地下水是可利用水资源的重要组成部分,全球三分之一的淡水资源来自于地下水,也是许多国家的主要饮水来源[1]。随着经济社会的发展,地下水环境面临越来越多的威胁,病原微生物成为影响地下水环境的重要污染物[2]。以地下水为媒介传播疾病的微生物主要包括细菌、病毒和寄生虫,能引发霍乱、伤寒、传染性肝炎、钩端螺旋体等疾病,会造成大规模传染病的暴发[3]。在发展中国家,约80%的疾病是由水生病原菌引起的,这些疾病造成每天约27 000人死亡[4];在美国,每年估计有590万人因暴露地下水病原菌而致病,造成9 400 人死亡[5]。
随着人口增长和土地利用扩展,病原菌污染源增加,增加了地下水污染的可能性,因此开展地下水中病原菌监测是保障饮水安全,保护人群健康的重要前提。目前中国地下水中病原菌的检测方法主要为传统微生物培养法[6],该方法存在操作繁琐、检测时间长、部分病原菌难以检测等问题,难以满足当前地下水环境管理对检测技术的需求。核酸扩增、生物芯片及生物传感器等检测技术是当前重要的水体病原菌检测手段。发展快速、精确的病原菌检测技术是世界各国研发的热点,该文对近年来国际上出现的地下水病原菌检测技术进行简要的介绍,以期为地下水中病原菌的有效监测和控制提供技术支持。
1 地下水中的病原菌
地下水中的病原菌微生物一般来自人与动物的排泄物,特别是粪便,其污染源有发生渗/泄漏的垃圾填埋场、畜禽养殖场、污水处理厂和接纳生活和畜禽污水的地表水体[7],通过土壤淋滤和地表水-地下水水力交换进入到地下水中。
地下水常见的污染致病菌主要为肠道菌如大肠杆菌(Escherichia Coli)、沙门氏菌(Salmonella)、空肠弯曲杆菌(Campylobacter)及水生细菌如嗜肺军团菌(Legionella pneumophila)等多种细菌,详见表 1[8-13]。
大多数通过人或动物的粪便进入水体的水源性病原菌不能在水中生长,但某些病原菌,如军团菌、非典型分支杆菌、类鼻疽伯克氏菌是环境微生物,可在水体或土壤中生长,增加人群暴露风险。除摄入途径外,有些病原菌还可通过吸入和皮肤接触暴露于人群,如军团菌和非典型分支杆菌可通过吸入途径引起呼吸道感染,类鼻疽伯克氏菌则可通过接触传播引起接触部位的感染。大部分病原菌可引起腹泻、呕吐、发热、头痛等轻微症状,少部分强致病菌在较低感染浓度下可引起严重的系统性疾病甚至死亡。

表1 地下水中常见病原菌
2 病原菌迁移转化
病原菌在地下水中的迁移转化受到表层土壤和包气带厚度、地质构造、水文地质状况和污染源类型的影响,见表2[14]。土壤和包气带通过过滤和吸附作用阻止病原菌向地下水中迁移。土壤粒径分布是影响过滤效果的重要因素,粒径越小,效果越好,因为细颗粒物的死端孔隙能有效过滤病原菌;吸附作用受到包气带疏水性、土壤和沉积物湿度、pH、离子强度的影响。相比于含水层,表层土壤和包气带具有更强的使病原菌衰减的能力,Schijven研究结果表明,在沙丘中传染性病毒用25 d从污染源迁移了30 m,病毒数量减少了8个数量级,在该场地的含水层中,要降低到相同的数量需要迁移 40 d[15]。
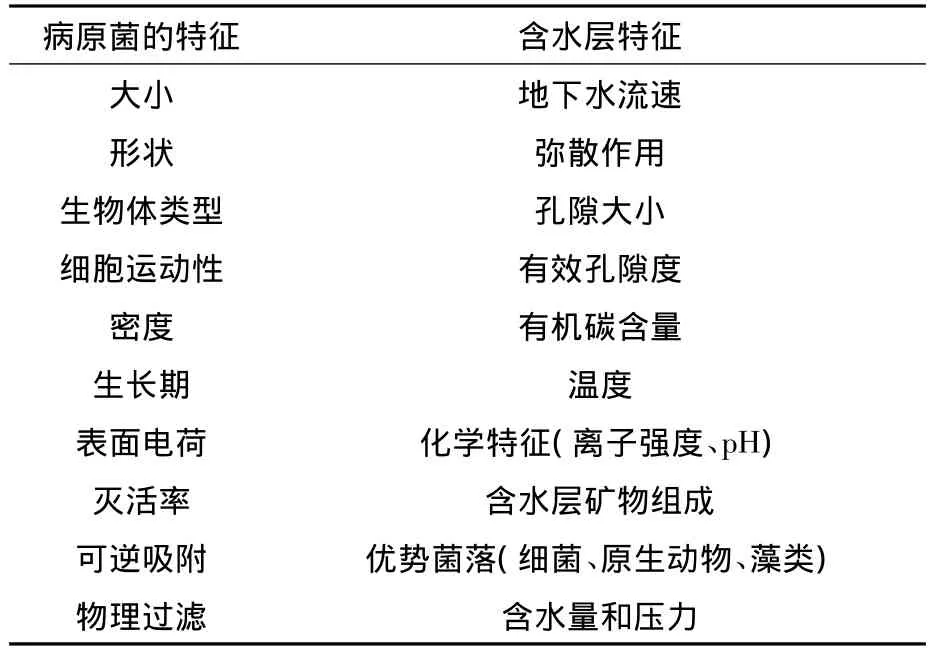
表2 地下水中影响病原菌迁移的因素
病原菌进入含水层后,含水层孔隙度和水流速度是影响病原菌衰减和迁移的重要因素。孔隙、岩溶和裂隙地下水具有不同的流速,对病原菌的衰减程度各不相同[16],在孔隙地下水中,地下水流速为每天十几米,病原菌不容易迁移,易发生衰减;在岩溶水中,由于较大的水流速度,病原菌可以每天迁移数公里,因此裸露的岩溶含水层易受病原菌的污染。大量结果表明,水源性疾病容易在非孔隙型地下水中爆发,特别是在基岩裂隙或者岩溶地下水中发生[17]。因此,应重视岩溶和裂隙地下水等环境敏感地区的地下水病原菌污染的监测工作。
3 地下水中病原菌检测现状
目前大多数国家和地区仍采用传统培养方法对环境中病原菌进行检测。由于传统培养方法获得检测结果的成本和时限问题,对于地下水中特殊病原菌的检测,目前一般仅限于确认是否污染或确证某种水处理工艺的有效性,而对于微生物的监测通常仅限于指示性微生物,如用总大肠菌群监测水处理效果,用埃希氏大肠杆菌指示水体是否被粪便污染。然而多数病原菌的存在条件与指示微生物的存在条件不是完全相关的,用传统的培养方法很难直接反映其污染情况,因此仅检测一种或少数几种病原菌的浓度并不能反映病原菌的污染情况,如流行病学数据表明总大肠菌数,粪大肠菌数等指示微生物与霍乱弧菌、志贺氏菌等引起的病理学发病率的相关性较差,这些情况都说明传统的检测方法已经过时,很难起到监测水体环境质量的作用[18]。
4 影响地下水中病原菌检测的因素
对地下水中病原菌检测影响因素的了解,是研发有针对性检测技术的基础。影响地下水中病原菌的检测因素主要包括以下3个方面:1)水体体积较大,水体中微生物浓度相对较低,很难直接从大量水中分离或检测到少量的特定病原菌[19];2)水体中营养物质匮乏,病原菌污染水体并经长时间的适应后易形成存活但不可培养的状态[20];3)随着人类活动的增加,病原菌污染水体的机会也相应增加,这也相应提高了对水体中病原菌监测的种类、场所及频次等要求。因此,快速、高通量、准确特异性是水体病原菌检测技术研究中需考虑的重要因素。
5 不同病原菌检测方法在地下水中的应用现状
病原菌检测技术主要分为以下几大类:传统平板培养法、免疫学方法、聚合酶链反应技术(PCR)、生物芯片法(biochip)、生物传感器(biosensor)等方法,如表3所示。平板培养法检测所需周期长,很多病原菌检测方法还未建立,更重要的是许多微生物不能在固体培养基上形成菌落,环境中物理或化学等因素也易影响病原菌的活性而形成不可培养状态,影响对水体病原微生物及时和有效的监测[21-22]。免疫学方法检测病原菌的假阳性或假阴性率较高,不能反映水体中病原菌实际污染情况。聚合酶链反应技术、生物芯片法、生物传感器等生物和生物物理方法,因为其快速灵敏、准确度高等特点被广泛关注。下面对上述3类新技术进行详细介绍。

表3 地下水中病原菌主要检测技术
5.1 聚合酶链式扩增反应技术在地下水中的应用现状
聚合酶链反应技术是针对目的基因设计特异寡核苷酸引物,并在反应体系中加入DNA聚合酶和4种dNTP,以目的基因为模板进行多个循环的DNA体外扩增的技术,在短时间内可通过扩增获得大量目的基因。PCR及其衍生的方法,如实时荧光定量PCR技术,已用于检测或鉴别不同类型水体中的一种或多种病原菌[23-24],但单独使用上述方法不能区分活菌和死菌,易造成假阳性,研究发现在荧光定量PCR或基因芯片等核酸扩增技术中利用叠氮溴化丙锭(propidium monoazide),可选择性结合修饰死菌细胞的DNA,从而间接计算出水体样本中活菌数量,提高PCR技术的检测准确度[25-26]。另外由于病原菌在水体中的浓度较低,可通过较长时间的前增菌或浓缩处理,增加目标活菌的浓度,稀释干扰杂质,复苏受损细菌[27-29],提高检测的准确性。为了避免长时间的增菌或浓缩步骤,He等[30]采用免疫磁珠(IMS)直接从水样中特异性捕获产志贺毒素大肠杆菌(STEC)。微生物的rRNA(pre-rRNA)是一种反映系统发育特性和指示生理状态的总rRNA的重要成分,Cangelosi等[31]通过反转录实时荧光定量PCR(RT-qPCR)分析pre-rRNA的变化情况达到检测活病原菌的目的。环介导等温扩增法(Lamp)是一种比PCR技术更灵敏,速度更快的核酸扩增技术,依赖于能够识别靶DNA上6个特定区域的4条引物和一种具有链置换活性的DNA聚合酶,在恒温条件下高效扩增核酸,1 h即可完成。Yamazaki等[32]用 Lamp检测产霍乱毒素的霍乱弧菌,大大缩短了检测时间,灵敏度提高10倍。为了改善Lamp的应用性,Chang等[33]将病原菌DNA磁珠提取组件及微流控LAMP反应芯片和光学检测系统整合成一台可快速、灵敏、方便的装置,用于水产养殖中水体中病原菌的检测。
5.2 生物芯片技术在地下水中的应用现状
生物芯片技术是采用原位合成或微矩阵点样等方法将大量的生物分子有序地固定在硅胶片等固化材料上,然后与已标记的待测样品进行作用,通过特定的仪器分析相应的作用情况而达到分析检测的目的。根据生物芯片所选用固化材料的不同,可将生物芯片分为基因芯片、蛋白芯片、细胞芯片及组织芯片等,各种生物芯片检测技术均具有检测通量和自动化程度高的特点,目前对水中病原菌检测应用较多的主要是基因芯片和微流控芯片。Zhou[34]等针对细菌的16S-23S rRNA 内部转录间隔区和促旋酶B基因序列设计寡核苷酸基因芯片,可同时检测水体中包括肺炎军团菌、沙门氏菌、致贺氏菌、肠炎耶尔森菌及霍乱弧菌等28种致病菌,重复性和特异性较好,在饮用水中添加阳性细菌后检测灵敏度最低可至0.1 ng DNA或104CFU/mL。微流控芯片(Microfluidic lab-on-a-chip)具有比表面积大、样品需要量小及检测通量高等多种优点,Agrawal等[35]将其同免疫磁纳米粒子及碲化镉量子点标记结合可快速灵敏地同时检测水中低浓度的大肠杆菌和沙门氏菌。Cichova等[36]在研究水中病原菌的检测过程中,应用微电化学技术开发了一种微流控生物芯片,可在线裂解病原菌并提取核酸,从而有利于后续的快速检测。Dharmasiri等[37]用O157多克隆抗体共价耦联修饰微流控芯片后,可快速富集水体中较低浓度(<100细胞/毫升)的E.coli O157:H7,然后将富集的菌体使用实时荧光定量PCR针对slt1基因扩增检测,结果表明,该方法的回收率可达到72%,检出限最低达到6 CFU/L。Liu等[38]用低蛋白结合膜获得菌体,然后直接提取RNA并进行RT-PCR后,利用芯片检测E.coli O157:H7的rfbE和fliC基因,该方法最低可检测到自来水中的3~4 CFU/L的E.coli O157:H7,河水中最低可检测7 CFU/L的E.coli O157:H7。
5.3 生物传感器检测技术在地下水中的应用现状
生物传感器起源于20世纪60年代,是将生物识别元件和信号转换元件紧密结合,从而检测目标物的分析装置。不同的生物识别元件和信号转换元件组成了不同的生物传感器,分类方式有多种,可根据生物识别元件或信号转换元件分。转化元件,目前的生物传感器主要包括电化学生物传感器、电流生物传感器、电位生物传感器、阻抗生物传感器、光学生物传感器和压电生物传感器等,目前在致病菌检测方面研究较多的是电化学生物传感器、光学生物传感器和压电生物传感器[39]。Simpson 等[40]直接利用光纤可视生物传感器在2 h内快速检测水体中的大肠杆菌,不但降低了基质中的PCR反应抑制剂,还减少了对样品进行前增菌处理过程,提高了检测速度。Chowdhury等[41]用基于共价连接在电极上多聚苯胺膜的抗原抗体反应制成的阻抗生物传感器检测水中的E.coliO157:H7。目前,用于水中致病菌检测的生物传感器种类很多,也有不少商品化的仪器问世,但其检测限还偏高,在实际检测中易出现假阳性、假阴性的结果。未来生物传感器的发展趋势和重点方向是微型化、多功能化、智能化和集成化,开发新一代低成本、高灵敏度、高稳定性和高寿命的生物传感器是目前研究的热点。
6 讨论
目前,中国对于水体中病原菌等微生物的检测种类相对于水体中日益增多的病原菌污染仍然比较少,主要限于菌落总数、大肠菌群和军团菌等。另外,检测手段也比较落后,主要包括微生物的分离纯化,生化鉴定、血清学鉴定、酶学鉴定等常规方法或普通PCR鉴定。随着分子生物学、物理、电子、材料等多种学科的发展,病原菌的检测手段也日益丰富,荧光定量PCR、生物芯片或生物传感器等技术手段得到广泛的研究和发展,水体中病原菌检测的准确性、检测速度、特异性和灵敏度都得到了极大的改善和提高。对于水中病原菌的检测,除了以上几种主要技术外还有几种技术值得研究。如Fan等[42]应用表面增强拉曼光谱结合纳米技术检测水中的多种致病菌,研究表明该技术有望进行快速灵敏的检测。Hunter等[43]应用死端超滤浓缩水中E.coli O157:H7后,可有效进行IMS/ATP(免疫磁珠捕获-ATP)的活菌检测。Keserue等[44]利用流式细胞仪结合膜浓缩技术、免疫磁珠分离技术研究对嗜肺军团菌的快速检测,灵敏度高,而且可识别细菌的不同状态。然而上述这些技术手段在中国还没得到有效应用,主要原因是这些检测技术的研究主要集中在对分离纯化好的病原菌的检测阶段,病原菌的前增菌和分离纯化仍然是这些检测技术难以突破的技术瓶颈,另外,这些技术的成本昂贵也是难以在实际监测中得到广泛应用的因素。
7 展望
各种致病菌检测技术各有所长,也有各自的缺陷。综上所述,目前病原菌检测技术的发展趋势是将PCR、生物芯片等检测技术同微流控分离、免疫磁珠分离、纳米材料标记等结合,利用不同技术的优势,达到快速、灵敏、经济、便携甚至在线监测的目的。因此,未来对地下水中病原菌的检测技术还要更多考虑多种技术平台或手段的整合,同时兼顾成本和实用性,以期为保障人类健康和饮用水安全提供更强有力的支撑。
[1]Danielopol D L,Griebler C,Gunatilak A,et al.Present state and future prospects for groundwater ecosystems[J].Environmental Conservation,2003,30:1-27.
[2]Bruce A,Macler J C,Merkle.Current knowledge on groundwater microbial pathogens and their control[J].Hydrogeology,2000,8(1):29-40.
[3]Craun G F,Brunkard J M,Yoder J S,et al.Causes of outbreaks associated with drinking water in the United States from 1971 to 2006[J].Clinical Microbiological Reviews,2010,23(3):507-528.
[4]Andrew S,Ferguson A,Alice C,et al.Comparison of fecal indicators with pathogenic bacteria and rotavirus in groundwater[J].Science of the Total Environment.2013,431:314-322.
[5]Paul D H,Thomas K M,Katarina D M,et al.Contamination of groundwater systems in the US and Canada by enteric pathogens,1990-2013:a review and pooled-analysis[J].Plos One,2014,9(5):1-12.
[6]赵仲麟,李淑英,李燕.水体病原微生物分子检测技术研究进展[J].生物技术通报,2012,1:41-44.
[7] Kirschner A K,Kavka G G,Velimirov B,et al.Microbiological water quality along the Danube River:integrating data from two whole-river surveys and a transnational monitoring network[J].Water Research,2009,43:673-3 684.
[8]Palmer S,Gully P,White J,et al.Water-borne outbreak of Campylobacter gastroenteritis[J].The Lancet,1983,321(8 319):287-290.
[9]Stout J, Yu V M. BestEcology ofLegionella pneumophila within water distribution systems[J].Applied and environmental microbiology,1985,49(1):221-228.
[10]Baird-Parker A.Foodborne salmonellosis[J].The Lancet,1990,336(8 725):1 231-1 235.
[11]Swerdlow D L,Woodruff B A,Brady R C,et al.A waterborne outbreak in Missouri of Escherichia coli O157:H7 associated with bloody diarrhea and death[J].Annals of Internal Medicine,1992,117(10):812-819.
[12] Bottone E J.Yersinia enterocolitica:overview and epidemiologic correlates[J].Microbes and Infection,1999,1(4):323-333.
[13]Edberg S,Rice E,Karlin R,et al.Escherichia coli:the best biological drinking water indicator for public health protection[J].Symposium Series(Society for Applied Microbiology),2000,(29):106S-116S.
[14]Chilton J.Assessment of aquifer pollution vulnerability and susceptibility to the impactsofabstraction.Protecting Groundwater for Health -Managing the Quality of Drinking Water Sources[R].World Health Organization,2006:199-241.
[15]Schijven J F,Bruin H A,Hassanizadeh S M,et al.Bacteriophages and Clostridium spores as indicator organisms for removal of pathogens by passage through saturated dune sand[J].Water Research,2000,37:2 186-2 194.
[16]Pang L.Microbial removal rates in subsurface media estimated from published studies of field experiments and large intact soil cores[J].J Environmental Quality,2009,38:1 531-1 559.
[17]Fong T T,Mansfield L S,Wilson D L,et al.(2007):Massive MicrobiologicalGroundwaterContamination Associated with a Waterborne Outbreak in Lake Erie,South Bass Island,Ohio[J].Environmental Health Perspective,2007,115(6):856-864.
[18]Lund V.Evaluation of E.coli as an indicator for the presence of Campylobacter jejuni and Yersinia enterocolitica in chlorinated and untreated oligotrophic lake water[J].Water Research,1996,30(6):1 528-1 534.
[19] Fleisher J M.Conducting recreational water quality surveys:someproblemsand suggested remedies.Marine pollution bulletin[J],1990,21(12):562-567.
[20]Cenciarini-Borde C,Courtois S,La S B.Nucleic acids asviability markers forbacteria detection using molecular tools[J].Future Microbiology,2009,4(1):45-64.
[21]Kong R,Lee S,Law T,et al.Rapid detection of six types of bacterial pathogens in marine waters by multiplex PCR.Water Research[J],2002,36(11):2 802-2 812.
[22]Tissie A,Denis M,Hartemann P,et al.Development of a Rapid and Sensitive Method Combining a Cellulose Ester Microfilter and a Real--Time Quantitative PCR Assay To Detect Campylobacter jejuni and Campylobacter coli in 20 Liters of Drinking Water or Low-Turbidity Waters[J].Applied and environmental microbiology,2012,78(3):839-845.
[23]Zhou G,Wen S,Liu Y,et al.Development of a DNA microarray for detection and identification of Legionella pneumophila and ten other pathogens in drinking water[J].International journal of food microbiology,2011,145(1):293-300.
[24] Dharmasiri U,Witek M A,Adams A A,et al.Enrichment and detection of Escherichia coli O157:H7 from watersamples using an antibody modified microfluidic chip[J].Analytical chemistry,2010,82(7):2 844-2 849.
[25]Liu Y,Gilchrist A,Zhang J,et al.Detection of viable but nonculturable Escherichia coli O157:H7 bacteria in drinking water and river water[J].Applied and environmental microbiology,2008,74(5):1 502-1 507.
[26]Fan C,Hu Z,Mustapha A,et al.Rapid detection of food-and waterborne bacteria using surface-enhanced Raman spectroscopy coupled with silver nanosubstrates[J].Applied microbiology and biotechnology,2011,92(5):1-9.
[27]Hunter D M,Leskinen S D,Maga˜na S,et al.Deadend Ultrafiltration Concentration and IMS/ATP-bioluminescence detection of Escherichia coli O157:H7 in recreational water and produce wash[J].Journal of microbiological methods,2011,87(3):338-342.
[28] Keserue H A,Baumgartner A,Felleisen R,et al.Rapid detection of total and viable Legionella pneumophila in tap water by immunomagnetic separation,double fluorescentstaining and flow cytometry[J].Microbial Biotechnology,2012,5(6):753-763.
[30]Roszak D,Colwell R.Survival strategies of bacteria in the natural environment[J].Microbiological Reviews,1987,51(3):365.
[31]Fukushima H,Tsunomori Y,Seki R.Duplex real-time SYBR green PCR assays for detection of 17 species of food-or waterborne pathogens in stools[J].Journal of Clinical Microbiology,2003,41(11):5 134-5 146.
[32] Bae S,Wuertz S.Discrimination of viable and dead fecal Bacteroidales bacteria by quantitative PCR with propidium monoazide[J].Applied and environmental microbiology,2009,75(9):2 940-2 944.
[33] Nocker A,Mazza A,Masson L,et al.Selective detection of live bacteria combining propidium monoazide sample treatment with microarray technology[J]. Journalofmicrobiologicalmethods,2009,76(3):253-261.
[34]Bonetta S,Borelli E,Conio O,et al.Development of a PCR protocol for the detection of Escherichia coli O157:H7 and Salmonella spp.in surface water[J].Environmentalmonitoring and assessment,2011,177(1):493-503.
[35]Leskinen S D,Kearns E A,Jones W L, et al.Automated dead-end ultrafiltration of large volume water samples to enable detection of low-level targets and reduce sample variability[J].Journal of applied microbiology,2012,113(2):351-360.
[36]He X,W Qi,Qui˜nones B,et al.Sensitive detection of Shiga Toxin 2 and some of its variants in environmental samples by a novel immuno-PCR assay[J].Applied and environmental microbiology,2011,77(11):3 558-3 564.
[37]Cangelosi G A,Weigel K M,Lefthand-Begay C,et al.Molecular detection of viable bacterial pathogens in water by ratiometric pre-rRNA analysis[J].Applied and environmental microbiology,2010,76(3):960-962.
[38]Yamazaki W,Seto K,Taguchi M,et al.Sensitive and rapid detection ofcholera toxin-producing Vibrio cholerae using a loop-mediated isothermal amplification[J].BMC microbiology,2008,8(1):94-102.
[39]Chang W H,Yang S Y,Wang C H,et al.Rapid isolation and detection of aquaculture pathogens in an integrated microfluidic system using loop-mediated isothermal amplification[J].Sensors and Actuators B:Chemical,2011,45(4):146-160.
[40]Agrawal S,Morarka A,Bodas D,et al.Multiplexed Detection of Waterborne Pathogens in Circular Microfluidics[J]. Applied biochemistry and biotechnology,2012,167(6):1-10.
[41]Cíchová M,ProkˇsováM,Tóthová L,et al.On-line cell lysis of bacteria and its spores using a microfluidic biochip[J].Central European Journal of Biology,2012,7(2):230-240.
[42]Palchetti I,Mascini M.Electroanalytical biosensors and their potential for food pathogen and toxin detection[J].Anal Bioanal Chem,2008,391(2):455-471.
[43]Simpson J M,Lim D V.Rapid PCR confirmation of E.coliO157:H7 after evanescent wave fiber optic biosensor detection[J].Biosens Bioelectron,2005,21(6):881-887.
[44]Chowdhury A D,De A,Chaudhuri C R,et al.Label free polyaniline based impedimetric biosensorfor detection of E.coliO157:H7 Bacteria[J].Sensors and Actuators B:Chemical,2012,171:916-923.
