16株中国东北地区临床孢子丝菌菌种分子生物分类学研究
2014-04-02黄梦雅刘婷婷杨致邦
黄梦雅,刘婷婷,杨致邦,周 汛
孢子丝菌是人兽共患性真菌[1],可引起人和哺乳动物的皮肤、皮下组织及周围淋巴管亚急性或慢性感染,偶可播散至全身如骨、脑,引起系统性损害,甚至死亡[2]。该菌分布于全球各地,以热带和亚热带地区为主[3],病人大多为接种感染[4],多见于园林、农业工作者,也可感染某些动物如猫、狗[1]和海豚[5]等。该菌是双相型真菌,在25℃为菌丝相,动物体内和37℃为酵母相。 它是美国人Shenk首次从孢子丝菌病人患病组织中分离出来,故命名为申克孢子丝菌[6]。长久以来我国各地区孢子丝菌病的病原菌也一直被认为是申克孢子丝菌[7]。近期有研究认为,孢子丝菌病病原菌并非申克孢子丝菌一种,而是由申克孢子丝菌、球形孢子丝菌、巴西孢子丝菌(Sporothrixbrasiliensis,S.brasiliensis)、墨西哥孢子丝菌(Sporothrixmexicana,S.mexicana)、白孢子丝菌(Sporothrixpallida,S.pallida)、申克孢子丝菌卢艾里变种(Sporothrixschenckiivarluriei,S.luriei)6个种构成[8-9,22]。不同菌种致病性、药物敏感性不同[10-11],因此,准确鉴定菌种有利于临床治疗与预后判断。
目前,国内传统的针对孢子丝菌病病原菌鉴定多通过皮损处组织块或浓汁培养,在25 ℃和37 ℃培养观察菌落形态以及血清学、免疫荧光染色辅助诊断[7,12],但这些方法特异性不高,不能准确鉴别6个菌种[13],且耗时长,需要2~3周时间。已报道钙调蛋白(Calmodulin,CAL)基因序列可以鉴定孢子丝菌6个菌种[9,14-15],但核糖体内转录间隔区(ITS)序列及β微管蛋白序列PCR扩增率更高,可用于快速、准确鉴定孢子丝菌菌种[16-17]。本文对16株既往根据组织病理鉴定为申克孢子丝菌的临床孢子丝菌株的孢子丝菌进行ITS及β微管蛋白序列PCR分子生物学鉴定,现报道如下。
1 材料与方法
1.1 材料
1.1.1菌株 16株孢子丝菌来自于吉林大学第二医院皮肤科孢子丝菌病患者组织,该菌曾用显微镜病理检查、PSA染色等方法鉴定为申克孢子丝菌[12]。本次实验将其分别编号为J29、J30、J31、J33、J34、J36、J37、J38、J39、J41、J42、J43、J44、J45、J46、J47。
1.1.2培养基 土豆葡萄糖琼脂培养基(Potato Dextrse Agar Medium,PDA), CTAB法提取基因组DNA相关试剂[18],PCR扩增试剂盒(TaKaRa,American)。
1.1.3引物(Beijing Genomics Institute ,BGI提供)
ITS4:5′-TCCTCCGCTTATTGATATGC-3′
ITS5:5′-GGAAGTAAAAGTCGTAACAAG
G-3′
β-tubulin 2a:5′-GGTAACCAAATCGGTG
CTGCTTTC-3′
β-tubulin 2b:5′-ACCCTCAGTGTAGTGA
CCCTTGGC-3′
1.2 方法
1.2.1真菌菌丝相细胞基因组DNA提取 将16株孢子丝菌菌株接种于PDA培养基,25 ℃下温箱孵育10 d,取孢子丝菌菌丝相菌体500 mg,按CTAB法提取真菌基因组DNA[18],1.2%琼脂糖凝胶电泳分析测定其完整性后,产物储存于-20 ℃冰箱中。
1.2.2PCR扩增孢子丝菌ITS及β-tubulin基因 分别使用ITS1/ITS4[28]和β-tubulin 2a/β-tubulin 2b[29],PCR采用50 μL体系,内含1 μL DNA模板,各2 μL 上下游引物(15 μm),10×PCR缓冲液5.0 μL,dNTP(各10 mm)4.0 μL,Taq聚合酶(5U/ μL)0.3 μL,双蒸水35.7 μL。扩增条件:预变性95℃ 3 min,1个循环:变性95 ℃ 30 s,退火52~54 ℃ 30 s,延伸72℃ 1 min,35个循环,最终72 ℃延伸10 min。
1.2.3基因组测序 分别将所获得的16株菌的ITS、β-tubulin扩增产物于1.2%琼脂糖凝胶电泳检测后送往北京六合华大基因科技股份有限公司正反向测序,得到PCR扩增片段原始序列。
1.2.4序列对比及系统进化分析 分别将所获得的ITS、β-tubulin正反向序列通过ContigExpress软件拼接得到共识序列,将此序列输入NCBI网站(http://blast.Ncbi.nlm.nih.gov/Blast)进行对比,并将序列存入基因库,合并ITS、β-tubulin序列,与已经发表在基因库内的其他地区的参考序列ITS、β-tubulin一起使用Clustalx 1.81进行对比[19],将对比后获得的序列利用MEGA4软件中的邻接(Neighbor-Joining,NJ)进行系统分析,系统树各分支的置信度以1000次自引导法(Bootstrap)重复检测[20]。
2 结 果
2.1扩增产物电泳 所提取的16株孢子丝菌基因组DNA分别经ITS、β-tubulin的PCR扩增,所得原液及相应引物经1.2%琼脂糖凝胶电泳检测如下(见图1、图2)。经ITS、β-tubulin扩增所得基因片段分别近似长600 bp和400 bp,同孢子丝菌相应基因片段长度相符。
2.2系统发育学分析 凝胶电泳初步检测后的16株孢子丝菌基因片段经正反向测序,获得的基因原始序列分析通过系统发育学分析得到以下系统进化树(图3)。
图1ITS扩增产物电泳图谱
Fig.1PCRproductionofITS
1: Marker 2000 bp; J29, J30, J31, J33, J34, J36, J37, J38, J39, J41, J42, J43, J44, J45, J46, J47: ITS genes.

图2β-tubulin扩增产物电泳图谱
Fig.2PCRproductionofβ-tubulin
1: Marker 2 000 bp; J29, J30, J31, J33, J34, J36, J37, J38, J39, J41, J42, J43, J44, J45, J46, J47: β-tubulin genes.
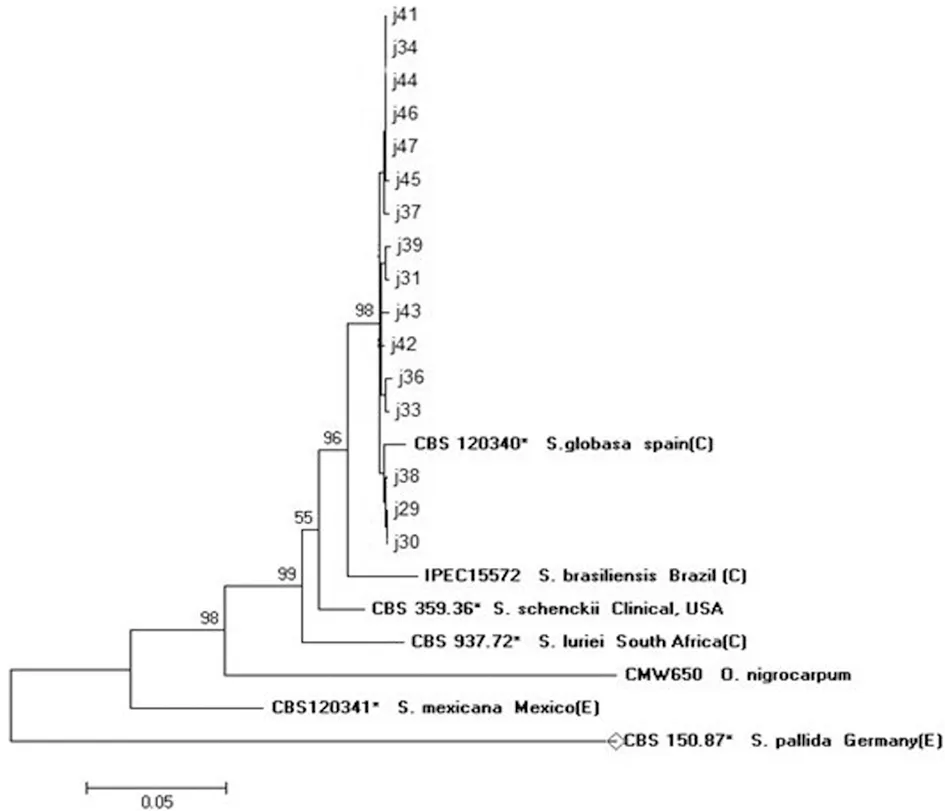
图316株孢子丝菌的系统进化树图谱
Fig.3Phylogenetictreeof16Sporothrixisolates
Bootstrap support values >50% are shown above the branches, and the scale bar indicates 0.05 changes. The tree is rooted withOphiostomanigrocarpum. References sequences are shown in bold. “C” and “E” indicate clinical strains and environmental strains, respectively. An asterisk (*) indicates the type strains.
此次鉴定的16株菌均与球形孢子丝菌(CBS 120340*)在同一分支上,其支持率为98%,这些进化枝与申克孢子丝菌(CBS 359.36*)有明显的距离,故鉴定为球形孢子丝菌。
3 讨 论
孢子丝菌菌株通过形态学鉴定提示不同的遗传特性和系统发育分组[21]。分子生物学系统发育的分析显示,孢子丝菌属下并非只有一个的种,它由6个物种构成。不同的物种分布不同,巴西孢子丝菌的感染和分布在巴西,申克孢子丝菌主要流行在美洲、非洲、亚洲[9]。球形孢子丝菌在亚洲、欧洲、美洲等国家均有分布[23]。目前体外培养表明不同菌种的菌株在环境和人类宿主相对频率的差异,导致了不同的致病性[24]。有小鼠模型的研究表明,不同孢子丝菌基因型的毒力、致病性不同导致孢子丝菌临床类型的不同[25]。在儿童中,多为固定型[26],成人中皮肤淋巴管型最为多见,其次为固定型,皮肤外型、播散型相对少见[27]。不同的临床类型提示不同的治疗、预后。巴西孢子丝菌和申克孢子丝菌为最致命的物种[11]。孢子丝菌的其中5个种在体外对12个抗真菌剂进行了敏感测试,不同种之间存在显著差异,巴西孢子丝菌对抗真菌药物最敏感,最不敏感的是墨西哥孢子丝菌,在一般情况下,特比萘芬是最有效的药物[10]。 因此,临床分离株的物种鉴定对孢子丝菌致病力、临床表现、药物敏感疗效评估及预后有明显意义。
现已经有5株中国环境株[9]、33株中国北方孢子丝菌病临床分离株[14]、74株中国东北孢子丝菌病临床分离株[15]通过CAL序列,6株中国孢子丝菌病临床分离株、64株中国孢子丝菌临床分离株[30]通过ITS序列和β-tubulin序列[17]鉴定为球形孢子丝菌。本次研究的16株菌既往依据病人临床表现、显微镜病理检查、PSA染色后鉴定为申克孢子丝菌,但该方法不能准确区分6个菌种。在分子生物学的基础上, ITS序列及β-tubulin序列比CAL序列的PCR扩增率更高,可快速、准确鉴定孢子丝菌菌种[16-17]。因此,我们将这16株临床分离菌株在ITS序列和β-tubulin序列处扩增,通过系统发育树分析证实这16株菌均为球形孢子丝菌,而非申克孢子丝菌。故此次收集的孢子丝菌病临床分离株虽然既往鉴定为申克孢子丝菌,但在分子生物学的角度更为准确地分析为球形孢子丝菌。 本次研究结果表明,6个孢子丝菌菌种通过体外培养后的肉眼观察和显微镜检并不能得到良好的区分。仅仅通过组织形态学、病理染色等方法鉴定孢子丝菌致病菌种存在一定的局限性。CAL基因序列可以鉴定孢子丝菌6个菌种,但有报道其PCR扩增率较其他常用序列如ITS序列及β-tubulin序列低,核糖体内转录间隔区序列虽然在鉴定某些真菌菌种时需要配合其他标记物,但其被广泛应用于真菌菌种鉴定,并建议作为鉴定的一般方法[16]。故通过应用ITS序列及β-tubulin序列对孢子丝菌DNA进行PCR高效扩增,测序后利用系统发育树分析可以得到有关物种鉴定的明确结果。
综上所述,本次研究的东北孢子丝菌鉴定为球形孢子丝菌,目前所有的研究表明,在分子生物学角度鉴定的中国东北甚至已研究的中国地区孢子丝菌病致病菌目前只找到球形孢子丝菌,故孢子丝菌病致病菌在中国东北地区乃至中国可能为球形孢子丝菌或并非单一申克孢子丝菌,但此结论还需要更多多中心大量样本的研究证明。本次研究表明,传统的形态学对于精确鉴定孢子丝菌菌种尚存在一定缺陷,利用提取孢子丝菌基因组DNA进行测序及构建系统发育树可以进一步完善鉴定方法,为下一步直接提取患者孢子丝菌DNA,PCR扩增鉴定菌种奠定了基础。为了提高临床效率,缩短诊断时间,我们下一步将研究直接从孢子丝菌病患者皮损组织直接提取致病菌DNA、利用ITS序列和β-tubulin分子生物学诊断技术去快速有效地鉴定物种,从而指导孢子丝菌病的临床治疗方案及预后判断。
参考文献:
[1]Dos Santos IB, Schubach TM, Leme LR, et al. Sporotrichosis: the main differential diagnosis withtegumentary leishmaniosis in dogs from Rio de Janeiro, Brazil[J]. Vet Parasitol, 2007, 143(1): 1-6. DOI: 10.1016/j.vetpar.2006.08.002
[2]Silva-Vergara ML, Maneira FR, De Oliveira RM, et al. Multifocal sporotrichosis with meningeal involvement in a patient with AIDS[J]. Med Mycol: Official Publication of the INT, 2005, 43(2): 187-190. DOI: 10.4269/ajtmh.2012.11-0441
[3]Ramos-E-Silva M, Vasconcelos C, Carneiro S, et al. Sporotrichosis[J]. Clin Dermatol, 2007, 25(2): 181-187.
[4]Sivagnanam S, Bannan AM, Chen SC, et al. Sporotrichosis (Sporothrixschenckiiinfection) in the New South Wales mid-north coast, 2000-2010[J]. Med J Australia, 2012, 196(9): 588-590. DOI: 10.5694/mja11.10755
[5]Migaki G, Font RL, Kaplan W, et al. Sporotrichosis in a pacific white-sided dolphin (lagenorhynchus obliquidens)[J]. Am J Vet Res, 1978, 39(12): 1916-1919. DOI: 10.1121/1.2932059
[6]Lopes-Bezerra LM, Schubach A, Costa RO.Sporothrixschenckiiand sporotrichosis[J]. Anais DA Academia Brasileira DE Ciencias, 2006, 78(2): 293-308. DOI: 10.2217/fmb.10.157
[7]Zhao B, Zhang ZK, Ni RZ, et al. Sporotrichosis[M]. In Clinical Dermatology, Nanjing: Jiangsu Science & Technology Press, 2001: 433-434. (in Chinese)
赵辨,张振楷,倪容之,等. 孢子丝菌病[M].临床皮肤病学,2001: 433-434 .
[8]Marimon R, Gene J, Cano J, et al. Molecular phylogeny ofSporothrixschenckii[J]. J Clin Microbiol, 2006, 44(9): 3251-3256. DOI: 10.1128/JCM.00081-06
[9]Marimon R, Cano J, Gene J, et al.Sporothrixbrasiliensis,S.globosa, andS.mexicana, three newSporothrixspeciesof clinical interest[J]. J Clin Microbiol, 2007, 45(10): 3198-3206. DOI: 10.1128/JCM.00808-07
[10]Marimon R, Serena C, Gene J, et al.Invitroantifungal susceptibilities of five species of sporothrix[J]. Antimicrob Agents Chemother, 2008, 52(2): 732-734. DOI: 10.1128/AAC.01012-07
[11]Arrillaga-Moncrieff I, Capilla J, Mayayo E, et al. Different virulence levels of the species ofSporothrixin a murine model[J]. Clin Microbiol Infect Official, 2009, 15(7): 651-655. DOI: 10.1111/j.1469-0691.2009.02824.x
[12]Mei XL, Xia JX, Wang JY, et al. Clinical and pathological analysis of 100 cases of sporotrichosis[J]. Chin J Mycol, 2011, 6(4): 203-206. (in Chinese)
梅向林,夏建新,王敬医,等.孢子丝菌病100例临床及病理分析[J] .中国真菌学杂志, 2011, 6(4):203-206.
[13]Schechtman RC. Sporotrichosis: part II[J]. Skinmed, 2010, 8(5): 275-280.
[14]Tan JW, Liu W, Wan Z. Reclassification of 33 clinical strains ofSporothrixfrom northern China based on phenotypic and molecular characters[J]. Mycosystema, 2013, 32(2): 161-167. (in Chinese)
谭静文,刘伟,万喆,等.来自中国北方部分地区33株孢子丝菌菌株的表型和基因型鉴定[J] .菌物学报, 2013,32(2): 161-167.
[15]Yu X, Wan Z, Zhang Z, et al. Phenotypic and molecular identification of Sporothrix isolates of clinical origin in Northeast China[J]. Mycopathologia, 2013, 176(1-2): 67-74. DOI: 10.1007/s11046-011-9437-3
[16]Schoch CL, Seifert KA, Huhndorf S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi[J]. Proc Nat Acad Sci U S A, 2012, 109(16): 6241-6246. DOI: 10.1073/pnas.1117018109
[17]Zhou X, Rodrigues AM, Feng P, et al. Global ITS diversity in theSporothrixschenckiicomplex[J]. Fungal Diversity, 2013: 1-13. DOI: 10.1007/s13225-013-0220-2
[18]Porebski S, Bailey LG, Baum RB. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J]. Plant Mol Biol Rep, 1997, 15: 8-15.
[19]Thompson JD, Gibson TJ, Plewniak F, et al. The CLUSTAL_X Windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4876-4882. DOI: 10.1093/nar/25.24.4876
[20]Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (mega) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599. DOI: 10.1093/molbev/msm092
[21]Mesa-Arango AC, Del Rocio Reyes-Montes M, Perez-Mejia A, et al. Phenotyping and genotyping ofSporothrixschenckiiisolates according to geographic origin and clinical form of Sporotrichosis[J]. J Clin Microbiol, 2002, 40(8): 3004-3011. DOI: 10.1128/JCM.40.8.3004-3011.2002
[22]Madrid H, Gene J, Cano J, et al.SporothrixbrunneoviolaceaandSporothrixdimorphospora, two new members of theOphiostomastenoceras-Sporothrixschenckiicomplex[J]. Mycologia, 2010, 102(5): 1193-1203. DOI: 10.3852/09-320
[23]Madrid H, Cano J, Gene J, et al.Sporothrixglobosa, a pathogenic fungus with widespread geographical distribution[J]. Revista Iberoamericana DE Micolog a, 2009, 26(3): 218-222.
[24]Rodrigues AM, De Hoog S, De Camargo ZP. Emergence of pathogenicity in theSporothrixschenckiicomplex[J]. Med Mycol, 2012, 51(4): 405-412. DOI: 10.3109/13693786.2012.719648
[25]Kong X, Xiao T, Lin J, et al. Relationships among genotypes, virulence and clinical forms ofSporothrixschenckiiinfection[J]. Clin Microbiol Infect Official, 2006, 12(11): 1077-1081. DOI: 10.1111/j.1469-0691.2006.01519.x
[26]Tlougan BE, Podjasek JO, Patel SP, et al. Neonatal sporotrichosis[J]. Pediatr Dermatol, 2009, 26(5): 563-565. DOI: 10.1111/j.1525-1470.2009.00986.x
[27]Devi KR, Devi MU, Singh TN, et al. Emergence of sporotrichosis in Manipur[J]. Indian J Med Microbiol, 2006, 24(3): 216-219.
[28]White TJ, Bruns T, Lee S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]. In:PCR protocols: a guide to methods and applications. Nutley: Hoffmann-La Roche Inc., 1990, 18: 315-322.
[29]Glass NL, Donaldson GC. Development of primer sets designed for use with the PCR to amplify conserved genes from filamentous ascomycetes[J]. Appl Environ Microbiol, 1995, 61(4): 1323-1330.
[30]Liu TT, Zhang K, Zhou X. Molecular identification of Sporothrix clinical isolates in China[J]. J Zhejiang Univ Sci B, 2014, 15(1): 100-108.
