鳜小肽转运载体PepT1基因分子特征及其表达研究
2014-03-29刘知行张健东刘小燕李虹辉王开卓陈琳褚武英张建社
刘知行张健东刘小燕李虹辉王开卓陈 琳褚武英张建社
(1.湖南农业大学动物科技学院, 长沙 410128; 2. 长沙大学生物工程与环境科学系, 长沙 410003; 3. 广东海洋大学水产学院, 湛江 524088)
鳜小肽转运载体PepT1基因分子特征及其表达研究
刘知行1,2张健东3刘小燕1李虹辉2王开卓2陈 琳2褚武英2张建社2
(1.湖南农业大学动物科技学院, 长沙 410128; 2. 长沙大学生物工程与环境科学系, 长沙 410003; 3. 广东海洋大学水产学院, 湛江 524088)
小肽转运载体(PepT1)是低亲和力、高容量的肽转运载体, 在小肽的吸收过程中发挥着重要的作用。研究采用同源克隆和RACE技术克隆了鳜鱼(Siniperca chuatsi) PepT1基因全长cDNA序列, 其cDNA序列全长为2480 bp, 包含43 bp的5′UTR序列, 232 bp的3′UTR序列, 以及2205 bp开放阅读框, 编码735个氨基酸。氨基酸序列同源性分析结果显示, 鳜鱼与石斑鱼(Epinephelus aeneus)、鲈鱼(Dicentrarchus labrax)PepT1间同源性均为89%, 与其他非鱼类物种的同源性则在46%—56%。经预测, 鳜鱼PepT1编码蛋白的分子量为64.8 kD,等电点为8.97, 该蛋白具有与哺乳动物同源蛋白相似的12 个螺旋跨膜结构, 并且在跨膜区9和10之间有一个大的外环; 跨膜区氨基酸高度保守, 并存在有5个膜外N-糖基化位点和3个膜内含蛋白激酶C基序的相同区域。实时荧光定量表达分析表明, 鳜鱼PepT1基因在前肠和中肠中表达量显著高于后肠(P<0.05), 这说明前、中肠是鳜鱼肠道吸收小肽的主要部位; 在胚后不同发育阶段鳜鱼前肠均能检测到 PepT1基因的表达,并且在10 g个体中表达量最高, 之后随着体重的增加其表达量维持在一个稳定水平。本研究结果首次报道了鳜鱼PepT1基因全序列及其分子表达特征, 为鱼类营养及生理学的研究提供有价值的参考资料。
鳜鱼; 小肽转运载体; 克隆; 表达分析
Agar, et al.[1]在1953年首次对小肽转运载体进行了研究, 然而直到1994年才有人首次成功克隆出兔的小肠小肽转运载体, 并将其命名为Ⅰ型肽转运载体(PepT1)[2]。目前对于PepT1的研究还是主要集中在高等脊椎动物PepT1基因的分子结构及其功能关系[3—9], 关于鱼类PepT1基因的研究还相对较少,已见的报道有斑马鱼(Danio rerio, AY300011)[10]、鲫鱼(Carassius auratus, AF295589.1)[11]和大西洋鳕(Gadus morhua, AY921634)[12]等。这些研究为探讨鱼类PepT1基因的分子结构和生理功能奠定了基础。在鱼类组织中PepT1基因的表达有着极其重要的意义,它的组织特异性表达水平不仅能反映组织中细胞的功能关系, 而且还能体现出PepT1的转运活性[13]。熊刚等[11]研究发现, PepT1基因在鲫鱼成鱼肠道前段的表达最高, 并沿着肠道纵轴方向从前往后的表达水平依次降低, 这为肠段分区即肠道前、中、后不同部位吸收蛋白质的强度不同提供了理论依据。但鱼类肠道PepT1在不同发育阶段的表达规律研究尚未见报道。对PepT1在不同发育阶段的表达进行研究,将有助于认识鱼类PepT1对小肽转运的分子机理。因此, 本研究采用RACE技术和同源克隆的方法首次克隆得到了鳜鱼肠道小肽转运载体PepT1 cDNA的全长序列(KF668018), 对其进行了生物信息学分析,并首次采用实时荧光定量PCR技术分析该基因在鳜鱼发育时期不同阶段的表达变化规律, 为研究小肽转运载体PepT1基因在生物体内转运小肽的分子机
1 材料与方法
理奠定了基础。1.1 试验材料
实验成鱼取自湖南省衡阳市湘鳜养殖公司。采集体重约500 g鳜鱼成体6尾, 分别解剖获取其前、中、后段肠道样本, 保存于液氮中。其他阶段实验鱼取自湖南省常德市兴达鳜鱼繁养场, 按体重不同将鳜鱼分为5、10、50、100和250 g五组, 每组分别采集实验鱼6尾, 解剖获取其前肠样本, 250 g组取心、肝、脑、肾、白肌和红肌样本, 迅速于液氮中冷冻, 稍后保存于–80℃冰箱备用。
1.2 总RNA提取和cDNA第一链的合成
取鳜鱼成鱼肠道不同部位以及不同发育阶段各组鳜鱼样品, 采用Trizol法提取总RNA。在1%琼脂糖凝胶中电泳检测总 RNA的浓度和纯度, 保证其A260/A280在1.8至2.0之间。以总RNA为模板, 利用反转录试剂盒按说明合成cDNA第一链。
1.3 PepT1 基因的克隆
从我们已构建的鳜鱼成鱼肌肉转录组文库中找到PepT1序列部分片段, 通过DNAMAN软件与多种相近鱼类进行比对分析其保守性。并用Premier5.0软件在保守区内设计特异性引物(表1), 由上海铂尚公司合成。PCR反应程序为95℃预变性3min; 94 ℃30s, 56 ℃ 30s, 72 ℃ 30s,共35个循环; 72℃延伸10min。共设计 4条引物完成对 3′端片段的克隆, F1S、F2R、3′RACE Olig(T)-Adaptor 和 3′RACE Adaptor, 根据已获得的中间片段序列设计特异性引物 3’F1和 3’F2, 且 3’F1在 3’F2的上游; 其中3′RACE Olig(T)-Adaptor和3′RACE Adaptor 是一对通用引物(表1)。反转录时, mRNA反转录试剂盒中的Olig(T)用3′RACE Olig(T)-Adaptor代替; 扩增时, 以 F1S和 3′RACE Adaptor为上下游引物进行第一轮PCR, 扩增条件为: 94℃预变性3 min;94℃ 30s, 58℃30s, 72℃ 30s,共30个循环; 72℃延伸10min。第一轮产物稀释50倍后再用F2R和3′RACE Adaptor为上下游引物做第二轮巢式 PCR, 反应条件除第三步变为60℃ 30s, 其余同上。通过已克隆得到的中间片段,在保守区内用 Premier5.0设计特异性引物 Pe-F2和Pe-R2, 得到结果后, 用样的方法设计引物 Pe-F3和Pe-R3, 其中Pe-F3在Pe-F2的上游。PCR扩增条件: 95℃预变性3min; 94℃ 30s, 58℃ 30s, 72℃ 1min30s,共35个循环; 72℃延伸10min。采用同源克隆的方法,从石斑鱼(Epinephelus aeneus)PepT1 5′端非编码区序列设计引物Pe-F4和从已克隆到的鳜鱼PepT1序列设计引物 Pe-R4克隆鳜鱼 PepT1 5′端非编码区部分序列。条件为 95℃预变性 3min; 94℃ 30s, 60℃ 30s, 72℃ 30s, 共35个循环; 72℃延伸10min。各段PCR产物通过 1.0%的琼脂糖凝胶电泳检测、切胶回收并纯化后过夜连接至 pGM-T载体, 转化到感受态Top10, 蓝白斑筛选通过菌液PCR检测为阳性克隆后,送至上海铂尚公司进行测序。

表1 PepT1 基因克隆引物Tab. 1 The primers of PepT1 gene
1.4 鳜鱼 PepT1基因的不同部位及不同发育阶段表达分析
分别以鳜鱼成鱼肠道不同部位(前肠, 中肠, 后肠)及不同发育阶段(5、10、50、100和250 g)cDNA为模板, 用Primer Primer 5.0软件根据本实验室已有的鳜鱼 RPL13 (ribosomal protein L13)基因序列(BC063378)设计荧光定量 PCR内参引物: RPL13-F(CACAAGAAGGAGAAGGCTCGGGT), RPL13-R (TTTGG-CTCTCTTGGCACGGAT), 产物长度为120 bp。在所得鳜鱼PepT1基因的ORF区域内设计引物, Pe-FA(GTGAAGATGGTGCTGAAGGTGC), Pe-FB(GAGAAATCCAAAGTCGCCGTT), 产物长度为126 bp; 采用RT-qPCR方法检测PepT1基因在不同部位及不同发育阶段的表达差异。实验中设置了 6个生物学样品重复, 并且每个生物学样品有 3个技术性重复。qPCR反应体系: SYBR®Premix Ex TaqTM(2×) 12.5 μL, dH2O 10 μL, cDNA 1 μL, ROX Reference Dye(50×) 0.5 μL, 引物各0.5 μL。反应条件为: 95 ℃ 3min; 95 ℃ 3s, 60 ℃ 25s, 40Cycles;绘制溶解曲线: (65—95)℃ , 每0.05 ℃ 读板1次; 反应结束后观察实验结果并进行数据处理。
对荧光定量PCR所得的实验数据用Excel 2007软件进行初步处理, 去掉一个最高值和最低值后,用公式计算目的基因的表达量: 2–ΔΔCt, 其中ΔΔCt=(Ct目的基因-Ct内参基因)-(Ct目的基因-Ct内参基因)对照, Ct表示定量PCR仪上达到阈值的循环数, 通过SPSS16.0软件对所得的表达丰度数据进行单因素方差分析(One-Way ANOVA)并采用最小显著差数法(LSD)进行多重比较(P<0.05表示显著性差异)。所有的结果都用平均值±标准差(mean±SD)来表示。
1.5 数据分析
采用DNA MAN软件对序列进行比对分析, 用MEGA 5.1制作系统进化树, 数据用平均值±标准差表示, 荧光定量数据采用 SPSS 16.0软件进行单因素方差分析与显著性检验。
2 结果
2.1 PepT1 cDNA序列和氨基酸序列分析
通过对鳜鱼肠道中 PepT1的克隆, 将克隆的PepT1基因序列进行拼接得到PepT1基因cDNA全序列, 通过 BLAST分析确认为目的基因鳜鱼PepT1基因。鳜鱼PepT1基因cDNA全长2480 bp (图1), 其中开放阅读框ORF区为2205 bp, 编码了734个氨基酸, 5′端非编码区长43 bp, 3′端非编码区长232 bp。
2.2 PepT1预测蛋白质生物学分析
预测鳜鱼PepT1蛋白质分子量大小64.8 kD, 理论等电点 PI值为 8.97。通过在线软件 TMpred和TMHMM-2.0进行鳜鱼 PepT1跨膜结构域预测, 其预测结果包含多个跨膜域。推荐模型为包含12个跨膜结构域, 它的理论得分值最高; PepT1二级结构预测发现, 有3个蛋白激酶C位点和5个N-糖基化位点。通过生物学信息分析软件的结果分析发现PepT1是一种含有多个跨膜结构域的跨膜蛋白, N端从第 16个氨基酸残基(Cys16)开始到第 674 (Tyr674)个氨基酸残基之间完成跨膜, 其一共进行了12次跨膜; 且在第九个和第十个跨膜结构域之间存在一个大的细胞外环, 这和其他物种的预测结果是一致的。
2.3 PepT1氨基酸同源性与进化分析
氨基酸序列的同源性结果分析表明, 鳜鱼PepT1氨基酸序列比较于其他物种的同源性相似度都很高, 其中与鲈鱼和石斑鱼的同源性最高均为89%, 其次是斑马鱼和 云纹平 鲉均为88%, 与人、兔和猪的同源性在50%—56%。运用DNAMAN软件把鳜鱼PepT1基因和其他物种该基因进行多序列比对结果显示, 鳜鱼和其他物种之间存在着高度保守,尤其是与其他鱼类。
2.4 种间系统进化树的构建
在对翘嘴鳜 PepT1基因进行进化树分析, 用NCBI BLASTn(核苷酸-核苷酸)和BLASTX(蛋白质-蛋白质)搜索同源序列, 选取的鱼类有: Epinephelus aeneus (JX122768.1), Danio rerio (AY300011.1), Cyprinus carpio (JN896885.1), Dicentrarchus labrax (FJ237043.2), Sebastes Nebulosus (EU160494.1), Perca flavescens (GQ906471.2), Chionodraco hamatus (AY-170828.2), Anguilla japonica (AB762417.1), Gadus morhua (AY921634.1), Oryctolagus cuniculus (U06467.1), Oreochromis niloticus (XM_003447363.1), Homo sapiens (AF043233.1), Mus musculus (AF205540.1), Sus scrofa (AY180903.1)。然后用MEGA4.1软件, NJ邻接法构建系统进化树结果如下图2。
2.5 鳜鱼 PepT1基因在鳜鱼不同部位以及不同发育阶段的表达差异
采用qPCR方法对鳜鱼PepT1基因在不同部位及不同发育阶段的表达进行分析, 用 SPSS 17.0软件对所得表达丰度数据进行单因素方差分析(P<0.05), 所有结果以平均值±标准差(mean±SD)表示。结果显示: (1)在250 g鳜鱼成鱼中, PepT1在前肠中表达水平最高, 中肠次之, 后肠最低(图3)。(2)在5 g、10 g、50 g、100 g、250 g和500 g不同发育时期个体中均检测到 PepT1基因的表达, 并且PepT1在5 g和10 g组中表达量较高, 随着体重的增加在50 g、100 g、250 g和500 g组中反而下降, 其中在10 g组表达量最高, 且与其他组存在显著性差异(P<0.05)(图4)。(3)250 g鳜鱼成鱼中, PepT1在不同组织中的表达量, 肠道显著高于其他组织, 脑次之, 在白肌和红肌中均检测到了表达量(P<0.05)。

图1 鳜鱼PepT1 cDNA全长序列及其推导的氨基酸序列Fig. 1 Full length cDNA sequences and the the deduced amino acid sequence of PepT1 fromS. chuatsi
3 讨论
PepT1是小肽转运家族中最先发现的一种小肽转运载体, 它是一种低亲和力、高容量的肽转运载体, 其在细胞中利用质子梯度来推动寡肽的吸收, PepT1基因在小肠中的表达最为显著, 吸收食物中分解的小肽[14]。目前PepT1在鱼类中的研究越来越受人们重视, 在斑马鱼(Danio rerio, AY300011)[10]、鲫鱼(Carassius auratus, AF295589.1)[11] 和大西洋鳕(Gadus morhua, AY921634)[12]等中都已经有过研究。通过对鳜鱼PepT1基因克隆和表达研究有利于从分子水平阐明鳜鱼吸收小肽的调控机理。
本研究从鳜鱼前肠组织中成功克隆PepT1基因,包括其ORF序列2480 bp, 共编码735个氨基酸。通过对氨基酸同源性进行比对, 结果发现 PepT1基因在跨膜结构域中具有高度保守, 在大的细胞外环之间的氨基酸序列同源性较低, 而以往的研究认为这些相似度较高的残基会在基因的功能和结构上发挥着重要的作用[12]。预测结果显示, 鳜鱼PepT1具有3个蛋白激酶C磷酸化位点(PKC), 5个N-糖基化位点, 在大西洋鲑鱼, 鲈鱼, 斑马鱼等其他物种中同样存在这些结构位点; 相关研究资料认为, 蛋白质功能位点区域的氨基酸结构大部分都具有高度保守, 当前已经有很多研究是关于功能位点区域氨基酸保守模式[15]。这些保守区域能够对PepT1的结构和功能调节起着重要的作用[16]。有研究发现 PepT1在Caco-2 (结肠腺癌细胞)细胞系中Gly-Sar 二肽的转运规律中, 使用佛波醇酯激活 PKC能够抑制PepT1的转运, 这是由于在Km不变的情况下, 最大速度降低了[17]。把鳜鱼和各物种的PepT1基因推测出的氨基酸序列进行树聚类分析, 结果显示鳜鱼PepT1氨基酸序列和其他物种的同源性在 46%至89%之间。利用PepT1基因XX序列构建的进化树符合系统发生规律: 鳜鱼与鲈鱼、石斑鱼同属于鲈形目, 聚为一枝; 鱼类物种之间有明显更近的系统发生关系, 而人、鼠、兔等哺乳动物另外聚为一类。

图2 鳜鱼PepT1基因与其他物种PepT1的系统进化分析Fig. 2 Phylogenetic analysis of S. chuatsi PepT1 with other species PepT1
PepT1不仅在不同种属动物体内分布不一致,而且在同一个体内不同器官的分布亦不一致[18]。有研究发现在哺乳动物中PepT1 mRNA主要在肾上皮细胞和小肠中表达[14], 特别是在肠道中前段的表达量很高, 在脑细胞、胆管上皮细胞、结肠、肾和肝中表达相对较少[19—20], Ogijara H, et al.对小鼠的研究表明在肠道中, 沿肠管垂直方向的表达规律是PepT1 的表达从绒毛顶部向隐窝逐渐下降; 沿肠管纵轴方向的表达规律是从十二指肠到回肠PepT1 逐渐降低等两种分布模式[21—23], 这种分布的差异性表明了对小肽利用程度和蛋白质消化吸收部位的差异。本研究发现小肽转运载体PepT1 基因在鳜鱼前肠、中肠、后肠等不同部位的表达, 且在鳜鱼前肠表达最高、中肠其次、后肠最低, 这与我们采用基因芯片技术所获得的鳜鱼小肽转运体基因表达结果相一致[24]。据此推断, PepT1 在鳜鱼肠道组织中的表达也是沿肠管纵轴方向从前肠到后肠的表达量逐渐降低, 这和其他的物种, 如虹鳟(O. mykiss)[25]、成体鸡[13]、泥鳅(Misgurnus mizolepis Gunther)[26]、欧洲黑鲈(Dicentrarchus labrax)[27]等的研究结果相同。鳜肠道小肽转运载体基因的组织表达特征, 为更好的分析小肽的利用和吸收规律提供基础。

图3 鳜鱼PepT1在肠道不同部位中的mRNA相对表达丰度Fig. 3 The abundance of PepT1 mRNA expression in different intestinal position of S. chuatsi (n=6)
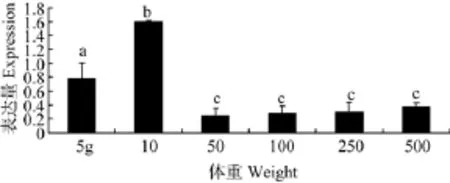
图4 鳜鱼PepT1在不同发育阶段的前肠mRNA相对表达丰度Fig. 4 The abundance of PepT1 mRNA expression in different developmental stages of S. chuatsi (n=6)

图5 鳜鱼PepT1在不同组织中mRNA相对表达丰度Fig. 5 The abundance of PepT1 mRNA expression in different tissues of S. chuatsi (n=6)
我们将鳜鱼不同发育阶段按体重分为6个不同组。PepT1在5、10 g中表达量较高, 随着体重的增加在50、100、250、500 g中反而下降且趋于稳定, 其中在10 g时表达量最高且与其他组存在显著性差异(P<0.05), 因此我们推测, 鳜鱼幼鱼在约 10 g左右时, 其吸收小肽能力比其他阶段更强, 有利于满足其快速生长需要。我们针对250 g成鱼中PepT1在不同组织的表达量发现, 肠道的表达量显著高于脑,而脑又高于其他组织, 在非肠道组织中的白肌、红肌中均有转录表达, 这为鱼类 PepT1在肌肉组织中表达提供了证据。
本研究结果第一次报道了鳜鱼PepTl 基因全长序列, 发现了 PepTl 基因在鳜鱼成鱼肠道的不同部位的表达规律, 以及鳜鱼幼鱼的成长 PepTl 基因的表达变化, 和其在氨基酸运转中潜在的生理学功能,为鱼类营养生理学的研究提供有价值的参考资料。
[1] Agar W T, Hird F J, Sidhu G S. The active absorption of amino-acids by the intestine [J]. The Journal of Physiology, 1953, 121(2): 255—263
[2] Fei Y J, Kanai Y, Nussberger S, et al. Expression cloning of a mammalian proton-coupled oligopeptide transporter [J]. Nature, 1994, 368(6471): 563—566
[3] Pan Y X, Wong E A, Bloomquist J R, et al. Poly (A) + RNA from sheep omasal epithelium induces expression of a peptide transport protein(s) in Xenopus laevis oocytes [J]. Journal of Animal Science, 1997, 75(12): 3323—3330
[4] Chen H, Wong E A, Webb K E. Tissue distribution of a peptide transporter mRNA in sheep, dairy cows, pigs and chickens [J]. Journal of Animal Science, 1999, 77(5): 1277—1283
[5] Wenzel U, Kuntz S, Daniel H. Flavonoids with epidermal growth factor-receptor tyrosine kinase inhibitory activity stimulate PepT1-mediated cefixime uptake into human intestinal epithelial cells [J]. Biotransporters, 2001, 299(1): 351—357
[6] Chen H, Pan Y X, Wong E A, et al. Dietary protein level and stage of development affect expression of an intestinal peptide transporter (cPepT1) in Chickens [J]. The Journal of Nutrition, 2005, 135(2): 193—198
[7] Gilbert E R, Li H, Emmerson D A, et al. Developmental Regulation of nutrient transporter and enzyme mRNA abundance in the small intestine of Broilers [J]. Poultry Science, 2007, 86(8): 1739—1753
[8] Fei Y J, Kanal Y, Nussbe S. Expression cloning of a mammalian proton coupled oligopeptide transporter [J]. Nature (Lood), 1994, 368(6471): 563—566
[9] Liang R, Fei Y J, Prasad P D, et al. Human intestinal H+/peptide cotransporter: Cloning functional expression, and chromosomal localization [J]. The Journal of biological chemistry, 1995, 270(12): 6456—6463
[10] Ye W L, Li Y W. Progress on intestinal oligopeptide transporter PepT1 of fishes [J]. Hebei Fisheries, 2009, (12): 50—54 [叶万里, 李英文. 鱼类肠道小肽转运载体 PepT1的研究进展. 河北渔业, 2009, (12): 50—54]
[11] Xiong G. Cloning, analysis of sequences and tissue expression of LAT2 and PepT1 genes in carp [D]. Hunan Agricultural University. Changsha. 2009 [熊刚. 鲫鱼LAT2和PepT1基因克隆、序列分析及组织表达研究.湖南农业大学. 长沙. 2009]
[12] Amaerg J J, Myr C, Kamisaka Y, et al. Expression of the oligopeptide transporter, PepT1, in larval Atlantic cod (Gadus morhua) [J]. Comparative Biochemisty and Physiology. Part B, Biochemisty & Molecular Biology, 2008, 150(2): 177—182
[13] Gilbert E R. Dietary and developmental regulation of nutrient transporter gene expression in the small intestine of two lines of broilers [J]. Animal and Poultry Sciences, 2008, 251—254
[14] Kottra G, Stamfort A, Daniel H. PepT1 as a paradigm for membranecarriers that mediate electrogenic bidirectional transport of anionic, cationic, and neutral substrates [J]. The Journal of Biological Chemistry, 2002, 277(326): 83—91
[15] Bause E. Structural requirements of N-glycosylation of proteins. Studies with proline peptides as conformational probes [J]. The Biochemical Journal, 1983, 209(2):331—336
[16] Xiang J, Jiang H, Hu Y, et al. KyotorpHin transport and metabolism in rat and mouse neonatal astrocytes [J]. Brain Research, 2010, 1347: 11—18
[17] Brandsch M, Miyamoto Y, Ganapathy V, et al. Expression and protein kinase C-dependent regulation of peptide/H+ co-transport system in the Caco-2 human colon carcinoma cell line [J]. The Biochemical Journal, 1994, 299(Pt 1): 253—260
[18] Feng J C, Liu Z, Lu S Q, et al. Cloning of the Pept1 gene and analysis of its expression in Cteno pharyngodon idellus tissues [J]. Journal of Fishery Sciences of China, 2013, (2) 276—285 [冯军厂, 刘臻, 鲁双庆, 等. 草鱼小肽转运载体PepT1基因的克隆与表达特征. 中国水产科学, 2013, (2): 276—285]
[19] Ford D, Howard A, Hirst B H. Expression of the peptide transporter hPepT1 in human colon: a potential route for colonic protein nitrogen and drug absorption [J]. Histochemistry and Cell Biology, 2003, 119(1): 37—43
[20] Knutter I, Rubio A I, Boll M, et al. H+peptide cotransport in the human bile duct epithelium cell line SKChA-1 [J]. American Journal of Physiology Gastrointestinal and Liver Physiology, 2002, 283(1): G222—G229
[21] Miyamoto K, Shiraga T, Morita K, et al. Sequence, tissuedistribution and developmental changes in rat intestinal oligopeptide transporter [J]. Biochimica et Biophysiac Acta, 1996, 1305(1—2): 34—38
[22] Ogijara H, Suzuki-T, Nagamachi Y, et al. Peptide transporter in the rat small intestine: uhrastructural localization and the effect of starvation and administration of amino acids [J]. Histochemical Journal, 1999, 31(3): 169—174
[23] Tanaka H, Miyamoto K I, Morita K, et al. Regulation of the PepT1 peptide transporter in the rat small intestine in response to 5-fluorouracil-inducedinjury [J]. Gastroenterology, 1998, 14(4): 714—723
[24] Zhang J S, Xia X J, Chu W Y, et al. Gene expression profiles of the muscle tissues of the Mandarin fish, Siniperca chuatsi [J]. Acta Hydrobiologica Sinica, 2009, 33(1): 46—53 [张建社, 夏新界, 褚武英, 等. 基于异源 cDNA基因芯片杂交的鳜鱼肌肉组织基因表达谱初步分析. 水生生物学报, 2009, 33(1): 46—53]
[25] Ostaszewska T, Kamaszewski M, Grochowski P, et al. The effect of peptide absorption on PepT1 gene expression and digestive system hormones in rainbow trout (Oncorhynchus mykiss) [J]. Comparative Biochemistry and Physiology. Part A, Molecular & Integrative Physiology, 2010, 155(1): 107—114
[26] Goncalves A, Cstro L, Pereira W C, et al. Is there a compromise between nutrient uptake and gas exchange in the gut of Misgurnus anguiIlicaudatus, an intesfinal airbreathing fish [J]? Comparative Biochemisty and Physiology. Part D, Genomics & Protemics, 2007, 2(4): 345—355
[27] Terova G, Cord S, Verri T, et al. Impact of feed availability on PepT1 mRNA expression levels in sea bass (Dicentrarchus labrax) [J]. Aquaculture, 2009, 294(3—4): 288—299
MOLECULAR CHARACTERIZATION AND EXPRESSION RESEARCH OF OLIGOPEPTIDE TRANSPORTER PEPT1 IN SINIPERCA CHUATSI
LIU Zhi-Xing1,2, ZHANG Jian-Dong3, LIU Xiao-Yan1, LI Hong-Hui2, WANG Kai-Zhuo2, CHEN Lin2, CHU Wu-Ying2and ZHANG Jian-She2
(1. College of Animal Science and Technology, Hunan Agricultural University, Changsha, 410128, China; 2. Department of Bioengineering and Environmental Science, Changsha University, Changsha 410003, China; 3. Fisheries College of Guangdong Ocean University, Zhanjiang 524088, China)
Small peptide transporter (PepT1) is a transporter with low-affinity and high capacity peptide, which plays an important role in the peptide absorption. In order to reveal the molecular mechanism of the small peptide transporter PepT1-mediated protein digestion and absorption, the small peptide transporter PepT1 gene of the Siniperca chuatsi was cloned by using RT-PCR and RACE techniques. The full-length cDNA sequence of the PepT1 was 2480 bp, including 43 nucleotides at 5′UTR and 232 nucleotides at 3′UTR and 2205 nucleotides as an open reading frame encoding a 735-amino-acid peptide. The PepT1 amino acid sequence was determined and it shared a similarity to those of other mammalian homolog protein, which included 12 helix trans-membrane regions and existed a large outer-ring between 9th and 10th of the transmembrane region. Homologous analysis of the PepT1 showed that the amino acid of the PepT1 was highly homologous to those of other vertebrates and it showed high percentages of similarities at 89% with Epinephelus aeneus and Dicentrarchus labrax. In addition, the PepT1 amino acid sequence varied in similarity to other vertebrates from 46% to 56%. The encoded protein molecular weight was predicted at 64.8 kD with pI at 8.97. The quantitative RT-PCR analysis demonstrated that the foregut and midgut may be the key parts of intestinal absorption of small peptides as the PepT1 expression in the foregut and midgut was significantly higher than the hindgut (P<0.05). The PepT1 gene expression was detected in all post-embryonic developmental stages, and it showed a peak expression the growing to about 10g in weight, then the expression remained stable in late developmental stages. The significance of the work first reported the PepT1 cDNA structure and its expression pattern, furthermore, it provided a valuable reference for research on fish nutrition and physiology.
Siniperca chuatsi; Peptide transporter (PepT1); Clone; mRNA expression
Q344+.1
A
1000-3207(2014)03-0556-07
10.7541/2014.78
2013-11-05;
2014-02-15
国家自然科学基金重点项目(31230076)资助
刘知行(1989—), 男, 湖南娄底人; 硕士; 研究方向水生生物学。E-mail: 85335251@qq.com
张建社 (1956— ), 男, 湖南长沙人; 博士, 教授; 研究方向鱼类发育分子生物学。E-mail: jzhang@ccsu.cn刘小燕(1965—), 女, 湖南益阳人; 博士, 教授; 研究方向鱼类病害。E-mail: liuxy186@163.com
