鲟分枝杆菌病及其病原研究
2014-03-29张德锋李爱华龚小宁
张德锋李爱华龚小宁
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
鲟分枝杆菌病及其病原研究
张德锋1,2李爱华1龚小宁1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
2009—2010年间, 我国人工养殖的中华鲟(Acipenser sinensis)、史氏鲟(Acipenser schrencki)和杂交鲟(hybrid sturgeon: A. baeri-A. gueldenstaedtii)暴发了细菌性疾病。患病鲟通过组织切片观察, 病原菌的分离、鉴定以及组织样品中病原菌的?检测, 结果显示从19条患病鲟中分离到49株分枝杆菌。病原菌经过多个保守基因的测序分析和部分生理生化特征的鉴定, 共发现有 7种分枝杆菌, 分别为龟分枝杆菌(Mycobacterium chelonae)、海分枝杆菌(Mycobacterium marinum)、戈登氏分枝杆菌(Mycobacterium gordonae)、偶发分枝杆菌(Mycobacterium fortuitum)、苏尔加分枝杆菌(Mycobacterium szulgai)、猪分枝杆菌 (Mycobacterium porcinum)和Mycobacterium arpuense。在诊断过程中发现两种或三种分枝杆菌同时存在于同一样品中, 分子生物学的诊断结果表明分枝杆菌复合感染十分常见, 而海分枝杆菌是分枝杆菌复合感染中最为常见的分枝杆菌。分离的病原菌对斑马鱼的攻毒试验结果表明在以上 7种分枝杆菌中海分枝杆菌的毒力最强。以上结果表明海分枝杆菌是鲟分枝杆菌病的主要致病菌, 分枝杆菌复合感染是鲟分枝杆菌病的主要感染形式。研究中史氏鲟和中华鲟的分枝杆菌病, 以及在病鱼体内分离的猪分枝杆菌和M. arupense在国内外均尚未见报道。
鲟; 分枝杆菌; 鉴定; 系统发育分析; 复合感染
分枝杆菌病是鱼类常见的细菌性疾病之一, 目前已经在 150多种鱼类中发现了分枝杆菌病[1], 它是由非结核分枝杆菌(Nontuberculous mycobacteria, NTM) 造成的长期性感染疾病, 尤其当鱼类个体的免疫系统处于较弱的时候更易感染[2]。非结核分枝杆菌广泛存在于水体、土壤、空气等自然环境中[3,4],它也是人的一种机会致病菌, 对人类尤其是渔民的健康构成威胁。非结核分枝杆菌可以分为速生型分枝杆菌(Rapidly growing mycobactreia, RGM)和缓慢生长型分枝杆菌(Slowly growing mycobacteria, SGM), 前者的培养时间一般少于 7d, 而后者的培养时间通常大于7d, 需要培养2—4周才形成清晰可见的菌落。在鱼类分枝杆菌中, 龟分枝杆菌(Mycobacterium chelonae)、偶发分枝杆菌(Mycobacterium fortuitum)和海分枝杆菌(Mycobacterium marinum)是鱼类常见的病原菌[5]。而且, 戈登分枝杆菌(Mycobacterium gordonae)和苏尔加分枝杆菌(Mycobacterium szulgai)等也常在病鱼中分离到[6—8]。然而, 鲟分枝杆菌病却很少有报道, 更没有关于鲟分枝杆菌病暴发和流行的相关报道。目前, Pate, et al.[9]在斯洛文尼亚对观赏鱼病原菌调查时发现小体鲟(Acipenser ruthenus)可以感染偶发分枝杆菌。Stragier, et al.[10]在比利时的杂交鲟(Acipenser baerii-Acipenser guldenstaedtii hybrid sturgeon)中分离到海分枝杆菌ITM06-3844。然而, 中华鲟(Acipenser sinensis)、史氏鲟(Acipenser schrencki)等鲟分枝杆菌病却没有相关报道。
中国作为渔业养殖大国, 渔业的健康养殖与经济发展息息相关。近年来中华鲟、史氏鲟、杂交鲟(hybrid sturgeon: A. baeri-A. gueldenstaedtii)、西伯利亚鲟(Acipenser baeri)等在中国已经大规模养殖, 在水产养殖中占有十分重要的地位。在过去的十年中,中国是世界上鲟产量最大的国家[11,12]。随着鲟的高度集约化人工养殖, 相关的疾病给鲟养殖业造成严重的经济损失[13,14]。2009—2010年, 我国湖北省宜昌市人工养殖的史氏鲟、中华鲟以及江苏省淮安市人工养殖的杂交鲟均出现了大规模的死亡, 经过症状诊断、病原菌的分离培养和分子生物学方法检测,结果发现鲟的病原菌是分枝杆菌。本文着重对我国鲟分枝杆菌病的病原生物学进行研究, 以期为今后研究其流行病学和防治对策奠定理论基础。
1 材料与方法
1.1 样品采集和患病鲟的症状
2009—2010年间, 共收集到19条患病鲟。患病鲟分别来自于湖北省宜昌市中华鲟研究所、湖北省宜昌市清江网箱和江苏省淮安市大棚等 3三个地区。其中湖北省清江网箱养殖的中华鲟为室外养殖,其他均为室内养殖。鲟发病时的水温为25—30℃。
对两年中8批次共19尾患病鲟的观察发现, 其典型症状主要表现为发病初期没有明显的症状特征,发病晚期表现出体表有斑点状溃烂(尤其在腹部和鳃盖), 腹部肿大, 沿壁上浮游动且游动迟缓, 厌食。解剖发现腹部有大量的腹水, 肝脏有乳白色或浅红色斑点(图1A), 脾脏有淤血斑。
1.2 病原菌的分离和培养
患病鲟的肝脏、脾脏或血液等样品进行无菌操作分离病原菌。病原菌接种到Lowenstein–Jensen 培养基(Difco, Detroit, MI, USA) 和Middlebrook 7H10 (Difco, Detroit, MI, USA)+OADC的琼脂培养基上进行分离培养, 30°C 培养箱中培养 30d, 为了保持培养过程中培养基的湿度, 首先培养箱的湿度保持在50%以上, 其次用封口膜将平板封口, 每隔3天打开一次。挑取单菌落在Middlebrook 7H10+OADC的琼脂培养基上进行二次划线培养以得到纯的单克隆菌落。每个样品选取2—3株菌用来鉴定和保种, 从每个样品中分离的不同种病原菌中各选一株为代表菌株作进一步研究。
1.3 分离菌株的形态学和生理生化特征
分枝杆菌选择性培养基上生长的病原菌首先经过Ziehl-Neelsen染色和HE染色, 观察细菌形态和Ziehl-Neelsen染色情况, 并观察细菌的生长速率,色素的产生和生长时间。再对分离菌株进行如下的生理生化测定: 5% NaCl 生长、硝酸盐还原、Tween-80水解、脲酶活性、接触酶活性和芳香硫酸酯酶活性。
1.4 细菌基因组DNA的提取
分离菌株的基因组 DNA使用细菌基因组提取试剂盒提取 (天根生化科技有限公司), 使用方法参见试剂盒说明书, 基因组DNA于–20℃保存备用。肝脏和脾脏组织中细菌的基因组提取方法如下: 取50—100 mg组织样品加入无菌 PBS稀释后匀浆, 3000 r/min 离心 1min, 取上清液加入溶菌酶于 37℃消化过夜。然后使用细菌基因组提取试剂盒提取组织中的细菌基因组DNA, 最后将基因组DNA保持于–20℃备用。
1.5 分离菌株的分子生物学鉴定
分离菌株首先通过分枝杆菌属特异引物进行检测, 即以细菌基因组 DNA为模板, 分枝杆菌属通用引物T39和T13 (第一轮PCR)和引物preT43和T531 (第二轮PCR) 进行巢式PCR检测[15](表1)。阳性菌株进行保守基因 16S rRNA、rpoB和hsp65测序, 所用的引物分别是: 27f和1492r[16]、Myco-F和Myco-R[17]、HspF和HspR[18](表1)。PCR产物经过1%琼脂糖凝胶电泳后切胶回收, 然后将回收产物连接到pMD18-T载体(TaKaRa, 日本), 再转化感受态细胞大肠杆菌DH5α, 最后挑取阳性克隆进行测序分析。
1.6 系统发育树的构建
将上述三个保守基因的序列通过Genbank在线Blastn比对分析, 同时 16S rRNA序列通过 RDP (Ribosomal database project)网站进行比对分析, 经过比对分析后选取并下载与分离菌株序列同源性较高的且具有代表性的菌株序列(如ATCC、CIP等典型代表菌株), 然后去除测序基因的引物序列, 并将代表菌株的相应基因序列也截取到与分离菌株基因序列相同的长度。通过Clustal X (1.81) 软件分别进行多重比对, 再通过MEGA 4.1软件分别构建系统发育树。最后可以通过分析构建的系统发育树来确定分离菌株的种属关系。
1.7 组织中病原菌的检测
由于分枝杆菌与其他快速生长的鱼类病原菌相比而言较难培养, 因为它不仅需要专用培养基, 而且培养时间较长。因此, 为了进一步确定患病鲟的病原菌为分枝杆菌, 检测样品组织中的分枝杆菌显得十分必要。本文通过rpoB文库法和 PCR-DGGE法对样品中的病原菌进行检测。

表1 本文中使用的引物Tab. 1 The primers used in this study
由于分枝杆菌 16S rRNA具有较高的保守性,用于分枝杆菌种的鉴定得到的结果常常不够理想,相比较而言, 分枝杆菌rpoB在种间具有更高的区分度, 所以通过PCR扩增样品组织中的分枝杆菌rpoB (引物为Myco-F和Myco-R)构建rpoB文库。即PCR产物电泳后切胶回收, 将回收产物连接到pMD18-T载体, 再转化感受态细胞大肠杆菌DH5α (即构建组织中病原菌rpoB文库), 最后每个样品挑取10个阳性克隆进行测序和分析。
其次, 使用PCR-DGGE技术检测鲟组织样品中的分枝杆菌多样性。因为通过构建患病鲟组织中细菌rpoB文库, 然后进行测序, 虽然可以得到较长的rpoB序列, 但是仍然有可能筛选不到病鱼组织中丰度较低的分枝杆菌。而PCR-DGGE方法非常适合用于检测组织中分枝杆菌的多样性。以组织中细菌基因组 DNA为模板, 以 F357-GC (F357-GC: 引物F357的5′ 端加上GC夹子5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGG-3′)和Myco-600R为引物进行PCR扩增: 94℃预变性4min, 然后94℃变性 0.5min, 56℃退火 0.5min, 每个循环退火温度降低0.5℃, 72℃延伸1min, 共10个循环。接着94℃变性0.5min, 51℃退火0.5min, 72℃延伸1min,共25个循环, 最后72℃终延伸8min。PCR-DGGE 产物进行变性梯度凝胶电泳[19]。然后切下所有条带, 将含有目的条带的凝胶加入 40 µL TE缓冲液并捣碎, 4℃放置过夜, 以上清液中的DNA作为模板, 进行再次PCR扩增(引物为F357和Myco600R)[20](表1), PCR产物如上所述连接到pMD18-T载体, 转化感受态细胞大肠杆菌DH5α, 最后挑取阳性克隆测序分析。
最后, 对患病鲟肝脏、脾脏组织病理进行切片检测。病鱼的组织抗酸染色是进一步证实鲟的病原菌是否为分枝杆菌的又一重要手段。通过将新鲜的肝脏、脾脏组织样品进行 10%的福尔马林固定, 然后进行HE染色和Ziehl–Neelsen染色观察组织病理切片[21]。将新鲜的肝脏组织于预冷的2.5%戊二醛中固定, 然后制备透射电镜样品, 最后观察和分析。
1.8 分离菌株对斑马鱼的致病性
斑马鱼被认为是研究分枝杆菌致病机理的模式动物之一[22]。为了获得不同种分离菌株的致病力,选取13株代表菌株进行斑马鱼的人工感染试验, 即CST-8.2b、CST-8.29、CST-9.1、ASCy-1.0、ASCw-1.3、HA-1、ASCr-1.1、ASCr-1.5、ASCr-1.6、ASCw-1.7、ASCr-1.41、ASCr-1.42和ASCw-1.43菌株。细菌在7H10+OADC琼脂培养基上30°C培养, 在无菌PBS中悬浮和稀释, 然后离心收集菌体, 再用无菌 PBS洗涤一次, 最后计算出菌液浓度(通过测定OD600)。腹腔注射成年斑马鱼, 每尾注射50 μL细菌悬浮液,每组注射16尾健康的斑马鱼(体长约3.5 cm), 对照组斑马鱼每尾人工腹腔注射50 μL无菌PBS。感染组和对照组均分别在相同的条件下饲养, 即每组分别在25℃的水温条件下饲养(装有18 L水的箱子)。每天早晚定期投喂, 在投喂后每天换新鲜水约 5 L,并做好相关记录, 连续观察 100d。当斑马鱼表现出发病症状时(垂死), 立即进行解剖和病原菌分离,当连续观察到 100d时未发病的斑马鱼也进行病原菌分离。分离菌株的鉴定方法如上所述。
2 结果
2.1 鲟分枝杆菌病的典型病理特征
患病鲟的典型症状表现体表有斑点状溃烂(尤其在腹部和鳃盖), 腹部肿大, 腹部有大量的腹水,解剖发现肝脏有乳白色或浅红色斑点, 脾脏有淤血斑。组织切片观察发现患病鲟的肝脏有典型的肉芽肿, 而且Ziehl–Neelsen染色为阳性(图1)。透射电镜观察发现患病鲟的肝脏和脾脏中均有杆状细菌, 在肝脏中呈现聚集状, 而且电镜观察的结果也表明患病鲟组织中没有病毒颗粒(图1)。以上特征符合鱼类分枝杆菌病的相关特征。
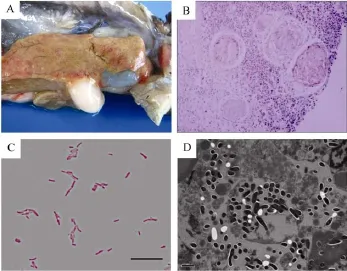
图1 患病鲟的肝脏病理特征和病原菌的显微观察Fig. 1 Pathological features of diseased Chinese sturgeon and microscopic-observation of the pathogens
2.2 分离菌株的生理生化特征
从患病鲟中分离到49株病原菌, 部分生理生化试验的结果显示分离菌株可分为 7种分枝杆菌(表2)。所有的病原菌均表现出分枝杆菌属的细菌生理生化特征: 需氧、不运动、生长缓慢、Ziehl–Neelsen染色阳性和某些分枝杆菌产生色素等。此外, 按其生长速率可分为速生型分枝杆菌(RGM)和缓慢生长型分枝杆菌(SGM), 按其有无色素的产生又可分为产色分枝杆菌和不产色分枝杆菌(表 2)。我们从 49株分离菌株中选取27株作为代表菌株, 即在每尾患病鲟分离到的病原菌中, 同种病原菌选取一个菌株作为代表菌株进一步研究。
菌株CSTy-8.29、ASCy-1.0、CSTy-9.1、ASCy-1.5、HA-1、ASCr-1.1、ASCr-1.2、ASCr-1.3、ASCr-1.41、ASCr-1.42、ASCr-1.5和 ASCr-1.6属于缓慢生长型分枝杆菌(需要生长7d以上才能观察到明显的菌落),且产生色素。菌株CSTy-8.29、ASCy-1.0、CSTy-9.1和ASCy-1.5在Lowenstein-Jensen培养基上生长(无光培养条件下)形成的菌落无色素, 当转移到光照条件下培养时则产生黄色色素。菌株ASCr-1.2、ASCr-1.3、ASCr-1.41、ASCr-1.42、ASCr-1.5和ASCr-1.6在黑暗条件下培养则产生橙黄色色素。其他分离菌株则无论光照培养还是黑暗培养条件下均不产生色素。菌株CST-7.3、CST-7.10、CST-8.2a、CST-8.2b、CST-8.10、CST-8.17、CST-8.29、CST-8.30、CST-8.31、ASCw-1.43、ASCw-1.6、ASCw-1.3和CST-9.1为速生型分枝杆菌, 菌株 ASCw-1.2 和 ASCw-1.7为缓慢生长型分枝杆菌。值得注意的是以上这些菌株中速生型分枝杆菌能够在 BHIA平板上生长, 缓慢生长型分枝杆菌则不能或生长不良(难形成可见的菌落)。因此, 在使用 BHIA平板分离分枝杆菌时, 通常只能够分离某些快速生长型分枝杆菌。
2.3 分离菌株的分子生物学鉴定
分离菌株经分枝杆菌属特异引物检测均为阳性。通过对分离菌株的3个保守基因16S rRNA、rpoB和 hsp65的测序和分析, 然后构建系统发育树。分离菌株的 16S rRNA系统发育树的结果表明: 分离菌株CST-7.3、CST-7.10、CST-8.2a、CST-8.2b、CST-8.10、CST-8.17、CST-8.29、CST-8.30、CST-8.31、ASCw-1.43和 ASCw-1.6属于龟分枝杆菌或脓肿分枝杆菌(Mycobacterium abscessus); 分离菌株 CSTy-8.29, CSTy-9.1, ASCy-1.0和ASCy-1.5属于海分枝杆菌或溃疡分枝杆菌(Mycobacterium ulcerans); 分离菌株HA-1、ASCr-1.1、ASCr-1.2、ASCr-1.3和ASCr-1.42属于戈登分枝杆菌; 分离菌株ASCr-1.41、ASCr-1.5和ASCr-1.6属于苏尔加分枝杆菌; 分离菌株ASCw-1.2和ASCw-1.7属于M. arupense; 分离菌株ASCw-1.3属于偶发分枝杆菌; 分离菌株 CST-9.1 属于猪分枝杆菌(Mycobacterium porcinum) (图2A)。
通过 16S rRNA系统发育树的分析, 结果发现海分枝杆菌和溃疡分枝杆菌不易区分。海分枝杆菌和溃疡分枝杆菌的基因组比较研究发现它们有 4000多个共同的基因, 它们的基因组相似度高达98.3%[28]。然而, 研究发现海分枝杆菌和溃疡分枝杆菌 16S rRNA的3′ 端序列存在变异区, 这一差异常常被用于作为区分它们的一种标准[29]。序列比对发现分离菌株CSTy-8.29、CSTy-9.1、ASCy-1.0和ASCy-1.5的16S rRNA序列在1249 bp位点处同海分枝杆菌的相同, 即为“A”碱基; 在1450—1452 bp位点处缺失“CTTT”四个碱基, 这与海分枝杆菌的16S rRNA序列相同。所以, 分离菌株 CSTy-8.29、CSTy-9.1、ASCy-1.0和ASCy-1.5属于海分枝杆菌。

表2 分枝杆菌分离菌株的几种关键生化特征Tab. 2 Key biochemical characteristics of Mycobacterium strains isolated from the diseased sturgeons
为了对分离菌株更为准确的鉴定, 又构建了rpoB (图2B)和hsp65 (图2C)的系统发育树。通过分析这两个基因的系统发育树, 结果表明分离菌株CST-7.3、CST-7.10、CST-8.2a、CST-8.2b、CST-8.10、CST-8.17、CST-8.29、CST-8.30、CST-8.31、ASCw-1.43和 ASCw-1.6属于龟分枝杆菌, 而其他分离菌株的鉴定结果表明同以上的鉴定结果相同。分离菌株的生理生化测定结果同样支持分子生物学鉴定的结果,这表明从患病鲟中分离到的分枝杆菌有 7种, 分别是: 海分枝杆菌、龟分枝杆菌、戈登分枝杆菌、苏尔加分枝杆菌、偶发分枝杆菌、猪分枝杆菌和 M. arupense (表3)。
2.4 鲟组织中分枝杆菌的分子生物学检测
鲟组织的分枝杆菌 rpoB文库测序结果表明除了014和016号鲟样品, 其他鲟组织样品中均检测到了与已分离的海分枝杆菌 rpoB完全相同的序列,而且该种序列在文库中所占测序的克隆数比例通常较高, 这表明海分枝杆菌存在于多个鲟组织样品中。而在样品010号中只发现与海分枝杆菌rpoB序列相同的rpoB序列, 这表明该鲟组织中可能只有一种海分枝杆菌。其他分离菌株的rpoB序列在相应的鲟组织样品的rpoB文库中也均被发现。这表明在部分鲟样品中, 虽然没有分离到海分枝杆菌, 但是在患病鲟组织中检测到了海分枝杆菌的存在。
PCR-DGGE电泳的条带显示检测样品中产生有1—3个电泳条带, 对每个电泳产生的每个条带进行切胶回收, 然后通过测序分析。测序结果表明除了样品014和样品016外所有鲟组织样品中均含有与海分枝杆菌16S rRNA序列相同的序列。并且在这些样品中也发现了相应分离菌株的16S rRNA序列。通过构建rpoB文库和PCR-DGGE方法的测序分析,结果表明所有已经分离的菌株在相应的组织中都能够检测到。虽然在一些鲟组织样品中未分离到海分枝杆菌, 但是可以检测到海分枝杆菌。rpoB文库和PCR-DGGE的结果也表明患病鲟为分枝杆菌复合感染, 而在复合感染中海分枝杆菌最为流行, 因为其存在于绝大多数样品中(表3)。
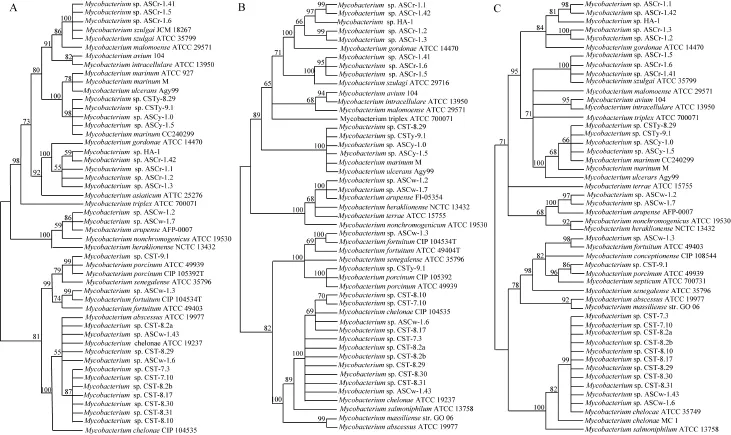
图2 患病鲟分离菌株与其他相关代表菌株的16S rRNA (A)、rpoB (B)和hsp65 (C)系统进化树分析(NJ法构建)Fig. 2 Phylogenetic trees based on sequences of 16S rRNA (A), rpoB (B) and hsp65 (C) constructed using the neighbor-joining method, which shows the relationships between isolates and other related species of Mycobacterium. Bootstrap was replicated 1000 times; only the values >50% are given at nodes
2.5 分枝杆菌对斑马鱼的致病性
选取 13株分枝杆菌为代表菌株对斑马鱼腹腔注射感染试验(表4), 结果表明海分枝杆菌的致死率最高, 当腹腔注射剂量为(5×105) CFU/尾时, 13d后死亡率为100%, 当注射剂量为(5×104) CFU/尾, 16d后死亡率为 100%; 当注射剂量降低为(5×103) CFU/尾时, 28d后死亡率为100%。注射海分枝杆菌10d后表现为皮肤溃烂、消瘦、腹部肿大, 而且能成功分离到海分枝杆菌。其他非海分枝杆菌对斑马鱼的致死率相对较低, 如在注射剂量为(5×105) CFU/尾时, 苏尔加分枝杆菌和M. arupens对斑马鱼的致死率为50%—62.5%。其他4种分枝杆菌(龟分枝杆菌、猪分枝杆菌、戈登分枝杆菌和偶发分枝杆菌)的致死率为0—37.5%, 明显低于海分枝杆菌的致死率。注射感染龟分枝杆菌、猪分枝杆菌、戈登分枝杆菌和偶发分枝杆菌斑马鱼在100d时没有明显的症状出现,但是注射的菌株都能够在100d后分离到(表4)。
3 讨论
鲟是我国重要的水产养殖品种之一, 具有十分重要的经济价值。然而, 关于鲟分枝杆菌病知之甚少, 更没有鲟分枝杆菌复合感染的相关报道。因此,对鲟分枝杆菌病的研究具有十分重要的意义。本文从19条患病鲟(中华鲟、史氏鲟和杂交鲟)中分离到了 49株分枝杆菌, 通过传统的培分离培养、Ziehl-Neelseen 染色、生理生化特征以及16S rRNA、rpoB和hsp65的亲缘关系进化树来确定病原菌的种属关系。鉴定结果表明在患病鲟中分离到海分枝杆菌、龟分枝杆菌、戈登分枝杆菌、苏尔加分枝杆菌、偶发分枝杆菌、猪分枝杆菌和M. arupense 7种分枝杆菌。
分析两年期间3个不同区域的患病鲟中分离到的分枝杆菌可知: 19条鲟中11条鲟分离出龟分枝杆菌(57.9%), 5条鲟分离出戈登分枝杆菌(26.3%), 4条鲟分离到海分枝杆菌(21.1%), 3条鲟分离出苏尔加分枝杆菌(15.8%), 2条鲟分离出M. arupense (10.5%), 1条鲟分离出偶发分枝杆菌(5%), 1条鲟分离出猪分枝杆菌(5%)。分离菌株中龟分枝杆菌的比例最高,而偶发分枝杆菌和猪分枝杆菌的比例最低(5%)。分离的结果表明在19条鲟中7条鲟为分枝杆菌复合感染(36.8%)。然而, 患病鲟组织中的病原菌分子生物学检测结果表明19条鲟中17条鲟(89.5%)检测到海分枝杆菌, 18条鲟(94.7%)为分枝杆菌复合感染。这与病原菌分离的结果不同, 海分枝杆菌的检出率明显高于分离的结果, 鲟复合感染的比例也明显提高。导致这一差异的原因在于部分样品中没有成功分离到海分枝杆菌却检测到海分枝杆菌, 这或许是因为在病原菌分离之前病鱼使用了某些抗生素(如恩诺沙星和氟苯尼考)所致, 或者因为其他未知原因导致海分枝杆菌培养为阴性。因此, 这提示我们在诊断鱼类分枝杆菌疾病时, 尤其是缓慢生长型的分枝杆菌, 不仅要注重病原菌的分离培养, 分子生物学诊断也十分重要。通过鲟分枝杆菌病的诊断和分析, 结果表明海分枝杆菌最为流行, 其次是龟分枝杆菌, 这一结果不同于同 Zanoni, et al.[30]的研究结果, 他们在观赏鱼分枝杆菌病的调查中发现龟分枝杆菌和偶发分枝杆菌比海分枝杆菌更加流行, 这或许是因为不同宿主的原因所致。
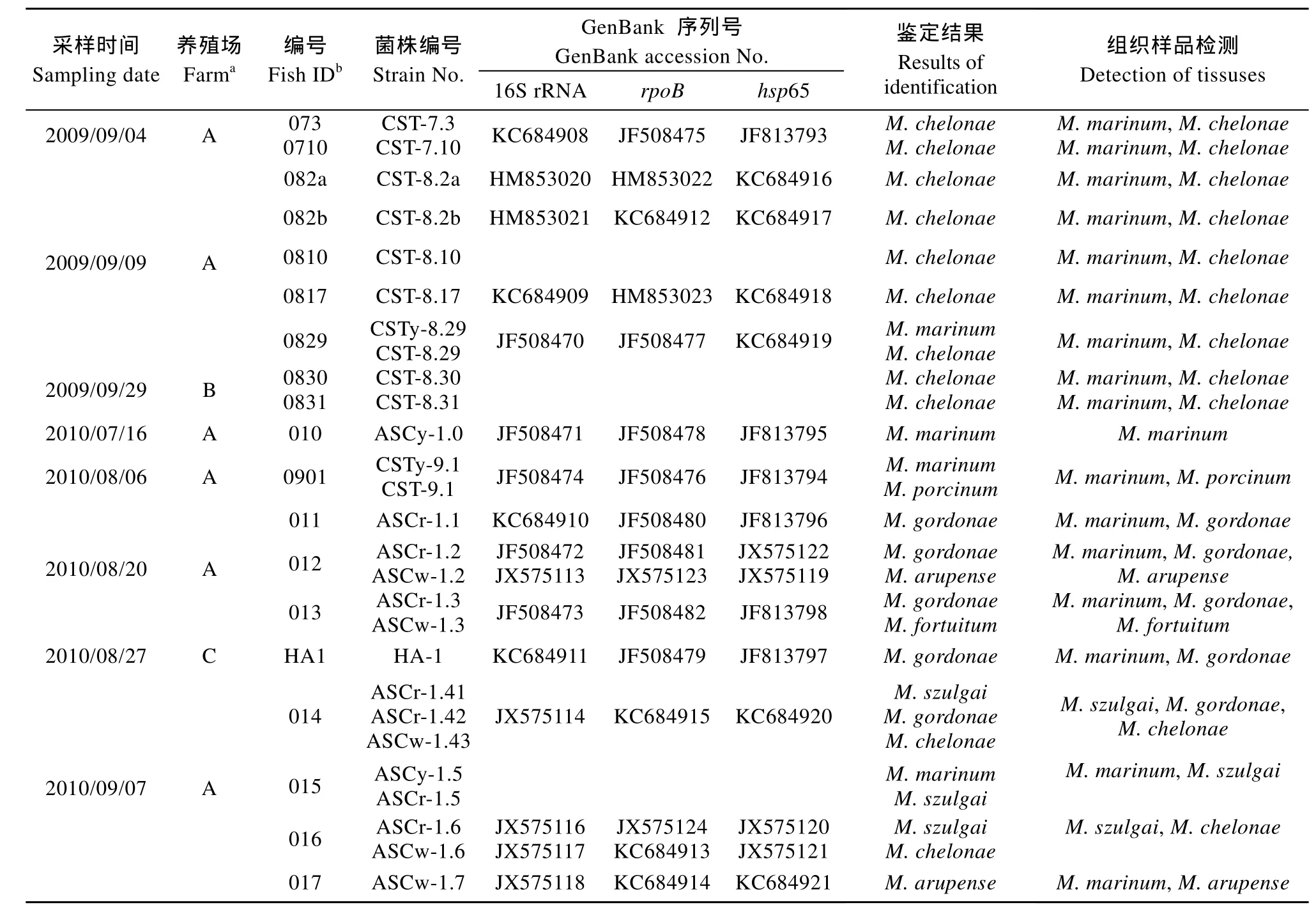
表3 分枝杆菌代表菌株信息Tab. 3 Details of the representative mycobacterial isolates collected in this study
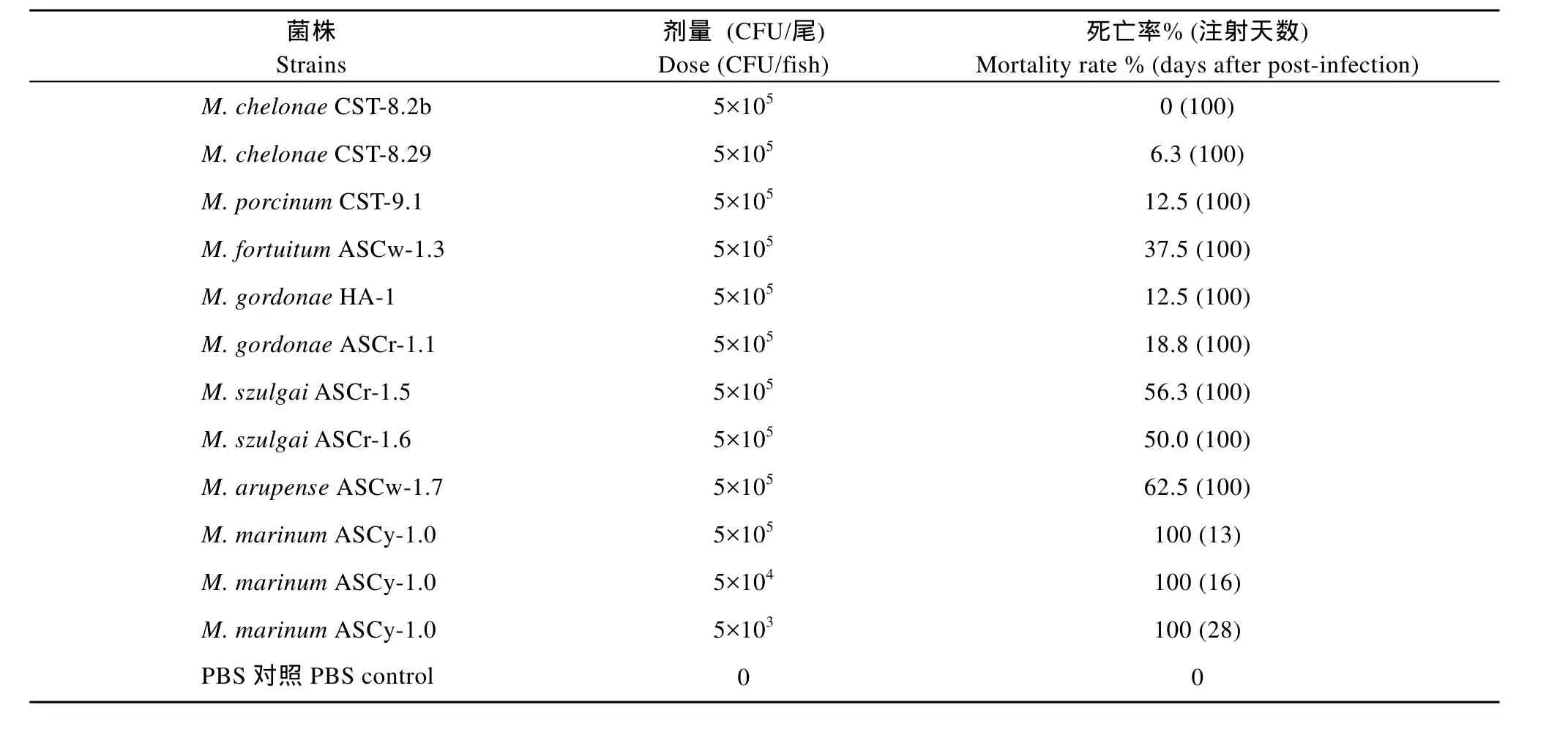
表4 分离菌株对斑马鱼的攻毒试验结果Tab. 4 Virulence assay of the representative Mycobacterium isolates against zebrafish intraperitoneally injected (N=16 for each group)
鲟可以被多种分枝杆菌感染。分离培养和分子生物学诊断结果均表明患病鲟存在分枝杆菌复合感染, 而且分子生物学诊断的结果显示分枝杆菌复合感染是患病鲟主要感染方式, 这种现象在鱼类分枝杆菌病中尚未见报道。虽然在鱼类分枝杆菌病中,分枝杆菌的复合感染也时常被报道, 如Cacatoo Dwarf Cichlid[31]、凤尾短鲷(Apistogramma cacatuoides)[32]、珍珠马甲(Trichogaster leeri)[30]、观赏鱼[9]等鱼类中均发现分枝杆菌复合感染, 但是, 令人惊奇的是本研究结果表明鲟分枝杆菌复合感染具有非常高的比例(94.7%)。这提示我们今后在鲟分枝杆菌病的诊断和防治时应注意分枝杆菌的复合感染情况。
目前, 鱼类分枝杆菌对斑马鱼的致病性研究很少有报道, 而且主要集中在海分枝杆菌对斑马鱼的致病力研究。本研究分离菌株对斑马鱼的攻毒试验结果表明, 海分枝杆菌的致病力最强(100%), 苏尔加分枝杆菌和 M. arupense对斑马鱼的毒力次之(50%— 62.5%), 偶发分枝杆菌、戈登分枝杆菌、猪分枝杆菌和龟分枝杆菌对斑马鱼的毒力最弱(0—37.5%)。这与Watral, et al.[33]的研究结果相似,即认为RGM是能够造成有长期的、亚致死的感染。值得注意的是, 本研究中发现复合感染的比例非常高, 这或许表明多种分枝杆菌共同感染或许可以提高它们的致病力, 因为我们在研究中也发现虽然单一非海分枝杆菌对斑马鱼的致病力不高, 而复合感染的致病力则会大大提高(结果未显示)。这一现象在一定程度上解释了为何在鲟中发现了如此高比例的分枝杆菌复合感染。在鲟分枝杆菌病中, 海分枝杆菌不仅检出的比例最高, 而且毒力最强, 因此,海分枝杆菌可能是鲟分枝杆菌复合感染过程中主要的致病菌。
鱼类中的分枝杆菌对人有一定的致病性。例如, Feng, et al.[34]在江苏省海安县发现有渔民感染海分枝杆菌。Slany, et al.[35]也发现从事渔业的人员感染海分枝杆菌, 尤其是导致皮肤感染。猪分枝杆菌常导致猪的下颌下淋巴炎, 并且能够在人和水环境中分离到[36,37]。Mycobacterium arupense多次在人的临床样品中被分离到[38—40]。有趣的是, 本研究在鲟中也分离到猪分枝杆菌和M. arupense。这提示我们鱼源分枝杆菌对人类的健康也存在威胁。因而, 深入研究鲟分枝杆菌病对鲟的健康养殖具有十分重要的意义。
[1] Nigrelli R F, Vogel H. Spontaneous tuberculosis in fishes and in other cold-blooded vertebrates with special reference to Mycobacterium fortuitum Cruz from fish and human lesions [J]. Zoologica, 1963, 48(9): 131—143
[2] Gauthier D T, Rhodes M W. Mycobacteriosis in fishes: a review [J]. The Veterinary Journal, 2009, 180(1): 33—47
[3] Falkinham J O. Nontuberculous mycobacteria in the environment [J]. Clinics in Chest Medicine, 2002, 23(3): 529—551
[4] Falkinham J O. Surrounded by mycobacteria: nontuberculous mycobacteria in the human environment [J]. Journal of Applied Microbiology, 2009, 107(2): 356—367
[5] Novotny L, Dvorska L, Lorencova A, et al. Fish: a potential source of bacterial pathogens for human beings [J]. Veterinarni Medicina, 2004, 49(9): 343—358
[6] Ross A J, Brancato F P. Mycobacterium fortuitum Cruz from the tropical fish Hyphessobrycon innesi [J]. Journal of Bacteriology, 1959, 78(3): 392—395
[7] Gauthier D T, Rhodes M W, Vogelbein W K, et al. Experimental mycobacteriosis in striped bass Morone saxatilis [J]. Diseases of Aquatic Organisms, 2003, 54(2): 105—117
[8] Abalain-Colloc M L, Guillerm D, Salun M, et al. Mycobacterium szulgai isolated from a patient, a tropical fish and aquarium water [J]. European Journal of ClinicalMicrobiology and Infectious Diseases, 2003, 22(12): 768—769
[9] Pate M, Jencic V, Zolnir-Dovc M, et al. Detection of mycobacteria in aquarium fish in Slovenia by culture and molecular methods [J]. Diseases of Aquatic Organisms, 2005, 64(1): 29—35
[10] Stragier P, Hermans K, Stinear T, et al. First report of a mycolactone-producing Mycobacterium infection in fish agriculture in Belgium [J]. FEMS Microbiology Letters, 2008, 286(1): 93—95
[11] Wei Q, He J, Yang D, et al. Status of sturgeon aquaculture and sturgeon trade in China: a review based on two recent nationwide surveys [J]. Journal of Applied Ichthyology, 2004, 20: 321—332
[12] Li B R, Zou Y, Wei Q. Sturgeon aquaculture in China: status of current difficulties as well as future strategies based on 2002-2006/2007 surveys in eleven provinces [J]. Journal of Applied Ichthyology, 2009, 25(6): 632—639
[13] Yang W, Li A. Isolation and characterization of Streptococcus dysgagalactiae from diseased Acipenser schrenckii [J]. Aquaculture, 2009, 294: 14—17
[14] Yang Y B, Xia Y T, Zheng W D, et al. Isolation and identification of Yersinia ruckeri from Acipenser baerii and its antibiotic sensitivity [J]. Acta Hydrobiology Sinica, 2013, 37(2): 393—398 [杨移斌, 夏永涛, 郑卫东, 等. 鲟源鲁氏耶尔森菌的分离鉴定及药敏特性研究. 水生生物学报, 2013, 37(2): 393—398]
[15] Talaat A M, Reimschuessel R, Trucksis M. Identification of mycobacteria infecting fish to the species level using polymerase chain reaction and restriction enzyme analysis [J]. Veterinary Microbiology, 1997, 58(2-4): 229—237
[16] Milinovich G J, Burrell P C, Pollitt C C, et al. Streptococcus henryi sp. nov. and Streptococcus caballi sp. nov., isolated from the hindgut of horses with oligofructose-induced laminitis [J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(Pt1): 262—266
[17] Adékambi T, Colson P, Drancourt M. rpoB-based identification of nonpigmented and late-pigmenting rapidly growing mycobacteria [J]. Journal of Clinical Microbiology, 2003, 41(12): 5699—5708
[18] Kim H, Kim S H, Shim T S, et al. Differentiation of Mycobacterium species by analysis of the heat-shock protein 65 gene (hsp65) [J]. International Journal of Systermatic and Evolutionary Microbiology, 2005, 55(Pt4): 1649—1656
[19] Florez A B, Mayo B. Microbial diversity and succession during the manufacture and ripening of traditional, Spanish, blue-veined Cabrales cheese, as determined by PCR-DGGE [J]. International Journal of Food Microbiology, 2006, 110(2): 165—171
[20] Leys NM, Ryngaert A, Bastiaens L, et al. Occurrence and community composition of fast-growing Mycobacterium in soils contaminated with polycyclic aromatic hydrocarbons [J]. FEMS Microbiology Ecology, 2005, 51(3): 375—388
[21] Allen J L. A modified Ziehl-Neelsen stain for mycobacteria [J]. Medical Laboratory Science, 1992, 49(2): 99—102
[22] Kanther M, Rawls J F. Host-microbe interactions in the developing zebrafish [J]. Current Opinion in Immunology, 2010, 22(1): 10—19
[23] Najiah M, Lee K L, Noorasikin H, et al. Phenotypic and genotypic characteristics of Mycobacterium isolates from fighting fish Betta spp. in Malaysia [J]. Research in Veterinary Science, 2011, 91(3): 342—345
[24] Mun H S, Park J H, Kim H, et al. Mycobacterium senuense sp. nov., a slowly growing, non-chromogenic species closely related to the Mycobacterium terrae complex [J]. International Journal of Systermatic and Evolutionary Microbiology, 2008, 58(Pt 3): 641—646
[25] Adékambi T, Stein A, Carvajal J, et al. Description of Mycobacterium conceptionense sp. nov., a Mycobacterium fortuitum group organism isolated from a posttraumatic osteitis inflammation [J]. Journal of Clinical Microbiology, 2006, 44(4): 1268—1273
[26] Fanti F, Tortoli E, Hall L, et al. Mycobacterium parmense sp. nov. [J]. International Journal of Systermatic and Evolutionary Microbiology, 2004, 54(Pt 4): 1123—1127
[27] Cloud J L, Meyer J J, Pounder J I, et al. Mycobacterium arupense sp. nov., a non-chromogenic bacterium isolated from clinical specimens [J]. International Journal of Systermatic and Evolutionary Microbiology, 2006, 56(Pt6): 1413—1418
[28] Pidot S J, Asiedu K, Kaser M, et al. Mycobacterium ulcerans and other mycolactone-producing mycobacteria should be considered a single species [J]. PloS Neglected Tropical Diseases, 2010, 4(7): e663. doi: 10.1371/journal.pntd.0000663
[29] Portaels F, Fonteyne P A, de Beenhouwer H, et al. Variability in 3′end of 16S rRNA sequence of Mycobacterium ulcerans is related to geographic origin of isolates [J]. Journal of Clinical Microbiology, 1996, 34(4): 962—965
[30] Zanoni R G, Florio D, Fioravanti ML, et al. Occurrence of Mycobacterium spp. in ornamental fish in Italy [J]. Journal of Fish Diseases, 2008, 31(6): 433—441
[31] Lescenko P, Matlova L, Dvorska L, et al. Mycobacterial infection in aquarium fish [J]. Veterinarni Medicina, 2003, 48(3): 71—78
[32] Novotny L, Halouzka R, Matlova L, et al. Morphology and distribution of granulomatous inflammation in freshwater ornamental fish infected with mycobacteria [J]. Journal of Fish Diseases, 2010, 33(12): 947—955
[33] Watral V, Kent M L. Pathogenesis of Mycobacterium spp. in zebrafish (Danio rerio) from research facilities [J]. Comparative Biochemistry and Physiology Part C, 2007, 145(1): 55—60
[34] Feng Y, Xu H, Wang H, et al. Outbreak of a cutanuousMycobacterium marinum infection in Jiangsu Haian, China [J]. Diagnostic Microbiology and Infectious Diseases, 2011, 71(3): 267—272
[35] Slany M, Jezek P, Fiserova V, et al. Mycobacterium marinum infections in humans and tracing of its possible environmental sources [J]. Canadian Journal of Microbiology, 2012, 58(1): 39—44
[36] Taddei R, Barbieri I, Pacciarini M L, et al. Mycobacterium porcinum strains isolated from bovine bulk milk: Implications for Mycobacterium avium subsp. paratuberculosis detection by PCR and culture [J]. Veterinary Microbiology, 2008, 130(3—4): 338—347
[37] Shojaei H, Heidarieh P, Hashemi A, et al. Species identification of neglected nontuberculous mycobacteria in a developing country [J]. Japanese Journal of Infection diseases, 2011, 64(4): 256—271
[38] Masaki T, Ohkusu K, Hata H, et al. Mycobacterium kumamotonense sp. nov. recovered from clinical specimen and the first isolation report of Mycobacterium arupense in Japan: Novel slowly growing, nonchromogenic clinical isolates related to Mycobacterium terrae complex [J]. Microbiology and Immunology, 2006, 50(11): 889—897
[39] Neonakis I K, Gitti Z, Kontos F, et al. Mycobacterium arupense pulmonary infection: antibiotic resistance and restriction fragment length polymorphism analysis [J]. Indian Journal of Medical Microbiology, 2010, 28(2): 173—176
[40] Slany M, Svobodova J, Ettlova A, et al. Mycobacterium arupense among the isolates of non-tuberculous mycobacteria from human, animal and environmental samples [J]. Veterinarni Medicina, 2010, 55(8): 369—376
RESEARCH ON THE MYCOBACTERIOSIS AND ITS PATHOGEN IN STURGEONS
ZHANG De-Feng1,2, LI Ai-Hua1and GONG Xiao-Ning1
(1. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
Mycobacteriosis in fish is a common chronic progressive disease spread worldwide. Several outbreaks of mycobacteriosis in reared sturgeons occurred in China in year 2009 and 2010, which affected Chinese sturgeon (Acipenser sinensis), Amur sturgeon (Acipenser schrencki) and hybrid sturgeon (Acipenser baeri-Acipenser gueldenstaedtii). Mycobacteria were identified as the pathongens of this disease based on a variety of observation, including the identification of the isolates, the detection of pathogens in tissues, the clinical signs and the histopathological examinations. Forty-nine isolates of nontuberculous mycobacteria were extracted from 19 infected sturgeons. We identified seven species of Mycobacterium from these isolates, namely, Mycobacterium chelonae, Mycobacterium marinum, Mycobacterium gordonae, Mycobacterium fortuitum, Mycobacterium szulgai, Mycobacterium porcinum, and Mycobacterium arupense. We found that two or three mycobacterial species co-existed in the same tissues. Our results also showed that M. marinum was more prevalent than other mycobacterial species. Virulence assay revealed that M. marinum showed the highest pathogenicity to zebrafish. These results suggested that M. marinum should be the major pathogenic bacteria in sturgeon mycobacteriosis, and that the mixed mycobacterial infection be the predominant form of sturgeon mycobacteriosis. To our best knowledge, this is the first report about the mycobacteriosis in farmed Chinese sturgeons and Amur sturgeons, and is the first report about the isolation of M. porcinum and M. arupense from the infected fish.
Sturgeon; Mycobacterium; Identification; Phylogenetic analysis; Mixed infections
S941
A
1000-3207(2014)03-0495-10
10.7541/2014.70
2013-04-11;
2014-01-21
国家自然科学基金项目(No. 30670112; No. 31070112)资助
张德锋(1985—), 男, 安徽阜阳人; 在读博士; 研究方向为鱼类病原微生物学。E-mail: zhangdefeng08@126.com
李爱华(1963—), 男, 研究员; E-mail: liaihua@ihb.ac.cn
