乙醇消耗菌的分离鉴定及其对基因工程集胞藻乙醇产量的影响
2014-03-29葛平平陈林张维董庆喆刘天中吕雪峰
葛平平陈 林张 维董庆喆刘天中吕雪峰
(1. 中国科学院青岛生物能源与过程研究所, 生物燃料重点实验室, 青岛 266101; 2. 中国科学院大学, 北京 100049)
乙醇消耗菌的分离鉴定及其对基因工程集胞藻乙醇产量的影响
葛平平1,2陈 林1张 维1董庆喆1刘天中1吕雪峰1
(1. 中国科学院青岛生物能源与过程研究所, 生物燃料重点实验室, 青岛 266101; 2. 中国科学院大学, 北京 100049)
为研究产乙醇基因工程集胞藻培养液中的乙醇消耗菌污染情况及其对乙醇产量的影响, 从污染的培养液中分离出4株乙醇消耗菌, 通过16S rDNA、26S rDNA序列分析对分离出的菌株进行鉴定, 并研究其乙醇消耗能力及对基因工程集胞藻乙醇产量的影响。结果表明, 分离出的 4株菌分别为红酵母(Rhodotorula sp.)、季也蒙酵母(Meyerozyma guilliermondii)、短波单胞菌(Brevundimonas sp.)、微杆菌(Microbacterium sp.)。其中乙醇消耗能力最强的是红酵母, 乙醇比消耗速率达到 391 g/(1015cfu⋅d); 其次为季也蒙酵母, 乙醇比消耗速率为80.1 g/(1015cfu⋅d); 短波单胞菌和微杆菌的乙醇比消耗速率远低于红酵母和季也蒙酵母。将分离出的菌株与产乙醇集胞藻共培养7d后, 污染红酵母、季也蒙酵母、短波单胞菌、微杆菌的实验组乙醇产量分别下降了 53.8%、23.6%、40.7%、27.3%。4株菌对基因工程集胞藻的生长无明显影响, 均通过直接消耗乙醇而降低集胞藻的乙醇产量。
乙醇消耗菌; 微生物污染; 生物乙醇; 基因工程集胞藻
生物乙醇是重要的生物液体燃料之一, 可作为汽油替代品或可再生交通燃料添加剂[1]。应用基因工程蓝藻直接利用太阳能和CO2一步法生物合成乙醇具有独特的优势, 因此成为各界关注的热点[2—5]。目前, 用于生产乙醇的基因工程蓝藻主要有聚球藻(Synechococcus sp. PCC 7942)[4]和集胞藻(Synechocystis sp. PCC 6803)[3,5]等, 最高乙醇产量达到5.5 g/L[5], 显示出太阳能生物转化直接合成乙醇的巨大潜力。然而, 有研究表明基因工程集胞藻的乙醇产量很不稳定, 在培养后期培养液中的乙醇浓度急剧下降[3,5], 因此研究确定影响乙醇产量稳定性的因素对生产工艺的建立有重要意义。
在我们的前期工作中发现, 基因工程集胞藻在培养过程中易发生食藻生物和微生物污染, 尤其是在放大培养阶段这种污染尤为严重。食藻生物会吞食藻细胞, 使生物量迅速下降, 从而降低乙醇产量。自然界中存在着多种能利用乙醇的微生物, 例如醋酸菌、面包酵母、嗜有机甲基杆菌、枯草芽孢杆菌、巴氏醋杆菌、门多萨假单胞菌等都可代谢乙醇[6—11],其中巴氏醋杆菌的乙醇消耗速率达到2.3 mL/d。而在管囊酵母[12]、酿酒酵母[13]中, 乙醇的合成代谢和分解代谢同时存在。如果污染了这些微生物, 也可能严重影响乙醇产量。目前, 关于微生物污染对基因工程集胞藻乙醇产量的影响还未有报道。
本研究从污染的产乙醇基因工程集胞藻的培养液中分离出 4株乙醇消耗菌, 评价各菌的乙醇消耗能力以及对基因工程集胞藻的生长和乙醇产量的影响, 并采用形态观察和基因序列分析手段鉴定污染菌的种类, 以期能为蓝藻乙醇的培养工艺及污染控制提供理论参考。
1 材料与方法
1.1 藻种及培养方法
产乙醇基因工程集胞藻 Synechocystis sp. PCC 6803 Syn-ZG25, 由中国科学院青岛生物能源与过程研究所构建[5]。集胞藻在250 mL三角摇瓶(装液量100 mL)或直径30 mm的气泡柱式光反应器(装液量200 mL)中培养, 通入含5% CO2的空气混合气,通气速率(10±2) L/h, 光强100 μmol/(m2⋅s), 培养温度(30±2) ℃。培养基为 BG11培养基, 其中含20 mg/L壮观霉素(Spectinomycin, Sigma)[14]。
1.2 乙醇消耗菌的分离纯化
培养基 (1)初筛培养基: 牛肉膏3 g/L, 蛋白胨10 g/L, NaCl 5 g/L, 乙醇2 g/L, 壮观霉素20 mg/L,琼脂 15 g/L; (2)复筛培养基: BG11培养基中添加2 g/L的乙醇, 20 mg/L的壮观霉素, 15 g/L的琼脂; (3)纯化培养基: 牛肉膏 3 g/L, 蛋白胨10 g/L, NaCl 5 g/L, 乙醇 2 g/L, 琼脂 15 g/L; (4)发酵培养基: 牛肉膏 3 g/L, 蛋白胨10 g/L, NaCl 5 g/L, 乙醇 2.3 g/L,壮观霉素 20 mg/L。
乙醇消耗菌的分离及纯化 (1)初筛: 集胞藻在气泡柱式反应器中通气培养 12—14d后, 经无菌检测为阳性, 作为染菌藻液。取染菌的Syn-ZG25藻液,经梯度稀释后涂布于初筛培养基平板上, 置于 30℃生化培养箱中培养。待平板上有单菌落长出后, 依据菌落形态和显微观察结果, 分别挑取多株菌落形态和显微形态相同的单菌落培养。(2)复筛: 将初筛得到的各菌株分别在复筛培养基平板上划线或涂布, 平板放置于 30℃生化培养箱培养 7—10d,长出的菌株在纯化培养基平板上经多次传代后作为复筛菌株。
1.3 乙醇消耗菌的鉴定
菌落及个体形态观察 在纯化培养基平板上,利用目测法观察乙醇消耗菌的菌落形态。挑取纯化后的单菌落接种于发酵培养基中, 培养 24h后取适量菌液低速离心, 收集菌体, 依次使用 2.5%戊二醛溶液和 1%锇酸固定, 经乙醇梯度脱水后喷金处理,然后用冷场发射扫描电子显微镜(S-4800型, 日本Hitachi公司)进行个体形态观察。
乙醇消耗菌的16S rDNA及26S rDNA序列测定根据各菌的菌落和个体形态分析, 4种菌可能分属细菌和酵母两大类, 因此同时进行了16S rDNA及26S rDNA序列测定。引物设计: 分别选用16S rDNA通用引物对 27F(5′-AGAGTTTGATCCTGGCTCAG-3′)/ 1492R(5′-TACCTTGTTACGACTT-3′)以及26S rDNA通用引物对NL1(5′-GCATATCAATAAGCGGAGGA AAAG-3′)/NL4(5′-GGTCCGTGTTTCAAGACGG-3′)。PCR反应体系与条件: PCR反应采用50 μL体系, 以提取的基因组 DNA为模板, 用高保真 Taq酶进行PCR扩增。反应条件为: 95 ℃ 5min; 94 ℃ 30s, 55℃1min, 72 ℃ 1min, 34个循环; 72 ℃ 10min 。PCR产物用0.8%琼脂糖凝胶电泳进行检验。目的基因片段克隆: 将PCR产物用DNA纯化试剂盒(Cycle-Pure Kit,美国Omega公司)进行纯化, 产物与pMD18-T Vector连接后转化到E. coli DH5α感受态细胞中, 涂布到Amp抗性平板上培养9—16h。挑取3个单克隆,扩繁后送上海桑尼生物科技有限公司测序, 将测序结果与GenBank中已有序列进行比对和同源性分析。
1.4 乙醇消耗菌的生长及乙醇消耗速率测定
乙醇消耗菌生长曲线的测定 挑取纯化后的单菌落接种于发酵培养基中培养 24h, 取适量培养液在 660 nm下测定其吸光度(A660), 然后稀释至A660= 0.05, 按10%接种量接种至发酵培养基中, 于30℃、180 r/min摇床中培养, 定时取样测定菌液A660。每实验组设 2个平行, 对照组中接种等体积的无菌培养液。
菌液细胞浓度的确定 取生长至对数期的菌液,测定其 A660。梯度稀释后采用平板活菌计数法测定细胞浓度, 计数时仅选取菌落数在20—200的平板,每株菌每浓度做3个平行。用Origin拟合出菌液A660与细胞浓度D (Cell density, cfu/mL)的关系, 结果列入表1。
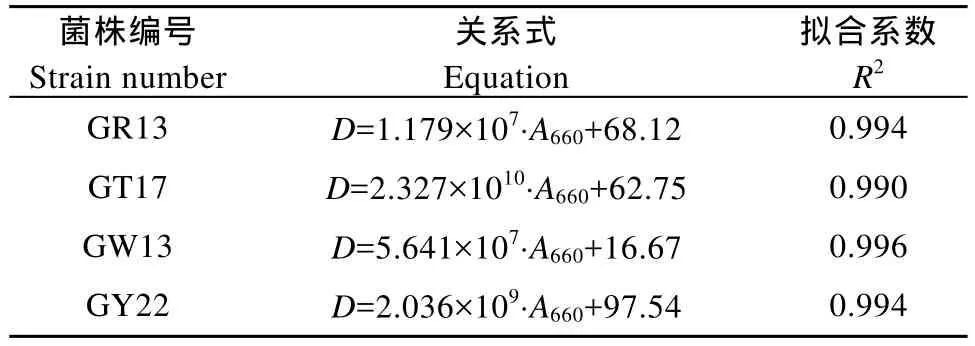
表1 各菌菌液A660与其细胞浓度D的关系Tab. 1 Relationship between A660and cell density
乙醇消耗速率的测定 将培养18—24h后的各菌接种到装有20 mL发酵培养基的锥形瓶中, 根据拟合得到的公式将培养液适当稀释, 使其初始细胞浓度维持在106cfu/mL, 将锥形瓶置于30℃、180 r/min摇床培养。以不接菌实验组为对照, 每组实验设 2个平行。每6h取样测定细胞浓度, 并用生物传感分析仪(SBA-40D, 山东省科学院生物研究所)测定培养液中乙醇浓度[14]。
乙醇的比消耗速率按公式(1)计算:
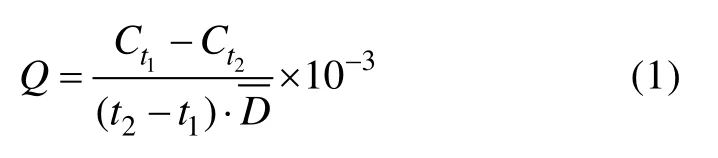
其中, Q为比消耗速率, 单位为g/(cfu⋅d); Ct1表示t1时刻的乙醇浓度, 单位为g/L;D为 t1到 t2时间段内的平均细胞浓度, 按照公式(2)计算:

1.5 乙醇消耗菌对基因工程集胞藻乙醇产量的影响
无菌藻种在三角瓶中培养至对数生长期后, 接种于气泡柱式光生物反应器(装液量200 mL)中, 每24h取样, 测定藻液的叶绿素a含量及乙醇浓度。自培养第 4天始, 向各实验组中分别接种稀释至一定细胞浓度的菌液2 mL, 使各乙醇消耗菌细胞终浓度为106cfu/mL, 对照组加入2 mL无菌水; 每组实验设2个平行。每24h取样, 测定藻液的叶绿素a含量及乙醇浓度。对照组及实验组每48h作无菌检测和采用平板活菌计数法测定菌的细胞密度。
1.6 基因工程集胞藻生长的测定
考虑到乙醇消耗菌在730 nm处也有光吸收, 因此以叶绿素a的含量计量集胞藻的生物量。取1 mL藻液, 离心收集藻细胞, 再加入1 mL甲醇重悬浸泡, 60℃水浴避光提取30min, 离心取上清液测定665 nm下的吸光度, 计算叶绿素a含量[15]。
1.7 统计分析
统计分析采用单因素方差分析(One-Way ANOVA)方法, 使用SPSS 11.0软件。
2 结果
2.1 乙醇消耗菌的分离及鉴定
从污染的基因工程集胞藻培养液中, 经过初筛、多次纯化及复筛, 得到 4株具有乙醇消耗能力的菌,分别编号为GR13、GW13、GT17、GY22。四株菌分别在30 ℃恒温箱中培养5d后的菌落特征列入表2。

表2 四株乙醇消耗菌的菌落特征Tab. 2 Colony characteristics of the four ethanol consuming microorganisms
扫描电镜观察显示(图1), GR13菌的细胞为球形或椭球形,细胞直径约2—4 μm; GW13菌的细胞呈卵球形, 长度约 1.5—3 μm, GT17菌的细胞呈杆状, 直径约为0.3 μm, 长度约1—2 μm。GY22菌的细胞呈短杆状, 直径约 0.3—0.4 μm, 长度约 0.8—1.2 μm。综合考虑四株菌的菌落和个体形态, 初步认为GR13和GW13属于酵母菌, GT17和GY22属于细菌。随后分别提取各菌的DNA, 并进行16S rDNA及26S rDNA序列测定, 将测得的序列在Genbank BLAST进行同源性比对(表3)。

图1 四株乙醇消耗菌的个体形态Fig. 1 The morphology of the four ethanol consuming microorganisms

表3 四株乙醇消耗菌的同源性比对结果Tab. 3 BLAST results of the four ethanol consuming microorganisms
从比对结果可以看出, 菌株GR13、GT17、GW13、GY22分别与黏质红酵母(Rhodotorula mucilaginosa)、缺陷短波单胞菌(Brevundimonas diminuta ATCC 11568)、季也蒙酵母(Meyerozyma guilliermondii strain KAML05)、人型微杆菌(Microbacterium hominis strain DSM 12509)同源性最高。
2.2 乙醇消耗菌的生长及乙醇消耗能力评价
在发酵培养基中, 测定四株乙醇消耗菌的生长速率(图2)。从图中可以看出, 菌株GW13在0—6h为迟滞期, 6—27h为对数生长期, 之后生长速度开始减缓并进入稳定生长期; 菌株 GR13的对数生长期能够持续更长时间, 至 42h后进入稳定生长期;而菌株GT17的对数生长期仅持续18h。菌株GY22在整个实验周期内一直匀速生长, 没有出现明显的稳定生长期。

图2 四株乙醇消耗菌的生长曲线Fig. 2 Growth curves of the four ethanol consuming microorganisms
4株菌的初始接种浓度均为 106cfu/mL, 在此条件下, 发酵培养基中乙醇浓度变化见图3。其中,对照组由于自然挥发的原因, 乙醇浓度略有下降。在扣除因自然挥发而消耗的乙醇后, 除了GY22外,其他三个实验组中的乙醇浓度均显著下降(P< 0.01)。其中乙醇消耗最快的为GW13菌株, 乙醇消耗速率达到2.05 g/(L⋅d); 其次为GR13、GT17, 消耗速率分别为1.7 g/(L⋅d)和0.725 g/(L⋅d); GY22菌株的乙醇消耗速率最低, 为0.025 g/(L⋅d)。目前文献报道的基因工程蓝藻的乙醇产率最高为 0.21 g/(L⋅d)[5],而本研究发现的三株乙醇消耗菌(GW13、GR13和GT17), 在本研究所设定的菌细胞浓度下, 其乙醇消耗速率均高于报道的最高乙醇产率, 都能将该浓度的乙醇消耗殆尽。即使是乙醇消耗速率最低的GY22菌株也将使乙醇产量下降约 8%。但是因为该菌株(GY22)在 48h后仍保持生长(图2), 其细胞浓度可能随时间延长而累积增加, 也可能消耗更多的乙醇。
图4显示菌液浓度的变化, 与图3对比可以发现, 乙醇消耗速率是与培养液中细菌浓度相关的。为了更清楚地研究各菌的乙醇消耗能力, 我们分别计算了四株菌的乙醇比消耗速率(图5)。从图中可以看出, GR13的乙醇消耗能力最强, 达到 391 g/(1015cfu⋅d); 其次为 GW13, 乙醇比消耗速率为80.1 g/(1015cfu⋅d); GT17、GY22的乙醇比消耗速率分别为0.44 g/(1015cfu⋅d)和0.43 g/(1015cfu⋅d), 远低于GR13和GW13两株菌。

图3 四株菌的乙醇消耗情况Fig. 3 Ethanol consuming condition of four microorganisms

图4 四株乙醇消耗菌在发酵液中的生长Fig. 4 Growth of four ethanol consuming microorganisms

图5 四株菌的乙醇比消耗速率Fig. 5 Specific ethanol consumption rates of the four microorganisms
2.3 四株乙醇消耗菌对基因工程集胞藻的生长及乙醇产量的影响
向无菌集胞藻培养液中分别加入不同的乙醇消耗菌, 使各菌的终浓度均为106cfu/mL。结果表明,各实验组在污染乙醇消耗菌后, 乙醇积累速率开始减缓, 而对照组的乙醇产量仍快速增长(图6)。在培养7d(接种乙醇消耗菌3d)后, 污染GR13菌的实验组乙醇浓度开始下降, 污染 GT17、GW13、GY22菌株的实验组均在第8天时开始下降。培养至第11天时, 污染GR13、GT17、GW13、GY22菌株的实验组乙醇浓度分别降至对照组的 46.2%、59.3%、76.4%和72.7%。此外, 由图6计算可知, 第11天时,污染GR13、GT17、GW13、GY22菌株的实验组中平均乙醇消耗速率分别为 0.051、0.038、0.022和0.026 g/(L⋅d)。与发酵培养基中的测定结果(图3)相比, GR13、GT17、GW13三个实验组的乙醇消耗速率均有所降低, 但仍然导致污染乙醇消耗菌的培养液中乙醇浓度明显下降。相比于发酵培养基, 集胞藻培养液中营养相对缺乏, 乙醇消耗菌的生长速度相对缓慢(第11天时, 菌细胞浓度约为 107cfu/mL,远低于发酵培养基中的浓度), 因此乙醇消耗速率有所降低。
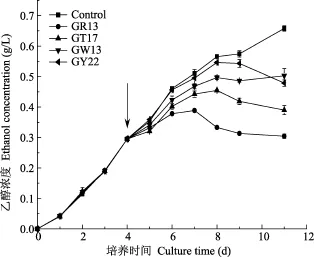
图6 各菌对集胞藻乙醇产量的影响Fig. 6 Effects of the four microorganisms on ethanol production of Synechocystis sp.
为考察污染微生物对集胞藻生长的影响, 以叶绿素a来计量培养过程中集胞藻生物量的变化。结果表明, 在污染乙醇消耗菌后, 实验组和对照组中集胞藻的生物量都保持同等速率的增长(图7), 各实验组间无明显差异(P>0.05)。由此可见, 在本实验条件下, 4株菌的存在虽然明显降低培养液中乙醇浓度(图6), 但对基因工程集胞藻的生长并无明显影响, 据此也能推测乙醇浓度的降低主要是由于污染的乙醇消耗菌对乙醇的直接消耗。

图7 四株乙醇消耗菌对集胞藻生长的影响Fig. 7 Effects of the four microorganisms on growth of Synechocystis sp.
3 讨论
基因工程集胞藻是在野生型集胞藻PCC 6803中转入表达丙酮酸脱羧酶和乙醇脱氢酶的基因, 构建出乙醇合成途径, 通过光合作用固定CO2直接合成乙醇。研究表明, 有很多种因素会影响乙醇合成途径中关键酶的活性, 从而影响藻细胞的乙醇产率。Gao, et al.的研究结果表明当培养基中存在较高浓度的Zn2+、Co2+等金属离子时, 乙醇脱氢酶的活性受抑制[5]。Lan, et al.发现富氧条件下相关酶的活性受抑制, 相应地, 藻细胞的产醇能力降低[16]。但在Dexer, et al.、Gao, et al.的研究中即使在上述抑制因素并不存在的情况下, 集胞藻培养液中的乙醇积累量仍然表现出快速下降的现象[3,5]。Gao, et al.认为因藻细胞的衰老而引起的乙醇产率降低是乙醇积累量迅速下降的主要原因[5]。而本研究发现即使在生长末期藻细胞不产生乙醇, 自然挥发也只是导致乙醇浓度略微下降(图3)。Dexer, et al.猜测是乙醇脱氢酶反向催化乙醇转变成乙醛, 但没有阐明导致代谢方向转变的原因[3]。
基因工程集胞藻的目的产物乙醇可通过自由扩散透过原生质膜, 直接分泌到胞外, 从而在培养液中积累一定浓度的乙醇。从产物回收的角度看, 从培养液中而非从藻细胞中回收目的产物相对较容易,成本也相应降低。而就培养本身而言, 乙醇的积累可能对藻细胞产生胁迫或导致培养液中滋生乙醇消耗菌。Kämäräinen, et al. 的研究证明培养液中乙醇浓度为4.5 g/L时, 可以对集胞藻PCC6803的生长产生明显抑制, 导致最高细胞浓度下降4.5%[17]。本研究发现基因工程集胞藻在培养过程中容易染菌, 并从中分离出4株能够代谢乙醇的菌(表2, 3)。图7的结果表明, 4株菌在BG11培养液中均表现出明显的乙醇消耗能力, GR13、GW13、GT17、GY22的乙醇消耗速率分别为0.051、0.022、0.038和0.026 g/(L⋅d), 在营养更丰富的发酵培养基中的乙醇消耗速率更高,分别达到1.7、2.05、0.725和0.025 g/(L⋅d) (图3)。在本研究中使用的基因工程藻株Synechocystis sp. PCC 6803 Syn-ZG25的乙醇产率为0.009—0.104 g/(L⋅d),与上述分离的菌株共培养7d后, 实验组的乙醇产量均显著低于无菌对照组(图6)。当培养液中无乙醇消耗菌时, 乙醇产量呈现增长趋势; 当培养液中污染少量乙醇消耗菌时, 乙醇积累速度减缓。随着培养液中乙醇消耗菌浓度的增加, 污染乙醇消耗速率较低的杂菌的实验组, 乙醇浓度不再增长; 污染乙醇消耗速率较快的杂菌的实验组, 乙醇浓度迅速下降。在本研究中菌株GR13的乙醇消耗能力最强, 其乙醇比消耗速率为391 g/(1015cfu⋅d)。目前报道的基因工程蓝藻的最高乙醇产量为0.21 g/(L⋅d)[5], 据此计算, 若要将菌株GR13对乙醇产量的影响控制在1%以内, 则其最高细胞浓度应控制在5.37×106cfu/mL以内。
一些研究表明, 某些细菌能通过分泌溶藻物质,直接破坏藻细胞[18]; 或者与藻细胞竞争营养和光照而抑制藻细胞的生长。而本研究中分离到的4株乙醇消耗菌与基因工程集胞藻共培养时, 并未对集胞藻的生长产生明显抑制(图7), 而与此同时, 培养液中乙醇的积累量出现明显差异, 说明本研究中的几株乙醇消耗菌并非通过抑制藻细胞的生理状态而影响其乙醇产量, 而是通过消耗产物乙醇而导致乙醇产量的降低。
经形态和分子鉴定, 本研究中分离的4株菌分别属于红酵母属、季也蒙酵母属、短波单胞菌属和微杆菌属。Okada, et al.、Verduyn, et al.的研究表明[19,20], 某些酵母兼有生产和消耗乙醇的能力, 两种代谢的方向取决于培养液中可利用糖的浓度, 当培养液中糖浓度较高时, 酵母发酵产乙醇; 当培养液中糖浓度较低时, 酵母就会消耗乙醇。而产乙醇集胞藻培养使用无机培养基, 有机碳相对匮乏, 因此污染的酵母可能从发酵产乙醇转变为消耗乙醇,从而降低培养液中的乙醇积累量。本研究还发现,短波单胞菌和微杆菌也能够消耗乙醇, 有文献报道显示短波单胞菌能够代谢利用乙醇[21], 而微杆菌消耗乙醇的报道还不多见。
在规模培养中, 由于反应器的非完全密闭性、培养环节众多和培养周期较长等原因, 很难做到绝对避免菌的污染。而且在不同培养情况下, 污染菌株的种类和对目标藻类和目标产物的影响机制也各有不同。本研究分离出的几株菌广泛存在于环境中,容易造成规模培养中的污染并大量消耗乙醇而影响乙醇产量, 尤其是短波单胞菌、微杆菌因为体积微小(直径约为 0.3 μm), 容易通过用于制备无菌空气的过滤器而造成污染, 在未来的研究和生产中应该加以重视。因此, 本研究通过对几株乙醇消耗菌的分离鉴定并评价其乙醇消耗速率, 有益于人们了解基因工程集胞藻培养过程中乙醇产量下降的原因, 也有助于认识微藻培养中污染菌的性状和污染途径,并就此可进一步发展针对性的污染控制方法。
[1] Balat M, Balat H. Recent trends in global production and utilization of bio-ethanol fuel [J]. Applied Energy, 2009, 86(11): 2273—2282
[2] Woods R P, Smith C R, Kramer D, et al. Genetically modified cyanobacteria for the production of ethanol [P]. United States Patent: 20100068776A1, 2010—03—18
[3] Dexter J, Fu P. Metabolic engineering of cyanobacteria for ethanol production [J]. Energy & Environmental Science, 2009, 2(8): 857—864
[4] Deng M D, Coleman J R. Ethanol synthesis by genetic engineering in cyanobacteria [J]. Applied and Environmental Microbiology, 1999, 65(2): 523—528
[5] Gao Z X, Zhao H, Li Z M, et al. Photosynthetic production of ethanol from carbon dioxide in genetically engineered cyanobacteria [J]. Energy & Environmental Science, 2012, 5(12): 9857—9865
[6] Wei K, Cao X H, Li X, et al. Genome shuffling to improve fermentation properties of acetic acid bacterium by the improvement of ethanol tolerance [J]. International Journal of Food Science & Technology, 2012, 47(10): 2184—2189
[7] Kometani T, Morita Y, Kiyama Y, et al. Relationship between ethanol consumption rate and prochiral ketone reduction rate in bakers’ yeast [J]. Journal of Fermentation and Bioengineering, 1995, 80(2): 208—210
[8] Chen L, Zheng J, Zhang Y X, et al. Isolation of an ethanol-utilizing strain and its application in determination of ethanol [J]. Chinese Journal of Applied & Environmental Biology, 2009, 15(4): 559—562 [陈亮, 郑军, 张益霞, 等.一株乙醇利用菌的分离及其在乙醇含量测定中的应用.应用与环境生物学报, 2009, 15(4): 559—562]
[9] Sun Z Y, Niu T G. Isolation and identification of a Bacillus sp. degrading alcohol [J]. Food Research and Development, 2006, 27(7): 77—79 [孙志一, 牛天贵. 一株能够有效解酒的芽孢杆菌的分离筛选与初步鉴定. 食品研究与开发, 2006, 27(7): 77—79]
[10] Lu C C, Li Z L, Li Y R, et al. Screening & identification of a strain with potentiality in ethanol detoxification and optimizition of its culture conditions [J]. Liquor-Making Science & Technolog, 2011, 6(204): 17—20 [陆晨晨, 李宗亮, 叡李盈 , 等. 一株乙醇降解菌株的筛选、鉴定及其培养条件的优化. 酿酒科技, 2011, 6(204): 17—20]
[11] Xiao J B, Jiang H X, Chu S Y. Isolation and Characterization of Denitrifying Bacterium Pseudomonas Mendocina aHD7 with Anaerobic Ammonium Oxidization [A]. In: Sustainable Environment and Transportation. Applied Mechanics and Materials, May 2012, Stafa, Zurich [C]. Trans Tech Publications. 2012, 699—703
[12] Maleszka R, Schneider H. Concurrent production and consumption of ethanol by cultures of Pachysolen Tannophilus growing on D-xylose [J]. Applied and Environmental Microbiology, 1982, 44(4): 909—912
[13] Pham H T, Larsson G, Enfors S O. Growth and energy metabolism in aerobic fed-batch cultures of Saccharomyces Cerevisiae: simulation and model verification [J]. Biotechnology and Bioengineering, 1998, 60(4): 474—482
[14] Ge P P, Chen L, Lü X F, et al. Response of genetically engineered Synechocystis sp. to salt concentration for production of ethanol [J]. The Chinese Journal of Process Engineering, 2013, 13(1): 129—133 [葛平平, 陈林, 吕雪峰, 等. 产乙醇基因工程集胞藻的盐胁迫响应. 过程工程学报, 2013, 13(1): 129—133]
[15] Wu Z X, Zeng B, Li R H, et al. Morphological and physiological characteristics and common aphanizomenon types in Chinese water bodies [J]. Acta Hydrobiologica Sinica, 2012, 36(2): 323—328 [吴中兴, 曾波, 李仁辉, 等.中国淡水水体常见束丝藻种类的形态及生理特性研究.水生生物学报, 2012, 36(2): 323—328]
[16] Lan E I, Liao J C. Metabolic engineering of cyanobacteria for 1-butanol production from carbon dioxide [J]. Metabolic Engineering, 2011, 13(4): 353—363
[17] Kämäräinen J, Knoop H, Stanford N J, et al. Physiological tolerance and stoichiometric potential of cyanobacteria for hydrocarbon fuel production [J]. Journal of Biotechnology, 2012, 162(1): 67—74
[18] Wang H, Liu L, Liu Z P, et al. Investigations of the characteristics and mode of action of an algalytic bacterium isolated from Tai Lake [J]. Journal of Applied Phycology, 2009, 22(4): 473—478
[19] Okada W, Fukuda H, Morikawa H. Kinetic expressions of ethanol production rate and ethanol consumption rate in baker's yeast cultivation [J]. Journal of Fermentation Technology, 1981, 59(2): 103—109
[20] Verduyn C, Zomerdijk T P L, Vandijken J P, et al. Continuous measurement of ethanol production by aerobic yeast suspensions with an enzyme electrode [J]. Applied Microbiology and Biotechnology, 1984, 19(3): 181—185
[21] Cápiro N L, Da Silva M L, Stafford B P, et al. Microbial community response to a release of neat ethanol onto residual hydrocarbons in a pilot-scale aquifer tank [J]. Environmental Microbiology, 2008, 10(9): 2236—2244
ISOLATION AND IDENTIFICATION OF ETHANOL CONSUMING MICROORGANISMS AND IMPACT ON ETHANOL PRODUCTION OF GENETICALLY ENGINEERED SYNECHOCYSTIS SP.
GE Ping-Ping1,2, CHEN Lin1, ZHANG Wei1, DONG Qing-Zhe1, LIU Tian-Zhong1and LÜ Xue-Feng1
(1. Key Lab. Biofuel, Qingdao Institute of Bioenergy and Bioprocess, Chinese Academy of Sciences, Qingdao 266101, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
Four ethanol consuming microorganisms (GR13, GW13, GT17, and GY22) were isolated from the contaminated culture of genetically engineered Synechocystis sp. and were utilized to evaluate the effects of these microorganisms on the ethanol consuming capabilities and ethanol productions of Synechocystis sp. Based on the morphological characteristics and 16S rDNA and 26S rDNA gene sequence, the four strains were identified as Rhodotorula sp., Meyerozyma sp., Brevundimonas sp., and Microbacterium sp., respectively. We observed that strain GR13 (Rhodotorula sp.) had the highest level of specific ethanol consumption rate [391 g/(1015cfu⋅d)], followed by strain GW13 (Meyerozyma sp., [ 80.1 g/(1015cfu⋅d)]. While strains GT17 (Brevundimonas sp.) and GY22 (Microbacterium sp.) had much lower ethanol consumption rate. All four strains did not significantly affect the growth of Synechocystis sp., however, they remarkably decreased the ethanol production of Synechocystis sp. The results of the co-culture experiment demonstrated that strains GR13, GW13, GT17, and GY22 diminished ethanol production of the genetically engineered Synechocystis sp. by 53.8%, 23.6%, 40.7% and 27.3%, respectively.
Ethanol consuming microorganism; Microorganism contamination; Bioethanol; Genetically engineered Synechocystis sp.
Q949.22
A
1000-3207(2014)03-0487-08
10.7541/2014.69
2013-04-10;
2013-12-25
国家高技术发展计划(863)(2012AA052103)资助
葛平平(1988—), 女, 河北保定人; 硕士研究生; 主要从事生物化工研究。E-mail: gepp@qibebt.ac.cn
陈林, Tel: 0532-80662737; E-mail: chenlin@qibebt.ac.cn
