琼胶酶产生菌Stenotrophomonas sp.Z705的发酵条件优化
2014-03-26缪伏荣董志岩
缪伏荣,董志岩,刘 景
(福建省农业科学院 畜牧兽医研究所,福建 福州 350013)
琼胶(琼脂)是存在于红藻细胞壁中的一类多糖,是自然界中存在的重要碳源之一,是我国产量最大的海洋三大多糖(褐藻胶、琼胶、卡拉胶)之一,广泛应用于食品、医药、生物技术等领域[1]。琼胶由中性的琼胶糖(agarose)和离子性的硫琼胶(agaropectin)组成,其中琼胶糖是由1,3连接的β-D-半乳吡喃糖和1,4连接的3,6-内醚-α-L-半乳吡喃糖残基反复交替连接的链状中性多糖[2]。琼胶黏度高、溶解度低,影响了其在各领域中的应用;但琼胶降解后可产生一定分子质量和聚合度的琼胶寡糖,这些寡糖具有良好的抗癌[3]、抗氧化[4]、抗病毒[5]、增强免疫[6]等药理作用和生物活性。但由于未能找到有效降解琼胶的途径[7],因此长期以来琼胶未能得到很好的开发利用。利用琼胶酶降解琼胶生产琼胶寡糖,被认为是当今最理想的绿色环保生产技术。但由于琼胶酶来源少、活性低、价格昂贵,难以满足琼胶寡糖生产的需要,因此,筛选高产琼胶酶的菌株及研究其发酵产酶具有重要的实际意义。
目前,已见报道的产琼胶酶菌属有Pseudomonas[8]、Pseudoalteromonas[9]、Streptomyces[10]、Alteromonas[11]、Vibrio[12]和Cytophaga[13]等。本试验从腐烂的紫菜中分离到1株高产琼胶酶的菌株,并将其初步鉴定为寡养单胞菌属Stenotrophomonassp.Z705[14], 目前国内外尚未见该菌属产琼胶酶的相关报道。本研究通过单因素试验和正交试验,对该菌株的发酵产酶条件和培养基组成进行优化,旨在为琼胶酶的深入研究以及琼胶低聚糖的开发应用奠定基础。
1 材料与方法
1.1 材 料
1.1.1 菌种来源 寡养单胞菌属Stenotrophomonassp.Z705,由福建省农业科学院畜牧兽医研究所动物营养室提供。
1.1.2 培养基及试剂 纯化培养基:牛肉膏5 g,酵母浸膏1.5 g,琼脂15 g,陈海水1 000 mL,pH 7.2;121 ℃灭菌30 min。
种子和发酵培养基:牛肉膏5 g,酵母浸膏1.5 g,琼脂3 g,陈海水1 000 mL,pH 7.2。
主要试剂有DNS试剂[15]、0.2 mol/L磷酸缓冲液[16]、琼脂粉、蛋白胨、酵母浸膏和牛肉膏等。
1.1.3 主要仪器 主要仪器有DY-89Ⅱ电动玻璃匀浆机、LRH-250A生化培养箱、SW-CJ-1FD单人超净工作台、热电multican fc 酶标仪、TDL-5000B高速冷冻离心机、DY-ⅢS水质分析仪、FW-80高速万能粉碎机、LDZS-30KBS立式高压灭菌器、CRY-200恒温摇床等。
1.2 菌株的培养
1.2.1 纯化培养 将Stenotrophomonassp.Z705划菌斜面置于28 ℃培养箱内培养2 d,然后置于4 ℃冰箱中保存。
1.2.2 种子培养 在250 mL三角瓶中装入25 mL种子培养基,121 ℃灭菌30 min后,取培养好的斜面挑取1环于种子培养基中,28 ℃、200 r/min摇床培养24 h。
1.2.3 发酵培养 在250 mL三角瓶中装入25 mL发酵培养基,121 ℃灭菌30 min后,按体积分数0.1%的接种量接入种子液,28 ℃、200 r/min摇床培养23 h。
1.3 琼胶酶活力的测定
发酵培养11 h开始每2 h对发酵液取样1次。发酵液经4 000 r/min离心5 min,其上清液即为粗酶液。根据Imoto等[17]的方法,通过DNS-还原糖法测定发酵液的酶活,用D-半乳糖绘制标准曲线[18]。取粗酶液1 mL加入磷酸缓冲液1 mL,再加入0.3 g/L的琼胶底物溶液1 mL,50 ℃水浴酶解30 min后再于沸水浴中灭活5 min,立即用冰水冷却至室温,然后加入1.5 mL的DNS试剂;在沸水浴中保温显色5 min后,于室温下定容至10 mL,20 ℃、2 000 r/min离心5 min得上清液,用酶标仪测其在520 nm波长处的吸光度;同时以灭活酶作对照。在上述反应条件下,以1 mL酶液1 min产生1 μg还原糖作为1个酶活(U)。
1.4 菌株产琼胶酶发酵条件的优化
在发酵培养基基础上,按表1中的设计方案对发酵条件进行单因素试验,分析不同因素对菌株发酵产琼胶酶活力的影响,其他培养条件和培养基相同,每组设置3个重复。
1.5 菌株产琼胶酶发酵培养基的优化
1.5.1 单因素试验 首先,在获得菌株产琼胶酶发酵最佳条件的基础上,分析不同氮源(牛肉膏、酵母浸膏、蛋白胨、NaNO3、(NH4)2SO4)和碳源(葡萄糖、D-半乳糖、琼胶、果糖、麦芽糖)对菌株发酵产琼胶酶活力的影响;在筛选到最佳氮源(牛肉膏、酵母浸膏)以及最佳碳源(琼胶)的基础上进行单因素试验,培养基中氮源和碳源添加水平见表1。
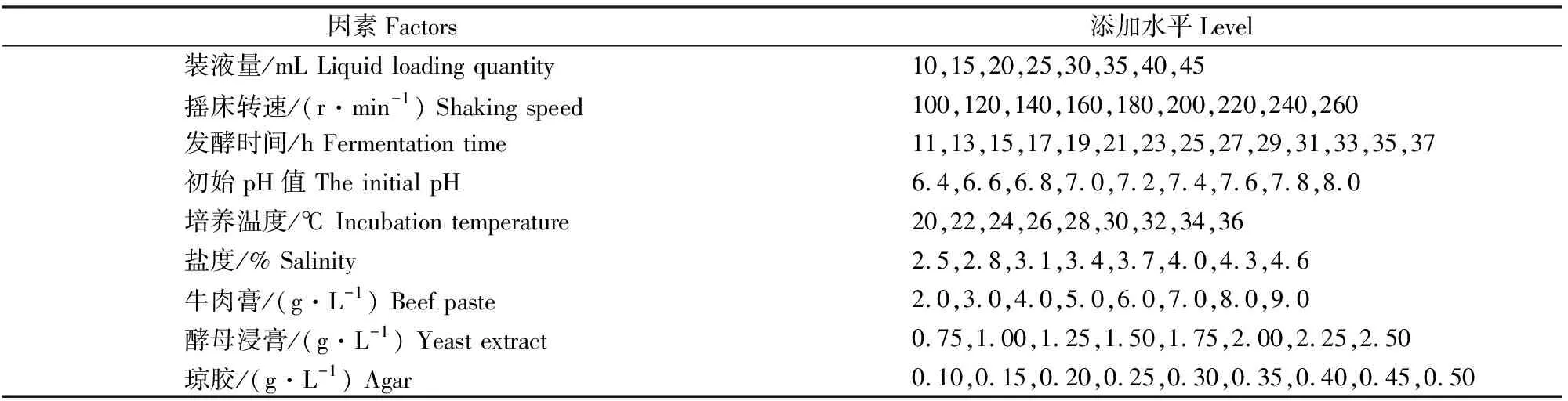
表1 Stenotrophomonas sp.Z705菌株产琼胶酶发酵条件和培养基组成优化的单因素试验
1.5.2 正交试验 在单因素试验基础上,选择最佳氮源(牛肉膏、酵母浸膏)以及最佳碳源(琼胶)等培养基组分,采用三因素三水平L9(33)的正交试验(表2)筛选最佳培养基组成。所有试验重复3次,分别平行取样。用SPSS 13.0进行数据分析。

表2 Stenotrophomonas sp.Z705菌株产琼胶酶发酵培养基组成优化的正交试验方案
2 结果与分析
2.1 菌株产琼胶酶发酵条件的优化
2.1.1 装液量和摇床转速对菌株发酵产酶的影响 在微生物发酵过程中,溶氧量对好氧细菌的生长和产酶具有十分重要的作用。减少装液量和增加摇床转速均可以达到增加溶氧量的目的,使菌株快速地利用营养物质,从而有利菌株产酶。但溶氧量必须适度,过量供给会造成酶活力的下降。由图1可以看出,在转速为200 r/min时,随着装液量的增加,琼胶酶活力随之升高,至装液量为25 mL时,琼胶酶活力最高(37.8 U/mL);然后随着装液量的增加,琼胶酶活力逐渐降低。从图2可知,在装液量为25 mL时,随着摇床转速的提高,琼胶酶活力随之升高,当摇床转速为200 r/min时,琼胶酶活力达到最高(35.6 U/mL);之后随着摇床转速的提高,琼胶酶活力逐渐降低。可知最佳装液量和摇床转速分别为25 mL和200 r/min。
2.1.2 发酵时间对菌株发酵产酶的影响 由图3可见,在装液量25 mL和摇床转速200 r/min的条件下,培养11 h时琼胶酶活力为5.6 U/mL;随着发酵时间的延长,琼胶酶活力逐渐提高,至23 h琼胶酶活力达到最高(45.2 U/mL);之后随着发酵时间增加,琼胶酶活力逐渐下降。表明最佳发酵时间为23 h。
2.1.3 初始pH值对菌株发酵产酶的影响 海水的pH为7.2~8.3,海洋细菌最适宜生长的pH值与此关系密切。培养物的pH不仅直接影响菌体对营养物的吸收及代谢产物的分泌,而且影响营养物的分解。从图4可以看出,在培养基的初始pH值为6.4时,琼胶酶活力为0.6 U/mL,随着培养基初始pH值的升高,Stenotrophomonassp.Z705菌株产琼胶酶活力逐渐提高,当初始pH值为7.2时达到最高(46.3 U/mL);之后随着培养基初始pH值的升高,琼胶酶活力逐渐下降。表明最佳初始pH值为7.2。
2.1.4 培养温度对菌株发酵产酶的影响 培养温度是影响微生物生长和代谢的最重要的环境因素之一。适当地提高培养温度有助于促进微生物的生长和酶促反应。从图5可以看出,在培养温度为20 ℃时,琼胶酶活力为17.53 U/mL,随着培养温度的升高,Stenotrophomonassp.Z705菌株产琼胶酶活力逐渐提高,当培养温度为28 ℃时,琼胶酶活力最高(43.8 U/mL);之后随着培养温度升高,琼胶酶活力下降。可知最佳培养温度为28 ℃。
2.1.5 盐度对菌株发酵产酶的影响 盐度与海洋细菌的生长密切相关,是区分海洋菌与淡水菌的重要指标。本试验改变培养基的盐度,来研究该菌株产酶最适的盐度。从图6可以看出,随着发酵培养基盐度的升高,Stenotrophomonassp.Z705菌株产琼胶酶活力逐渐提高,至发酵盐度为3.4%时达最高(43.2 U/mL);之后随着发酵液盐度的升高,琼胶酶活力逐渐下降。表明此菌株是典型的海洋细菌,具有耐高渗透压的特性,发酵培养的最佳盐度为3.4%。

图1 装液量对Stenotrophomonas sp.Z705菌株发酵产琼胶酶的影响

图3 发酵时间对Stenotrophomonas sp.Z705菌株产琼胶酶的影响

图5 培养温度对Stenotrophomonas sp.Z705菌株发酵产琼胶酶的影响
2.2 菌株产琼胶酶发酵培养基组成优化的单因素试验
2.2.1 氮源对菌株发酵产酶的影响 图7显示,以牛肉膏作为氮源时,Stenotrophomonassp.Z705菌株产琼胶酶活力最大,其次为酵母浸膏。因此,分别选用牛肉膏和酵母浸膏作为氮源,研究其对Stenotrophomonassp.Z705菌株产琼胶酶活力的影响。从图8可以看出,随着牛肉膏添加量的增加,琼胶酶活力逐渐提高,至牛肉膏添加量为5.0 g/L时,琼胶酶活力最高(45.6 U/mL);之后随着牛肉膏添加量升高,琼胶酶活力下降。由图9可见,随着酵母浸膏添加量增加,琼胶酶活力逐渐提高,当酵母浸膏添加量为1.50 g/L时,琼胶酶活力最高(41.8 U/mL);之后随着酵母浸膏添加量的增加,琼胶酶活力下降。

图7 不同氮源对Stenotrophomonas sp.Z705菌株发酵产琼胶酶的影响

图8 牛肉膏添加量对Stenotrophomonas sp.Z705菌株发酵产琼胶酶的影响
2.2.2 碳源对菌株发酵产酶的影响 碳源是微生物生长和发酵过程中必需的营养物。它不仅可为微生物生长繁殖提供能量,还可为菌体合成目标产物提供所需的碳成分。本研究中,不同碳源对Stenotrophomonassp.Z705菌株发酵产琼胶酶的影响见图10和图11。

图10 不同碳源对Stenotrophomonas sp.Z705菌株发酵产琼胶酶的影响
图10显示,以琼胶作为惟一碳源时,发酵液培养Stenotrophomonassp.Z705菌株产琼胶酶的活力最高。因此,选用琼胶作为碳源,研究其对Stenotrophomonassp.Z705菌株产琼胶酶活力的影响。从图11可以看出,随着琼胶添加量的升高,琼胶酶活力逐渐提高,琼胶添加量为0.30 g/L时琼胶酶活力达到最高(43.5 U/mL);之后随着琼胶添加量的升高,琼胶酶活力逐渐降低。这可能是因为随着琼胶添加量的增加,培养基的流动性变差,发酵液通气量降低,从而影响了菌株的发酵产酶性能。
2.3 菌株产琼胶酶发酵培养基组成优化的正交试验
从单因素试验结果可以看出,Stenotrophomonassp.Z705菌株产琼胶酶的最佳发酵条件为:装液量25 mL、摇床转速200 r/min、发酵时间23 h、初始pH 7.2、发酵温度28 ℃、盐度3.4%;发酵培养基中的牛肉膏、酵母浸膏和琼胶对该菌株的产酶影响较大。从表3和表4可见,影响Stenotrophomonassp.Z705菌株产琼胶酶活力的因素顺序为A(牛肉膏)>B(酵母浸膏)>C(琼胶);同时各因素对该菌产酶水平均有显著的影响(P<0.05),确定Stenotrophomonassp.Z705菌株发酵的最佳培养基组成为A2B2C3,即牛肉膏5.0 g/L、酵母浸膏1.50 g/L、琼胶0.30 g/L。结合最佳发酵条件,经多次发酵试验,该菌株发酵产琼胶酶活力稳定在87.1 U/mL左右。

表3 Stenotrophomonas sp.Z705菌株产琼胶酶发酵培养基组成优化的正交试验结果

表4 Stenotrophomonas sp.Z705菌株产琼胶酶发酵培养基优化的正交试验结果的方差分析
3 讨 论
微生物发酵产酶是一个复杂的生理生化过程,菌体不同的营养条件会直接影响菌体的代谢,从而影响酶的产量。要提高酶的产量,就必须对发酵产酶的条件进行优化[19]。不同的琼胶酶产生菌的培养条件不相同,各种因素如培养基的组成、培养温度、初始pH值、培养时间等都对产酶有很大的影响。本研究以来源于腐烂紫菜且具有较高琼胶降解能力的野生菌株Stenotrophomonassp.Z705为出发菌株,以提高菌株的产酶活性为目的,对其产酶发酵条件和培养基组成进行了优化,确定Stenotrophomonassp.Z705菌株的最佳发酵条件为:装液量25 mL、摇床转速200 r/min、发酵时间23 h、初始pH 7.2、发酵温度28 ℃、盐度3.4%;最佳培养基组成为:牛肉膏5.0 g/L、酵母浸膏1.50 g/L、琼胶0.30 g/L,在此条件下该菌株产酶效果最好,琼胶酶活力最高。经多次发酵试验,该菌株发酵产琼胶酶活力稳定在87.1 U/mL左右,与优化前(平均酶活力为34.49 U/mL)相比提高了60.4%。
与国内外已报道的琼胶酶产生菌相比,首先从酶活力方面而言,菌株NBRC102603[20]为56.89 U/mL,菌株HF3-04[21]为80.7 U/mL,Bacillussp.MK03[22]为14.1 U/mL,Vibriosp.JT0107[23]为66 U/mL,AgarivoransalbusYKW-34[24]为83 U/mL,均低于本研究中的Stenotrophomonassp.Z705菌株;其次从琼胶酶活力达到最高时的发酵时间而言,MA-B22[25]需要60 h,Halomonassp.DT-3[26]需要36 h,而本研究供试菌株仅需23 h。目前我国尚未有工业化产琼胶酶的菌株,所需产品均来自国外,价格昂贵,而本研究供试菌株发酵培养基组分简单低价、培养条件简便,稳定性好,具有一定的生产潜力,可为琼胶酶的深入研究及琼胶低聚糖的开发应用奠定基础。
[参考文献]
[1] 王选良.一株新的具有脱色能力的琼脂分解菌 [J].微生物学报,1985,25(4):289-293.
Wang X L.A novel decolorizing ability of agar-decomposing microorganisms [J].Journal of Microbiology,1985,25(4):289-293.(in Chinese)
[2] 王静雪,江晓路,胡晓珂.细菌降解琼胶的研究进展 [J].中国水产科学,2001,8(3):94-96.
Wang J X,Jiang X L,Hu X K.Research advance on agar degradation by bacteria [J].Journal of Fishery Sciences of China,2001,8(3):94-96.(in Chinese)
[3] Wang J X,Mou H J,Jiang X L.Biological activities of a neutral water-soluble agar polysaccharide prepared by agarase degradation [J].High Tech Lett,2005,11(4):415-420.
[4] 徐 强,薛长湖,赵 雪,等.酸法制备琼胶低聚糖及其抗氧化性评价 [J].中国海洋药物,2002,21(1):19-22.
Xu Q,Xue C H,Zhao X,et al.Preparation of agar oligosaccharides by acid hydrolysis and determination of their antioxidative effect [J].Chinese Journal of Marine Drugs,2002,21(1):19-22.(in Chinese)
[5] 毛文君,林 洪,管华诗.琼胶寡糖的制备及其 C-NMR研究 [J].水产学报,2001,25(6):582-584.
Mao W J,Lin H,Guan H S.Preparation and C-NMR investigation of agarose oligosaccharides [J].Journal of Fisheries of China,2001,25(6):582-584.(in Chinese)
[6] 刘江涛,蔡俊鹏,吴 冰.琼胶酶及其综合应用的研究概况 [J].现代食品技术,2005,21(1):177-179.
Liu J T,Cai J P,Wu B.Progress of studies on agarase and its applications [J].Modern Food Science and Technology,2005,21(1):177-179.(in Chinese)
[7] 陈海敏,严小军,郑 立,等.琼胶的降解及其产物的分析 [J].郑州工业学院学报,2003,24(3):41-44.
Chen H M,Yan X J,Zheng L,et al.Preparation of agaros oligosaccharides [J].Journal of Zhengzhou Institute of Technology,2003,24(3):41-44.(in Chinese)
[8] Groleau D,Yaphe W.Enzymatic hydrolysis of agar purification and characterization of beta neoagaro hydrolase fromPseudomonasatlantica[J].Can J Microbiol,1977,23(6):672-679.
[9] Vera J,Alvarez R,Murano E,et al.Identification of a marine agarolyticPseudoalteromonasand characterization of its extracellular agarase [J].Appl Environ Microbiol,1998,64(11):4378-4383.
[10] Buttner M J,Feamley I M,Bibb M.The agarasegene(dagA) ofStreptamycescoelicor A3(2):Nuelotide sequence and transcriptional analysis [J].Mol Gen Genet,1987,209(1):101-109.
[11] Potin P,Richard C,Rochas C,et al.Purification and characterization of the alpha-agarase fromAlteromonasagarlyticus(Cataldi)comb.nov.,strain GJ1B [J].Eur J Biochem,1993,214(2):599-607.
[12] Aoki T,Araki T,Kitam ikado M.Purification and characterization of a novel beta-agarase fromVibriosp. AP-2 [J].Eur J Biochem,1990,187(2):461-465.
[13] Qhta Y,Nogi Y.Enzymatic properties and nucleotide and amino acid sequences of a thermostable β-agarase from the novel marine isolate,JAMB-A94 [J].Biosci Biotechnol Biochem,2004,68(5):1973-2081.
[14] 缪伏荣.一株琼胶酶产生菌的筛选与鉴定 [J].中国农学通报,2013,29(21):88-91.
Miao F R.Screening and molecular identification of a new agarase-producing bacterium [J].Chinese Agricultural Science Bulletin,2013,29(21):88-91.(in Chinese)
[15] 张信旭,高海波.3,5-二硝基水杨酸测定古尼虫中多糖含量方法 [J].贵州化工,2002,27(3):23-38.
Zhang X X,Gao H B.Determination of polyose in Chinese guni caterpillar fungus by spectrophotometry [J].Guizhou Chemical Industry,2002,27(3):23-38.(in Chinese)
[16] 林加涵,魏文铃.现代生物学实验:上册 [M].北京:高等教育出版社,2000:243.
Lin J H,Wei W L.Modern biology experiments:First volume [M].Beijing:Higher Education Press,2000:243.(in Chinese)
[17] Imoto T,Yegishita K.A simple activity measurement of lysozyme [J].Agr Biol Chem,1971,35:1154-1156.
[18] Kohtaro K,Noriyoshi M,Yousuke L,et al.Purication and characterization of a novel β-agarase from an alkalophilic bacterium,Alteromonassp.E-1 [J].Journal of Bioscience and Bioengineering,1999,87(4):436-441.
[19] 付万冬.高产琼胶酶菌株的筛选、发酵条件优化及琼胶酶酶学性质的研究 [D].山东青岛:中国海洋大学,2006.
Fu W D.Study on screening and selecting an effectively agarase-producing strain,its optimal cultivation and characteristics of agarase [D].Qingdao,Shandong:Ocean University of China,2006.(in Chinese)
[20] 朱启加,朱慧文,孙艳娜,等.海洋细菌NBRC102603产琼胶酶发酵条件优化 [J].中国酿造,2011,230(5):53-55.
Zhu Q J,Zhu H W,Sun Y N,et al.Optimization of fermentation conditions for agarase production by a marine bacterium NBRC102603 [J].China Brewing,2011,230(5):53-55.(in Chinese)
[21] 马悦欣,安 军,刘双连,等.仿刺参消化道内产琼胶酶菌株的选育及培养条件优化 [J].大连水产学院学报,2007,22(2):86-91.
Ma Y X,An J,Liu S L,et al.Breeding and its optimal culture of bacterial and agarase-producing strain from digestive tracts in sea cucumberApostichopusjaponicu[J].Journal of Dalian Fisheries University,2007,22(2):86-91.(in Chinese)
[22] Hisashi S,Yoshinori S,Tohru S,et al.Purification and characterization of an extracellular β-agarase fromBacillussp.MK03 [J].Journal of Bioscience and Bioengineering,2003,95(4):328-334.
[23] Sugano Y,Terada I,Arita M,et al.Purification and characterization of a new agaarase from a marine bacterium,Vibriosp.strain JT0107 [J].Applied and Environmental Microbiology,1993,59(5):1549-1554.
[24] Fu X T,Lin H,Kin S M.Purification and characterization of a novel β-agarase agaA34 fromAgarivoransalbusYKW-34 [J].Appl Microbiol Biotechnol,2008,78:265-273.
[25] 卢 斌,柯才焕,杨 明,等.一株高产琼胶酶菌株MA-B22的分子鉴定与产酶条件优化 [J].水产学报,2009,33(6):1037-1043.
Lu B,Ke C H,Yang M,et al.Isolation and identification of a bacterium MA-B22 producing agarase and the optimal cultivation of enzyme production [J].Journal of Fisheries of China,2009,33(6):1037-1043.(in Chinese)
[26] 王晓燕,桑卫国.SPSS正交设计优化琼胶酶产生菌的发酵条件 [J].宁波大学学报,2010,23(2):11-16.
Wang X Y,Sang W G.Optimization of fermentation conditions of an agarase-producing bacterium by SPSS orthogonal design [J].Journal of Ningbo University,2010,23(2):11-16.(in Chinese)
