亚心型扁藻叶绿体psbA基因的克隆及序列分析
2014-03-22侯士昌崔玉琳温少红唐志红
侯士昌, 崔玉琳, 温少红, 唐志红, 秦 松
(1. 烟台大学 生命科学学院, 山东 烟台 264003; 2. 中国科学院 烟台海岸带研究所, 山东 烟台 264003)
亚心型扁藻(Platymonussubcordiformis) 是一种海洋产的单细胞浮游藻类,属衣藻科、扁藻属。亚心型扁藻因具有适应能力强,繁殖速度快,适口性好,必需氨基酸含量高及自身能够合成并富集高浓度多不饱和脂肪酸(polyunsaturated fatty acids, PUFA)[1]等特点,是生产中人们广泛采用的优良海产饵料[2],在水产经济动物育苗中广为应用。此外,亚心型扁藻在药用研究[3]、化合物毒性评估[4]及光合放氢[5]等方面同样具有广阔的开发空间。
psbA基因是光合作物叶绿体基因组中的一个重要光调控基因,编码光系统Ⅱ反应中心的核心蛋白D1,介导光合作用中的电子传递。psbA基因在不同的光合作物之间具有高度的同源性[6]。到目前为止,psbA基因在植物(如水稻、小麦、烟草、菠菜等)[7]和藻类(如冈村枝管藻[8]、小球藻[9]等)里都有报道研究,但是在亚心型扁藻里却鲜有报道。本文以亚心型扁藻为材料,采用基因步移的手段克隆了叶绿体基因psbA并对其序列进行了分析,以期为亚心型扁藻的分子生物学研究提供理论参考。
1 材料与方法
1.1 材料
亚心型扁藻藻株为中国科学院烟台海岸带研究所保存与培养。
1.2 方法
1.2.1 藻体的培养与收集
亚心型扁藻采用f/2培养基,光照强度80~90 μmolm-2s-1,于23℃以12 h/12 h的光暗周期进行静止培养。10 d左右以6000 r/min离心5 min收集藻体,备用。
1.2.2 总DNA的提取
DNA提取依据CTAB法进行。取100 mg样品置于1.5 mL的离心管中,液氮冷却10 min,充分研磨后加入900 μL CTAB,震荡混匀后加入35 μL β-巯基乙醇,65℃温浴1 h;加入等体积苯酚/氯仿/异戊醇(25∶24∶1),混匀后12000 r/min离心15 min,取出上清液,加入等体积的苯酚/氯仿/异戊醇(25∶24∶1),重复2次;向上清液中加入0.8倍体积异丙醇,-20℃放置3 h,12000 r/min离心15 min,弃上清;用70%乙醇洗涤DNA沉淀2次,晾干,将DNA溶解于30 μL的TE缓冲液中,加入3 μL RNase于37℃处理0.5 h,电泳检测。
1.2.3psbA基因的克隆和分析
根据GenBank上收录的莱茵衣藻psbA基因序列(基因登录号:HQ667991.1),人工设计合成6段寡聚核苷酸片段作为引物,结果如表1。以提取的亚心型扁藻总DNA为模板,配合Takara公司基因步移试剂盒里的兼并引物AP1,分别对亚心型扁藻叶绿体基因psbA的5′端和3′端进行SP1、SP2及SP3步移PCR反应。
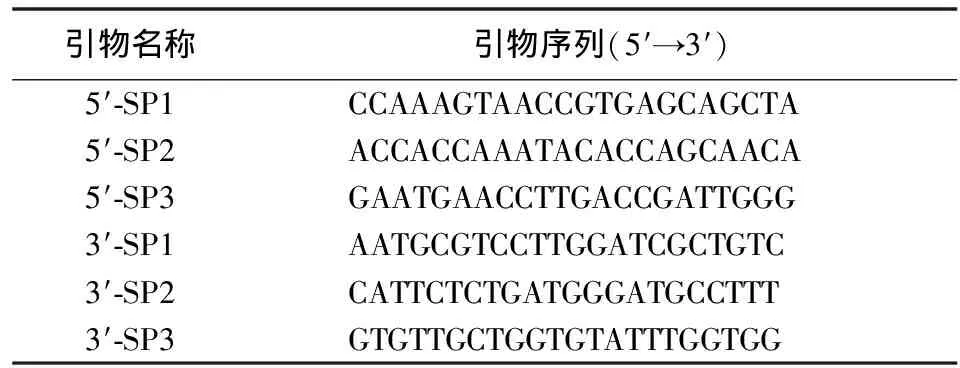
表1亚心型扁藻psbA基因步移反应设计引物
PCR产物在1.2%的琼脂糖凝胶上电泳,目的片段DNA用Takara公司胶回收试剂盒进行回收与纯化,利用pMD-18T载体进行体外重组,再转化到感受态大肠杆菌体内。对挑取的单克隆进行测序,然后对该序列用NCBI数据库中的BLAST进行同源性比对。
2 结果与分析
2.1 总DNA的提取
对提取的基因组DNA用1.2%的琼脂糖凝胶电泳分析,结果如图1。获得的基因组DNA条带明亮且比较集中,说明提取的基因组DNA浓度高、纯度好,可用于PCR实验。

M—DNA分子质量标准;1、2—基因组DNA。
2.2 PCR产物的分析
以提取到的基因组DNA为模板,分别从5′端和3′端进行SP1、SP2及SP3步移PCR,结果如图2和图3。回收目的片段后进行DNA拼接和测序,结果如图4。在所测序列中,psbA基因全长1939 bp,编码区片段大小为1066 bp,为单外显子基因,编码353个氨基酸;5′端非翻译区片段长429 bp,转录起始位点位于翻译起始密码子ATG上游第108个核苷酸G;3′端非翻译区片段长444 bp,具有多个多聚A和多聚T以及短的回文结构。此外,编码区T、C、A、G的平均含量分别为36%、20%、25%及19%;其中,A+T的含量为61%,明显高于G+C的含量,表现出很强的碱基(A+T)组成偏向性。

M—DNA分子质量标准;1、2—SP3 PCR步移结果。
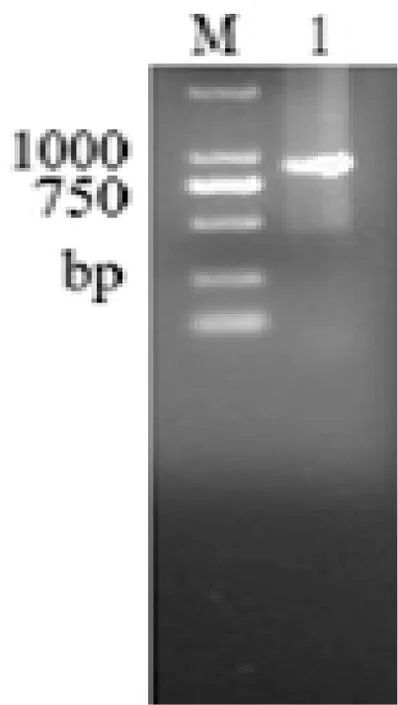
M—DNA分子质量标准;1—SP3 PCR步移结果。

图4 亚心型扁藻psbA基因编码区序列
叶绿体的基因组序列高度保守。psbA基因编码区的核苷酸序列同源性在双子叶植物之间超过90%,其编码的氨基酸序列在大豆、烟草、水稻等植物之间的保守性超过98%。在藻类等低等植物之间,psbA的同源性在84%~90%[10]之间。对得到的基因片段与莱茵衣藻psbA序列(基因登录号:HQ667991.1)进行比对,核苷酸序列同源性达到87.92%,翻译成的氨基酸序列相似性为91.79%。这一方面说明采用PCR步移扩增成功得到亚心型扁藻psbA基因(基因登录号:KF528742),另一方面,psbA基因在亚心型扁藻体内是比较保守的,这可能与D1蛋白的电子传递功能有关[9]。
此外,钟珍萍等[10]研究发现光合植物叶绿体psbA基因内没有或很少编码赖氨酸残基的密码子,特别是在陆生植物体内。藻类中,纤细裸藻有5个密码子,鱼腥藻有2个密码子,莱茵衣藻无此密码子,但是在推导的亚心型扁藻所编码的氨基酸中含有4个赖氨酸残基。
目前利用L-赖氨酸为配基制作的HyperD亲和层析填料含有一个较长的间隔键,使得该填料更容易结合赖氨酸配基,具有稳定性高、亲和力强的特点,可以快速高效地纯化含有赖氨酸的蛋白质,这或许为亚心型扁藻光合蛋白D1的分离纯化提供了有效地方法。
2.3 有效密码子数和密码子第3位碱基中G+C含量分析
有效密码子数(Effective number of codons,ENC)代表着某一基因中各同义密码子之间偏好使用的程度,可以为密码子的使用偏好性提供客观的判断[11]。ENC值的范围从20到61之间,分别表示着某一个氨基酸只使用一个密码子和平均使用各个密码子的两种极端情况,与20越接近则偏好性越强。密码子第3位碱基中G+C含量(GC3s)表示G+C的含量与第3位碱基总量的比值。
应用在线程序EMBOSS[12]计算亚心型扁藻叶绿体基因psbA的ENC值和GC3s值分别为39.871和0.3183,ENC值偏小,表明亚心型扁藻psbA基因具有明显的密码子使用偏好性;亚心型扁藻psbA基因编码区G+C含量低,而GC3s值更低,说明psbA基因偏好使用以A、T结尾的密码子。钟珍萍等[10]通过对叶绿体基因psbA的表达调控研究认为,在光合植物体内psbA基因对NNC的选择优于NNT,这与亚心型扁藻叶绿体基因psbA的表达不一致。
2.4 密码子偏好性分析
相对同义密码子使用度(Relative synonymous codon usage,RSCU)可以直观地反应密码子使用的偏好程度,指在编码特定氨基酸的密码子中,某一密码子在同义密码子中使用的相对概率[13]。RSCU值等于1表示密码子没有使用偏好,大于1则表明密码子的使用偏好性较强,反之亦然。比例(Fraction)表示在编码该氨基酸的密码子中某一密码子所占的比例,所有比例相加总和是1。频率(Frequency)代表某一密码子在1000个基因编码总密码子中出现的次数。

表2 EMBOSS和CodonW 程序分析亚心型扁藻psbA基因的密码子偏好性
续表2 Table 2(continued)

(f/‰)TTTF0.42330.986110.85GGAG0.0775.63420.31GGCG0.0775.63420.31GGGG0.0775.63420.31GGTG0.76956.338203.08CACH0.71414.08551.43CATH0.2865.63420.57ATAI0.0715.63420.21ATCI0.57145.070161.71ATTI0.35728.169101.07AAAK1.00011.26842.00AAGK0.0000.00000.00CTAL0.0000.00000.00CTCL0.0615.63420.36CTGL0.0000.00000.00CTTL0.24222.53581.45TTAL0.60656.338203.64TTGL0.0918.45130.55ATGM1.00030.986111.00AACN0.56225.35291.12AATN0.43819.71870.88CCAP0.47122.53581.88CCCP0.0592.81710.24CCGP0.0592.81710.24CCTP0.41219.71871.65CAAQ0.90928.169101.82CAGQ0.0912.81710.18AGAR0.26314.08551.58AGGR0.1588.45130.95CGAR0.0532.81710.32CGCR0.1055.63420.63CGGR0.0532.81710.32CGTR0.36819.71872.21AGCS0.10811.26840.65AGTS0.13514.08550.81TCAS0.29730.986111.78TCCS0.0272.81710.16TCGS0.10811.26840.65TCTS0.32433.803121.95ACAT0.26711.26841.07ACCT0.0000.00000.00ACGT0.0672.81710.27ACTT0.66728.169102.67GTAV0.47425.35291.89GTCV0.0000.00000.00GTGV0.0532.81710.21GTTV0.47425.35291.89TGGW1.00025.35291.00TACY0.52925.35291.06TATY0.47122.53580.94TAG∗1.00014.0851∗TAA∗0.0000.0000∗TGA∗0.0000.0000∗
*—代表终止密码子。
CodonW在线程序[12]计算结果如表5,亚心型扁藻psbA基因密码子中,25个密码子的RSCU值大于l,具有一定的偏好性(表2以下划线标出)。其中,GCA、GCT、TCT、GAT、GAA、GGT、ATT、AAA、CTT、TTA、CCT、CAA、AGA、CGT、TCA、TCT、ACA、ACT、GTA及GTT共20个密码子是以A或T碱基结尾的,且Fraction值也较大,为亚心型扁藻psbA基因的偏好密码子。大多数C或G碱基结尾的密码子的RSCU值较低,表明这些密码子在该基因中的使用次数较少;其终止密码子使用了TAG。
3 结论
psbA基因的核苷酸序列具有高度保守性,国内外很多研究者利用这一特性直接从基因组中扩增出psbA基因。本实验利用PCR步移法从亚心型扁藻(Platymonassubcordiformis)基因组DNA中克隆得到psbA基因,获得的psbA核苷酸序列与衣藻中该基因序列的同源性达到87.92%,二者编码的氨基酸残基同源性高达91.79%。
密码子的使用偏好性是在进化过程中内外因素的综合作用下演变来的,不同物种体内密码子的偏好性不同,特定氨基酸对同义密码子的选择也不一样[14]。亚心型扁藻叶绿体基因psbA的ENC值和GC3s值都较低,表明psbA基因具有明显的密码子偏好性,偏好使用以A/T结尾的密码子;RSCU值显示25个psbA的偏好密码子中有20个是以A或T碱基结尾的,同样,psbA基因的碱基组成分析显示psbA基因表现出很强的碱基(A+T)组成偏向性。另外,亚心型扁藻psbA基因偏好使用以A/T结尾的密码子,与之前报道的光合植物体内psbA基因对NNC的选择优于NNT不一致[10]。亚心型扁藻的这种特征可能和它是一种低等的藻类有关,生长过程中容易受到各种影响而表现在对密码子的选择偏好性上。目前课题组正在克隆亚心型扁藻叶绿体的其余基因,以期为亚心型扁藻叶绿体基因的深入探究提供理论支持。
到目前为止,psbA基因在多种植物和藻类中都有研究,但是在亚心型扁藻里却鲜有报道。本文克隆得到亚心型扁藻叶绿体基因psbA序列,一方面为研究藻类psbA基因的特点及编码序列提供理论依据,另一方面为实现不同藻类之间甚至藻类和高等植物之间的叶绿体基因工程提供序列重组的参考。
参考文献:
[1]韩立中, 曾庆波, 孙 丽, 等. 亚心型扁藻培养技术要点[J]. 天津水产, 2006(04): 40-22.
[2]张华军, 李元广, 魏晓东, 等. 亚心形扁藻培养基的筛选与优化[J]. 海洋科学, 2003, 27(5): 37-41.
[3]刘 艳, 杨海波, 于 媛, 等. 扁藻多糖的性质及结构特征的研究[J]. 生物学杂志, 2007, 24(3): 29-31.
[4]李 坤, 李 琳, 侯和胜, 等. Cu2+, Cd2+, Zn2+对两种单胞藻的毒害作用[J]. 应用与环境生物学报, 2002, 8(4): 395-398.
[5]Guan Y F, Deng M C, Yu X J, et al. Two-stage photo-biological production of hydrogen by marine green algaePlatymonassubcordiformis[J]. Biochemical Engineering Journal, 2004, 19(1): 69-73.
[6]Hirose T, Sugiura M. Cis-acting elements and trans-acting factors for accurate translation of chloroplastpsbAmRNAs: development of an in vitro translation system fromTobaccochloroplasts[J]. The EMBO Journal, 1996, 15(7): 1687.
[7]晁岳恩, 吴政卿, 杨会民, 等. 11种植物psbA基因的密码子偏好性及聚类分析[J]. 核农学报, 2011, 25(5): 927-932.
[8]朱清华, 张学成, 李玉晖. 冈村枝管藻叶绿体psbA基因的克隆及序列分析[J]. 海洋科学, 2009, 33(9): 20-24.
[9]吴晓微, 孙 雪, 陆开形, 等. 小球藻psbA基因的克隆与序列分析[J]. 水产科学, 2008, 27(7): 360-362.
[10]钟珍萍, 吴乃虎. 植物光合基因psbA的结构及表达调控综述[J]. 福建农业大学学报, 1997, 26(3): 257-261.
[11]Wright F. The ‘effective number of codons’ used in a gene[J]. Gene, 1990, 87(1): 23-29.
[12]李 平, 白云凤, 冯瑞云, 等. 籽粒苋苹果酸酶 (NAD-ME) 基因密码子偏好性分析[J]. 应用与环境生物学报, 2011, 17(1): 12-17.
[13]Sharp P M, Li W H. An evolutionary perspective on synonymous codon usage in unicellular organisms[J]. Journal of Molecular Evolution, 1986, 24(1-2): 28-38.
[14]Wong G K, Wang J, Tao L, et al. Compositional gradients inGramineaegenes[J]. Genome Research, 2002, 12(6): 851-856.
