自养条件下高氯酸盐降解细菌群落研究
2014-03-17谢宇轩关翔宇于丽莎刘菲
谢宇轩关翔宇于丽莎刘菲
(1.中国地质大学水资源与环境学院,北京 100083;2.中国地质大学海洋学院,北京 100083)
自养条件下高氯酸盐降解细菌群落研究
谢宇轩1关翔宇2于丽莎1刘菲1
(1.中国地质大学水资源与环境学院,北京 100083;2.中国地质大学海洋学院,北京 100083)
旨在研究自养条件下以氢气作为电子供体高氯酸根离子(ClO4-)的微生物降解机制,利用HiSeq 2000对微生物群落结构及多样性进行高通量测序及分析。结果表明,添加氢气的HD(hydrogen degradation)体系将10 mg/L ClO4-降至检出限以下共经历71 d。ClO4-完全降解后HD体系中总细菌的相对丰度为84.96%,绝大多数降解细菌分布的变形菌门相对丰度为68.11%,占总细菌数的比例达到80.16%。其中属于β-变形杆菌纲的ClO4-代表性降解细菌(perchlorate-reducing bacteria,PRB)Dechloromonas的相对丰度为2.7%,另一种Azospira为3.1%。通过KEGG注释对菌种功能进行分析。参与糖代谢的基因相对丰度为4.75%,参与能量代谢的基因相对丰度为3.35%,其中参与氮循环的基因相对丰度为0.72%,参与氯代物降解和氯循环的基因相对丰度为0.83%,补充氢气对反应体系内生物群落结构变化起到选择纯化作用,使优势菌群趋于单一。
高氯酸根离子(ClO4-) 电子供体 氢气 细菌群落结构 高通量测序
作为一种环境污染物,高氯酸根(ClO4-)最早于1997年被美国加利福尼亚州饮用水卫生监测部门发现,当地278个检测井的109口中有高氯酸盐的检出(包括地下水和地表水)[1,2]。至2007年美国已有超过400个位点的地下水,地表水,饮用水和土壤中都有高氯酸盐污染的检出发现。目前已
有报道的高氯酸盐污染最大质量浓度,饮用水中为811 μg/L;地下水中为3.7×106μg/L,地表水中为1.2×105μg/L[3]。2011年美国EPA制定的最新规划将高氯酸盐正式纳入饮用水安全标准(http://water. epa.gov/drink/contaminants/unregulated/perchlorate. cfm)并将于随后的24个月内公布最大污染界限值(MCL)[4]。
高氯酸盐的分子结构决定了其化学性质较稳定,通常情况下很难去除[5-7]。目前常用的去除高氯酸盐的方法有离子交换法(IX)和生物降解法。生物降解作用的本质是由参与高氯酸盐降解细菌(perchlorate reducing bacteria,PRB)的活性决定的。添加无机电子供体实现自养条件下高氯酸盐降解的研究及应用相对较多的是H2,其理论依据是许多自养型细菌可以利用氢作为电子供体满足自身生长需求。自然界中的高氯酸盐去除往往并非单一菌种的作用,而是多种细菌共同作用,而自养型细菌的种群密度正是决定氢还原体系下高氯酸盐降解速率的关键[8-10]。参与高氯酸盐降解的细菌可分为4类:高氯酸盐降解细菌,如Dechloromonas和Dechlorosoma(Azospira);氯酸盐降解细菌(chlorate reducing bacteria,CRB),如Ideonella dechloratans;高浓度氯酸盐耐受条件下的高氯酸盐降解细菌(high chlorate accumulating perchlorate reducing bacteria,HCAP),如DechloromonasPC1;一些脱氮细菌(Denitrifying bacteria),如Pseudomonas[11]、Rhodobacter sphaeroides[12]等。最常见的参与高氯酸盐降解作用的细菌是Azospira[13]和Dechloromonas[14]。以往的研究往往针对单一菌种或在特定的培养条件下进行,少有对复杂介质中生物降解高氯酸盐菌种及功能的分析。
本研究选用常见的厌氧生物反应器作为高氯酸盐降解的培养基质,添加氢气作为电子供体支持ClO4
-的完全降解,采用高通量测序的方法对该体系内菌种的结构和功能进行分析。该方法可以更详尽地把握环境中的微生物群落结构及其功能,进一步反映电子供体的存在对反应基质的选择性影响,阐释混合体系中高氯酸盐生物降解作用的本质,旨在为原位或异位高氯酸盐生物修复技术的强化提供理论支持和现实依据。
1 材料与方法
1.1 高氯酸盐降解试验体系的设置
用于高氯酸盐降解的厌氧生物反应器培养条件为pH值6.92-7.35,温度30℃,测定溶解氧(DO)质量浓度为0.46 mg/L[15]。生物培养采用的基础营养液成分参考王蕊等[16]研究内容。
高氯酸盐降解试验体系的设置如下所示:
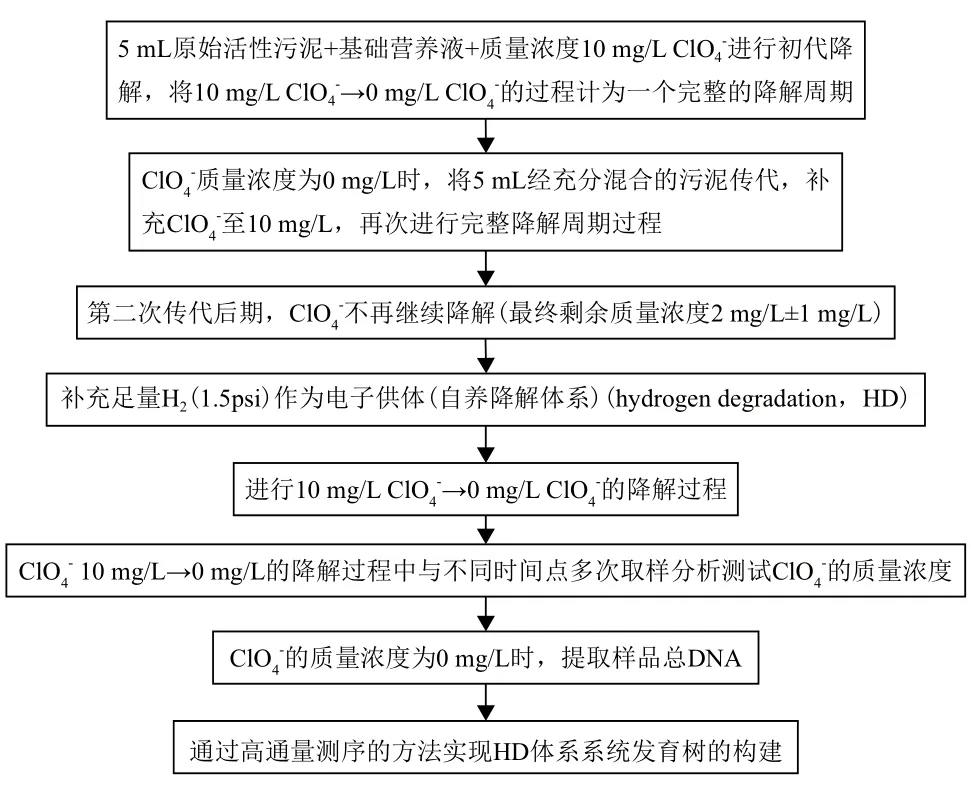
以上各反应体系的条件为:DO<2.0 mg/L,pH:7.0-8.0,温度:30℃。
1.2 微生物数据分析方法
1.2.1 样品总DNA的提取 使用细菌基因组提取试剂盒(天根,TIANGEN,北京)对HD体系降解完毕的样品进行总DNA提取,产物保存于-20℃冰箱以备后续生物群落分析研究。
1.2.2 高通量测序分析 通过高通量测序(High-Throughput Sequencing)的方法获得生物群落的信息。通过Stratagene Mx3000P Real-time PCR Cycler(Agilent,Santa Clara,CA,USA)分析仪进行定量分析[Qubit Fluorometer(Invitrogen,Life Technologies,Grand Island,NY,USA)作为染料]。使用Illumina HiSeq 2000高通量测试仪上机测序(Sequencing),所得的碱基对末端序列(PE)需进行筛选,排列(6-40 bp)和拼接(Data Analysis)。整理所得序列与BLASTX(Basic Local Alignment Search Tool)进行比对和注释,并与NCBI(National Center for Biotechnology Information,http://www.ncbi.nlm.nih.gov/)的NR
(non-redundant)数据库进行比对,最后通过MEGANv4.0软件构建HD体系系统发育树。
2 结果
2.1 HD体系ClO4-降解与菌种多样性分析
研究中HD体系ClO4-初始质量浓度为10 mg/L。反应开始较长一段时间(约15 d)内,排除仪器测试误差等因素外,ClO4-的质量浓度基本无变化。反应进行至20 d后,经历了降解延滞期的ClO4-质量浓度缓慢下降,至ClO4-质量浓度达到2 mg/L左右时,如图1所示,60 d后ClO4-的降解出现加速的过程,并于70 d时降至检出限以下。ClO4-降解完全后,对体系内生物群落构成进行系统发育树的构建分析,高通量测序所得数据大小为1.13 GB。MEGAN(version 4.70.4)软件比对结果表明,HD体系中总细菌相对丰度为84.96%,HD体系系统发育树如图2所示。
变形杆菌门细菌在总细菌群落中所占比例最大,达到80.16%。属于变形杆菌门的细菌结构比例如图3所示。
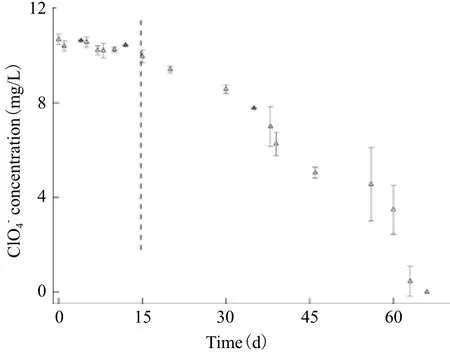
图1 HD体系高氯酸盐降解图
属于α-变形杆菌纲的细菌占体系内总细菌数的比例为18.62%,β-变形杆菌纲的细菌相对丰度为34.17%,占总细菌数的比例为40.22%,而γ-变形杆菌纲的细菌占总细菌数的比例为11.12%。除δ-proteobacteria变形杆菌纲所占比例极少外,HD体系中的α-变形杆菌纲、β-变形杆菌纲和γ-变形杆菌纲的细菌分布相对较均匀。其中代表性的高氯酸盐(氯酸盐)降解细菌,如Dechloromonas,Azospira和一部分可以参与ClO4-降解的反硝化细菌如Pseudomonas[17-20]等均有检出,且相对丰度较大。该体系内主要以β-变形杆菌纲的细菌为主。
2.2 HD体系基因功能性分析
HD体系降解完毕后,通过高通量测序和KEGG注释的功能分析可知,HD体系中参与物质代谢过程的基因总相对丰度为17.67%。维持生物体正常生命活动和能量代谢常见的几种重要功能基因的相对丰度较大,如参与氨基酸类物质代谢的基因相对丰度最大为5.46%,其次为参与生物体内糖代谢的基因,其相对丰度为4.75%,参与能量代谢的基因相对丰度为3.35%。对于环境中能够降解某些特定污染物的功能基因分析结果表明,可参与氮循环的基因相对丰度为0.72%,参与氯循环的基因丰度为0.83%。部分基因KEGG功能分析结果如图4所示。
3 讨论
研究所选用的降解基质本身经传代过程验证,具有降解ClO4-的潜在能力,说明该厌氧反应器自身的细菌群落组成中即有高氯酸盐降解细菌的存在。
添加氢气作为电子供体支持高氯酸盐降解的生物修复过程较缓慢,整个反应周期共需要约71 d。王蕊等[16]研究表明,降解初始质量浓度为9 mg/L的ClO4-,ClO4-初期的降解速率低,当ClO4-降至2 mg/L时,降解过程可提速2倍,延滞期的时长占整个降解周期的约1/4,此现象与Nozawa-Inoue等[21]利用氢气作为电子供体降解高氯酸盐反应初期延滞期较长(41 d)的特点一致。造成HD体系延滞期较长的原因:(1)由于H2本身在水中的溶解度很低(0.08 mg/L at 5% H2)。根据亨利定律,在等温等压的条件下,某种气体在溶液中的溶解度与液面上该气体的平衡压力成正比。本研究中当通入气体流量一致时,H2在溶液中的溶解度应与液面上该气体的平衡压力成正比。因此,通入H2的时间越长,则H2的溶解度应当越高,故反应初期H2在反应溶液中的低溶解度会影响自养型降解细菌的适应和发育;(2)由于HD体系的培养基质是由原始厌氧生物反应器传代而来,整个反应体系内的细菌相当于经历了一次异养降解过程,导致系统初始细菌种群密度有所变化,故将此体系转为由H2提供电子供体的自养降解环境时,细菌的适应期会更长,从而对后续的试验研究造成影响;(3)当H2存在时,反
应体系内同时发生电子供体的竞争作用。NO3-、O2、ClO4-三者间电子利用的优先级顺序为:O2>NO3->ClO4-[22,23]。综合以上3点,HD体系中ClO4-降解初期反应速率很低。随着反应的进行,经历延滞期的细菌开始加速高氯酸盐的降解。也有相关研究表明,用Dechloromonassp. PC1细菌降解高氯酸盐,反应后期高氯酸盐降解速率增加,有一部分原因是当体系内的ClO4-质量浓度变低时,高氯酸盐降解细菌在整个生物群落中所占的比例增加,并逐渐成为体系内的优势种群[18,24]。该降解过程最终降解周期为65 d。2000年Miller等[8]利用污水处理厂的污泥作为菌源和反应基质,同样通过通入H2作为电子供体,进行高氯酸盐的降解柱试验研究。145 d的时间内,高氯酸盐降解率达45%。当利用H2作为电子供体进行高氯酸盐的降解研究时,其反应时间往往较
长,是由于自养细菌本身对能量的利用方式在与异养型细菌共存竞争时往往处于劣势。
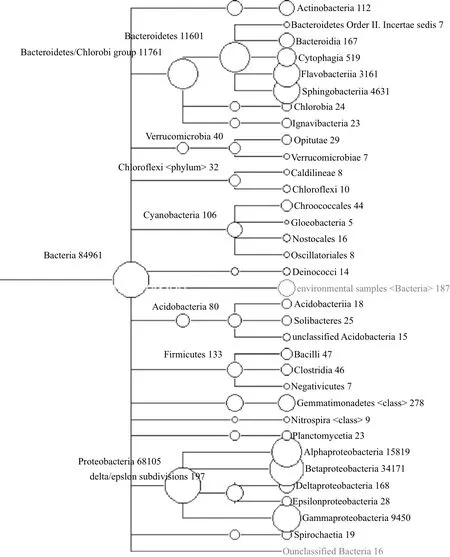
图2 HD体系基于reads序列的系统发育树
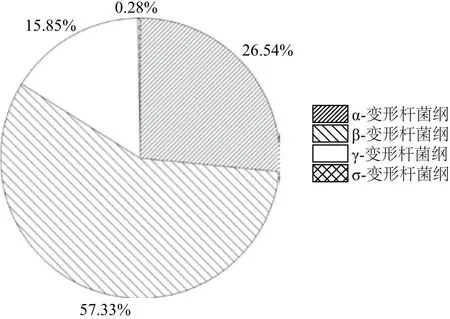
图3 HD降解体系中变形杆菌门细菌分布比例图
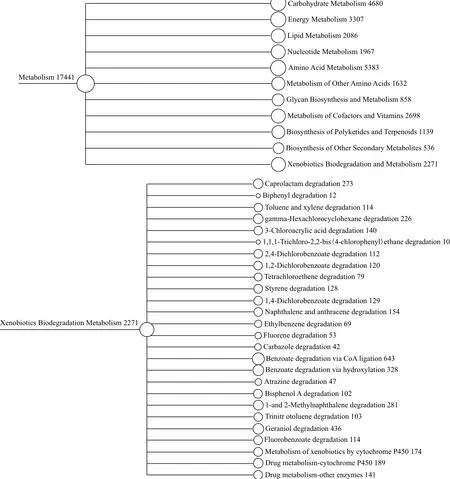
图4 KEGG注释功能分析图
HD体系中总细菌相对丰度为84.96%,导致该体系内菌种丰度较大的两个因素:一是由于添加氢气促进了反应体系内所有可利用氢气作为电子供体的细菌生长,另一方面则是由于该反应的降解周期较长且用于生物多样性分析的样品取自ClO4-刚刚降解完毕,培养时长会导致该体系内菌种数量明显增加。大部分的高氯酸盐降解细菌(PRB)分布于β-变形杆菌纲,而氯酸盐降解细菌(CRB)则主要分布于γ-变形杆菌纲[12]。HD体系中Dechloromonas的相对丰度为2.7%,证明了作为高氯酸盐降解最具代表性的细菌,Dechloromonas广泛存在于自养条件下的高氯酸盐降解体系中[7,17,18,25,26]。另一种常见的高氯酸盐降解细菌Azospira的相对丰度也有3.1%。有文献[27-29]表明,许多已经分离得到的高氯酸盐降解菌都同时具有脱氮作用。本研究HD体系中也有较多既参与高氯酸盐还原又可以参与反
硝化过程的细菌,如Pseudomonas[11]、Rhodobacter sphaeroides[12]和Acinetobacter[29]的检出,其相对丰度分别0.01%、4.4%和3.5%,证明HD体系由于电子供体氢气的加入,不仅促进了反应体系内某些代表性的高氯酸盐降解细菌的生长,同时也促进了其他一些可以参与高氯酸盐(或氯酸盐)还原过程的细菌发育。对HD体系内检出基因的功能分析结果证明参与氮循环的基因丰度和参与降解氯代物的基因相对丰度比例较为相似,推测这些参与氮类物质和氯循环的细菌共同作用实现了HD体系中ClO4-的还原降解。虽然通过KEGG注释分析功能基因具有一定的局限性,但亦能说明复杂条件下高氯酸盐污染的降解是多种细菌共同作用的结果。并且由于添加了氢气使得厌氧生物反应器本身的菌种结构发生改变,成为适合高氯酸盐降解的体系。
4 结论
高氯酸盐污染在添加了电子供体氢气后可以实现完全降解。10 mg/L ClO4-降至检出限以下共经历71 d,降解速率较慢。ClO4-降解完全后,HD体系中总细菌相对丰度为84.96%,大多高氯酸盐降解细菌分布的变形杆菌门细菌占总细菌的比例较大,达到80.16%。如Azospira和Dechloromonas等代表性的高氯酸盐降解细菌相对丰度也有一定的检出。此外,可同时参与高氯酸盐降解和反硝化作用的细菌如Pseudomonas相对丰度为4.4%。功能分析表明高氯酸盐的降解是多种细菌共同作用的结果。额外添加电子供体使HD体系内的细菌种群结构简单化,参与某种特定污染物降解的细菌所占比例增加,从而使混合降解基质本身的功能趋于专一。
[1] Urbansky ET. “Perchlorate chemistry:implications for analysis and remediation.”[J]. Bioremed J, 1998, 2(2):81-95.
[2] Tikkanen MW. Development of a drinking water regulation for perchlorate in California[J]. Analytica Chimica Acta, 2006, 567(1):20-25.
[3] Bardiya N, Bae JH. Dissimilatory perchlorate reduction:A review[J]. Microbiological Research, 2011, 166(4):237-254.
[4] Richardson SD, Ternes TA. Water analysis:emerging contaminants and current issues[J]. Anal Chem, 2011, 83(12):4614-4648.
[5] Motzer WE. Perchlorate:problems, detection, and solutions[J]. Environmental Forensics, 2001, 2(4):301-311.
[6] Sorial GA. The perchlorate dilemma in drinking water[J]. Journal of Environmental Engineering, 2004, 130(1):1-2.
[7] Stetson SJ, Wanty RB, Helsel DR, et al. Stability of low levels of perchlorate in drinking water and natural water samples[J]. Analytica Chimica Acta, 2006, 567(1):108-113.
[8] Miller JP, Logan BE. Sustained perchlorate degradation in an autotrophic, gas-phase, packed-bed bioreactor[J]. Environ Sci Technol, 2000, 34(14):3018-3022.
[9] Wu D, He P, Xu X, et al. The effect of various reaction parameters on bioremediation of perchlorate-contaminated water[J]. Journal of Hazardous Materials, 2008, 150(2):419-423.
[10] Shrout JD, Scheetz TE, Casavant TL, et al. Isolation and characterization of autotrophic, hydrogen-utilizing, perchloratereducing bacteria[J]. Appl Microbiol Biotechnol, 2005, 67(2):261-268.
[11] Coates JD, Achenbach LA. Microbial perchlorate reduction:rocket fuelled metabolism[J]. Nat Rev Microbiol, 2004, 2(7):569-580.
[12] Roldan MD, Reyes F, Morenovivian C, et al. Chlorate and nitrate reduction in the phototrophic bacteria Rhodobacter capsulatus and Rhodobacter sphaeroides[J]. Curr Microbiol, 1994, 29(4):241-245.
[13] Tan Z, Reinhold-Hurek B. Dechlorosoma suillum Achenbach et al. 2001 is a later subjective synonym of Azospira oryzae Reinhold-Hurek and Hurek 2000[J]. Int J Syst Evol Microbiol, 2003, 53(4):1139-1142.
[14] Wolterink A, Kim S, Muusse M, et al. Dechloromonas hortensis sp. nov. and strain ASK-1, two novel(per)chlorate-reducing bacteria, and taxonomic description of strain GR-1[J]. Int J Syst Evol Microbiol, 2005, 55(5):2063-2068.
[15] 李广.预处理后剩余污泥中温两相厌氧消化效能研究[D],北京:中国地质大学, 2012.
[16] 王蕊. 生物法去除地下水中高氯酸盐的实验研究[D].北京:中国地质大学, 2012.
[17] Bruce RA, Achenbach LA, Coates JD. Reduction of(per)chlorate by a novel organism isolated from a paper mill waste[J]. Environ Microbiol, 1999, 1(4):319-329.
[18] Coates JD, Michaelidou U, Bruce RA, et al. The ubiquity and diversity of dissimilatory perchlorate reducing bacteria[J]. Appl Environ Microbiol, 1999, 65(12):5234-5241.
[19] Nerenberg R, Kawagoshi Y, Rittnann BE. Kinetics of a hydrogenoxidizing, perchorate-reducing bacterium[J]. Water Research, 2006, 40(17):3290-3296.
[20] Wallace W, Ward T, Breen A, et al. Identification of an anaerobic bacterium which reduces perchlorate and chlorate as Wolinella succinogenes[J]. Ind Microbiol Biotechnol, 1996, 16(1):68-72.
[21] Nozawa-Inoue M, Scow KM, Rolston DE. Reduction of perchlorate and nitrate by microbial communities in vadose soil[J]. Appl Environ Microbiol, 2005, 71(7):3928-3934.
[22] Nerenberg R, Rittmann BE, Najm I. Perchlorate reduction in a hydrogen-based membrane-biofilm reactor[J]. J Am Water Works Assoc, 2002, 94(11):103-114.
[23] Tang Y, Zhao H, Marcus AK, et al. A steady-state biofilm model for simultaneous reduction of nitrate and perchlorate, part 1:model development and numerical solution[J]. Environ Sci Technol, 2012, 46(3):1598-1607.
[24] Nerenberg R, Kawagoshi Y, Rittnann BE. Microbial ecology of a perchlrate-reducing hydrogen-based membrane biofilm reactor[J]. Water Research, 2008, 42(4-5):1151-1159.
[25] Logan BE, Zhang H, Mulvaney P, et al. Kinetics of perchlorateand chlorate-respiring bacteria[J]. Applied and Environmental Microbiology, 2001, 67(6):2499-2506.
[26] Zhang H, Bruns MA, Logan BE. Perchlorate reduction by a novel chemolithoautotrophic hydrogen-oxidizing bacterium[J]. Environ Microbiol, 2002, 4(10):570-576.
[27] Choi H, Silverstein JA. Inhibition of perchlorate reduction by nitrate in a fixed biofilm reactor[J]. Journal of Hazardous Materials, 2008, 159(30):440-445.
[28] Okeke BC, Giblin T, Frankenberger WT Jr. Reduction of perchlorate and nitrate by salt tolerant bacteria[J]. Environ Pollut, 2002, 118(3):357-363.
[29] Krauter P, Daily B Jr, Dibley V, et al. Perchlorate and nitrate remediation efficiency and microbial diversity in a containerized wetland bioreactor[J]. Int J Phytoremediation, 2005, 7(2):113-128.
(责任编辑 马鑫)
The Study of the Structure of Perchlorate(ClO4-)-degrading Bacterial Communities Under Autotrophic Conditions
Xie Yuxuan1Guan Xiangyu2Yu Lisha1Liu Fei1
(1. School of Water Resources and Environment,China University of Geosciences,Beijing 100083;2. School of Ocean Sciences,China University of Geosciences,Beijing 100083)
For the purpose of further investigating the biological degradation under an autotrophic condition and well understanding the microbial community structures in a complex environment, hydrogen was used as an electron donor to completely reduce perchlorate(ClO4-)in this study. The composition of microbial communities after degradation was analyzed via the construction of a cloning library by using the High-Throughput Sequencing method(HiSeq 2000). 71 days were needed to completely degrade 10 mg/L ClO4-. Microbial phylogenic analysis of HD(hydrogen degradation)after degradation indicated that the relative abundance of total bacteria in the HD was 84.96% whereas the relative abundance of Proteobacteria was 68.11%, whose percentage accounting for the total bacteria reached to 80.16%. The relative abundance of Dechloromonas which is representative in PRB was 2.7% in the HD. Simultaneously, the relative abundance of Azospira was 3.1%. KEGG was used to analyze the function of bacteria in HD. The relative abundance of genes which engaged in carbohydrate metabolism was 4.75%, and the genes included in energy metabolism was 3.35%, whereas the genes participated in nitrogen cycle was 0.72%, and the genes involving chloride transformation was 0.83%. It was demonstrated that degradation of ClO4-in a complicated condition was achieved by various kinds of microbes rather than a single one. Adding hydrogen as an electron donor to change microbial community played a role in the purification or selection process in the system, which allowed the complex systems to have the specific capacity to remove given contaminates.
Perchlorate(ClO4-) Electron donor Hydrogen The composition of microbial communities High-Throughput Sequencing
2013-10-31
国家自然科学基金项目(41272268),中国地质调查局项目(1212011121171),中央高校基本科研业务费专项资金(2652012071)
谢宇轩,男,博士研究生,研究方向:地下水污染生物修复;E-mail:yuxuanxie@163.com
关翔宇,女,博士,讲师,研究方向:环境微生物;E-mail:guanxy@cugb.edu.cn
