异育银鲫消化道微生物群落对恢复投喂的响应
2014-03-17李星浩颜庆云胡红娟李金金王纯余育和
李星浩颜庆云胡红娟李金金王 纯余育和
(1. 中国科学院水生生物研究所, 中国科学院水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
异育银鲫消化道微生物群落对恢复投喂的响应
李星浩1,2颜庆云1胡红娟1,2李金金1,2王 纯1,2余育和1
(1. 中国科学院水生生物研究所, 中国科学院水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
以实验室养殖的异育银鲫(Carassius auratus gibelio)“中科3号”成鱼为对象, 利用PCR-DGGE技术比较研究了其经饥饿与恢复投喂后消化道微生物多样性的变化。实验共检测到23条谱带, 其中饥饿组、投喂组谱带数分别为18条和17条(共有谱带12条), 投喂组大部分谱带亮度高于饥饿组。基于PCR-DGGE所得到的谱带进行的 CA (Correspondence Analysis)分析和聚类都表明饥饿组与投喂组消化道微生物存在明显不同。结果表明, 在充分饥饿处理后, 恢复饲料投喂可导致消化道微生物种类减少及微生物平均密度的增加,同时对消化道微生态系统内微生物种类组成有显著影响。研究为进一步阐述消化道微生物功能基因奠定了基础, 也为水产鱼类肠道菌群研究提供一些基础数据。
异育银鲫; 消化道微生物群落; PCR-DGGE; 肠道微生态系统
肠道对于动物来说就像是内在生命线, 而肠道内微生物影响着肠道众多功能的发挥, 因此关于肠道内微生物的研究近几年也越来越被重视[1]。已有研究表明, 鱼类消化道微生物对鱼的免疫、营养等都有重要影响[2,3], 消化道内微生物群落的平衡稳定可以促进鱼体健康的生长。鱼类消化道微生物在影响着宿主鱼的同时, 其组成也受着宿主自身与外界因素的影响[4—6], Romero等发现银大麻哈鱼(Oncorhynchus keta)消化道微生物群落在第一投喂阶段后才稳定[7], 而李学梅等对室内养殖的斑点叉尾、银鲫和异育银鲫的消化道微生物进行了比较,发现不同种鱼的消化道微生物的种类组成有显著差异[8], 同时尹军霞等研究发现同种鱼消化道不同区段的微生物组成也存在很大不同[9], 另外, Margolis早在 1952年用传统培养法对经过饥饿处理的大头鱼(Ameiurus nebulosus)及斑点鳟(Salaeliruts fontinali)的消化道细菌进行了研究, 发现饥饿使消化道微生物种类减少[10]。以上研究表明消化道微生物与宿主鱼之间存在密切关系。对于外界条件变化,消化道微生物自身是否存在响应机制呢?本研究选用消化道微生物群落已相对稳定的异育银鲫“中科3号”[11]成鱼作为研究对象, 并将其消化道作为相对封闭的微型生态系统来研究[12], 旨在进一步阐述饲料这一鱼类营养源在作为消化道微生态系统外界干扰时对其内部物种组成的影响, 为今后研究消化道微生物功能基因指明了方向, 同时为水产鱼类肠道菌群研究提供一些基础数据。
1 材料与方法
1.1 实验材料与样品采集
实验鱼饲养于中国科学院水生生物研究所鱼类生理生态学学科组循环水系统中, 水温为 24—28℃,期间投喂人工配合饲料, 待性成熟后进行饥饿处理30d, 随机抽取3尾同规格的鱼, 在编号A、B和C后解剖取样, 取样时用 75%的酒精擦拭肠外壁, 分前肠、中肠和后肠取样, 取得 9份样品分别编号H1-H9 (H1、H2、H3代表A的前肠、中肠和后肠, H4-H6及H7-H9对应代表B和C的前肠、中肠和后肠), 保存用于微生物 DNA的提取; 再给剩下的鱼投喂相同人工配合饲料, 25d后再次随机抽取3尾同规格的鱼编号D、E和F, 解剖取样得9份样品, 分别编号 F1-F9(F1、F2、F3代表D的前肠、中肠和后肠, F4-F6及F7-F9对应代表E和F的前肠、中肠和后肠), 保存用于微生物DNA的提取。
1.2 消化道微生物总DNA的提取
实验采用传统的酚-氯仿法提取肠道微生物总DNA[8,13], 即: 向每管样品中加入 1.2 mL裂解液(0.5% SDS; 10 mmol/L Tris-Cl, pH8.0; 10 mmol/L EDTA, pH8.0; 100 mmol/L NaCl; 蛋白酶K 100 μg/mL; RNase A, 50 μg/mL), 37℃水浴 1h后, 加入 PK (20 mg/mL) 6 μL, 55℃过夜(12h, 至清亮); 取上清液进行DNA抽提, DNA在4℃下经24h沉淀后, 离心干燥并加入100 mL无菌水溶解, –20℃保存备用;总DNA 用0.7%的琼脂糖凝胶(含EB)进行电泳以检测提取效果[14,15]。
1.3 PCR-DGGE分析
采用细菌16S rRNA基因V3区通用引物(F357-GC和R518)对所得18个样品中的肠道细菌DNA进行 PCR扩增, 将扩增得产物进行DGGE分析[16,17],变性剂浓度范围为40%—70%, 凝胶在110V下经过12h的电泳后紫外照相, 由于投喂组F5和F6未能得到相应的PCR扩增产物, 样品F5和F6不参与数据分析, 使用Quantity One (Bio-Rad Laboratories, CA, USA)软件对谱带进行分析。
1.4 数据分析
在得到各泳道及谱带亮度等详细信息并转化为0/1矩阵后, 使用Canoco 4.5对数据进行CA (Correspondence Analysis)分析, 通过R 3.0.0对各泳道数据进行基于 Jaccard指数的相似性分析并绘制聚类图[18], 通过Microsoft Excel 2010对谱带数量及亮度值进行分析并绘图, 同时计算谱带亮度值的方差值以比较两组中各物种分布密度的变异程度大小。
2 结果
2.1 消化道微生物群落PCR-DGGE指纹分析
针对细菌16S rRNA基因进行的PCR-DGGE指纹分析共检测到 23条不同的谱带(图 1), 其中饥饿组谱带数为18条, 投喂组谱带数为17条, 两组共有谱带12条, 由图上可以看出饥饿组特异性谱带稳定且与投喂组存在明显差别, 以实验鱼为操作单元,基于全肠微生物种类分布的相似性聚类分析表明(图2), 饥饿组A、B和C聚为一枝, D、F聚为一枝。

图1 16S rDNA变性梯度凝胶(DGGE)电泳图Fig. 1 DGGE patterns of 16S rDNA
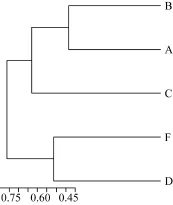
图2 16S rDNA图谱相似性比较Fig. 2 Comparison of similarity based on DGGE fingerprints of 16S rDNA
2.2 消化道微生物群落结构相似性
CA结果显示除样品F9外, 投喂组(F1- F4, F7, F8)与饥饿处理组(H1- H9)消化道存在明显差异, 投喂组样品主要存在于第二、三象限, 饥饿组主要存在于第一象限, 且投喂组各样品总体聚集较饥饿组更紧密(图3), 表明投喂组各样品间消化道微生物群落种类组成相似性高于饥饿组; 为了更直观的显示各消化道微生物群落的差别, 基于 Jaccard指数相似性聚类结果显示除F1、F2样品外, 饥饿组和投喂组大部分分别聚为两大枝(图4)。

图3 基因16S rDNA指纹图谱的CA分析Fig. 3 Correspondence analysis based on PCR-DGGE fingerprints of 16S rDNA
2.3 消化道微生物群落结构比较
对两组样品中的各个谱带进行比较显示, 饥饿组拥有谱带数为18, 投喂组拥有谱带数为17, 两对照组共同拥有谱带B3、B4、B8、B9、B10、B12、B13、B15、B18、B19、B20和B22, 共计12条(图5); 其中B3、B13、B15和B20在饥饿组和投喂组中出现频率较高, 分别为77.8%与100%、100%与71.4%、77.8%与57.1%、77.8%与71.4%, 属高频率共有谱带; 结果还显示, 饥饿组中不存在的谱带为B11、B14、B16、B17和B21, 投喂组中不存在的条带为B1、B2、B5、B6、B7和B23, 两者存在明显差异。
为更清晰的了解两组中各谱带的存在以及分布情况, 我们计算并比较了两对照组中各条带的平均亮度, 结果显示(图 6), 投喂组 56.5%的谱带平均亮度大于饥饿组, 且投喂组中最大亮度与最小亮度之差(2901.99)远大于饥饿组最大亮度与最小亮度之差(1508.55); 对两组方差值进行计算, 结果显示, 饥饿组方差值(365.79)小于投喂组方差值(885.67), 表明饥饿组各样品间各微生物分布密度的变异程度小于投喂组。

图4 基于16S rDNA指纹图谱的聚类分析Fig. 4 Clustering on the basis of 16S rDNA bands
3 讨论
鱼类消化道作为一个微型生态系统, 其内部生活的微生物对营养物质有适应性反应[19], 同时鱼类肠道的生物性结构差异为生活于其内的微生物造就了不同的生境, 这对其内微生物分布以及扩散有很大的影响[5,20]。对同一种鱼来说, 成鱼的消化道较幼鱼的消化道内环境稳定, 受到水中微生物以及温度等的影响较幼鱼的小[21]。因此, 本研究选用成鱼消化道作为研究肠道微型生态系统的对象, 以饲料作为这一微型生态系统的“干扰”具有很高的可行性。

图5 各谱带在两对照组中出现的频率比较Fig. 5 Comparison based on the frequency of bands in 2 groups

图6 两对照组中各谱带亮度值的比较Fig. 6 Comparison on the intensity of bands between 2 groups
异育银鲫(Carassius auratus gibelio)自1985 年由中国科学院水生生物研究所育成以来, 迅速被众多养殖户以及大众接受[22], 而“中科3号 ”品系目前已成为为我国大宗水产养殖品种, 对该鱼的研究具有很好的实际意义。基于前人的探索, 本研究选用了异育银鲫成鱼作为研究对象, 用饲料作为干扰因素以探索消化道这一微型生态系统中微生物群落对恢复饲料投喂的响应机制。本研究发现充分饥饿后恢复投喂导致肠道微生物发生明显变化, 首先是肠道微生物种类数量下降, 并且 CA分析显示投喂组样品间微生物群落物种组成相似性高于饥饿组, 然而Manuel等认为, 在宏观生态系统中, 更多的能量来源可以支持更多的生物种群的存活[23], 推测本研究这一结果可能与Bitry[24]研究有关, 饲料等营养物质可在一定程度上影响前、中和后肠的差异[25—26], 尽管恢复饲料投喂增加了消化道微型生态系统的能量来源, 但相比于空肠, 恢复投喂后, 同质的饲料充满了投喂组实验鱼的肠道, 在降低了实验鱼前肠、中肠、后肠的“生境”差异并导致微生物种类的下降, 同时还使得使得各肠道样品之间的总差异减少。
在分析比较了两组条带的丰度和亮度后, 发现投喂组样品平均亮度高于饥饿组样品平均亮度, 表明饥饿后恢复投喂使得肠道微生物总体分布密度上升。投喂组中最大亮度(B20)与最小亮度(B18)之差(2901.99)远大于饥饿组最大亮度(B13)与最小亮度(B4)之差(1508.55), 且对比饥饿组方差值(365.79)与投喂组方差值(885.67)表明投喂组肠道样品中微生物的种间密度差异远大于饥饿组, 推测该结果与Kamada等研究有关。Kamada等用小鼠研究表明消化道微生物物种间存在生存竞争关系[27]; 本实验饲料的恢复投喂在增加了能量来源并使得微生物数量增加的同时, 还使得微生物种间竞争加剧, 加速形成“优势种”并导致各微生物密度向两极分化, 微生物物种间竞争强度的增加使得有限的生境内能容纳的微生物种类数量减少, 这一结果在宏观生态系统研究中也得到证实[28,29]。伴随着B20密度在投喂后急剧增加以及B1、B2、B5、B6、B7和B23这五个低亮度条带的消失, 新出现了B11、B14、B16、B17和B21这五个条带平均亮度较高的谱带, 其代表的是肠道内物种的更替, 推测其与饲料中携带的微生物有关[30,31]。
以上研究结果表明, 充分饥饿后恢复投喂会导致肠道微生物发生明显变化, 本实验以恢复投喂饲料作为这一微型生态系统的“外界干扰”, 在其带入新的微生物同时, 使得微生物密度增加并导致种间竞争加剧, 说明在鱼类消化道中存在一个完整的微型生态系统。本研究为下一步研究这一现象作用机制是否与微生物或鱼肠道本身的功能基因有关指明了方向, 也为研究鱼类肠道内具有特定功能的菌群奠定了基础。
[1] Mueller K, Ash C, Pennisi E, et al. The gut microbiota [J]. Science, 2012, 336: 1245
[2] Gómez G D, Balcázar J L. A review on the interactions between gut microbiota and innate immunity of fish [J]. FEMS Immunology and Medical Microbiology, 2008, 52: 145—154
[3] Lü X R, Xiao K Y. Research of intestinal flora of fish [J]. Jiangxi Fishery Sciences, 2008, 2: 12—18 [吕欣荣, 肖克宇.鱼类肠道菌群的研究现状. 江西水产科技, 2008, 2: 12—18]
[4] Wang H N, He M Q, Liu P, et al. Study on the intestinal microflora of carp in freshwater culture ponds [J]. ActaHydrobiologica Sinica, 1994, 18(4): 354—359 [王红宁, 何明清, 柳苹, 等. 鲤肠道正常菌群的研究. 水生生物学报, 1994, 18(4): 354—359]
[5] John F R, Buck S S, Jeffrey I G. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota [J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(13): 4596—4601
[6] Bell T, Ager D, Song J I, et al. Larger islands house more bacterial taxa [J]. Science, 2005, 308: 1884
[7] Romero J, Navarrete P. 16S rDNA-Based analysis of dominant bacterial populations associated with early life stages of coho salmon (Oncorhynchus kisutch) [J]. Microbial Ecology, 2006, 51: 422—430
[8] Li X M, Yu Y H, Xie S Q, et al. PCR-DGGE fingerprinting Analysis on intestinal microbial community of three indoor rearing fishes [J]. Acta Hydrobiologica Sinica, 2011, 35(3): 423—429 [李学梅, 余育和, 解绶启, 等. 三种室内饲养鱼类肠道微生物群落PCR-DGGE指纹分析. 水生生物学报, 2011, 35(3): 423—429]
[9] Yin J X, Zhang J L, Shen W Y, et al. Study on the Relation between eating patterns of fishes and intestinal microflora [J]. Fisheries Science, 2004, 23(3): 4—6 [尹军霞, 张建龙, 沈文英, 等. 鱼食性与肠道菌群关系的初步研究. 水产科学, 2004, 23(3): 4—6]
[10] Margolis L. The effect of fasting on the bacterical flora of the intestine of fish [J]. Journal of the Fisheries Research Board of Canada, 1953, 10(2): 95—104
[11] Gui J F, Zhou L. Genetic basis and breeding application of clonal diversity and dual reproduction modes in polyploid Carassius auratus gibelio (review) [J]. Science China Life Sciences, 2010, 53(4): 409—415
[12] Costello E K, Stagaman K, Dethlefsen L, et al. The application of ecological theory toward an understanding of the human microbiome [J]. Science, 2012, 336: 1266—1262 [13] Yang D J, Wu J, Liu Y, et al. A rapid method for total DNA extraction from microorganisms of intestinal tract [J]. Chinese Journal of Microecology, 2006, 18(2): 91—93 [杨德君, 吴襟, 刘毅, 等. 一种快速提取肠道微生物总DNA的方法, 中国微生态学杂志, 2006, 18(2): 91—93]
[14] Yan Q Y, Yu Y H, Zhang T L, et al. PCR-DGGE fingerprinting analysis on intestinal contents of filter-feeding fishes (Hypophthalmichthys molirix and Aristicthys nobilis) [J]. Journal of Fisheries of China, 2009, 33(6): 972—979 [颜庆云, 余育和, 张堂林, 等. 滤食性鲢、鳙肠含物PCR-DGG指纹分析. 水产学报, 2009, 33(6): 972—979]
[15] Yan Q Y, Yu Y H, Feng W S, et al. Genetic diversity of plankton community as depicted by PCR-DGGE fingerprinting and its relation to morphological composition and environmental factors in Lake Donghu [J]. Microbial Ecology, 2007, 54(2): 290—297
[16] Li X M, Yu Y H, Feng W S, et al. Host species as a strong determinant of the intestinal microbiota of fish larvae [J]. The Journal of Microbiology, 2012, 50: 29—37
[17] Yu Y H, Zhang W J, Yan Q Y. The feasibility for application of DNA fngerprinting to community-level life system [J]. Acta Hydrobiologica Sinica, 2004, 28(5): 457—463 [余育和,张文静, 颜庆云. DNA指纹分析技术在群落级生命系统应用的可能性. 水生生物学报, 2004, 28(5): 457—463]
[18] Ma Z X, Shen H, Ning Y Z, et al. Community characteristics of soil ciliates in Taohuagou Forest Park in Xiaolongshan Mountains of Gansu Province [J]. Chinese Journal of Ecology, 2008, 27(2): 208—212 [马正学, 申海, 宁应之,等. 甘肃小陇山桃花沟森林公园土壤纤毛虫的群落特征.生态学杂志, 2008, 27(2): 208—212]
[19] Feng X, Wu Z X, Zhu D M, et al. Study on digestive enzyme-producing bacteria from the digestive tract of Ctenopharyngodon idellus and Carassius auratus gibelio [J]. Freshwater Fisheries, 2008, 38(3): 51—57 [冯雪, 吴志新,祝东梅, 等. 草鱼和银鲫肠道产消化酶细菌的研究. 淡水渔业, 2008, 38(3): 51—57]
[20] Yi Z H, Wang X X, Ji C, et al. Modulation of gastrointestinal microflora to amino acid homeostasis of the host animals [J]. Chinese Journal of Animal Science, 2005, 41(9): 64—66 [易中华, 王晓霞, 计成, 等. 消化道微生物对宿主动物氨基酸的自平衡调节作用. 中国畜牧杂志, 2005, 41(9): 64—66]
[21] Sun Y Z, Yang H L. A Review: Distribution and manipulation of fish gut microflora [J]. Fisheries Science, 2008, 17(5): 257—261 [孙云章, 杨红玲. 浅谈鱼类消化道微生物分布及调控. 水产科学, 2008, 17(5): 257—261]
[22] Chang Z W, Yan W H, Wang C H. High-density breeding technology for juvenile of allogynogenetic crucian carp (Carassius auratus gibelio) [J]. Journal of Aquaculture, 2010, 11: 39—40 [常智伟, 严维辉, 王崇华. 异育银鲫高密度发花技术. 水产养殖, 2010, 11: 39—40]
[23] Manuel C. Molles Jr. Ecology: Concepts & Applications [M]. Beijing: Higher Education Press. 2007, 413
[24] Bitry G. Study of digestive enzyme of two kinds filteringfeeding fish, Hypophthalmichthys molitrix and Aristichthys nobilis [J]. Fisheries Science (UK), 1985, 27(2): 102—107
[25] Gu J W. Effects of starvation on biochemical composition and digestive system histology in juvenile Pampus argenteus [D]. Thesis for Master of Science. Ningbo University, Ningbo. 2011 [谷江稳. 饥饿胁迫对银鲳幼鱼体成分及消化系统组织学的影响. 硕士学位论文, 宁波大学, 宁波. 2011]
[26] Wang Y L. Effects of four kinds of plant protein sources and different addition levels on intestinal structure of Carassius auratus gibelio [D]. Thesis for Master of Science. Soochow University, Suzhou. 2011 [王永玲. 四种植物蛋白源及其不同添加水平对异育银鲫肠道组织结构的影响. 硕士学位论文, 苏州大学, 苏州. 2011]
[27] Kamada N, Kim Y G, Sham H P, et al. Regulated virulence controls the ability of a pathogen to compete with the gut microbiota [J]. Science, 2012, 336: 1325—1329
[28] Tan X. Reseacrhes on several biological character of Microcystis during water bloom dominant species forming [D]. Thesis for Master of Science. Nanjing Normal University, Nanjing, 2006 [谭啸. 微囊藻在水华优势种形成过程中若干生物学特性的研究. 硕士学位论文, 南京师范大学, 南京, 2006]
[29] Zheng T L, Su J Q. The role of marien microorganisms in the occurence and declination of red-tide [J]. Acta Hydrobiologica Sinica, 2003, 27(3): 291—295 [郑天凌, 苏建强. 海洋微生物在赤潮生消过程中的作用. 水生生物学报, 2003, 27(3): 291—295]
[30] Sullam K E, Essinger S D, Lozupone C A, et al. Environmental and ecological factors that shape the gut bacterial communities of fsh: a meta-analysis [J]. Molecular Ecology, 2012, 21: 3363—3378
[31] Chen X X. Influences of feed and jejunum on the anount of intestinal microflora from grass carp [J]. Reservoir Fisheries, 1996, 6: 18—19 [陈孝煊. 草鱼鱼种饱食及空肠状态下肠道细菌数量变化的研究. 水利渔业, 1996, 6: 18—19]
EFFECTS OF REFEEDING ON THE DIVERSITY OF INTESTINAL MICROFLORA IN ALLOGYNOGENETIC CRUCIAL CARP (CARASSIUS AURATUS GIBELIO)
LI Xing-Hao1,2, YAN Qing-Yun1, HU Hong-Juan1,2, LI Jin-Jin1,2, WANG Chun1,2and YU Yu-He1
(1. Key Laboratory of Biodiversity and Conservation of Aquatic Organisms, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
In this study, we applied PCR-DGGE fingerprinting to investigate the effects of refeeding on the diversity of intestinal microflora in allogynogenetic crucian carp. Fishes were reared in a semi-recirculating system until maturation at Institute of Hydrobiology. After the 30-day starvation, fishes were divided into two groups (the hungry group and the feed group). Three fishes were blindly selected from the hungry group for the extraction of the microbial DNA with the traditional Phenol-chloroform method. In the meanwhile, fishes in the feed group resumed to be fed. Three fishes in the feed group were blindly selected for extraction of the microbial DNA after 25 days. The PCR-generated bacterial DNA products were separated using a polyacrylamide gel as it exhibited increasingly higher concentrations of chemical denaturant. Correspondence Analysis (CA) and clustering based on Jaccard index was used to analyze the similarity of the samples. The results showed that the feed group displayed less bands than the hungry group. There was significant difference in the deleted bands between the feed group (B1, B2, B5, B6, B7 and B23) and the hungry group (B11, B14, B16, B17 and B21), so we reasoned that the similarity of microflora between the two groups was low. The variance analysis showed that the variance within the feed group was larger than that within the hungry group. In conclusion, our results suggested that refeeding had strong effects on the diversity of intestinal microflora in allogynogenetic crucian carp.
Carassius auratus gibelio; Intestinal Microflora; PCR-DGGE Fingerprinting; Intestinal Micro-Ecosystem
S965.1
A
1000-3207(2014)05-0854-06
10.7541/2014.128
2013-07-22;
2014-02-12
国家重点基础研究发展计划(2009CB118705); 国家自然科学基金(31172084)资助
李星浩(1990—), 男, 江西新建人; 硕士研究生; 主要从事淡水鱼类消化道微生物研究。E-mail: lixhao90@163.com
余育和(1956—), 男, 湖南汨罗人; 研究员; 主要从事原生动物学及鱼类消化道微生物研究。E-mail: yhyu@ihb.ac.cn
