高通量二代测序基因条码技术在油料作物种类鉴别中的应用
2014-03-08吴亚君杨艳歌刘鸣畅
吴亚君,杨艳歌,李 莉,王 斌,刘鸣畅,陈 颖*
(中国检验检疫科学研究院,北京 100123)
高通量二代测序基因条码技术在油料作物种类鉴别中的应用
吴亚君,杨艳歌,李 莉,王 斌,刘鸣畅,陈 颖*
(中国检验检疫科学研究院,北京 100123)
对高通量二代测序基因条码技术在油料作物种类鉴别中的应用进行探索。采用Ion Torrent PGMTM测序技术,分别对橄榄、花生、大豆、油葵、玉米等14 种油料作物和小麦、大米的叶绿体核酮糖二磷酸羧化酶/氧化酶大亚基(ribulose-1,5-bisphosphate carboxylase/oxygenase large subunit,rbcL)基因246 bp聚合酶链式反应(polymerase chain reaction,PCR)产物混合物,及上述作物DNA混合液扩增得到的rbcL基因片段进行测序。结果表明,除小麦外,所有其他作物成分均获得了鉴定。其中,PCR产物混合物中每种作物成分的测序读数数量比例和该成分PCR产物在总产物中的比例接近,说明该技术可以用于定量分析。实验结果初步证实了该技术对混合样品中各作物种类鉴别的准确性,并具有检测效率、流程简便的特点,有望在未来成为食用油标识符合性查验的有力工具。
二代测序;基因条码;油料;检测
食用油作为关系国计民生的大宗食品,质量监管始终受到社会普遍关注。随着各类高端小品种食用油的快速发展,一方面满足了现代消费的需求,另一方面也受到掺假造假等市场乱象的困扰[1-3]。因此,建立快速准确的食用油身份鉴定方法,对完善现有的管理体系具有重要的现实意义。目前国内外对食用油的质量鉴别方法主要有两种,第一种是感官判断,这种方法虽然简单,但缺乏严格标准,具有主观性;第二种是理化指标,包括质谱、核磁共振、光谱及高效液相色谱等技术,主要依靠食用油中脂肪酸等化学组分判定食用油品种,虽然较为快速,但许多食用油品种之间缺乏显著的特征成分指标,化学计量学方法依赖庞大的数据收集和处理,且不同加工工艺和产地来源样品数据差异容易带来判别不确定性。
在物种鉴别方面,基于基因序列的检测方法相比于其他方法具有唯一性、稳定性、易于标准化等优点。目前,聚合酶链式反应(polymerase chain reaction,PCR)和实时荧光PCR技术已经成为食品质量安全关键技术,应用于食品真伪鉴别[4-9]、微生物快速检测[10-11]、饲料中动物成分监控[12-13]、过敏原检测[14-15]等。近年来随着测序技术的发展,基因条码成为全球分类学、生态学、进化学等领域的研究热点[16-17]。该技术是利用标准的、具有足够变异、易扩增且相对较短的DNA片段自身在物种内的特异性和种间的多样性而创建的一种新的生物身份识别系统,从而实现对物种的快速自动鉴定[18]。与传统的Sanger测序技术相比,二代测序结合模板分离和快速测序,摆脱了混合样品克隆分离等复杂步骤的限制,可以在一次实验中实现对复杂样品中各种成分的高通量检测[19-20]。本研究采用PGM芯片二代测序仪开展了14 种油料作物和小麦、大米核酮糖二磷酸羧化酶/氧化酶大亚基(ribulose-1,5-bisphosphate carboxylase/oxygenase large subunit,rbcL)基因小片段快速测定方法研究,以期为进一步开展混合食用油中油料成分的高通量检测提供参考依据。
1 材料与方法
1.1 材料与试剂
14 种油料材料橄榄、花生、大豆、油葵、玉米、芝麻、山茶、菜籽、棕榈粕、松子、腰果、榛子、棉籽、葵花籽以及小麦、大米,阴性材料猪、羊均为实验室保存材料。
NucleoSpin®Food核酸提取试剂盒 德国Macherey-Nagel公司;Wizard®Magnetic核酸提取试剂盒 美国Promega公司;末端修复DNA纯化试剂盒(gencourt®AMPure®XP Kit)、Ion Xpress Barcode Adapters试剂盒、Ion Library定量试剂盒、One Touch、Ion Torrent 314芯片和200bp读长测序试剂盒 美国Life Technologies公司。
1.2 仪器与设备
Invitrogen Qubit®2.0、PGM芯片二代测序仪 美国Life Technologies公司;DU®640核酸蛋白分析仪 德国Beckman公司;2100 Bioanalyzer毛细管电泳仪 美国安捷伦公司;Veriti™ 96梯度PCR仪 美国Applied Biosystems公司。
1.3 方法
1.3.1 引物设计
以植物叶绿体rbcL基因为目标序列,在GenBank数据库内搜索多种植物的rbcL序列并用软件进行比对,找到既具有种内保守性又具种间特异性的区域并设计植物通用引物,引物序列为F:5’ TTGGCAGCATTCCGAGTAAC-3’,R:5’-AGTAAACATGTTAGTAACAG -3’。
1.3.2 DNA提取
棕榈粕采用NucleoSpin®Food核酸提取试剂盒提取DNA,其余样品均采用Wizard®Magnetic核酸提取试剂盒提取DNA。通过DU®640核酸蛋白分析仪测定提取的DNA浓度和纯度,每个样品重复3次取平均值,并用无菌水将质量浓度调整至5 ng/μL,存于-20 ℃备用。
1.3.3 PCR及电泳检测
PCR扩增体系:10×Multi HotStart Buffer 5 μL,dNTP(2.5 mmol/L)2 μL,MgCl2(25 mmol/L)1.5 μL,上、下游引物(10 μmol/L)各0.5 μL,Multi HotStart Taq酶(5 U/L)0.2 μL,模板DNA(10 ng/L)5 μL,加灭菌双蒸水(dd H2O)补齐至总体积25 μL。
PCR扩增程序:95 ℃预变性10 min;95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸30 s,35个循环,72 ℃再延伸5 min;4 ℃保存。PCR产物用2100毛细管电泳仪进行分析。
1.3.4 PCR产物处理
采用Invitrogen Qubit®2.0测定PCR产物浓度。
PCR产物的DNA末端修复按如下步骤进行:50 ng PCR产物,20 øL 5×末端修复Buffer,1 øL末端修复酶,加灭菌双蒸水(dd H2O)补齐至总体积100 øL,室温(25 ℃)静置20 min。
DNA纯化步骤为:在修复后的PCR产物中加入180 øL(1.8 倍)Agencourt®AMPure®Kit磁珠,混匀,室温放置5 min,置于磁力架上至溶液澄清,去除上清液,加入新鲜配制的70%乙醇溶液,充分洗脱2次,待乙醇充分挥发后加入25 øL 低盐 TE缓冲液 (pH 8.0),涡旋10 s,置磁力架上约2~3 min,转移上清液至1.5 mL离心管。
采用Ion Xpress Plus Fragment Library Kit进行接头连接和缺口修复。反应体系为DNA 25 øL;10×Ligase Buffer 10 øL;Adapters 2 øL;dNTP Mix 2 øL;去除核酸酶的水51 øL;DNA Ligase 2 øL;缺口修复酶 8 øL;共100 øL。置PCR仪上25℃保持15 min;72℃保持5 min;4 ℃保存。
采用Ion Xpress Barcode Adapters 1-16 Kit分别加标签以区分样品,标签1、2分别对应DNA mix和PCR mix。反应体系:DNA 25 øL,10×Ligase Buffer 10 øL,Ion P1 Adapter 2 øL,Ion Xpress™ Barcode X 2 øL,dNTP Mix 2 øL,去除核酸酶的水 49 øL,DNA 连接酶 2 øL,缺口修复酶8 øL,共100 øL。加入140 øL(1.4 倍)Agencourt®AMPure®Kit磁珠,室温放置5 min,置磁力架上重复DNA纯化过程。用20 øL低盐TE缓冲液(pH 8.0)重悬。采用Ion Library定量试剂盒进行文库定量。
1.3.5 测序
分别采用Ion template preparation kit、One Touch以及Ion Xpress template kit MyOne streptavidin C1 beads进行PCR乳化和ISPs富集。
Ion Torrent PGM平台的序列检测:采用Ion PGM Sequencing kit及Ion Torrent 314芯片,进行测序反应共65 个测序循环,使用电阻率为18.2 MΩ•cm纯化水标准压缩氩气驱动PGM内液体的流动,每个样品设置重复3 次。
1.3.6 数据处理
采用Clustal Omega[21]进行DNA靶序列比对。根据Ion torrent PGMTM服务器自带分析软件对测序结果初步分析,得到整体数据量平均测序深度和参考序列匹配程度等数据,并生成FASTA文件,采用NextGENe软件(DEMO v 2.3.0)进行后续的序列数据结果分析。
2 结果与分析
2.1 通用引物的设计及PCR扩增
基因条码技术一般通过1对通用引物,或多对通用引物的混合物,实现对广谱物种的扩增。本研究对14 种油料和小麦、大米rbcL靶序列246 bp长度的基因片段进行序列比对,根据保守区域设计了1 对植物扩增通用引物(图1)。并验证其对14 种油料和小麦、大米的DNA以及DNA混合物的扩增情况。扩增结果表明:所有样品均获得明亮的条带,产物目的片段在243~253 bp之间,除玉米、芝麻有微弱的非目标条带外,其他物种均无非特异扩增(图2)。说明所设计的通用引物对目标作物具有较好的特异性和覆盖性。

图1 14 种油料和小麦、大米rrbbccLL靶序列的比对结果Fig.1 Alignment of target sequences of rbcL genes in wheat, rice and 14 kinds of oil crops
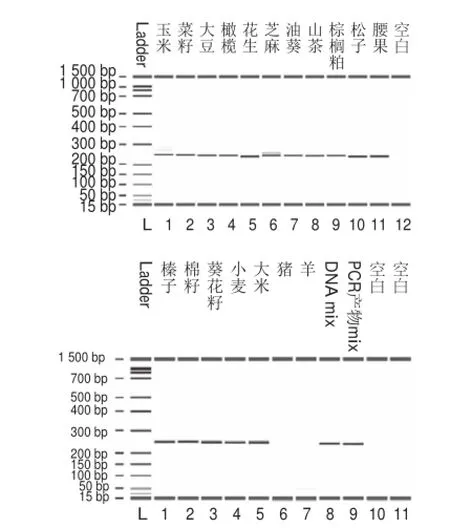
图2 通用引物rrbbccLL扩增产物2100毛细管电泳结果Fig.2 Amplification results of the rbcL universal primers by using 2100 capillary electrophoresis
2.2 测序分析
在序列匹配过程中,需要对物种的匹配率阈值做一个统一限制,故本研究对14 种油料和小麦、大米rbcL靶序列之间匹配率进行了两两比对。比对结果表明,靶序列平均相似度91.30%,其中油葵和普通葵花相似性最大,相似度为97.56%,其次为大豆和橄榄,相似度为96.75%(表1),据此将本实验测序的物种匹配阈值定为98%。
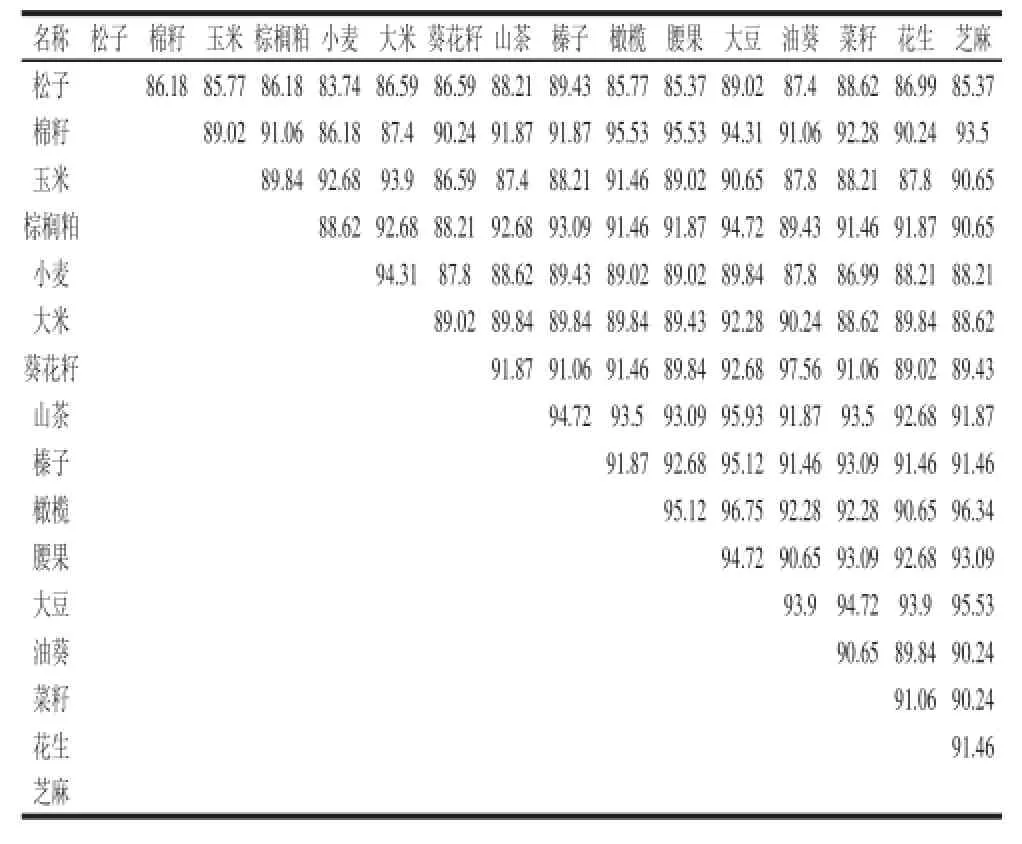
表1 14 种油料和小麦、大米rbcL靶序列两两比对结果(相似度)Table 1 Results of pairwise sequence alignment of rbcL gene-target sequences in wheat, rice and 14 kinds of oil crops%
测序实验针对4 个样品分别进行:将橄榄、花生、大豆、油葵、玉米、芝麻、山茶、菜籽、棕榈粕9 种油料PCR产物等体积混合,得到样品1;将上述9种油料DNA混合扩增后得到的PCR产物为样品2;将小麦、大米和14 种油料及2种阴性样品的PCR产物等体积混合,得到样品3;将上述和14 种油料以及小麦、大米、2 种阴性对照DNA混合扩增后的PCR产物为样品4。按照1.3.4~1.3.6节的方法对4个样品进行前处理和测序。其中第1次实验包括样品1和2,第2次实验包括样品3和4,2 次实验分别在两张芯片上进行。根据原始数据统计(图谱未示),第1次实验总通量为116.12 Mb,样品1和样品2的通量分别为60.4 Mb和54.5 Mb,平均读长大于200 bp,最长读长达330 bp。第2次实验总通量为79.7 Mb,样品3通量为37.1 Mb,样品4通量为39.39 Mb,平均长度为179 bp。2次实验4 个样品的数据质控情况显示,成功率99%以上(结果未示)。
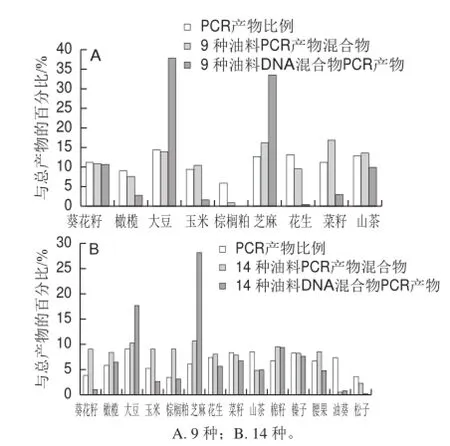
图3 油料成分和小麦、大米序列读数统计结果Fig.3 Statistical results of components of wheat, rice and the oil crops by reading sequences
对测序结果进行数据分析,图3是2 次实验样品序列与数据库序列比对情况,包括等量模板扩增后PCR产物的比例,实验样品的PCR产物的等量混合物以及其DNA混合物的PCR产物的各成分读数占所有读数的比例,这里只计算与数据库序列匹配率达到98%以上读数的总和。其中读数数量指每个物种匹配序列的数量,读数比例指该物种匹配序列总和占一个芯片上所有有效序列的比例。从图3可以看出,除了小麦的读数数量太低不做计算外,其他所有物种均有匹配读数,但不同物种的读数数量相差较大。
如图3A显示,除棕榈粕外,PCR产物混合物(斜纹)中各油料成分的读数比例与PCR产物比例数值比较接近,说明各个成分的PCR产物在芯片上分布均匀,对微孔的占有率和产物比例相当,芯片微孔与各种PCR产物的结合选择差异性很小。油料DNA混合物的PCR产物(方块)测序结果显示各个成分读数比例与PCR产物比例差异较大,说明通用引物对混合物中不同模板的扩增有选择性,模板之间存在竞争或相互干扰,导致一些成分优先扩增,例如大豆和芝麻,其序列在总体序列中比例较高,而其他成分序列比例明显下降。
为检验样品数的容量对测序是否有影响,本实验将检测物种增加至16 种(图3B),PCR产物混合物(斜纹)的测序结果显示,与相应PCR产物比例相比,棕榈粕和油葵所占比例较低,葵花籽和芝麻比例较高,其他成分的读数比例和PCR产物比例接近;而DNA混合物的PCR产物(方块)中仍以大豆和芝麻居高,各个物种读数比例与PCR产物比例差异较大。两次检测结果说明,在对混合成分的检测中,由于不同物种扩增效率的差异和竞争等因素的存在,各成分的检出率受影响,读数比例与各个成分的实际比例之间的关系也变得复杂。
以上数据是针对14 种油料作物和小麦、大米的已知序列进行匹配的结果。而在食用油实际检测过程中,掺假物种的成分及含量未知,因此将本研究中测得的序列直接在NCBI GenBank进行搜索,以验证在物种身份未知的情况下样品鉴定的准确性(结果未示)。除小麦和橄榄外,其他物种的测序序列均在NCBI数据库中得到最佳匹配。但这种针对大数据库的搜索受多种因素干扰,主要是植物基因组复杂,有关植物基因条码的候选基因很多,目前还没有定论。本研究尝试了246 bp的rbcL靶序列,数据库的检索显示许多无关物种也得到很高的匹配率,提示在油料物种的鉴定中,可以采取两种方式提高鉴别准确性,第一是建立油料物种的小数据库,减少无关物种的干扰;第二是增加基因数量,提高匹配的特异性。
3 讨 论
基因条码的概念由加拿大圭尔夫大学教授Hebert等[22]于2003年首次提出,即利用一段短的标准DNA序列对物种进行快速、准确的识别和鉴定,并希望以此建立起物种名称和生物实体之间一一对应的关系,其原理与零售业中对商品进行辨认的商品条形码相似,故而称为基因条码,代表了生物分类学研究的一个新方向。基因条码通过对一组来自不同生物个体的短的同源DNA序列进行PCR扩增和测序,随后对测得的序列进行多重序列比对和聚类分析,从而将某个体精确定位到一个已描述过的分类群中。序列的选择需要满足两个条件:1)必须具有相对的保守性,便于用通用引物扩增出来;2)要有足够的变异能够将物种区别开来。目前国际上针对动物基因条码有比较一致的标准,即线粒体细胞色素C氧化酶亚基Ⅰ(cytochrome c oxidaseⅠ,COI)基因进行物种鉴定和发现隐种或新物种。相对于动物,COI基因在高等植物中进化速率较慢,因此植物条形码研究以叶绿体基因组作为重点,但研究进展相对缓慢,目前尚处于对所提议各片段的比较和评价阶段,还未获得一致的标准片段,候选片段包括叶绿体上的rpoB、rpoC1、matK、rbcL、trnH-PSA及核基因组中ITS区域编码序列等[23]。
本研究证实了二代测序技术对14 种油料作物和小麦、大米DNA混合物的检测能力,并验证了该技术的准确性和对油料物种的覆盖度。虽然还有一些技术的细节有待进一步验证,包括序列长度、成分数量的容量、样品数的容量、序列匹配率阈值、食用油样品DNA提取的效率等对准确性的影响,但总体上,基于二代测序的短片段基因条码技术完全适用于油料混合成分的鉴别。下一步将开展该技术对其他基因检测的有效性,探讨该技术对食用油样品的适用性。
[1] 丁俊. 进口橄榄油市场起风波[N]. 江苏经济报, 2012-01-10(A02).
[2] 姚芃. 进口橄榄油并非都货真价实[N]. 中国国门时报, 2006-09-18(008).
[3] PEÑA F, CÓRDENAS S, GALLEGO M, et al. Direct olive oil authentication: detection of adulteration of olive oil with hazelnut oil by direct coupling of headspace and mass spectrometry, and multivariate regression techniques[J]. Journal of Chromatography A, 2005, 1074(1/2): 215-221.
[4] WU Yajun, CHEN Ying, GE Yiqiang, et al. Detection of olive oil using the Evagreen real-time PCR method[J]. European Food Research and Technology, 2008, 227(4): 1117-1124.
[5] BOTTERO M T, CIVERA T, ANASTASIO A, et al. Identification of cow’s milk in “Buffalo” cheese by duplex polymerase chain reaction[J]. Journal of Food Protection, 2002, 65(2): 362-366.
[6] DE S, BRAHMA B, POLLEY S, et al. Simplex and duplex PCR assays for species specific identification of cattle and buffalo milk and cheese[J]. Food Control, 2011, 22(5): 690-696.
[7] DALMASSO A, CICERA T, NEVE F L, et al. Simultaneous detection of cow and buffalo milk in mozzarella cheese by real-time PCR assay[J]. Food Chemistry, 2011, 124(1): 362-366.
[8] COSTA J, MAFRA I, AMARAL J S, et al. Monitoring genetically modi ed soybean along the industrial soybean oil extraction and re ning processes by polymerase chain reaction techniques[J]. Food Research International, 2010, 43(1): 301-306.
[9] DĄBROWSKA A, WAŁECKA E, BANIA J, et al. Quality of UHT goat’s milk in Poland evaluated by real-time PCR[J]. Small Ruminant Research, 2010, 94(1/3): 32-37.
[10] 许业莉, 相大鹏, 蔡颖. 食源性致病微生物中应用于PCR检测的靶基因[J]. 检验检疫科学, 2003, 13(6): 52-54.
[11] YU Shuijing, CHEN Wanyi, WANG Dapeng, et al. Species-specific PCR detection of the food-borne pathogen Vibrio parahaemolyticus using the irgB gene identified by comparative genomic analysis[J]. FEMS Microbiology Letters, 2010, 307(1): 65-71.
[12] CHEN Ying, WU Yajun, WANG Jing, et al. Identification of cervidae DNA in feedstuff using a real-time polymerase chain reaction method with the new fluorescence intercalating dye Evagreen[J]. Journal of AOAC International, 2008, 92(1): 175-180.
[13] MANE B G, MENDIRATTA S K, TIWARI A K, et al. Polymerase chain reaction assay for identification of chicken in meat and meat products[J]. Food Chemistry, 2009, 116(3): 806-810.
[14] NÉMEDI E, UJHELYI G, GELENCSER É. Detection of gluten contamination with PCR method[J]. Acta Alimentaria, 2007, 36(2): 241-248.
[15] DEMMEL A, HUPFER C, HAMPE E I, et al. Development of a realtime PCR for the detection of lupine DNA (Lupinus Species) in foods[J]. Journal of Agricultural Food Chemistry, 2008, 56(12): 4328-4332.
[16] KERR K C R, STOECKLE M Y, DOVE C J, et al. Comprehensive DNA barcode coverage of North American birds[J]. Molecular Ecology Notes, 2007, 7(4): 535-543.
[17] NEWMASTER S G, FAZEKAS A J, STEEVES R A D, et al. Testing candidate plant barcode regions in the Myristicaceae[J]. Molecular Ecology Resources, 2007, 8(3): 480-490.
[18] 任保青, 陈之端. 植物DNA条形码技术[J]. 植物学报, 2010, 45(1): 1-12.
[19] YANG Lin, WANG Huijun, WU Bailin, et al. Preliminary application of next-generation ion torrent PMGTM platform on genetic diseases in pediatrics[J]. Chinese Journal of Evidence-Based Pediatric, 2013, 8(3): 210-215.
[20] LI Yuanyuan, WU Yajun, HAN Jianxun, et al. Species-specific identification of seven vegetable oils based on suspension bead array[J]. Journal of Agricultural Food Chemistry, 2012, 60(9): 2362-2367.
[21] Clustal Omega Cookies on EMBL-EBI website[EB/OL]. http://www. ebi.ac.uk/Tools/msa/clustalo/.
[22] HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society Biological Sciences, 2003, 270: 313-321.
[23] HOLLINGSWORTH P M, FORREST L L, SPOUGE J L, et al. A DNA barcode for land plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(31): 12794-12797.
Application of High Throughput Next-Generation Sequencing Based on DNA Barcoding Technology in Species Identification of Edible Oils
WU Ya-jun, YANG Yan-ge, LI Li, WANG Bin, LIU Ming-chang, CHEN Ying*
(Chinese Academy of Inspection and Quarantine, Beijing 100123, China)
This study aimed to testify the application of second-generation sequencing based on DNA barcoding technology in the species identification of edible oils. Totally 14 species of oil crops including olive, peanut, soybean, oil sunflower, maize, sesame, camellia, rapeseed, palm and pine nut as well as rice and wheat were selected. Mixtures of the polymerase chain reaction (PCR) products of the 240 bp ribulose-1,5-bisphosphate carboxylase/oxygenase large subunit (rbcL) genes and the rbcL genes amplified from DNA mixtures of these 16 crop species were sequenced by Ion Torrent PGMTMrespectively. The results demonstrated that all crop species except wheat were successfully identified. As for two mixture samples, the reading ratio of each plant was consistent with the proportion of its PCR product. This study preliminarily proved that the combined technique is capable of identifying the plant species in blended oils accurately and efficiently. The procedure could be streamlined to be a promising detection method for oil product supervision in the near future.
next-generation sequencing; DNA barcoding; edible oil crops; detection
TS227
B
1002-6630(2014)24-0348-05
10.7506/spkx1002-6630-201424067
2014-03-26
国家高技术研究发展计划(863计划)项目(2011AA100807);公益性(质检)行业科研专项(201110015)
吴亚君(1975—),女,副研究员,博士,研究方向为食品安全。E-mail:wuyajuncaiq@163.com
*通信作者:陈颖(1972—),女,研究员,博士,研究方向为食品安全。E-mail:chenyingcaiq@163.com
