绿海龟α-actin基因的cDNA克隆与序列分析
2014-03-07陶翠花刘莹莹赵丽媛
陶翠花, 刘莹莹, 赵丽媛, 许 敏,, 祝 茜,
(1. 国家海洋局 第三海洋研究所, 福建 厦门 361005; 2. 山东大学(威海) 海洋学院, 山东 威海 264209)
绿海龟α-actin基因的cDNA克隆与序列分析
陶翠花1, 刘莹莹2, 赵丽媛1, 许 敏1,2, 祝 茜1,2
(1. 国家海洋局 第三海洋研究所, 福建 厦门 361005; 2. 山东大学(威海) 海洋学院, 山东 威海 264209)
为探究绿海龟(Chelonia mydas)α-actin基因序列的相关信息, 作者利用RT-PCR和RACE方法从绿海龟肌肉组织中获得了α-actin基因的cDNA全长序列, 共1347bp(GenBank登录号为JX073650)。所得序列包含一个1134 bp的开放阅读框, 编码由377个氨基酸组成的蛋白, 该蛋白7~377位为Actin结构域, 14~17位有一个糖基化位点, 无信号肽; 预测分子量为42.0 kDa, 理论等电点为5.23。将编码区序列与 GenBank上同源序列进行比对发现, 核苷酸序列相似性均在 85.4%以上, 氨基酸序列相似性均在98.9%以上, 说明α-actin基因作为编码蛋白是高度保守的。
绿海龟(Chelonia mydas); α-actin基因; RACE技术
绿海龟(Chelonia mydas)隶属于爬行纲(Reptilia),龟鳖目(Testudinata), 海龟科(Cheloniidae), 海龟属(Chelonia), 是在中国沿海分布的 5种海龟之一, 也是中国二级重点保护野生动物[1-2]。近百年来, 由于人类对海龟进行了掠夺性地乱捕滥杀、盗挖龟卵, 加上对其生存环境的破坏和污染, 海龟的数量在全世界范围内急剧下降[3-7]。目前, 世界自然保护联盟(IUCN)和濒危野生动植物种国际贸易公约(CITES)均将其确定为濒危物种[8-9]。
肌动蛋白(Actin)是一类存在于所有真核细胞中, 在进化过程中高度保守的蛋白质超家族, 与细胞内许多重要的功能活动有关。在高等动物细胞内, Actin大致分为6种, 其中4种具有肌肉组织特异性, 包括α-心肌型、α-骨骼肌型、α-血管平滑肌型和 γ-内脏平滑肌型; 其余两种广泛分布于各个组织中, 包括 β和 γ亚型, 均为细胞质型[10-11]。actin基因作为管家基因, 在生物体内生理或病理状态下都持续恒量表达, 因而在很多量化的试验中, 被当作内参(Internal control)基因。鉴于绿海龟分子方面的研究主要集中在种群遗传结构和系统发生关系[12-23]方面, 蛋白编码基因研究较少[24],肌动蛋白尤其是 α-actin基因的研究尚未见报道,因此, 作者对绿海龟的 α-actin基因的序列及结构进行了初步探究, 以期为今后进一步研究 actin基因结构、功能和表达分析以及绿海龟的遗传学研究积累基础资料。
1 材料和方法
1.1 材料
试验材料为采自中国南海的绿海龟的肌肉组织样品, 液氮速冻后于-70℃冰箱保存备用。总 RNA提取试剂盒(PureLinkTMRNA Mini)购于Invitrogen公司, 反转录试剂(SuperScript Ⅱ Reverse Transcriptase)、pMD18-T载体购于Takara公司, 胶回收纯化试剂盒(E.Z.N.A.®Gel Extraction Kit)购于Omega公司, 其余试剂为上海生工产品。
1.2 总RNA提取
将超低温保存的样品放入组织匀浆器, 加入Trizol reagent冰浴匀浆。其余步骤参照Invitrogen公司试剂盒说明书进行组织总 RNA的提取。用 0.8%的琼脂糖凝胶电泳鉴定总RNA, 用英国Picodrop公司Pico100型超微量紫外分光光度计测定总RNA的浓度及纯度。
1.3 cDNA的合成
根据CLONTECH公司SMART(Switching mec-hanism at 5’ end of RNA transcript)技术略加改动, 合成 cDNA。第一链 cDNA 合成所用引物为: Oligo-anchor R: 5’-GACCACGCGTATCGATGTCGA CT16(A/C/G)-3’和Smart F: 5’-TACGGCTGCGAGAA GACGACAGAAGGG-3’。取总RNA(1-5 µg)4 µL加入Smart F和Oligo anchor R引物各100 µmol/L, 混匀; 70℃水浴5 min, 立即置于冰上冷却2 min; 瞬时离心, 使各成分集合于管底; 再于冰盒中加入以下试剂: 5×反应缓冲液5 µL, RNA酶抑制剂25 U, 10 mmol/L dNTP混合物1.25 µL; 反转录酶1 µL(200 U/µL); 最后加DEPC水至25 µL, 42℃水浴60 min; 然后70℃温浴15 min, 冰浴10 min, 然后分装保存于-20℃。
1.4 绿海龟α-actin基因片段的克隆
根据 GenBank上的相关序列(登录号分别为AF416707, NM_203763, NM_001086591, AY986486), 采用Primer premier 5.0软件辅以人工方法设计兼并引物, 委托Invitrogen公司进行合成。引物序列为P1: 5’ATRCCAGCAGAYTCCATACC 3’, P2: 5’ CTGTSTT GTCCCTRTACGC 3’。
取2 µL 1.3中的反转录产物进行PCR扩增, 反应体系为: PCR mix kit 12.5 µL, Taq 酶0.2 µL(5 U/µL), cDNA模板1 µL, Primer P1/P2(10 µmol/L)各1 µL,灭菌蒸馏水9.3 µL。反应条件为: 94℃预变性10 min; 94℃变性40 s, 52℃退火40 s, 72℃延伸1 min, 30个循环; 最后72℃延伸10 min。PCR反应在Bio-Rad C1000型梯度PCR仪上进行。PCR产物使用DNA快速纯化回收试剂盒(Omega公司)经 1.5%琼脂糖凝胶进行回收、纯化, 纯化产物与pMD18-T载体(TaKaRa公司)连接, 转化感受态大肠杆菌, 用含有氨苄青霉素的LB固定培养基进行蓝白斑筛选, 挑取阳性克隆进行鉴定后, 送至上海生工生物技术有限公司测序。
1.5 5’和3’RACE扩增
根据测序结果设计 RACE 引物: GSP1: 5’AGCGTGGCTACTCCTTTGT3’, GSP2: 5’AATG AAGGATGGCTGGAAG3’, 分别与通用引物 3’anchorR: 5’GACCACGCGTATCGATGTCGAC3’和 5’primer: 5’TACGGCTGCGAGAAGACGACAG AA3’配对。PCR扩增反应条件均为: 94℃预变性10 min; 94℃变性40 s, 55℃退火40 s, 72℃延伸1 min, 30个循环; 最后72℃延伸10 min。5’和3’端获得的片段按照克隆部分片段的方法进行克隆测序。
1.6 序列分析
使用DNAman、Clustal W、Bioedit软件并结合人工校对将部分片段、5’端和3’端进行序列拼接, 获得全长 cDNA 序列, 并设计引物(F1: 5’CTGA ACCCTAAAGCTAACCG3’, R1: 5’CTTGCGATGGA CAATGGATG3)进行序列验证。通过NCBI的ORF程序获得开放阅读框(ORF)信息, 利用ExPASy在线软件获得其相应的氨基酸序列, 并对氨基酸进行性质(分子量、等电点)预测。利用SignalP 3.0(http://www. cbs.dtu.dk/services /SignalP/)进行信号肽的预测。利用SMART (http://smart. embl heidelberg.de/)进行结构域的预测。蛋白的三级结构预测在 SWISS-MODEL (http://swissmodel.expasy.org/)上进行, 利用PyMol软件进行三维结构的建构与标注。
2 结果
2.1 绿海龟α-actin基因扩增结果
经过序列的拼接、整理后, 获得 α-actin基因全长 cDNA序列, 共 1347 bp(GenBank登录号为JX073650)。该序列包含8 bp的5’端非编码区序列, 205bp的3’端非编码区序列, 以及1134 bp的完整开放阅读框序列。序列 3’端有一典型加尾信号(AATAAA), 其后24 bp处找到poly(A)尾巴。该基因共编码377个氨基酸, 其中第14-17位为糖基化位点NGSG(图1)。
2.2 绿海龟α-actin结构预测
根据在线软件分析, 该基因编码的蛋白分子量为42.0 kDa, 理论等电点(pI)为5.23。经预测, 该蛋白第7-377位氨基酸为Actin的结构域(图2), 高保守,无信号肽。
将推定的氨基酸序列进行三维结构预测, 获得的三级结构模型如图3所示, 结果发现, 绿海龟的α-actin蛋白与人(Homo sapiens)的α-actin 蛋白结构[25-27]很吻合, 主要由α-螺旋、β-转角、随机卷曲和延伸链组成。
2.3 核苷酸及氨基酸序列相似性比对
将该片段在 GenBank上进行比较, 结果显示:与热带爪蟾(Xenopus tropicalis)、非洲爪蟾(Xenopus laevis)、牛蛙(Rana catesbeiana)、牛(Bos taurus)等的α-actin基因核苷酸序列的同源性在 85.4%以上; 与热带爪蟾、牛蛙、牛、人(H. sapiens)、野猪(Sus scrofa)、小白鼠(Mus musculus)等物种氨基酸序列的同源性达100.0%, 与其他物种氨基酸序列的同源性达 98.9%以上(表1)。总体上, 绿海龟α-actin的保守性非常高。

图1 绿海龟α-actin基因的核苷酸序列和推导的氨基酸序列Fig. 1 The nucleotide and deduced amino acid sequences of α-actin gene from C. mydas
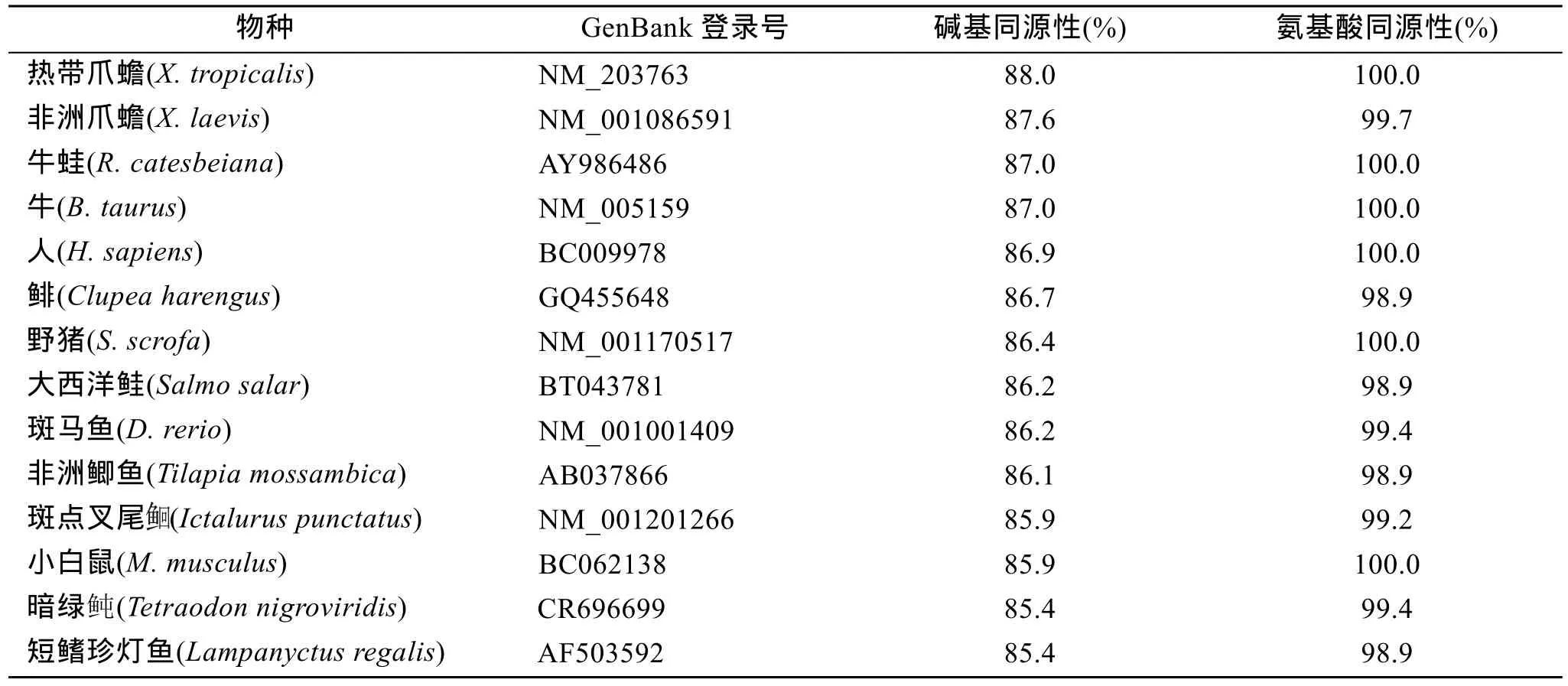
表1 绿海龟与其他动物α-actin基因全长cDNA序列的相似性比较Tab.1 Similarity comparisons of full-length cDNA of α-actin gene between C. mydas and other species
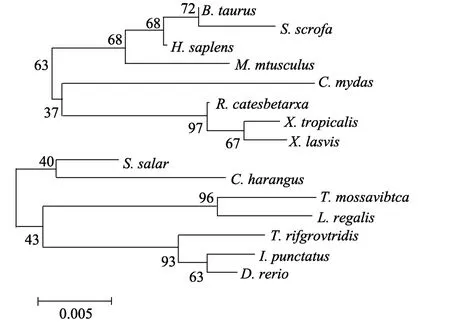
图4 根据α-actin基因核苷酸序列构建的系统进化树Fig. 4 Phylogenetic tree of α-actin gene based on nucleotide sequences

图2 α-actin蛋白结构域Fig. 2 Structural domain of C. mydas α-actin
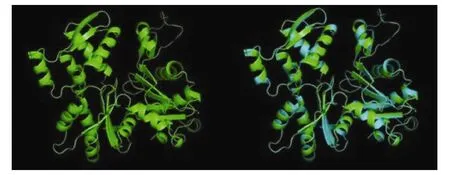
图3 理论预测的绿海龟α-actin蛋白及与人α-actin蛋白(3dawA)的结构叠合Fig. 3 Three-dimensional structure of modeled α-actin protein from C.mydas and its structural superposition of α-actin protein with H. sapiens (3dawA)
2.4 系统进化树的构建
利用Clustal W程序对表1中物种的α-actin基因核苷酸序列进行多序列比对, 并将比对结果用MEGA4.0生成系统进化树(Neighbor-Joining method),结果表明(图4), 绿海龟的α-actin基因序列先与热带爪蟾、非洲爪蟾和牛蛙聚类在一起, 再与牛、野猪、人和小白鼠聚类, 而与鱼类亲缘关系较远, 基本与传统分类相一致。
3 讨论
Actin普遍存在于真核生物中, 在转录、转录调控、RNA的加工和运输、维持细胞结构和细胞内运动等过程中发挥着重要作用, 是在进化中高度保守的蛋白质家族[28]。作者所获得的绿海龟α-actin基因核苷酸序列与热带爪蟾、非洲爪蟾、牛蛙等物种的同源性为 85.4%以上, 其所编码的氨基酸序列与其他物种的同源性达 98.9%以上, 表明 α-actin基因的核苷酸序列虽然在不同物种中存在较大变异, 但氨基酸序列却保持了高度的稳定性和一致性。蛋白质的进化速率主要取决于功能上的需要或选择压力[29], Actin蛋白的高度保守性暗示着其在进化过程中承受着较大的选择压力, 在生物体内执行着重要的生物学功能。在脊椎动物中, 存在于肌肉组织中的 Actin蛋白是可收缩器官的重要组成[30], 绿海龟的Actin蛋白对其生长发育的具体作用目前还没有报道, 需要进一步的研究。
脊椎动物的 actin基因在很多物种中已被克隆,如人、斑马鱼、原鸡、非洲爪蟾等, 也有学者克隆了绿海龟的β-actin基因片段, 但α-actin基因目前尚未见公开报道。本文利用RT-PCR和RACE方法首次克隆出绿海龟的α-actin基因全长cDNA序列, 丰富了 actin基因的资料库, 并对该基因的结构进行了初步探究, 为进一步研究相关基因的表达及功能奠定了基础。
致谢: 感谢海南师范大学陈忠教授、山东大学威海分校海洋学院李玉春教授及孔令明在收集样品过程中提供的帮助, 感谢山东大学威海分校海洋学院杨慧在软件分析中提供的帮助。
[1] 中华人民共和国濒危物种进出口管理办公室. 常见龟鳖类识别手册[M]. 北京: 中国林业出版社, 2002: 112-113.
[2] 张孟闻, 宗愉, 马积藩. 中国动物志, 爬行纲(第一卷)[M]. 北京: 科学出版社, 1998: 75-84.
[3] IUCN Marine Turtle Specialist Group. About marine turtles: Hazards[EB/OL].[2011.02]. http: //www.iucnmtsg.org/hazards.
[4] Asrar F F. Decline of marine turtle nesting populations in Pakistan[J]. Marine Turtle Newsletter, 1999, 83: 13-14.
[5] Spotila J R, Reina R D, Steyermark A C, et al. Pacific leatherback turtles face extinction[J]. Nature, 2000, 405: 529-530.
[6] Anne B M, Marydele D. Status justification for listing the Hawksbill turtle (Eretmochelys imbricata) as critically endangered on the 1996 IUCN red list of threatened animals[J]. Chelonian Conservation and Biology, 1999, 3(2): 200-224.
[7] Sella I. Sea turtles in the Eastern Mediterranean and Northern Red Sea[C]//World Conference on Sea Turtle Conservation- Biology and Conservation of Sea Turtles, Smithsonian Institution Press, Washington, D.C. Bjorndal K. A. 1982, 417-423.
[8] IUCN. IUCN red list of threatened species [EB/
OL].[2011.02]. http://www.iucnredlist.org/apps/redlist.
[9] CITES. The CITES appendices [EB/OL]. [2012.4.3]. http://www.cites.org/eng/app/appendices.shtml.
[10] Vandekerchkove J, Weber K. At least six different actins are expressed in a higher mammal: an analysis based on the amino acid sequences of the aminoterminal tryptic peptide[J]. Journal of Molecular Biology, 1978, 126: 783-802.
[11] Vandekerchkove J, Weber K. The complete amino acid sequence of actins from bovine aorta, bovine heart, bovine fast skeletal muscle, and rabbit, slow skeletal muscle: a protein-chemical analysis of muscle actin differentiation[J]. Differentiation, 1979, 14: 123-133.
[12] Bowen B W, Meylan A B, Ross J P, et al. Global population structure and natural history of the green turtle (Chelonia mydas) in terms of matriarchal phylogeny[J]. Evolution, 1992, 46(4): 865-881.
[13] Karl S A, Bowen B W, Avise J C. Global population genetic structure and male-mediated gene flow in the green turtle (Chelonia mydas): RFLP analyses of anonymous nuclear Loci[J]. Genetics, 1992, 131: 163-173.
[14] Lahanas P N, Miyamoto M M, Bjorndal K A, et al. Molecular evolution and population genetics of greater caribbean green turtles(Chelonia mydas) as inferred from mitochondrial DNA control region[J]. Genetica, 1994, 94: 57-67.
[15] Encalada S E, Lahanas P N, Bjorndal K A, et al. Phylogeography and population structure of the atlantic and mediterranean green turtleChelonia mydas: a mitochondrial DNA control region sequence assessment[J]. Molecular Ecology, 1996, 5(4): 473-483.
[16] Peare T, Parker P G. Local genetic structure within two rookeries ofChelonia mydas(the green turtle)[J]. Heredity, 1996, 77:619-628.
[17] Lahanas P N, Bjorndal K A, Encalada S E, et al. Genetic composition of a green turtle (Chelonia mydas) feeding ground population: evidence for multiple origins[J]. Marine Biology, 1998, 130: 345-352.
[18] Bass A L, Witzell W N. Demographic composition of immature green turtles (Chelonia mydas) from the East Central Florida coast: evidence from mtDNA markers[J]. Herpetologica, 2000, 56(3):357-367.
[19] Roberts M A, Schwartz T S, Karl S A. Global population genetic structure and male-mediated gene flow in the green turtle (Chelonia mydas): analyses of microsatellite loci[J]. Genetics, 2004, 166(4): 1857-1870.
[20] Noria O C, Grobois A A, Dutton P H, et al. Conservation genetics of the East Pacific green turtle (Chelonia mydas) in Michoacan, Mexico[J]. Genetica, 2004, 121: 195-206.
[21] Dethmers K M, Broderick D, Moritz C, et al. The genetic structure of Australasian green turtles (Chelonia mydas): exploring the geographical scale of genetic exchange[J]. Molecular Ecology, 2006, 15(13): 3931-3946.
[22] Formia A, Godley B J, Dontaine J F, et al. Mitochondrial DNA diversity and phylogeography of endangered green turtle (Chelonia mydas) populations in Africa[J]. Conservation Genetics, 2006, 7: 353-369. [23] Bourjea J, Lapegue S, Gagnevin L, et al. Phylogeography of the green turtle,Chelonia mydas, in the Southwest Indian Ocean[J]. Molecular Ecology, 2007, 16(1): 175-186.
[24] Yasuda A, Yamaguchi K, Papkoff H, et al. The complete amino acid sequence of growth hormone from the sea turtle (Chelonia mydas)[J]. General and Comparative Endocrinology, 1989, 73(2): 242-251.
[25] Arnold K, Bordoli L, Kopp J, et al. The Swiss-model workspace: a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22: 195-201.
[26] Schwede T, Kopp J, Guex N, et al. Swill-model: An automated protein homology-modeling server[J]. Nucleic Acids Research, 2003, 31: 3381-3385.
[27] Guex N, Peitsch M C. Swiss-model and the Swiss-PdbViewer: an environment for comparative protein modelling[J]. Electrophoresis, 1997, 18: 2714-2723.
[28] Wilma A H. Cell and molecular biology of nuclear actin[J]. International Review of Cell and Molecular Biology, 2009, 273: 219-263.
[29] Tourasse N J, Li W H. Selective constraints, amino acid composition, and the rate of protein evolution[J]. Molecular Biology and Evolution, 2000, 17(4): 656-664.
[30] Anna F, Ines D R, Francesca S, et al. Actin isoforms in amphioxusBranchiostoma lanceolatum[J]. Cell and Tissue Research, 1998, 292(1): 173-176.
(本文编辑: 谭雪静)
Cloning and sequence analysis of full-length cDNA of α-actin gene fromChelonia mydas
TAO Cui-hua1, LIU Ying-ying2, ZHAO Li-yuan1, XU Min1,2, ZHU Qian1,2
(1. Third Institute of Oceanography, State Oceanic Administration, Xiamen 361005, China; 2. Ocean College, Shandong University (Weihai), Weihai 264209, China)
Oct., 6, 2012
Chelonia mydas; α-actin gene; RACE
To explore the sequence and characteristic of α-actin gene fromChelonia mydas, the full-length cDNA sequence of α-actin gene was cloned using RT-PCR and RACE technique, which was consisted of 1347 bp nucleotides (GenBank accession number: JX073650), with a putative open reading frame (ORF) of 1134 bp encoding a deduced 377 amino acid protein containing a glycosylation site (from 14 to 17) and an Actin domain (from 7 to 377). The molecular weight of the protein was 42.0 kDa and the isoelectric point (pI) was 5.23. The nucleotide sequence similarity of α-actin gene betweenC. mydasand other species was above 85.4%, while the similarity of amino acid sequence was more than 98.9%, suggesting that α-actin gene was highly conserved. This study has enriched the Actin gene database and provided basic data for further studies on expression and function of relevant genes.
Q781
A
1000-3096(2014)03-0098-06
10.11759/hykx20121006001
2012-10-06;
2013-04-10
国家海洋局第三海洋研究所基本科研业务费专项资金项目(2010006); 国家海洋公益性行业科研专项(201105011)
陶翠花(1984-), 女, 新疆阜康人, 助理研究员, 硕士, 主要从事海洋生物学研究, 电话: 0592-2195211, E-mail: taocuihua@163.com祝茜, 通信作者, 电话: 0592-2191907, E-mail: qianzhu@sdu.edu.cn
