微生物中氧化态硒还原酶的研究进展
2014-03-07王东亮毛贝蓓
王东亮,毛贝蓓,张 路,肖 敏
(1.山东东阿阿胶股份有限公司,山东聊城252201;2.国家胶类中药工程技术研究中心,山东聊城252201;3.山东大学生命科学学院,山东济南250012)
·综 述·
微生物中氧化态硒还原酶的研究进展
王东亮1,2,毛贝蓓1,张 路1,2,肖 敏3
(1.山东东阿阿胶股份有限公司,山东聊城252201;2.国家胶类中药工程技术研究中心,山东聊城252201;3.山东大学生命科学学院,山东济南250012)
微生物还原氧化态硒对于硒在自然界的循环有重要作用,目前发现的还原方式主要有三种,即硒酸盐/亚硒酸盐经甲基转移酶或谷胱甘肽(GSH)作用后成为可挥发性的甲基硒化物;在细胞内的与含巯基的化合物发生化学反应,生成硒中间产物,进入各种代谢途径,或被巯基还原酶还原为Se0;反硝化细菌内硝酸还原酶具有还原氧化态硒的功能,发现另一种氧化态硒还原酶可以专门地还原氧化态硒为低价态或单质,该酶被发现同属于Ⅱ型钼酶家族。本文综述了氧化态硒还原酶的研究进展,以及在生化、遗传、基因组和蛋白质组学等方法研究中的应用。
氧化态硒还原酶;钼酶;硒酸盐;亚硒酸盐
硒是生物体必需的微量元素,其在生物体中主要以硒半胱氨酸、硒蛋氨酸等有机形式存在,作为机体中许多重要的酶—如谷胱甘肽还原酶、硫氧还蛋白还原酶、过氧化物酶、SOD以及甲状腺素脱碘酶等—的反应中心而参与各种反应[1]。在低浓度时具有抗诱变、抗致癌作用。高浓度时又能致突变及可能的致癌作用。硒的毒性大小与其化学状态和浓度有关,硒的安全浓度范围非常狭窄,成人建议摄入量55μg·d-1,最高安全摄入量400μg·d-1[2]。轻度缺硒会降低免疫力,导致关节炎症、脱发脱甲等,重度可能影响心肌和骨骼功能等,甚至导致基因的变异和断裂[3]。
硒的分布具有明显的地区差异性,除湖北恩施等个别地方外,世界上人口密集的地区硒含量往往很低。人体缺硒成为一种普遍现象。然而环境中硒元素主要以氧化态硒(SeO32-和SeO42-)的形式存在,这两种可溶性的无机物具有很高的毒性。因此,把高毒性硒转化成低毒、可有效利用的硒成为研究的热点。
另一方面,随着人类的发展,活动范围扩大,硒污染的程度也在加重。冶金工业、原油精炼排出的废水中含有大量的SeO32-,它是硒的主要污染源之一。此外,燃煤[4]是硒的最主要污染源,中国煤矿中硒平均含量(5.6 mg·kg-1)高于世界煤中硒平均含量(3.0 mg·kg-1)。燃煤产生的SeO2易溶于水形成SeO32-和SeO42-。硒污染的治理在国内外已经得
到广泛的重视。
目前,国内有关硒的研究主要集中在微生物富集氧化态硒的能力大小、硒化合物合成、食品中硒添加剂以及化学方法还原氧化态硒等表面研究上,在氧化态硒的转化机制上研究不深入。而在国际上,微生物转化硒的机制研究日益增多,对微生物初步还原氧化态硒形成了多种推测及观点。鉴于硒还原研究的工作对今后了解复杂的非金属氧化物代谢的研究意义以及环境治理、安全有效补硒的应用意义,本文就氧化态硒初步转化机制以及其中涉及的重要酶的关键问题进行了系统的分析和综述。
1 硒还原研究
越来越多的微生物被发现可以耐受高浓度的氧化态硒(亚硒酸盐和硒酸盐),并转化为有机硒和红色硒颗粒。1981年,Sarathchandra等[5]利用Bacillus megaterium进行硒的生物转化时,发现红色硒单质的生成现象。此后更多的菌株被发现具有生成红色单质硒的能力[6~9]。亚硒酸盐或硒酸盐在不同微生物中的可利用性不同,有的微生物两者都可以利用,如Escherichia coli[10]、Thauera.Selenatis[11]和Enterobacter cloacae SLD1a-1[12]。但有的微生物偏好利用亚硒酸盐,如Rhodobacter spheroids[13],Ralstonia metallidurans[14]。不同微生物耐受氧化态硒的能力不同,Rhizobium sp.[8]和Azospira oryzae[15],其最低抑制生长浓度(MIC)分别为16 mmol·L-1和50mmol·L-1,最高转化量分别为8mmol·L-1和4mmol·L-1;Tetrathiobacter kashmirensis[16]的MIC为64 mmol·L-1,是目前报道的耐受硒的能力最高的菌株。微生物高的硒耐受能力对于硒污染的环境治理有重要意义。此外,在真菌、藻类以及植物中也有发现氧化态硒耐受性,正在进行各种应用研究。
2 氧化态硒初步还原的途径
目前推定的细胞内氧化态硒初步还原的途径,主要有以下三种。
2.1 亚硒酸盐甲基化 亚硒酸盐甲基化后转变为挥发性气体,主要有二甲基硒(DMSe)和二甲基二硒(DMDSe)等,参与反应的酶是甲基转移酶。Ranjard等[17]在Escherichia coli中证实硫嘌呤甲基转移酶参与了氧化态硒和有机硒(如(Met-)Se-Cys)的甲基化过程,产物有DMSe和DMDSe,这个过程涉及几个还原和甲基化步骤。这是首次发现甲基转移酶具有硒甲基化功能。Ranjard等[18]在淡水细菌中又发现了一组硒甲基转移酶系,命名为Mm tA,系统发育分析把类似MmtA的序列分为两个簇,一个组是S-和O-甲基转移酶;另一组是UbiE C-甲基转移酶,该酶参与泛醌和甲基萘醌的合成,这组酶系与硫嘌呤甲基转移酶作用过程不同,它不需要还原步骤,可以直接把氧化态硒和有机硒转化为挥发性硒化物。Swearingen等[19]通过实验证实了UbiE C-甲基转移酶的硒甲基化功能,他们把Geobacillus stearother-mophilus来源的ubie基因转入ubie缺陷型Escherichia coli中,发现该基因编码的产物除了具有典型的C-甲基化活性以外,还有硒生物甲基化的功能。
此外,研究发现GSH可能也参与了挥发性硒化物的生成。Kessi[20]研究加硒培养的紫色非硫细菌发现有挥发性硒化物产生,但当加入GSH的合成酶抑制剂(BSO)后,检测不到挥发性硒化物,挥发性硫化物的代谢也受到影响,推测谷胱甘肽可能与气态硒化物的合成有关系,而且挥发性硒、硫化合物的代谢可能有同样的酶参与,但还没有在理论上证实。随着研究的深入,将有更多的具有硒甲基化功能的酶或其他化合物被发现,而且其多样化的甲基化代谢途径也将逐渐被阐明。
2.2 细胞内的化学还原途径 参与这类反应的主要是含巯基的化合物或蛋白,如谷胱甘肽/谷胱甘肽还原酶,硫氧还蛋白/硫氧还蛋白还原酶等,它们是原核和真核细胞中最主要的硫醇之一,可以迅速与亚硒酸盐反应。Ganther[21]发现哺乳动物组织中谷胱甘肽参与了亚硒酸盐的还原。二者的反应方程:
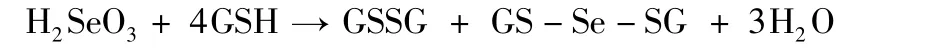
中间体GS-Se-SG可以进入各种代谢途径,被生物体同化[10],同时它也是含巯基蛋白及其还原酶的高效氧化剂,可以被还原酶进一步还原为Se0,伴随产物有H O和O′-。
222后来,该反应在Escherichia coli[22]转化硒的实验中利用同位素77Se和核磁分析证实该过程的存在。而且,Gabel-Jensen C[23]研究了猪小肠的上皮细胞代谢亚硒酸盐过程,发现当L-Cys、L-GSH和亚硒酸盐一起存在时,代谢物迅速形成并在15 min后消失,副产物挥发或沉淀,该实验现象支持了“2.1”中GSH参与甲基化的观点。利用LC-ESI-MS技术检测到的含硒中间物只有Cys-Se-Cys、Cys-Se-SG和GS-Se-SG。此外,在Rb.sphaeroides和E.coli[24,25]中发现加硒培养可以诱导谷胱甘肽还原酶的合成。同时,Bébien等[25]发现硫氧还蛋白/硫氧还蛋白还原酶和谷胱甘肽系统,在与亚硒酸盐反应时,具有相同的性质。并且在E.coli中硫氧还蛋白/硫氧还蛋白还原酶更容易被诱导合成。细胞中还原态巯基化合物可以减弱氧化态硒毒性带来的脂质类氧化性损伤,因此,其含量是细胞的硒耐受能力的重要影响之一,这也说明了细胞内化学还原和酶催化还原反应不是孤立的,而是相互联系、相互影响的。
2.3 细胞内的酶参与的氧化态硒还原反应 除了化学还原方式以外,酶促反应也逐渐被揭示。一种方式是氧化态硒的还原与反硝化作用有关,另一种方式是特有的氧化态硒还原酶参与了催化反应。这两种还原方式中参与的主要酶都是Ⅱ型钼酶类。下面将详细阐述。
3 氧化态硒还原酶的分类
3.1 硝酸盐和亚硝酸盐还原酶催化氧化态硒的还原 反硝化菌是其中的代表,它有两种酶类,即周质空间酶和膜结合蛋白酶,Rb.sphaeroides[13]位于周质空间的硝酸盐还原酶(NAR)及E.coli[26]膜结合状态的硝酸盐还原酶,在以苄基紫精为电子供体时可以在体外还原硒酸盐。后来研究发现这是反硝化细菌的普遍特征。Ralstonia eutropha,Paracoccus denitrificans,Paracoccus pantotrophus内的硝酸盐还原酶都可
以以硒酸盐为电子受体[27]。硝酸盐还原酶含有钼辅因子活性中心,是Ⅱ型钼酶的一种[28]。
3.2 氧化态硒还原的钼依赖性还原酶 目前发现的这类酶有一个共同的特点,即酶活性中心都有含钼辅因子。按底物不同分为硒酸盐还原酶和亚硒酸盐还原酶。硒酸盐还原酶专门催化硒酸盐为亚硒酸盐。在厌氧微生物中,以氧化态硒为最终的电子受体,细胞通过厌氧呼吸得到能量。从受硒污染严重的圣华金河谷地筛选出的Thauera.selenatis[11]中,首次发现细胞周质空间有一个特异的硒酸盐还原酶,而其硝酸盐还原酶则位于细胞质膜上,这说明硒酸盐和硝酸盐还原可能是由两个独立的酶催化完成的。硒酸盐还原酶只能以硒酸盐为底物反应生成亚硒酸盐,不能以亚硝酸盐、硝酸盐、氯酸盐和硫酸盐为底物。而亚硒酸盐的还原则是通过亚硝酸盐还原酶催化转化为硒颗粒。研究同时也显示该酶不是其他氧化物还原途径的一部分。硒酸盐的还原过程与细胞生长偶连,硒酸盐还原酶(SER)可能是电子传递的一部分,通过质膜产生电化学势能。纯化后得到该酶的组成信息,分子量为180 kDa,由SerA、SerB和SerC 3个异源亚基组成(96 kDa,40 kDa,23 kDa)。首次对硒酸盐还原酶的三个亚基进行了N端测序。SerA亚基N末端富含Cys结构域,可能含有一个[4Fe-4S]簇,同时含有一个以钼元素为活性位点的钼蝶呤辅因子。SerB也含有富含Cys结构域,可能也含有[Fe-S]簇。光谱分析发现SerC含有一个细胞色素b。对T.selenatis[29]的硒酸盐还原酶基因进行DNA序列分析还发现了第四个组成元件SerD,它可能的作用是作为特异的分子伴侣使钼蝶呤辅因子正确插入SerA。ERP谱和序列比对表明SER是Ⅱ型钼酶的一个成员[28],SER是目前了解比较清楚的酶之一。
另一个了解比较清楚的SER是在Enterobacter cloacae SLD1a-1质膜上[30]。该菌也是在圣华金河谷地发现的,首次纯化出了膜结合的硒酸还原酶。细胞质膜上定位着两个不同的酶:硒酸盐还原酶(SER)和硝酸盐还原酶(NAP),前者只能以硒酸盐和氯酸盐为底物。而后者只能以硝酸盐和氯酸盐为底物。E.cloacae的SER与T.selenatis中的SER组成有相似之处。其表观分子量为~600 kDa,是一个异源三聚体复合物,大小分别为~100 kDa(α)、~55 kDa(β)、~36 kDa(γ)。预想的整体组成为α3β3γ3,该酶也含有钼、亚铁血红素、非亚铁血红素铁离子作为辅基因子,电子吸收光谱显示在活性中心有一个细胞色素b。经SDS-PAGE后分离的亚基不能成功进行N-末端序列分析,阻碍了对其结构的进一步认识。因此该酶的基因序列还不清楚。不同的还原酶的好处在于,硒酸盐不会与硝酸盐竞争活性位点,从而有利于细胞在高浓度硒酸盐的环境中存活。
另一种酶是亚硒酸钠还原酶,催化亚硒酸盐转化为硒颗粒Se0,该酶主要位于细胞质内。已在球形红细菌和根瘤菌中发现。球形红细菌中亚硒酸盐还原酶可能是一个由亚硒酸盐诱导表达的蛋白,两个影响钼辅因子合成的突变株中亚硒酸盐的还原速率降低了,而且细胞培养基中加入钼酶的抑制剂钨酸盐后,亚硒酸盐的还原速率降低了同样程度,减少了原速率的40%[31]。这说明亚硒酸盐还原酶中可能含有钼元素,且承担了细胞还原亚硒酸盐能力的40%。同时说明该微生物细胞中亚硒酸盐还原还有其他未知的途径(约占60%)。研究发现该亚硒酸盐还原酶与其他Ⅱ型钼酶(如硝酸盐、生物素亚砜和二甲基亚砜的还原酶)不同。但酶的组成及性质还不清楚。
Rhizobium sp.可以在有氧条件下转化亚硒酸钠为硒颗粒,钨酸盐可以抑制细胞内亚硒酸盐的还原,说明还原途径中涉及到钼酶的催化作用[8]。非变形梯度电泳得到两条有亚硒酸盐还原活性蛋白条带,分别为~100 kDa和~40 kDa。尽管还原机制还不清楚,这是第一次报道一株菌中有两种还原亚硒酸盐的蛋白。在Azospira oryzae中发现同时含有还原硒酸盐和亚硒酸盐的酶,大小分别为~500 kDa和~55 kDa[9]。还原硒酸盐的酶和有还原硫酸盐活性的酶不同。亚硒酸盐还原也和硒酸盐、硫酸盐、亚硝酸盐及二甲基亚砜明显不同。同时含有两种硒还原酶微生物,对受硒污染的水域治理将有很大帮助。因此两个酶的酶学性质还值得深入研究。
4 酶的初步研究方法
4.1 生化分析 有氧或厌氧条件下建立氧化态硒还原曲线,通过添加可能对硒还原有影响的化合物,检测加入的化合物对氧化态硒还原影响。例如在细胞培养基里添加硝酸盐或亚硝酸盐,钼酶的底物,钼酶的抑制剂[8,13,31]等。进而推测可能的还原途径及相互关系。
4.2 构建基因文库或转座子文库来获得相关影响基因 将E.cloacae的基因文库[32]转导进无还原硒能力的E.coli SK17-1中,突变子涂于含抗生素和亚硒酸钠的平板上,筛选红色菌落,分析基因序列,最终获得使SK17-1还原亚硒酸盐的基因序列。也可以从转座子文库里筛选出影响氧化态硒还原的突变子。利用Tn5[31]或Tn10[33]转座子构建文库,通过反向PCR定位Tn5转座子的插入位点,然后测定基因序列。选择Tn5单插入的突变子序列分析,发现了一些与氧化态硒还原有关的调控基因或者对含钼辅因子合成有关系的基因片段。虽然试验繁琐,但是可以较快得到与硒还原相关的基因信息。
4.3 蛋白质组学方法 主要通过添加氧化态硒培养后,比较细胞中蛋白的二维图谱变化,选择有明显变化的蛋白质点进行质谱分析,确定功能分类。在Stenotrophomonas maltophilia SeITE02[34]培养基中添加亚硒酸盐和亚硝酸盐离子后,细胞质内可溶性蛋白做二维电泳,获得蛋白图谱,与正常培养获得的二维图谱比较,挑出图谱中异常表达的蛋白。经质谱分析把有明显变化的蛋白点归为以下几类:受损蛋白降解类、DNA代谢类、细胞分裂类和氧化应激反应类。被诱导表达的蛋白有:过氧化氢酶、谷氨酸-半胱氨酸连接酶前体和GSH合成酶。H2O2是亚硒酸盐被GSH非生物还原的副产物,支持了GSH参与细胞内第一步还原反应的假设。谷氨酸-半胱氨酸连接酶作用是驱动真菌、蓝细菌、紫细菌等
的GSH生物合成通路,这些都是为了抵御外界高浓度亚硒酸盐环境所作出的反应,从而可以推测细胞代谢硒的各种通路。
4.5 酶活测定方法 基于甲基紫精微孔板法测定氧化态硒还原酶酶活[35]。这种方法适用于酶在细胞内表达量低、酶活也相对较低的情况。它要求的酶样品量低(<5μL),可以同时测定不同底物的专一性试验。在酶活动力学参数测定、最佳pH、抑制剂等方面灵活准确性好。与其他氧化酶相比,膜蛋白酶及周质空间酶的酶活测定特别适用。
5 氧化态硒还原酶的调控基因
氧化态硒在自然界中循环受环境微生物代谢影响生成有机硒或还原为硒颗粒。但氧化态硒还原涉及的基因调控机制还不清楚。目前只在Enterobacter cloacae SLD1a-1[32]发现了一个调控基因——延胡索酸还原反应调控基因fnr,该基因可以控制硒酸盐还原酶的活性。研究发现在无还原氧化态硒现象的E.coli S17-1中异源表达fnr后,Escherichia coli S17-1具有了和Enterobacter cloacae SLD1a-1同样的还原氧化态硒的能力,通过构建基因文库对阳性突变子进行定向克隆找到可能相关的基因fnr。在Enterobacter cloacae SLD1a-1中fnr缺失突变,发现突变子失去产生红色硒单质的能力。两者共同证实了fnr调控硒酸盐还原酶的功能。其具体的调控机制不清楚。全基因组转录数据显示FNR蛋白调控至少103个操纵子。在E.coli[36]中,FNR蛋白调控着厌氧呼吸酶的表达,包括硝酸盐还原酶、延胡索酸还原酶及二甲基亚砜还原酶。FNR蛋白位于细胞质,含有一个[4Fe-4S]2+簇连接4个重要的半胱氨酸残基,是专一结合DNA的活性位点。FNR对O2敏感,有氧气存在时,[4Fe-4S]2+簇被迅速氧化为[2Fe-2S]2+,不能结合DNA。因此FNR在厌氧条件下才有调节活性,控制硒酸盐还原酶的表达量。氧化态硒还原酶的遗传鉴定和表达动力学测定,有助于深入地阐明微生物中硒代谢机制,揭开地球化学对微生物的影响。
6 展望
氧化态硒的多种多样的还原方式有利于微生物灵活地抵制外界环境的氧化压力。但还原酶的研究进度较慢,可能与这类酶在细胞中的含量、组成复杂性以及体外提取的稳定性和可行性等有关。目前仅有个别菌株的还原酶得到提纯和遗传组成信息,更多高耐受硒能力的微生物的还原酶仍需弄清楚,这将对高效还原氧化态硒有很大帮助。不同微生物中该还原酶性质可能差别很大,有些酶的活力测定要在厌氧条件下长时间反应,反应速度慢。氧化还原酶还易受其他底物影响。因此酶蛋白提取方法及灵敏的测定方法还需要摸索,这可能是它在很长一段时间里进展缓慢的原因之一。
此外,氧化态硒还原过程中的调控机制以及酶的氧化还原中心及能量传递等细节尚不清楚,是亟待解决的问题。随着人们对环境治理的重视,氧化态硒还原的机制研究日益被人们重视。利用生化分析、基因组学、蛋白质组学、遗传分析等方法,有助于微生物代谢硒的研究,对解决硒污染水体的环境治理及安全补硒有重要意义。
[1]Stadtman TC.Biosynthesis and function of selenocysteine-containing enzymes[J].JBiol Chem,1991,266(25):16257-16260.
[2]Whanger PD.Selenium and its relationship to cancer:an update[J].Br JNutr,2004,91(1):11-28.
[3]LetavayováL,Vlasáková D,Spallholz JE,et al.Toxicity and mutagenicity of selenium compounds in Saccharomyces cerevisiae[J].Mutat Res,2008,638(1-2):1-10.
[4]张莹,刘桂建,郑刘根,等.中国煤中硒的环境地球化学[J].矿物岩石地球化学通报,2007,26(4):389-398.
[5]Sarathchandra SU,Watkinson JH.Oxidation of elemental selenium to selenite by Bacillus megaterium[J].Science,1981,211(4482):600-601.
[6]Garbisu C,Carlson D,Adamkiewicz M,et al.Morphological and biochemical responses of Bacillus subtilis to selenite stress[J].Biofactors,1999,10(4):311-319.
[7]Kessi J,Ramuz M,Wehrli E,et al.Reduction of selenite and detoxification of elemental selenium by the phototrophic bacterium Rhodospirillum rubrum[J].Appl Environ Microbiol,1999,65(11):4734-4740.
[8]Hunter WJ,Kuykendall LD.Reduction of selenite to elemental red selenium by Rhizobium sp.strain B1[J].Curr Microbiol,2007,55(4):344-349.
[9]Hunter WJ.An Azospira oryzae(syn Dechlorosoma suillum)strain that reduces selenate and selenite to elemental red selenium[J].Curr Microbiol,2007,54(5):376-381.
[10]Turner RJ,Weiner JH,Taylor DE.Selenium metabolism in Escherichia coli[J].Biometals,1998,11(3):223-227.
[11]Schröder I,Rech S,Krafft T,et al.Purification and characterization of the selenate reductase from Thauera selenatis[J].J Biol Chem,1997,272(38):23765-23768.
[12]Losi ME,Frankenberger WT.Reduction of selenium oxyanions by enterobacter cloacae SLD1a-1:isolation and growth of the bacterium and its expulsion of selenium particles[J].Appl Environ Microbiol,1997,63(8):3079-3084.
[13]Van Fleet-Stalder V,Chasteen TG,Pickering IJ,et al. Fate of selenate and selenite metabolized by Rhodobacter sphaeroides[J].Appl Environ Microbiol,2000,66(11):4849-4853.
[14]Roux M,Sarret G,Pignot-Paintrand I,et al.Mobilization of selenite by Ralstonia metallidurans CH34[J].Appl Environ Microbiol,2001,67(2):769-773.
[15]Hunter WJ.An Azospira oryzae(syn Dechlorosoma suillum)strain that reduces selenate and selenite to elemental red selenium[J].Curr Microbiol,2007,54(5):376-381.
[16]Hunter WJ,Manter DK.Bio-reduction of selenite to elemental red selenium by Tetrathiobacter kashmirensis[J]. Curr Microbiol,2008,57(1):83-88.
[17]Ranjard L,Prigent-Combaret C,Nazaret S,et al.Methylation of inorganic and organic selenium by the bacterial thiopurine methyltransferase[J].J Bacteriol,2002,184(11):3146-3149.
[18]Ranjard L,Prigent-Combaret C,Favre-BontéS,et al. Characterization of a novel selenium methyltransferase from freshwater bacteria showing strong similarities with the calicheamicin methyltransferase[J].Biochim Biophys Acta,2004,1679(1):80-85.
[19]Swearingen JW Jr,Fuentes DE,Araya MA,et al.Expression of the ubiE Gene of Geobacillus stearothermophilus V in Escherichia coli K-12 Mediates the Evolution of selenium compounds into the headspace of selenite-and selenate-amended cultures[J].Appl Environ Microbiol,2006,72(1):963-967.
[20]Kessi J.Enzymic systems proposed to be involved in the dissimilatory reduction of selenite in the purple non-sulfur bacteria Rhodospirillum rubrum and Rhodobacter capsulatus[J].Microbiology,2006,152(Pt3):731-743.
[21]Ganther HE.Selenotrisulfides.Formation by the reaction of thiols with selenious acid[J].Biochemistry,1968,7(8):2898-2905.
[22]Rabenstein DL,Tan KS.77Se NMR studies of bis(alkylthio)selenides of biological thiols[J].Magn Resonance Chem,1988,26(12):1079-1085.
[23]Gabel-Jensen C,Gammelgaard B,Bendahl L,et al.Separation and identification of selenotrisulfides in epithelial cell homogenates by LC-ICP-MS and LC-ESI-MS after incubation with selenite[J].Anal Bioanal Chem,2006,384(3):697-702.
[24]Bébien M,Chauvin JP,Adriano JM,et al.Effect of selenite on growth and protein synthesis in the phototrophic bacterium Rhodobacter sphaeroides[J].Appl Environ Microbiol,2001,67(10):4440-4447.
[25]Bébien M,Lagniel G,Garin J,et al.Involvement of superoxide dismutases in the response of Escherichia coli to selenium oxides[J].J Bacteriol,2002,184(6):1556-1564.
[26]Avazeri C,Pommier J,et al.Reduction of oxyanions by photosynthetic bacteria and Escherichia coli:role of the nitrate reductase in the reduction of tellurite and selenate[A].Proceedings of The International Photosynthesis Congress[C].1995:423-426.
[27]Sabaty M,Avazeri C,Pignol D,et al.Characterization of the reduction of selenate and tellurite by nitrate reductases[J].Appl Environ Microbiol,2001,67(11):5122-5126.
[28]Dridge EJ,Watts CA,Jepson BJ,et al.Investigation of the redox centres of periplasmic selenate reductase from Thauera selenatis by EPR spectroscopy[J].Biochem J,2007,408(1):19-28.
[29]Krafft T,Bowen A,Theis F,et al.Cloning and sequencing of the genes encoding the periplasmic-cytochrome B-containing selenate reductase of Thauera selenatis[J]. DNA Seq,2000,10(6):365-377.
[30]Ridley H,Watts CA,Richardson DJ,et al.Resolution of distinct membrane-bound enzymes from enterobacter cloacae SLD1a-1 that are responsible for selective reduction of nitrate and selenate oxyanions[J].Appl Environ Microbiol,2006,72(8):5173-5180.
[31]Pierru B,Grosse S,Pignol D,et al.Genetic and biochemical evidence for the involvement of a molybdenum-dependent enzyme in one of the selenite reduction pathways of Rhodobacter sphaeroides f.sp.denitrificans IL106[J]. Appl Environ Microbiol,2006,72(5):3147-3153.
[32]Yee N,Ma J,Dalia A,et al.Se(VI)reduction and the precipitation of Se(0)by the facultative bacterium Enterobacter cloacae SLD1a-1 are regulated by FNR[J].Appl Environ Microbiol.2007,73(6):1914-1920.
[33]Bébien M,Kirsch J,Méjean V,et al.Involvement of a putative molybdenum enzyme in the reduction of selenate by Escherichia coli[J].Microbiology,2002,148(Pt12):3865-3872.
[34]Antonioli P,Lampis S,Chesini I,et al.Stenotrophomonas maltophilia SeITE02,a new bacterial strain suitable for bioremediation of selenite-contaminated environmental matrices[J].Appl Environ Microbiol,2007,73(21):6854-6863.
[35]Ridley H,Watts CA,Richardson DJ,et al.Development of a viologen-based microtiter plate assay for the analysis of oxyanion reductase activity:application to the membrane-bound selenate reductase from Enterobacter cloacae SLD1a-1[J].Anal Biochem,2006,358(2):289-294.
[36]Spiro S,Guest JR.FNR and its role in oxygen-regulated gene expression in Escherichia coli[J].FEMS Microbiol Rev,1990,6(4):399-428.
Research Progress on the selenium oxyanions reductase in microorganisms
WANG Dong-liang1,2,MAO Bei-bei1,ZHANG Lu1,2,XIAO Min3
(1.Shandong Dong-E E-Jiao Co.,Ltd.,Liaocheng 252201,China;2.National Engineering Technology Research Center of Glue of Traditional Medicine,Liaocheng 252201,China;3.College of Life Sciences,Shandong University,Jinan 250012,China)
The reduction of selenium oxyanions by microorganisms plays an important role in the circulation of nature.By now,3 mainly reduction ways had been discovered:selenate/selenite turns into volatile dhimethyl selenide by methyltransferase or glutathione(GSH);It will react with mercapto compounds,and turn into selenium intermediate product,and then goes into different metabolic pathways,or reduce into Se0by thiol reductase;The nitrate reductase in denitrifying bacteria has the function of reducing selenium oxyanions.The selenium oxyanions can be reduced to low state or Se0by the other kind of the selenium oxyanions reductase,the enzyme belongs to molybdenum enzymesⅡ.This text summarized the progress in research of the selenium oxyanions reductase,and application in biochemistry,genetics,genomics and proteomics.
Selenium oxyanions reductase;Molybdenum enzymes;Selenate;Selenite
S154.3
A
2095-5375(2014)01-0034-006
国家自然基金项目(糖工程技术与铂类抗肿瘤药物的靶向性改造,No.21172135)
王东亮,男,博士研究生,研究方向:中药药理,E-mail:wangdl@dongeejiao.com
