海芦笋内生真菌基于PKS Ⅰ型功能基因的分离与鉴定
2014-02-13刘怡君王明洋辛志宏
刘怡君,潘 沐,王明洋,王 惠,辛志宏
(南京农业大学食品科技学院,江苏 南京 210095)
植物内生菌为那些在生活史中的某一阶段生活在植物组织内、对植物组织没有引起明显病害症状的菌[1]。大量研究表明[2-3],植物内生菌能够产生结构新颖、活性独特的次级代谢产物,在生物制药、环境生态、农业生产等领域具有重要的应用价值,已经成为寻找和发现各种生物活性物质的重要资源。
在内生真菌的研究过程中,菌株来源和有效的筛选方法是决定能否发现新型生物活性物质的关键。常用的筛选方法是基于活性筛选和化学筛选或者二者的组合,这些方法在微生物次级代谢产物研究的过程中,曾经发挥过巨大的作用,筛选到许多盛产活性物质的菌株,但是随着研究的深入,这些筛选方法固有的缺陷也逐渐显现出来,最为突出的问题是在新化合物筛选的过程中,已知微生物和已知化合物不断被重复发现,大大增加了新化合物的发现难度,造成了巨大浪费,这已经成为植物内生真菌研究过程中的最主要的制约因素。
近年来,随着分子生物信息学的发展,部分微生物次级代谢产物合成酶系及其相关基因测序完成,生物合成机理越来越清晰,新的微生物筛选方法应运而生。
基于聚酮合成酶(polyketide synthase,PKS)功能基因定向筛选,目标直接指向合成聚酮化合物的目标基因,成为发现新型天然活性物质的重要策略之一,许多新发现的抑菌、抗肿瘤和抗病毒化合物都是由PKS合成的[4-6]。聚酮化合物的合成是以一个启动单元和若干延展单元为原料,经反复醇醛缩合和延伸而成,其过程与脂肪酸的合成类似。在合成途径中它们均会产生含有多个酮基的中间代谢产物。催化合成这些中间产物的关键酶称为聚酮合酶。PKS是生物合成次级代谢产物过程中的关键酶,对其合成基因深入研究不仅能够大大提高发现新化合物的概率,而且可以预测产生化合物的类型[7-8]。已经发现的聚酮合酶按结构和催化机制大致可分为Ⅰ型(又称模件型)、Ⅱ型(又称重复型)及Ⅲ型(又称查尔酮型)三大类[9]。真菌来源的PKS常属Ⅰ型,细菌PKS常属于Ⅰ和Ⅱ型,植物PKS属于Ⅲ型。PKS编码基因具有同源性和许多保守区域,为基因筛选提供了分子基础。PKS由载体蛋白(acylcarrier protein,CP)、酰基转移酶(acyltransferase,AT)、酮合成酶(ketosynthase,KS)、酮还原酶、脱水酶、烯脂酰还原酶等模块组成,以β-酮酸为单体按顺序依次添加到碳骨架上。PKS基因是模块化的,众多模块连在一起组成多功能酶或多酶体系,模块类型、数量和排列顺序决定了产物的多样化和功能的多样化[10-11]。基因组的常用研究方法包括免疫筛选法、遗传互补法、异源探针法和同源探针法等[12-13],其中最常用的是基于简并引物PCR扩增的同源探针法,扩增结果往往对应菌株产生的化合物类型。Bingle等[14]根据真菌PKS中最为保守的功能区域——KS单元的保守序列设计出两对简并引物(LC1/LC2c和LC3和LC5c),从不同真菌中扩增得到了苯并吡喃酮聚酮合酶(WA)型和甲基水杨酸合成酶(MSAS)型的PKS片段。WA型涉及色素和黄曲霉毒素的生物合成,MSAS型则主要参与6-甲基水杨酸的生物合成。Nicholson等[15]根据真菌PKS催化聚酮化合物合成过程中还原程度不同将其分为3类:非还原型或芬芳(non-reduced,NR)PKS,即催化过程中无还原步骤的真菌PKS;部分还原型(partially reduced,PR)PKS,即只有一个或几个还原步骤的真菌PKS;高度还原型(highly reduced,HR)PKS,即有多个还原步骤的真菌PKS。根据此分类方法,上述WA和MSAS型的PKS可分别归入NR、PR和HR 3种类型。其中WA型属于非还原型 PKS,MSAS型大多数属于部分还原型PKS,而有一部分MSAS型PKS则属于高度还原型PKS。还原型PKS通常以硫酯化羧酸辅酶A为起始单元,催化合成线性聚酮化合物,如洛伐他汀、烟曲霉毒素等,而非还原型PKS一般以乙酰辅酶A和丙二酰辅酶A为起始单元,合成环状化合物,如黄曲霉毒素、桔霉素等。
目前,通过跟踪PKS基因筛选目标菌株,大多集中在细菌与宿主的关系及其生态作用的报道,有关真菌PKS基因筛选罕见报道。
本实验以采自新疆盐湖的野生海芦笋(Salicornia bigelovii)为研究对象,采用平板划线分离、斜面分离纯化技术分离海芦笋内生真菌,以PKSⅠ型功能基因为指标,筛选出具有PKSⅠ型基因的真菌,在此基础上,提取内生菌DNA,PCR扩增18S rDNA和rDNA ITS目标序列,在NCBI上进行同源性分析,构建系统发育树,结合形态学观察,确定其种属地位,以期筛选到具有PKSⅠ型功能基因的内生真菌,提高新化合物的发现机率。
1 材料与方法
1.1 试剂与培养基
野生盐生海芦笋采自新疆盐湖。
D3390-01 E.Z.N.A真菌DNA微量提取试剂盒 美国Omega公司;DR001AM PCR试剂、Ver.3.0 D823A琼脂糖凝胶DNA提取试剂盒、D102A pMD19-T载体 日本TaKaRa公司。
PDA固体培养基(g/L):土豆200、葡萄糖20、琼脂20、NaCl 30、海水素10、海芦笋提取液250,自来水配制。
真菌发酵液体培养基(g/L):土豆200、麦芽糖20、甘露醇20、葡萄糖10、味精5、蛋白胨5、酵母膏3,pH 6.0。
LB液体培养基(g/L):胰蛋白胨10、酵母粉5、NaCl 10,pH 7.2。
1.2 仪器与设备
ECLIPSE TE2000-S 荧光倒置显微镜 日本Nikon株式会社;Microfuge 22R台式微量冷冻离心机 美国Beckman公司;TP600型梯度PCR仪 日本TaKaRa公司;DYCP-31DN电泳仪 北京市六一仪器厂;JS-380C全自动数码凝胶成像分析仪 上海培清科技有限公司。
1.3 内生真菌的分离培养
菌株Salix 1是从新疆盐湖野生海芦笋中分离得到,菌株分离采用PDA固体培养基,在无菌操作下,取1 根野生海芦笋,依次经过75%乙醇、1%次氯酸钠、75%乙醇分别浸泡1 min,然后用无菌水漂洗3 次,无菌吸水纸干燥。用灭菌后的剪刀将其剪成7~9 mm小段,放在PDA平板上,每板3~5 根,于28 ℃恒温培养至植物组织内部长出菌丝。将真菌转移到新的平板培养基上划线分离得到单个菌落,然后转移到PDA斜面试管中,4 ℃保存。
1.4 内生真菌的形态学观察
从保藏菌种的斜面培养基中挑取菌丝转接到PDA平板培养基中间位置,28 ℃培养3~4 d,观察菌落大小、颜色、质地等形态特征,记录生长速率。
1.5 基因组DNA的提取
从保藏菌种的斜面培养基中挑取菌丝转接到PDA平板上,28 ℃培养4~5 d。基因组DNA的提取采用真菌DNA微量提取试剂盒进行。称取40 mg新鲜样品至2 mL离心管,加600 μL FG1,玻璃棒研磨4 min;加5 μL 20 mg/mL RNA酶,65 ℃水浴20 min;加140 μL FG2、13 000×g离心10 min;吸600 μL上清液,加300 μL FG3和600 μL无水乙醇;将全部溶液移至HiBind DNA管,10 000×g离心1 min,弃废液。换一新2 mL收集管,加700 μL DNA Wash Buffer(已加乙醇),10 000×g离心1 min,弃废液。将HiBind DNA管放入1.5 mL离心管,加50 μL Elution Buffer,室温放置5 min,10 000×g离心1 min,收集DNA滤液。提取的基因组DNA在1%琼脂糖凝胶中电泳检测(120 V、30 min),溴化乙锭染色。4 ℃保存备用,或于-20 ℃长期保存。
1.6 PKS基因片段的PCR 扩增
PKS区域的扩增选择真核生物PKS通用的3对引物:KAF1/KAR1、KAF1/KAR2、LC1/LC2C。 KAF1 (5’-GAR KSI CAY GGI ACI GGI AC-3’);KAR1 (5’-CCA YTG IGC ICC RTG ICC IGA RAA-3’);KAR2 (5’-CCA YTG IGC ICC YTG ICC IGT RAA-3’);LC1 (5’-GAY CCI MGI TTY TTY AAY ATG-3’);LC2C (5’-GTI CCI GTI CCR TGC ATY TC-3’)。PCR反应条件为:预变性95 ℃、4 min;变性95 ℃、30 s,退火50 ℃、1 min,延伸72 ℃、2 min,共35 个循环;最后延伸72 ℃、10 min[14-15]。
1.7 18S rDNA基因片段的PCR扩增
18S rDNA区域的扩增选择真核生物18S rDNA的通用扩增引物:NS1(5’-GTAGTCATATGCTTGTCTC-3’),NS8(5’-TCCGCAGGTTCACCTACGGA-3’)。PCR反应条件为:94 ℃预变性4 min;94 ℃变性45 s,50 ℃退火1 min,72 ℃延伸1.75 min,共35 个循环;最后72 ℃延伸10 min[16]。
1.8 rDNA ITS基因片段的PCR扩增
ITS区域的扩增选择真核生物ITS通用扩增引物:ITS1(5’-TCCGTAGGTGAACCTGCGG-3’),ITS4(5’-TCCTCCGCTTATTGATATGC-3’)。PCR反应条件为:预变性94 ℃、2 min;变性94 ℃、30 s,退火59 ℃、30 s,延伸72 ℃、90 s,共35 个循环;最后延伸72 ℃、7 min[16]。
1.9 PCR产物的回收和克隆
采用琼脂糖凝胶DNA提取试剂盒回收PCR产物,纯化的PCR产物与pMD19-T载体连接,然后转化至EscherichiacoliDH5α感受态细胞中。在-70 ℃保存的100 μL感受态细胞E.coliDH5α中加入10 μL连接产物,冰中放置30 min,取出后42 ℃热激90 s,加入890 μL LB液体培养基37 ℃振荡培养1 h。菌液涂布于含100 μg/mL氨苄青霉素的LB培养基平板,倒置过夜培养,挑取白色单菌落培养。取0.5 μL菌液直接做PCR,引物为M13RV(5’-CAGGAAACAGCTATGAC-3’)和M13-47(5’-CGCCAGGGTTTTCCCAGTCACGAC-3’),电泳检测是否含有目的片段。PCR采用25 μL的反应体系,包括ddH2O 13.75 μL、10×PCR Buffer(Mg2+)5 μL、25 mmol/L MgCl22.5 μL、2.5 mmol/L dNTP 2 μL、20 μmol/L Primer-F 0.5 μL、20 μmol/L Primer-R 0.5 μL、DNA 0.5 μL、5 U/μLTaq聚合酶0.25 μL。
2 结果与分析
2.1 菌株的形态学鉴定
分离菌株Salix 1在PDA培养基上28 ℃培养4 d后,菌落呈丝绒状,直径为15~17 mm,培养初期菌落正面呈白色后转绿色直至暗褐色,气生菌丝体呈浅褐粉红色,有辐射状沟纹,菌落反面自近于无色至淡黄褐色,色素扩散于生长基质。
2.2 系统发育学分析
2.2.1 菌株Salix 1 PKS系统发育树的构建与分析
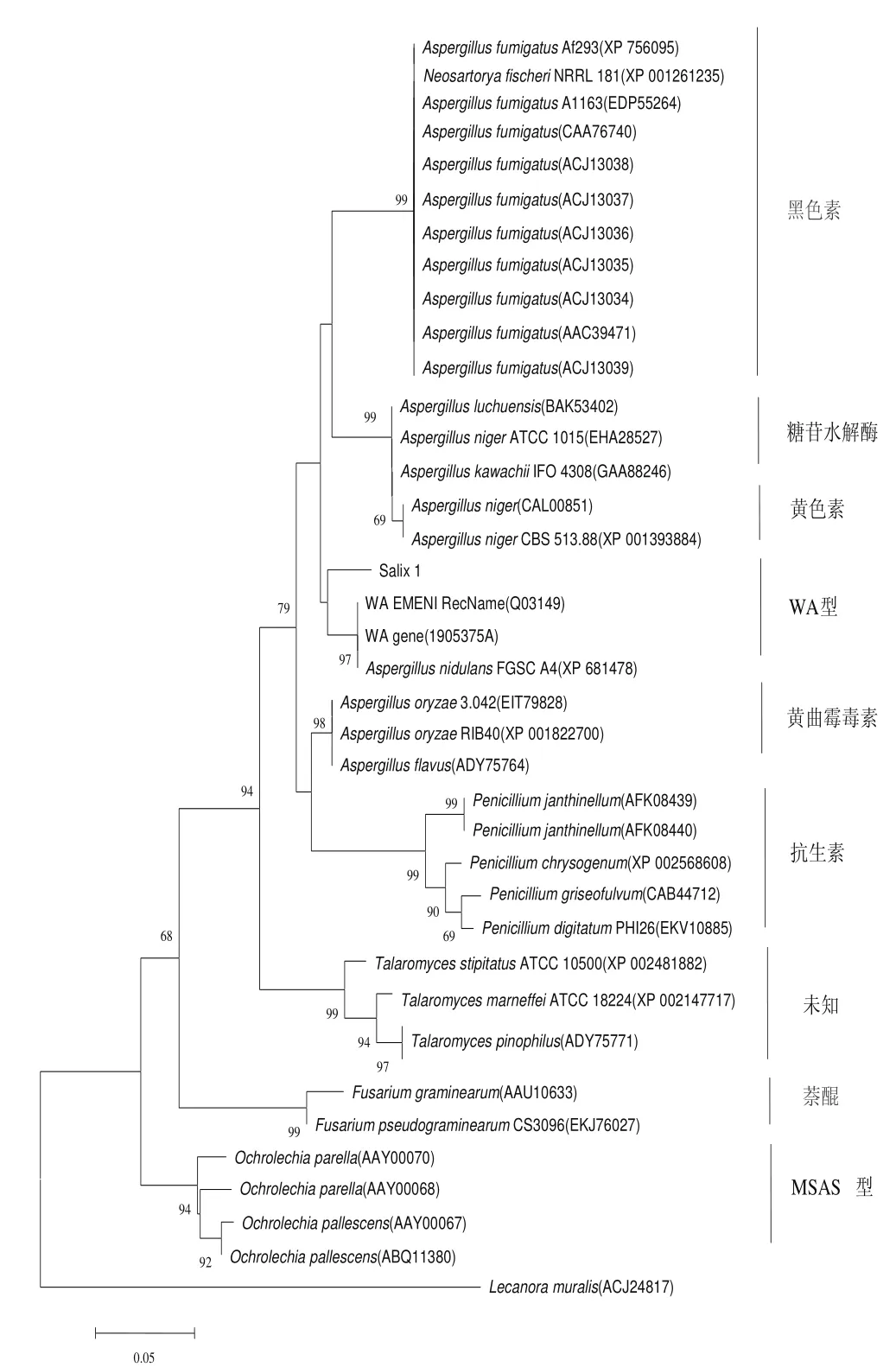
图1 基于PKS基因LC系列引物序列构建的邻接树Fig.1 Neighbor-joining tree based on LC series of primers for PKS gene sequences
分别选用KAF1/KAR1、KAF1/KAR2、LC1/LC2C3对简并引物对其PKS基因序列进行扩增。提取真菌Salix 1 DNA后进行PCR扩增,仅有LC1/LC2C引物扩增得到732 bp的目的条带并测序,该序列在NCBI蛋白数据库将核酸序列转化为蛋白序列并BLAST搜索,从中下载同源性最高的蛋白序列,采用MEGA5.0软件,将Salix 1对应的蛋白序列与37株下载的蛋白序列进行比对,以邻接法(Neighbor-Joining)构建PKS进化树,如图1所示。能够产生相同化学类型化合物的真菌分别聚为一枝,说明同种真菌的不同菌株或不同真菌能够产生相同类型的化合物,如不同的烟曲霉菌株能够产生黑色素,而黄曲霉和米曲霉都能产生黄曲霉毒素。Salix 1蛋白序列单独聚为一枝,但与WA型PKS比较接近,提示该菌株可能产生具有苯并吡喃酮聚酮的新化合物[14-15]。
2.2.2 菌株Salix 1 18S rDNA和ITS系统发育树的构建与分析
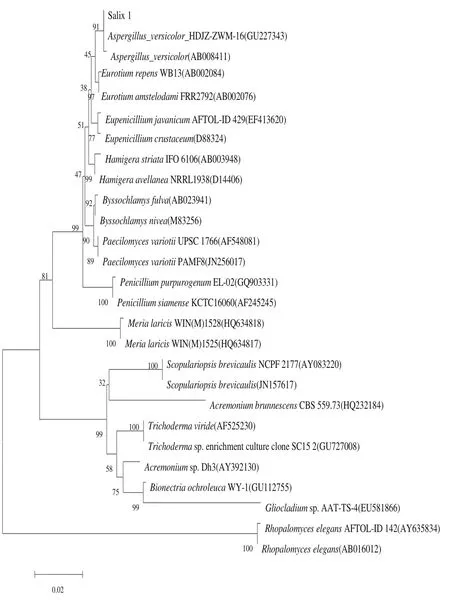
图2 基于18S rDNA基因序列构建的邻接树Fig.2 Neighbor-joining tree based on 18S rDNA gene sequences
菌株Salix 1经PCR扩增获得的18S rDNA序列全长为1 705 bp,在NCBI中进行BLAST比对,该菌株18S rDNA序列与Aspergillussp.同源性最高,相似度达99%。根据18S rDNA序列构建系统发育树,由图2可以看出,基本上不同的种形成不同的独立分枝,相同种聚为一枝,菌株Salix 1与Aspergillus versicolorHDJZ-ZWM-16(GU227343)和Aspergillus versicolor(AB008411)聚为一枝,自展值为91,表现出非常近的亲缘关系。获得的菌株ITS序列全长为573 bp,在NCBI中进行BLAST比对,该菌株ITS序列与Aspergillus versicolor的ITS序列同源性最高,相似度达99%。根据ITS序列构建系统发育树图3可以看出,基本上不同的种形成不同的独立分枝,相同种聚为一枝,菌株Salix 1与Aspergillus versicolorSGE24(JX232271)和Aspergillus versicolorRF6(GU232767)聚为一枝,自展值为89,表现出非常近的亲缘关系。综上所述,鉴定菌株Salix 1为子囊菌纲Ascomycetes、散囊菌目Eurotiales、发菌科Trichocomaceae、曲菌属Aspergillus、杂色曲霉Aspergillus versicolor。
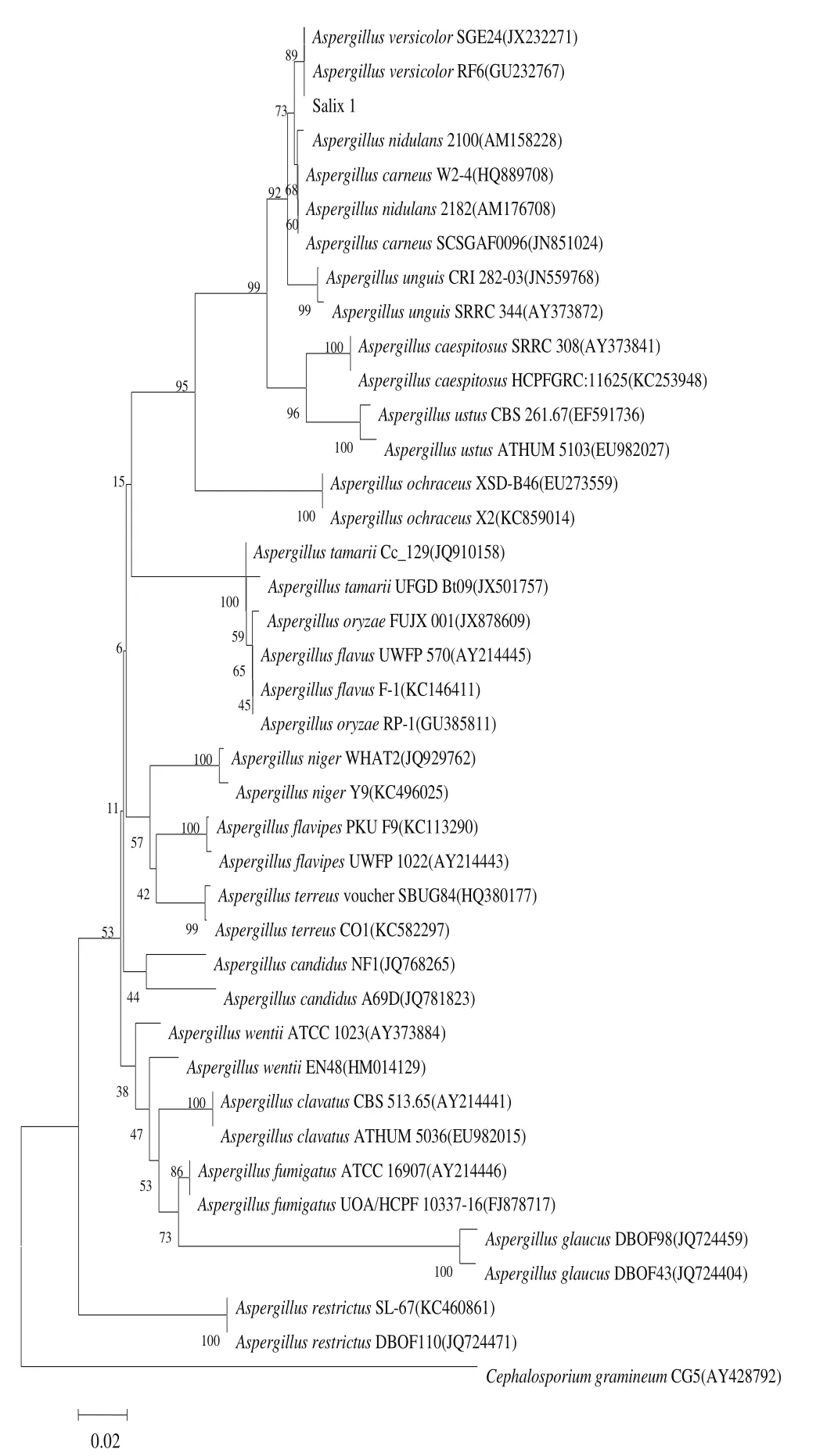
图3 基于rDNA ITS基因序列构建的邻接树Fig.3 Neighbor-joining tree based on ITS gene sequences
3 讨 论
微生物基因组中是否含有编码次级代谢产物合成的基因簇是产生相应活性产物的分子基础,分子生物学的快速发展使合成微生物次级代谢产物的合成酶系、相关基因序列及生物合成途径的逐步阐明,奠定了从基因到新化合物预测的理论基础,成为现代天然产物研究与开发的有效手段。基于PKSⅠ型基因分析方法在产生聚酮化合物真菌菌株的筛选过程中,目标直接指向新化合物,大大降低了传统活性追踪方法研究过程中的重复性,提高了工作效率。
本实验以PKSⅠ型功能基因为导向,从采自新疆盐湖的野生海芦笋中分离到1株编号为Salix 1的内生真菌含有PKSⅠ型基因,PCR扩增该菌的18S rDNA和ITS1-5.8SITS2 rDNA目标序列,经系统发育树分析结合形态学观察,将菌株Salix 1鉴定为杂色曲霉。已有研究证实,杂色曲霉能够产生结构新颖的活性次级代谢产物。Lin wenhan等[17-18]先后从海绵Xestospongia exigua中分离得到杂色曲霉,对其进行发酵培养,从发酵产物中得到13个新的次级代谢产物,利用二维核磁共振、质谱、紫外光谱等手段鉴定了这些化合物都为具有苯并吡喃酮结构的色原酮类化合物,这些化合物具有相似的化学骨架,只是取代基存在差异,这说明微生物产生的次级代谢产物大都是结构上相近的系列衍生产物,这可能是因为对于同一种菌株的次级代谢产物具有基本相同的生物合成途径的缘故。另外,不同种属的真菌也能够产生结构类似或相同的化合物,这对于筛选有价值的活性化合物高产菌株具有重要的实际应用价值。
迄今为止,还未见有关盐生海芦笋中分离出杂色曲霉的报道。尽管对杂色曲霉的次级代谢产物已有报道,但是微生物基因组中常常存在暂时丧失表达活性而静默的沉默基因,使得一些有价值的化合物在常规实验条件下产量很低或无法产生[19-20],如何通过环境刺激、培养条件优化、异源表达以及遗传学和表观遗传学手段对隐藏的次级代谢途径进行活化、上调和操纵,对于新型化合物的深入挖掘具有重要意义。今后将采用多种培养方式对该菌的发酵条件进一步优化,系统全面研究该菌的次级代谢产物与生物活性,为丰富和完善真菌功能基因的基础理论提供依据。
[1]PETRINI O.Fungal endophytes of tree leaves[M]//ANDREWS J,HIRANO S.Microbial ecology of leaves.New York: Springer New York, 1991: 179-197.
[2]RODRIGUEZ R J, WHITE J F, Jr, ARNOLD A E, et al.Fungal endophytes: diversity and functional roles[J].New Phytologist, 2009,182(2): 314-330.
[3]GAZIS R, CHAVERRI P.Diversity of fungal endophytes in leaves and stems of wild rubber trees (Hevea brasiliensis) in Peru[J].Fungal Ecology, 2010, 3(3): 240-254.
[4]SUN Wei, PENG Chongsheng, ZHAO Yunyu, et al.Functional geneguided discovery of type Ⅱ polyketides from culturable actinomycetes associated with soft coralScleronephthyasp.[J].PLoS One, 2012,7(8): e42847.
[5]ZHOU Kang, ZHANG Xia, ZHANG Fengli, et al.Phylogenetically diverse cultivable fungal community and polyketide synthase (PKS),non-ribosomal peptide synthase (NRPS) genes associated with the South China Sea sponges[J].Microbial Ecology, 2011, 62(3): 644-654.
[6]NIKOLOULI K, MOSSIALOS D.Bioactive compounds synthesized by non-ribosomal peptide synthetases and type-Ⅰ polyketide synthases discovered through genome-mining and metagenomics[J].Biotechnology Letters, 2012, 34(8): 1393-1403.
[7]SCHNEEMANN I, NAGEL K, KAJAHN I, et al.Comprehensive investigation of marine Actinobacteria associated with the spongeHalichondria panicea[J].Applied and Environmental Microbiology,2010, 76(11): 3702-3714.
[8]WATANABE A, FUJII I, SANKAWA U, et al.Re-identi fication ofAspergillus nidulanswA gene to code for a polyketide synthase of naphthopyrone[J].Tetrahedron Letters, 1999, 40(1): 91-94.
[9]SHEN Ben.Polyketide biosynthesis beyond the type Ⅰ, Ⅱ and Ⅲpolyketide synthase paradigms[J].Current Opinion in Chemical Biology, 2003, 7(2): 285-295.
[10]COX R J, SIMPSON T J.Fungal type I polyketide synthases[J].Methods in Enzymology, 2009, 459: 49-78.
[11]CHENG Y Q, COUGHLIN J M, LIM S K, et al.Type Ⅰ polyketide synthases that require discrete acyltransferases[J].Methods in Enzymology, 2009, 459: 165-186.
[12]AMNUAYKANJANASIN A, PUNYA J, PAUNGMOUNG P, et al.Diversity of type Ⅰ polyketide synthase genes in the wood-decay fungusXylariasp.BCC 1067[J].FEMS Microbiology Letters, 2005,251(1): 125-136.
[13]SCHUMANN J, HERTWECK C.Advances in cloning, functional analysis and heterologous expression of fungal polyketide synthase genes[J].Journal of Biotechnology2006, 124(4): 690-703.
[14]BINGLE L E H, SIMPSON T J, LAZARUS C M.Ketosynthase domain probes identify two subclasses of fungal polyketide synthase genes[J].Fungal Genetics and Biology,1999, 26(3): 209-223.
[15]NICHOLSON T P, RUDD B A, DAWSON M, et al. Design and utility of oligonucleotide gene probes for fungal polyketide synthases[J]. Chemistry & Biology2001, 8(2): 157-178.
[16]王惠, 湛东锐, 李连强, 等.18S rDNA和ITS相结合鉴定一株盐生海芦笋内生真菌的研究[J].食品科学, 2012, 33(21): 173-176.
[17] LIN Wenhan, LI Jun, FU Hongzheng, et al. Four novel hydropyranoindeno-derivatives from marine fungusAspergillus versicolor[J]. Chinese Chemical Letters, 2001, 12(5): 435-438.
[18] LIN Wenhan, BRAUERS G, EBEL R, et al. Novel chromone derivatives from the fungusAspergillus versicolorisolated from the marine spongeXestospongia exigua[J]. Journal of Natural Products, 2003, 66(1): 57-61.
[19] BRAKHAGE A A, SCHUEMANN J, BERGMANN S, et al. Activation of fungal silent gene clusters: a new avenue to drug discovery[J]. Progress in Drug Research, 2008, 66: 1; 3-12.
[20] NUMATA A, TAKAHASHI C, ITO Y, et al. Communesins, cytotoxic metabolites of a fungus isolated from a marine alga[J]. Tetrahedron Letters, 1993, 34(14): 2355-2358.
