亚细胞结构的超高分辨成像研究
2014-02-11贾思思邓素辉侯尚国柳华杰樊春海中国科学院上海应用物理研究所物理生物学室嘉定园区上海20800
贾思思 梁 乐 邓素辉,2 侯尚国 柳华杰,2 黄 庆,2 樊春海,2(中国科学院上海应用物理研究所物理生物学室 嘉定园区 上海 20800)
2(中国科学院上海应用物理研究所光源生物成像中心 嘉定园区 上海 201800)
亚细胞结构的超高分辨成像研究
贾思思1梁 乐1邓素辉1,2侯尚国1柳华杰1,2黄 庆1,2樊春海1,21(中国科学院上海应用物理研究所物理生物学室 嘉定园区 上海 201800)
2(中国科学院上海应用物理研究所光源生物成像中心 嘉定园区 上海 201800)
本文介绍了3种常用的亚细胞结构成像技术包括荧光超分辨显微成像术、软X-射线显微镜和三维冷冻电镜的成像原理,细胞成像应用及所存在的问题。并提出多种显微镜联用的关联显微术由于结合了多种显微镜的优势,能够提供多方位信息,丰富细胞生物学的研究,有可能给细胞学带来全新的认识。
亚细胞显微成像,荧光超分辨显微成像术,软X-射线显微镜,三维冷冻电镜,关联显微术
作为生命活动的基本单元,细胞无疑是生物学家最重要的研究对象。细胞是一个内部高度有序,组织复杂的结构,据空间分布和功能不同,可以分成不同细胞器或细胞区域,如细胞核、高尔基体、内质网、线粒体、胞浆和细胞膜等。研究细胞内这些亚细胞结构生物体的空间分布、结构、功能及相互作用是揭示生命本质的重要途径。例如,蛋白质作为生命活动的执行者,构成了细胞中所有的组织结构,并执行了所有细胞活动。蛋白质在核糖体中合成后,经蛋白质分选信号引导被运转到特定的细胞器中,因此蛋白质的胞内定位对于该蛋白质功能分析非常重要[1]。事实上,这些亚细胞结构大多处于纳米尺度,如细胞器核糖体直径在25 nm,蛋白质分子及复合物则是在10 nm以下[2]。因此,细胞学的进展很大程度上依赖于显微成像技术的进步。只有当显微镜分辨率达到这些细胞器相当的尺度时,这些结构才能被分辨出来。然而,光学显微镜的分辨率并不能无限制的提高。早在1873年,德国科学家阿贝(Ernst Abbe)[3]就指出,由于光的波动性和光的衍射效应的影响,成像系统存在分辨率极限。当使用波长为λ的单色光,数值孔径为 An=nsinα的单透镜进行成像时,该系统的横向和纵向分辨率极限的关系式见公式(1)和(2)。
dx,y=0.61λ/(nsinα) (1)
dz=2λ/(nsin2α) (2)
以上表达式表明,分辨率受到用于成像的光波波长和物镜数值孔径的限制,显微镜所能看清的物体最小尺寸约为光波长的一半。对于可见光波段,显微镜只能对亚微米级的结构进行观察。即使是利用波长较短的紫外光显微镜,极限分辨率提升不足一倍,仍然不优于200 nm,远远不能满足细胞生物学中对亚细胞结构的分辨率要求[3]。为了看清楚这些结构,就必须使用突破衍射极限的荧光成像方法或者选择更短波长的光源来提高显微镜的分辨率。X-射线是一种波长很短的电磁辐射,波长范围在0.01-10 nm。另外,电子束的波长也很短,其波长与发射电子束的电压平方根成反比,电压越高波长越短。当加速电压高达30 keV,电子的德布洛意波长为0.007 nm。因此,当如此短波长的X-射线和电子束用于成像时,衍射受限的分辨率可达到几个纳米量级。
由于能够提供超高分辨率,荧光超分辨成像技术,基于第三代同步辐射光源的软X-射线成像技术和三维冷冻电镜成像技术是研究细胞内蛋白质活动和亚细胞器结构的有力工具。这3种技术有着各自不同的特点和物理原理,应用范围也有所不同。本文重点概述细胞领域成像常用的这3种技术的成像原理,细胞成像应用及研究现状,希望结合3种技术的优势,相辅相成,给细胞生物学亚细胞成像带来全新的发展前景。
1 荧光超分辨显微成像术
荧光光学显微镜由于其特异性好,灵敏度高,不会对细胞造成损伤,可探测样品内部的三维活细胞成像等优势,一直是生物学研究的最常用工具之一。近20年来,利用光学非线性效应,出现了一系列打破光学衍射极限的荧光超分辨技术,其分辨率提高到几十纳米,为研究亚细胞结构与组织提供了前所未有的手段[4]。目前,超分辨显微镜技术大概分为两类,一类是以受激发射损耗显微镜(Stimulated emission depletion microscopy, STED)为代表的模式光照明成像方法;另一类是基于单分子定位的成像技术。这两类方法的共同点是避免衍射极限范围内,相互位置过于靠近而无法被传统光学显微镜同时分辨的荧光分子集体发射荧光,通过一些光学技巧或光开关荧光分子来控制部分或单个荧光分子激发,按时间先后有序或无序的被记录成像。
第一类方法通常使用精心设计的结构光构造出小于衍射极限的光点照明样品,来获得远小于衍射极限的荧光发射。例如,1994年,德国马普研究所Hell[5]提出的STED方法中,使用两束光来构造荧光发光点,即一束聚焦的激发光与另一束中空环形焦斑的损耗光重合,利用环形焦斑将激发态上的荧光分子发生受激发射过程从激发态回到基态而剥夺周边的荧光发射能力,从而将荧光光点局限在中心很小的区域。该方法中,随着损耗光增强,中心的荧光分子发射面积可以被进一步的压缩,实现极高的分辨率。利用无漂白的钻石中氮晶格空位色心为样品,STED显微镜报道的最好的空间横向分辨率已达到2.4 nm,如图1a所示[6]。
目前,STED显微镜已经成功应用于神经生物学、细胞膜生物学和细胞内动力学的观察。普遍情况下,亚细胞内的蛋白成像能达到30 nm横向分辨率和50 nm的纵向分辨率[7]。2013年最新的研究结果表明,STED技术已实现100,000面包圈点同时成像,此方法大大提高了成像速度,在不到1 s的时间内,获得了一幅120 μm × 100 μm大尺寸的活细胞高分辨率图片[8]。作为一个生物学应用的典型特例,STED显微镜成功地对线粒体内膜组织系统(Mitochondrial Inner Membrane Organizing System, MINOS)在线粒体内的空间分布进行了探索。我们知道MINOS是一个保守的异质低聚的蛋白复合物,主要作用是维持线粒体嵴形态及控制线粒体的功能与结构,与一些人类疾病相关。长久以来,由于缺乏超高分辨率的有效手段,对于MINOS在线粒体内的空间分布一直不清楚。2013年,Jans等[9]结合STED技术与免疫电镜相,揭示了MINOS的3种已知蛋白(Mitoflin、ATPs和TOM20)的具体分布,如图1b所示。该图表明3种亚基在线粒体内形成了类似的团簇,并且Mitofilin倾向于定位在线粒体嵴结合处,靠近细胞核的线粒体内的MINOS含量明显高过外周的线粒体。这些超分辨图片说明了MINOS复合物在纳米尺度的不可思议的高度有序分布。
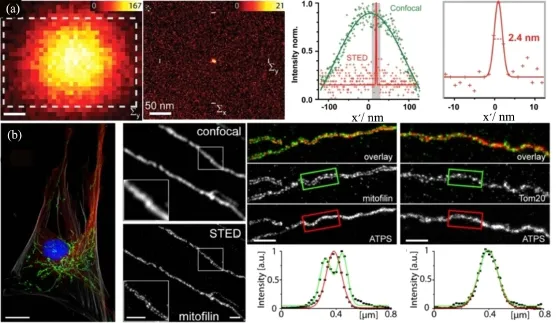
图1 受激发射损耗(STED)显微术突破光学衍射极限: (a) 利用钻石氮晶格色心为样品, STED实现(2.4 ±0.3) nm的分辨率[6]; (b) STED给出了线粒体内膜组织系统(MINOS)的3种已知蛋白(Mitoflin、ATPs和TOM200的胞内分布[9]Fig.1 STED breaks the diffraction barrier: (a) STED provides a resolution down to (2.4 ±0.3) nm using the samples of the diamond nitrogen vacancy centers[6]; (b) by using the STED, the distribution of three proteins of MINOS (Mitofilin, ATPs, and TOM20) in mammalian cells are clearly displayed[9]
这一类方法还包括基态损耗显微镜(Ground statedepletion microscopy, GSD)和饱和结构光照明显微镜(Saturated structured-illumination microscopy, SSIM)等,其优势在于提供高分辨率的同时也能高速成像,其缺点在于光路复杂,对光学系统的构建和维护提出了较高的要求。同时,使用的激光功率较高,会造成生物样品的光毒性等。
由于单分子定位不受衍射极限的限制,定位精度只与荧光强度相关,可达1 nm级别[10]。利用这个优势,第二类基于单分子成像的方法获得了超高分辨率图像。该方法使用光控荧光分子或者光敏蛋白作为探针,每一帧图像中随机的唤醒距离大于衍射斑大小的多个单分子,收集荧光并进行单分子定位。重复这个过程,进行多帧拍摄并采集了足够多的单分子位置信息后,将这些位置进行叠加,数模模拟重构出分辨率可与单分子定位精度媲美的超分辨图像。其典型代表有庄小威小组,Eric Betzig小组和Samuel Hess小组几乎同时发展的随机光重建显微术(Stochastic optical reconstruction microscopy, STORM)[11]、光敏定位显微术(Photoactivated localization microscopy, PALM)[12]和荧光光敏定位显微术(Fluorescence photo-activation localization microscopy, fPALM)[13]。例如,在PALM成像(图2a)中,首先采用极弱的敏化激光辐照光敏蛋白标记的样品,再使用激发光激发直至漂白。在此过程中,发荧光的单分子位置被记录。最后,将所有记录的单分子位置叠加,重构出超分辨图像[12]。目前,这一类方法应用于细胞成像中,已获得了约20 nm的横向分辨率,50 nm的纵向分辨率。利用该技术的超高分辨率,实现了对亚细胞组织结构的观察。例如在2013年,STORM超分辨显微术可视化的解析了细胞内端粒的结构功能[14],并且在去除端粒的帽蛋白情况下,进一步开展了端粒功能异常的结构状态的研究。同年,庄小威小组利用STORM技术对神经元中发挥重要作用的肌动蛋白和血影蛋白在轴突和树突的组织结构进行了高分辨成像[15]。结果表明肌动蛋白形成了180-190 nm指环状周期性结构,并均匀的包裹着轴突。这个周期性结构并未出现在树突,取而代之的是沿着树突形成肌动蛋白纤维。这些超分辨图像也展示了覆盖蛋白Adducin定位在肌动蛋白指环结构上,Spectrin蛋白与Actin和Adducin形成的结构交替形成周期性结构,并且两个相邻的Actin-Adducin复合物的距离与Spertrin四聚体的长度一致,如图 2b所示[15]。这一类超分辨技术能够提供极高的空间分辨率。但如此之高的空间分辨率要求定位精度极高,因此单分子成像时的荧光强度要足够强。为了收集足够的光子数,每一帧图像曝光时间较长,即时间分辨率较差。有报道的STORM最好成像速度约0.5 s/帧[16]。因此,该类技术还无法对快速的细胞内动力学行为开展超分辨成像。
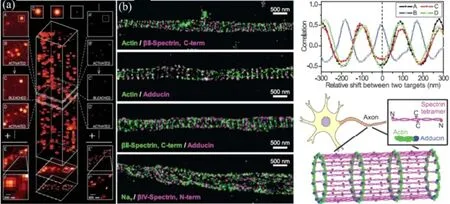
图2 (a)光敏定位显微术(PALM)原理示意图[12]; (b) STORM超分辨图像表明Spectrin蛋白与Actin和Adducin形成的结构交替形成周期性结构[15]Fig.2 (a) The principle behind PALM[12]; (b) the data of STORM images show that actin, spectrin, and adducinform a coordinated, quasi-1D lattice structure in axons, and sodium channels are distributed in a periodic pattern in coordination with the actin-spectrin based submembrane cytoskeleton[15]
2 同步辐射软X-射线成像
X-射线波长比可见光短了3个数量级,在理论上可以实现几个纳米量级的分辨率。与电子束相比,X-射线具有大得多的穿透能力。因此,X-射线显微镜不仅具有对厚样品进行纳米分辨成像的能力,也具有三维分辨的巨大潜力[17]。在X-射线显微术中,当 X-射线与物质相互作用时发生光电吸收、散射等,其振幅和相位等会发生改变,利用这种振幅和相位的变化,建立起物体图像。另外还有荧光、化学态、自旋等多种成像方式[18]。碳水化合物、蛋白质和水是构成生物样品的主要物质。对于以轻元素构成的生物样品,由于对硬X-射线的吸收较少或者吸收差别比较小,一般得到衬度很差的吸收像。而使用波长较长的软X射线(2.3-4.4 nm)作为光源时,蛋白质的吸收系数比水高一个数量级,水的吸收系数较小,可以认为水是透明的。利用水窗波段的软X-射线对水的高穿透性特点,生物样品不必进行脱水处理,观测时也无需处于高真空的状态,因此,软X-射线能提供自然状态下的清晰的细胞图像,成为纳米级别亚细胞结构成像的有力工具。另外,在软X-射线波长范围内,存在一些元素的吸收边,在这些吸收边附近稍微改变波长,吸收系数会有很大的改变。利用这一特性,就可以开展近边吸收精细结构谱学的研究[19],即调整X-射线波长到吸收边两侧获得两个不同的吸收谱,将这两个图进行比较并相减,就可以加强该元素的衬度而做出元素的微区分布图。
在X-射线波段,各种透明材料的折射率小于并接近1,常规光学元件难以使X-射线弯曲聚焦。长期以来,由于缺乏对X-射线精确聚焦的光学元件和高亮度 X-射线源的因素,制约了 X-射线的细胞成像技术的发展。波带片是根据菲涅尔衍射原理制成的一种透射衍射光栅,它是由线密度径向增加的透明和吸收材料相间的同心圆环带组成,它既是色散元件也能聚焦,是实现X-射线成像的理想元件。透射式X-射线显微镜成像原理如图3a所示[20],先使用会聚波带片将软X-射线会聚并投射到样品上,然后再通过微波带片等光学元件作为物镜成像到像平面上。放大的软X-射线显微图就能直接在像平面上的照相底片或 CCD上呈现。波带片成像的分辨率近似等于波带片最外环宽度。目前国际上制作的波带片最外环宽度已达15 nm,如图3b所示的扫描电镜表征的波带片[20]。利用此波带片,劳伦斯伯克利国家实验室清晰分辨出了 15.1 nm的周期性样品(图3c)[20],实现了15 nm的软X-射线空间分辨率,为在纳米尺度上观察细胞内的生物大分子提供了可能。
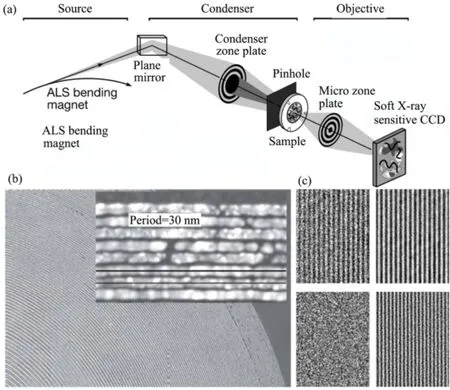
图3 (a)同步辐射软X-射线显微术示意图; (b)扫描电镜表征最外环宽度是15 nm的微波带片; (c)分别用25 nm(左边)和15 nm(右边)的波带片,软X-射线显微镜实现了对间隔19 nm (上面)和15.1 nm(下面)的周期性样品的高分辨成像[20]Fig.3 (a) The diagram of the soft X-ray microscope; (b) scanning electron micrograph of a zone plate with 15 nm outermost zone; (c) soft X-ray images of the 19.5 nm (above) and 15.1 nm (below) half-period test objects, using the zone plates with outer zone widths of 25 nm (left) and15 nm (right), respectively[20]
实际生物样品成像中,软X-射线成像分辨率还受辐射剂量和样品厚度的影响。为了避免辐射损伤对分辨率的影响,成像必须在损伤前完成,即要求在较短时间内有大通量的软X-射线入射到样品。考虑到这些因素,透射软X-射线显微术的分辨率约为50-60 nm。
另一个长期制约 X-射线的细胞成像技术因素是传统的X-射线源的亮度低。同步辐射光源由于能够提供高亮度,高准直性,高偏振,窄脉冲等优异性能的X光,是X-射线源的最佳选择。例如,第三代同步辐射光源的X-射线亮度是普通X光机的上千亿倍[21]。因此,基于同步辐射的X-射线显微镜将进一步推动其在细胞生物学的研究。
利用软X-射线显微镜,科学家们在细胞成像领域做了大量的工作。Larabell等首先在劳伦斯伯克利国家实验室的先进光源(Advanced Light Source, ALS)线上做了很多细胞三维结构观察方面的开创性工作。2001年,该小组利用金纳米颗粒的吸收衬度,通过免疫组化,获得了如图4a所示的纳米金标记的整细胞微管的空间分布图[22]。进一步的,将低温成像技术和断层扫描技术与 X-射线显微成像技术相结合,Uchida等[23]发展了冷冻三维断层扫描X-射线显微术,该方法降低了X-射线对辐射敏感的样品(特别是生物样品)的损伤,并通过一系列细胞断层的图片重构得到酵母细胞的三维成像,如图4b所示。该图中,细胞核、细胞胞质、类脂滴和液泡可以被清楚的观察到,空间分辨率达到了60 nm。Müller等[24]使用三维软 X-射线显微镜对 6-12 μm厚的小鼠腺癌细胞进行成像,获得了细胞核、核仁、核膜、核膜通道、线粒体、溶酶体、内质网、高尔基体、细胞微绒毛和核膜泡等细胞内亚细胞结构的高分辨图片。2012年,McDermott等[25]系统的概述了利用软 X-射线三维断层显微术能可视化并定量研究细胞表型差异的研究进展,表明软X-射线三维断层显微术能被用于对内部完全水合的结构,完整的真核和原核细胞成像,可实现50 nm或更好的空间分辨率。
利用不同种类的亚细胞组织的衬度差异,软X-射线显微镜能够实现非标记的亚细胞成像。但我们也注意到,影响X-射线进行细胞显微成像效果的一个重要因素是辐射损伤。当 X-射线与物质相互作用,会引起吸收物质结构的永久性变化,容易发生细胞死亡,化学破坏、或者形态改变等,限制了其在活细胞成像中的应用。
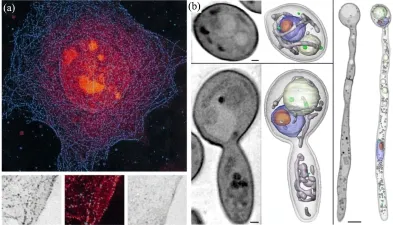
图4 (a)同步辐射软X-射线显微镜对纳米金标记的整个细胞微管网络成像[22]; (b)同步辐射软X-射线显微镜对不同形态的酵母细胞进行断层扫描重构[23]Fig.4 (a) Soft X-ray microscope image of the nano gold-labelled microtubule network (blue) in a whole cell[22]; (b) soft X-ray tomographic reconstruction of phenotypically distinct C. albicans cells[23]
3 三维冷冻电镜成像术
电子的德布罗意波长由加速电压决定,电子波长与电子加速电压的关系为速电压为100 keV时,电子波长为0.037 埃,远小于可见光的波长。因此,电子显微镜能达到很高的分辨率,与可见光显微镜分辨率(200 nm)相比,其分辨率提高了1000倍,可达0.1 nm,被广泛应用于亚细胞组织和蛋白的空间定位和结构解析。由于电子散射对于蛋白质和水没有太大的差异,利用电子显微镜研究细胞时,需要对样品进行脱水和样品染色增加衬度等处理,同时常规的电镜观测样本必须在真空中,这些因素导致电子显微镜难以研究活的生物细胞。另外,透射电子显微镜观测到的是三维物体的二维投影,不能获得样品内部的三维结构。
为了解决上述问题,1968年,剑桥大学的Klug小组[26]将电子显微镜技术与计算机图像处理技术结合发展了三维重构技术,通过获取同一物体的多个连续角度下的二维投影图,再使用傅里叶变换及反傅立叶变换来反向重构它的三维结构(图 5a),并且首次用三维重构技术得到了 T4噬菌体尾部的三维结构,如图 5b所示[27]。为了克服传统的生物样品电镜的缺陷,1974年,Taylor等[28]开创性的用冷冻电镜技术维持了生物样品的水,并减小了辐照损伤。随后,Dubochet等[29]对冷冻电镜技术进行改进,使生物冷冻电镜技术成为生物学研究的常规技术。冷冻电镜与传统电镜主要优势在于以下3个方面:(1) 冷冻制样,当水以超过105K/s的速度冷却时,不会形成晶体冰,而避免对细胞结构和蛋白质形成损伤。(2) 低温冷台,其稳定性关系到成像质量。(3) 低曝光剂量,减少对生物样品的损害。
近年来,借助于计算机硬件的发展和数据处理能力的增强,及电子显微镜的发展,冷冻电镜方法结合三维重构技术在结构生物学领域和细胞成像应用领域发展迅速并正在取得重要突破,三维冷冻电镜成像技术已经成为蛋白及病毒结构,分布与功能研究的重要工具手段,已经成功解析了众多蛋白的三维结构信息,得到很多详细的资料[30]。例如,2009年北京大学的尹长城等[31]使用三维冷冻电镜成功解析了戊型肝炎病毒样颗粒的结构,分辨率达到0.35 nm,如图5c所示。该图表明每个病毒衣壳蛋白有3个线性结构域构成及功能区域。该研究的结构模型的建立使人们极大的了解了戊型肝炎病毒如何入侵细胞的过程,并能为发展相关疫苗和抗病毒医疗的研究提供指导。2010年,周正洪等利用冷冻电镜实现了0.36 nm的分辨率,解析了3个主要病毒衣壳蛋白(IIIa, VIII和IX)的结构[32],研究了这3种蛋白相互作用的3个网络,结果表明腺病毒导致的癌症的最佳治疗方案是基因靶向这几个蛋白相互作用网络,使腺病毒失去侵染功能。传统的研究方法对于研究无包被病毒是如何改变形态进入细胞和如何穿透细胞膜的机制了解很少。Zhang等[33]通过冷冻电镜在原子水平揭示病毒颗粒的结构,发现病毒在入侵过程中涉及到膜穿透蛋白的自剪裁过程,暴露它们的细胞膜插入结构域,实现以0.33 nm的分辨率研究无包被病毒入侵细胞的机制。
电子的散射系数较小,对于电子显微镜来说,其穿透深度有限,约0.1 μm。因此电子显微镜不能用于观察较厚的样品,必须采用切片、固化等技术,可能破坏样本原有的结构,造成样品的破坏。同时,由于特殊的制样要求,导致冷冻电镜无法对活细胞内的蛋白质运输和相互作用进行实时观察。
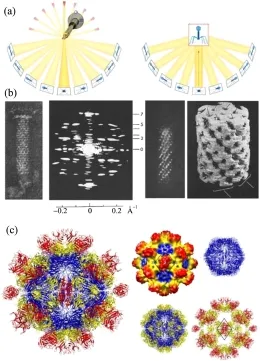
图5 (a)电镜三维重构原理示意图[26];(b)三维冷冻电镜技术得到了T4噬菌体尾部的三维结构[27];(c)三维冷冻电镜技术重构戊型肝炎病毒样颗粒的结构[31]Fig.5 (a) Principles of electron tomography[26]; (b) reconstruction of three dimensional structures of a bacteriophage T4 tail[27]using the three dimensional cryo-electron microscopy; (c) structure of the hepatitis E virus-like particle[33]using the threedimensional cryo-electron microscopy
4 结论与展望
本文概述了当前常用的3种高分辨率的亚细胞结构成像的显微镜技术的发展和各自的适用范围,这3种高分辨率显微镜在研究蛋白质的空间分布及结构功能都具有独特的优势。首先荧光超分辨显微镜能够用于活细胞成像,可实时动态观察生物样品,并且是对样品损伤最小,成像速度最快的的一种成像技术。但分辨率相对较低,也伴随着高功率激光带来的光毒性,光路系统复杂,或时间分辨率较低等问题。受多种荧光标记技术的发展和针对不同波长有不同的光照方案的限制,可见光的超分辨多色成像相对较难实现。同步辐射软X-射线显微镜利用短波长的X-射线进行照明,空间分辨率较高,而且利用样品对X-射线的吸收不同,实现无标记的细胞结构的多色成像相对容易。相对于电子显微镜,由于生物样品对 X-射线的吸收截面远大于对电子的散射截面,因此软X-射线成像的穿透深度较大,可以实现比电镜厚的样品,可达10 μm,非常适合对完整的细胞进行成像。但由于需要依托于同步辐射光源,能应用于软X-射线成像的第三代同步辐射装置有限,实际应用会受到限制。X-射线固有的辐射损伤也限制其对活细胞成像的应用研究。三维冷冻电镜能达到 0.1 nm的分辨率,远高于以上两种技术,是唯一能对蛋白质实现原子水平三维结构成像的技术,对于解析那些无法结晶的蛋白质有着重大意义。碍于电子有限的穿透深度,三维冷冻电镜不能用于较厚的样品成像,同时样品的制备过程非常复杂,并且对样品的制备有特殊要求,对生物样品有一定的损伤,无法开展活细胞研究。
最近的研究报道表明,结合不同显微镜方法的优势,“关联显微成像技术”(Correlative microscopy)联合多种显微镜共同使用,成为新的研究热点和发展趋势。例如,2011年Nature Methods报道了联用了多种超分辨光学显微镜和电子显微镜[34],开展了多种蛋白在亚细胞水平的定位的研究,获得了组蛋白H2B在细胞核中的高分辨定位和TOM20蛋白在线粒体的定位,如图6所示。该项研究利用荧光成像的特异性和电镜提供的整个亚细胞结构的超高分辨率成像,给出了特定蛋白在亚细胞水平的具体定位和分布。2014年,Sochacki等[35]将超分辨单分子定位显微术与扫描透射电镜关联使用,以20 nm的分辨率揭示了细胞膜上蛋白的三维结构,获得了内吞过程中网格蛋白小泡上的Epsin蛋白在细胞质膜的三维结构分布。因此,多种显微镜技术的结合与联用,多方位信息互相补充,有可能为细胞的研究带来全新的认识。
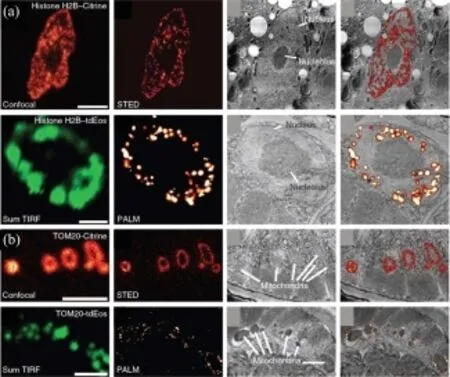
图6 关联超分辨显微术[34]联用光学超分辨显微镜和电子显微镜获得了组蛋白H2B在细胞核中的定位分布(a)和TOM20蛋白在线粒体的高分辨定位分布(b)Fig.6 The distribution of histone H2B fusion proteins in the cell nucleus (a)and the distribution of TOM20 fusion proteins in the mitochondriaby(b) correlative fluorescence and electron microscopy[34]
1 Eisenberg D, Marcotte E M, Xenarios I, et al. Protein function in the post-genomic era [J]. Nature, 2000, 405 (6788): 823-826.
2 Gitai Z. The new bacterial cell biology: moving parts and subcellular architecture [J]. Cell, 2005, 120(5): 577-586.
3 Abbe E. Beiträge zur Theorie des Mikroskops und der mikroskopischen Wahrnehmung [J]. Archiv für mikroskopische Anatomie, 1873, 9(1): 413-418.
4 Hell S W. Far-field optical nanoscopy [J]. Science, 2007, 316(5828): 1153-1158.
5 Hell S W, Wichmann J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emissiondepletion fluorescence microscopy [J]. Optics Letters, 1994, 19(11): 780-782.
6 Wildanger D, Patton B R, Schill H, et al. Solid immersion facilitates fluorescence microscopy with nanometer resolution and sub-Ångström emitter localization [J]. Advanced Materials, 2012, 24(44): OP309-OP313.
7 Huang B, Babcock H, Zhuang X. Breaking the diffraction barrier: super-resolution imaging of cells [J]. Cell, 2010, 143(7): 1047-1058.
8 Chmyrov A, Keller J, Grotjohann T, et al. Nanoscopy with more than 100,000 'doughnuts' [J]. Nature Methods, 2013, 10(8): 737-740.
9 Jans D C, Wurm C A, Riedel D, et al. STED super-resolution microscopy reveals an array of MINOS clusters along human mitochondria [J]. Proceedings of the National Academy of Sciences, 2013, 110(22): 8936-8941.
10 Yildiz A, Forkey J N, McKinney S A, et al. Myosin V walks hand-over-hand: single fluorophore imaging with 1.5-nm localization [J]. Science, 2003, 300(5628): 2061-2065.
11 Rust M J, Bates M, Zhuang X. Sub-diffraction-limitimaging by stochastic optical reconstruction microscopy (STORM) [J]. Nature Methods, 2006, 3(10): 793-796.
12 Betzig E, Patterson G H, Sougrat R, et al. Imaging intracellular fluorescent proteins at nanometer resolution [J]. Science, 2006, 313(5793): 1642-1645.
13 Hess S T, Girirajan T P K, Mason M D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy [J]. Biophysical Journal, 2006, 91(11): 4258-4272.
14 Doksani Y, Wu J Y, de Lange T, et al. Super-resolution fluorescence imaging of telomeres reveals TRF2-dependent T-loop formation [J]. Cell, 2013, 155(2): 345-356.
15 Xu K, Zhong G, Zhuang X. Actin, spectrin, and associated proteins form a periodic cytoskeletal structure in axons [J]. Science, 2013, 339(6118): 452-456.
16 Jones S A, Shim S H, He J, et al. Fast, three-dimensional super-resolution imaging of live cells [J]. Nature Methods, 2011, 8(6): 499-505.
17 Fahrni C J. Biological applications of X-ray fluorescence microscopy: exploring the subcellular topography and speciation of transition metals [J]. Current Opinion in Chemical Biology, 2007, 11(2): 121-127.
18 Chen H, Rogalski M M, Anker J N. Advances in functional X-ray imaging techniques and contrast agents [J]. Physical Chemistry Chemical Physics, 2012, 14(39): 13469-13486.
19 Ade H, Zhang X, Cameron S, et al. Chemical contrast in X-ray microscopy and spatially resolved XANES spectroscopy of organic specimens [J]. Science, 1992, 258(5084): 972-975.
20 Chao W, Harteneck B D, Liddle J A, et al. Soft X-ray microscopy at a spatial resolution better than 15 nm [J]. Nature, 2005, 435(7046): 1210-1213.
21 冼鼎昌. 同步辐射的现状和发展 [J]. 中国科学基金2005, 19(6): 321-325. XIAN Dingchang. Present status and development of synchrotron radiation [J]. Bulletin of national natural foundation of China, 2005, 19(6): 321-325.
22 Meyer-Ilse W, Hamamoto D, Nair A, et al. High resolution protein localization using soft X-ray microscopy [J]. Journal of Microscopy, 2001, 201(3): 395-403.
23 Uchida M, McDermott G, Wetzler M, et al. Soft X-ray tomography of phenotypic switching and the cellular response to antifungal peptoids in Candida albicans [J]. Proceedings of the National Academy of Sciences, 2009, 106(46): 19375-19380.
24 Muller W G, Heymann J B, Nagashima K, et al. Towards an atlas of mammalian cell ultrastructure by cryo soft X-ray tomography [J]. Journal of Structural Biology, 2012, 177(2): 179-192.
25 McDermott G, Fox D M, Epperly L, et al. Visualizing and quantifying cell phenotype using soft X-ray tomography [J]. Bio Essays, 2012, 34(4): 320-327.
26 McIntosh R, Nicastro D, Mastronarde D. New views of cells in 3D: an introduction to electron tomography [J]. Trends in Cell Biology, 2005, 15(1): 43-51.
27 De Rosier D J, Klug A. Reconstruction of three dimensional structures from electron micrographs [J]. Nature 1968, 217(5124): 130-134.
28 Taylor K A, Glaeser R M. Electron diffraction of frozen, hydrated protein crystals [J]. Science, 1974, 186(4168): 1036-1037.
29 Dubochet J, Adrian M, Chang J J, et al. Cryo-electron microscopy of vitrified specimens [J]. Quarterly Reviews of Biophysics, 1988, 21(2): 129-228.
30 Stark H, Dube P, Luhrmann R, et al. Arrangement of RNA and proteins in the spliceosomal U1 small nuclear ribonucleoprotein particle [J]. Nature 2001, 409(6819): 539-542.
31 Guu T S Y, Liu Z, Ye Q, et al. Structure of the hepatitis E virus-like particle suggests mechanisms for virus assembly and receptor binding [J]. Proceedings of the National Academy of Sciences, 2009, 10(1073): 09048-48106.
32 Liu H, Jin L, Koh S B S, et al. Atomic structure of human adenovirus by cryo-EM reveals interactions among protein networks [J]. Science, 2010, 329(5995): 1038-1043.
33 Zhang X, Jin L, Fang Q, et al. 3.3 angstrom cryo-EM structure of a nonenveloped virus reveals a priming mechanism for cell entry [J]. Cell, 2010, 141(3): 472-482.
34 Watanabe S, Punge A, Hollopeter G, et al. Protein localization in electron micrographs using fluorescence nanoscopy [J]. Nature Methods, 2011, 8(1): 80-84.
35 Sochacki K A, Shtengel G, van Engelenburg S B, et al. Correlative super-resolution fluorescence and metal-replica transmission electron microscopy [J]. Nature methods, 2014, 11(3): 305-308.
Study on the imaging of subcellular organelles with superresolution microscopy
JIA Sisi1LIANG Le1DENG Suhui1,2HOU Shangguo1LIU Huajie1,2HUANG Qing1,2FAN Chunhai1,21(Laboratory of Physical Biology, Shanghai Institute of Applied Physics, Chinese Academy of Sciences, Jiading Campus, Shanghai 201800, China)
2(Center of Bio-imaging, Shanghai Sychrotron Radiation Facility, Chinese Academy of Sciences, Jiading Campus, Shanghai 201800, China)
This review focused on the developments of three common microscopes in cell biology including far-field light nanoscopy, soft X-ray microscopy based on the synchrotron radiation light source, and three-dimensional cryo-electron microscopy. Their mechanisms, applications in cell imaging and the existing problems are discussed. The correlative microscopy combining several imaging methods can supply more information and may provide a new insight into cell biology in the future.
Subcellular imaging, Fluorescence super-resolution microscopy, Soft X-ray microscopy, Threedimensionalcryo-electronmicroscopy, Correlative microscopy
Q2-33,TL11
10.11889/j.1000-3436.2014.rrj.32.040101
国家自然科学基金项目(61378062, 21227804)资助
贾思思,女,1987年1月出生,上海应用物理研究所物理生物学实验室在读博士生,E-mail: jiasisi@sinap.ac.cn通讯作者:邓素辉,博士,副研究员,E-mail: dengsuhui@sinap.ac.cn
收稿2014-03-20;修回2014-04-18
CLCQ2-33, TL11
