观光木cpDNA非编码序列PCR反应体系优化及引物筛选
2014-01-02何长青王红霞黄芳芳闫丽君徐刚标
何长青,王红霞,付 甜,黄芳芳,闫丽君,徐刚标
(1.中南林业科技大学 林木遗传育种实验室,湖南 长沙 410004;2.国家林业局 林产工业规划设计院,北京 100010)
观光木cpDNA非编码序列PCR反应体系优化及引物筛选
何长青1,王红霞2,付 甜1,黄芳芳1,闫丽君1,徐刚标1
(1.中南林业科技大学 林木遗传育种实验室,湖南 长沙 410004;2.国家林业局 林产工业规划设计院,北京 100010)
利用单因素与正交设计试验,对影响观光木cpDNA-PCR扩增的主要因子进行优化,建立了观光木cpDNA-PCR 最 适 反 应 体 系(20 μL), 为:50 ng 模 板 DNA、1×PCR buffer、0.2 μmol·L-1引 物、2 mmol·L-1MgCl2、0.3 mmol·L-1dNTP以及1 UTaqDNA 聚合酶;筛选出了适合观光木分子谱系地理学研究的非编码区序列引物,为rpl32-trnL、psbJ-petA、3′rps16-5′trnK、atpI-atpH、petL-psbE。
观光木;cpDNA;非编码序列PCR;体系优化;引物筛选
观光木Tsoongiodendron odorum系木兰科常绿乔木,虫媒传粉[1],为我国珍稀孑遗树种,属国家II级重点保护植物[2],对被子植物系统发育以及系统分类方面具有重要的科学意义。在我国,观光木零散分布于长江流域以南海拔300~1 100 m 的山地常绿阔叶林中[3-4],天然种群很小,为我国极小种群野生植物[5]。
观光木的研究主要集中在观光木的生物学特性、生态学特征、引种繁殖、化学成分和遗传多样性等方面[5-6]。分子谱系地理学是揭示种群遗传多样性、推测种群进化过程中的历史事件、探讨近缘物种亲缘关系的重要手段,已引起国内外学者的广泛重视。叶绿体DNA(Chrloroplast DNA,cpDNA)非编码序列具有较高的核苷酸置换率,能揭示种间和种内遗传变异,多态性丰富,能提供对系统发育研究的信息位点[7-8]。本研究通过对影响观光木cpDNA标记PCR反应体系主要因子进行优化,筛选出适宜的非编码区序列引物,为进一步开展濒危植物观光木分子谱系地理学研究奠定基础。
1 材料与方法
1.1 材 料
材料采集于贵州省从江县。新鲜叶片经硅胶干燥后,带回实验室于-70℃冰箱中保存。cpDNA非编码序列标记通用引物序列参考文献 [9-11],由北京六合华大基因科技股份有限公司合成。
1.2 基因组DNA提取与检测
采用改良的CTAB法提取观光木总DNA[12]。用Eppendorf 公司生产的 Biophotometer 核酸蛋白分析仪检测 DNA 浓度和纯度,用 0.8% 琼脂糖凝胶对提取的总 DNA 进行电泳,用Syngene公司生产的 G-BOX 紫外凝胶成像系统观察 DNA 是否污染。
1.3 PCR扩增产物检测
[9-10]。观光木DNA PCR扩增初始反应体系(20 μL)为:50 ng模板DNA,1×PCR buffer,0.1 μmol·L-1引物,3.0 mmol·L-1MgCl2,0.2 mmol·L-1dNTP,1 UTaqDNA聚合酶。PCR反应程序为:80 ℃预变性5 min;95℃变性1 min,退火1 min,72 ℃延伸1 min,共30个循环;最后72℃延伸7 min,4 ℃保存。
PCR扩增产物采用1.5%琼脂糖凝胶电泳检测。电泳缓冲液为1×TAE,电压5 V/cm。在G-BOX紫外凝胶成像系统拍照记录。
1.4 PCR反应体系优化与引物筛选
以petL-psbE为引物,参考文献[13-14],进行6个浓度水平DNA模板用量试验,以确定最适DNA模板用量。对影响PCR反应扩增产物的特异性和产量的引物、Mg2+、dNTPs、TaqDNA 浓度进行单因素梯度试验(见表1)。根据单因子确定的各因素最适浓度范围,进行4因素4水平正交试验(见表2)。
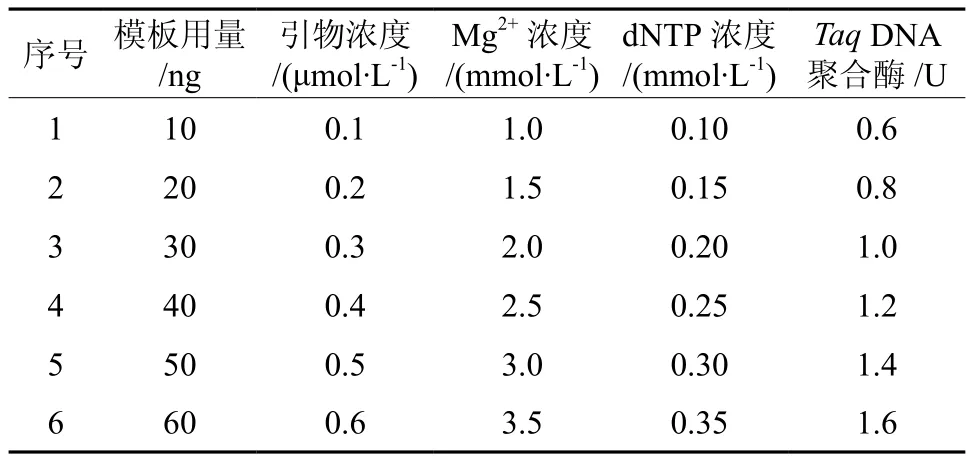
表1 PCR反应体系优化的因素与水平Table 1 Factors and levels of PCR reaction system
利用优化的PCR反应体系,对文献[9-11]中的cpDNA非编码序列通用引物进行筛选。对筛选出的最适cpDNA非编码序列引物,以Tm值计算理论退火温度,以理论值为中心,设置梯度为1℃的6个退火温度,最终确定引物的最佳退火温度。

表2 PCR正交设计Table 2 PCR orthogonal design
1.5 最适反应体系的验证
利用筛选出的最适引物对从江种群17株观光木进行PCR扩增,对优化的PCR反应体系及反应参数的稳定性进行检测。
2 结果与分析
2.1 DNA纯度与浓度
观光木样本提取总DNA,通过核酸蛋白分析仪检测 DNA 浓度和纯度。结果表明,提取的DNA的OD260nm/ OD280nm值在1.6~2.0之间,符合PCR扩增的要求。
2.2 PCR反应体系单因子试验
6个水平浓度的DNA模板扩增结果见图1。由图1可知,不同浓度DNA模板都能扩增出产物,但随着DNA模板用量的增加,PCR扩增条带越来越亮,DNA模板用量为50 ng时,扩增带条最明亮,之后,随浓度增加,扩增条带变暗。由此可见,观光木cpDNA PCR扩增体系的DNA模板最适浓度为50 ng。
引物、Mg2+、dNTP、TaqDNA聚合酶6种不同浓度的PCR扩增结果如图2所示。由图2可知,引物浓度为 0.3 μmol·L-1、Mg2+浓度为 2.5 mmol·L-1、dNTP 浓 度 为 0.30 mmol·L-1、TaqDNA聚合酶用量为1.0 U时,PCR扩增的条带最为清晰。
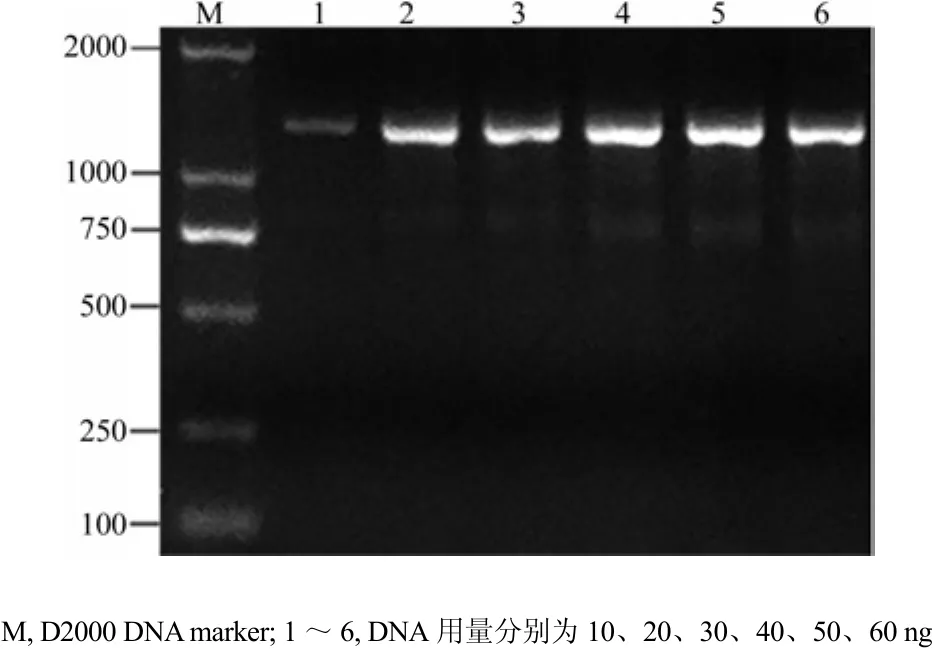
图1 不同DNA用量对PCR的影响Fig.1 Effects of different template DNA concentrations on PCR
2.3 PCR反应体系正交试验
根据单因素试验结果,设定上、下游引物浓度 为 0.1 ~ 0.5 μmol·L-1, Mg2+浓 度 为 0.1 ~ 2.5 mmol·L-1, dNTP 浓 度 为 0.15 ~ 0.30 mmol·L-1,TaqDNA聚合酶用量为0.6~1.4 U。采用4因素4水平的正交试验,对PCR反应体系进一步优化。正交试验结果如图3所示。由图3可知,8、12和15组试验的PCR扩增条带清晰明亮,但第8组合的结果稳定性较差。因此,最终确定观光木cpDNA非编码序列PCR最佳反应体系(20 μL)为:50 ng模板DNA、1×PCR buffer、引物各0.2 μmol·L-1、2.0 mmol·L-1MgCl2、0.3 mmol·L-1dNTP、1 UTaqDNA 聚合酶。

图2 各单因素用量对PCR的影响Fig.2 Effects of different factor concentrations on PCR

图3 PCR正交试验电泳图谱Fig.3 Electrophoretogram for an orthogonal design of PCR amplif i cation
2.4 观光木cpDNA引物筛选
根据正交试验结果,对文献[9-11]中trnK内含 子3914-2R序 列,rpl32-trnL、trnQ-5′rps16、3′trnV-ndhC、ndhF-rpl32、psbD-trnT、psbJ-petA、3′rps16-5′trnK、atpI-atpH、petL-psbE、trnTUGU-trnFGAA、trnCGCA-rpoB、trnDGUC-trnTGGU、trnSGCU-tnGUUC基因间隔序列共14对引物进行筛选。筛选出PCR扩增产物单一、清晰、明亮、稳定的5对引物(见表3)。图4是5对引物PCR圹增结果。

表3 筛选的5对引物Table 3 Selected five pairs of primers
利用筛选出的5对cpDNA非编码序列引物,对观光木从江天然种群17株个体进行PCR 扩增,凝胶电泳结果见图5。由图5可知,5对cpDNA非编码序列引物能扩增出清晰的目的片段,表明筛选出的PCR扩增体系适合于观光木分子谱系地理学研究。

图4 不同退火温度对PCR的影响Fig.4 Effects of different annealing temperature on PCR
3 结 论
PCR扩增结果受反应体系中模板DNA、引物、TaqDNA 聚合酶、Mg2+及dNTP浓度的影响[12-16]。本研究通过单因素与正交设计试验相结合的方法,得到了观光木cpDNA非编码区序列引物最适PCR扩增反应体系。利用优化的PCR反应体系,筛选出适合观光木分子谱系地理学研究的rpl32-trnL、psbJ-petA、3′rps16-5′trnK、atpI-atpH和petL-psbE5对cpDNA非编码区序列引物。其中,rpl32-trnL扩增的目的片段长度为1 200~1 400 bp,psbJ-petA扩增的目的片段长度为1 100~1 300 bp,3′rps16-5′trnK扩增的目的片段长度为750~850 bp,atpI-atpH扩增的目的片段长度为1 100~1 300 bp,petL-psbE扩增的目的片段长度为1 100~1 300 bp。本研究为后续扩增产物的回收、测序以及进一步开展观光木分子谱系地理学研究奠定了基础。

图5 各引物PCR扩增产物电泳图谱Fig.5 Electrophoretogram for PCR amplif i cation products of each primer
参考文献:
通盘考察,财政学专业的课程设置,应以学生综合发展为根本导向,若是以直接进入就业市场为主,则在课程设置上强化实务、实践课程学习,适度安排、设置语言表达、沟通交流课程。若是以继续攻读硕士学位为主,则在课程设置上以深化理论学习为主,强化数据分析和文献资料处理能力的培养。
[1] 曾庆文, 高泽正, 张奠湘, 等. 濒危植物观光木传粉生物学的初步研究[C]// 中国生物多样性保护与研究进展—第五届全国生物多样性保护与持续利用研讨会论文集.2002:199-204.
[2] 傅立国, 金鉴明. 中国植物红皮书— —稀有濒危植物 : 第一册 [M]. 北京 : 科学出版社 ,1992:454-455.
[3] 刘玉壶. 中国植物志[M]. 北京: 科学出版社, 1996.
[4] 黄松殿, 覃 静, 秦武明, 等. 珍稀树种观光木生物学特性及综合利用研究进展[J]. 南方农业学报, 2011, 42(10):1251-1254.
[5] 李松海, 谢安德, 贲丽云, 等. 珍贵树种观光木研究现状及展望[J]. 南方农业学报, 2011, 42(8): 968-971.
[6] 谢宝多, 曹跃进, 唐明武, 等. 观光木青枯病的研究[J]. 中南林学院学报, 1985, 5(1): 5.
[7] Taberlet P, Gielly L, Pautou G,et al. Universal primers for amplif i cation of three non-coding regions of chloroplast DNA[J].Plant molecular biology, 1991, 17(5): 1105-1109.
[9] Shaw J, Lickey E B, Beck J T,et al.The tortoise and the hare II: relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis[J]. American Journal of Botany, 2005,92(1): 142-166.
[10] Shaw J, Lickey E B, Schilling E E,et al. Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: the tortoise and the hare III[J]. American Journal of Botany, 2007, 94(3): 275-288.
[11] Cieslak T, Polepalli J S, White A,et al.Phylogenetic analysis of Pinguicula (Lent-ibulariaceae): chloroplast DNA sequences and morphology support several geographically distinct radiations[J].American journal of botany, 2005, 92(10): 1723-1736.
[12] 吴雪琴, 徐刚标, 梁 艳, 等. 观光木 ISSR-PCR 反应体系的建立及优化[J]. 中南林业科技大学学报, 2012, 32(7): 76-79.
[13] 胡尚力, 徐刚标, 梁 艳, 等. 伯乐树 cpDNA-PCR 反应体系的优化与引物筛选[J]. 中南林业科技大学学报,2013,33(7):67-71.
[14] 张玉梅, 徐刚标, 谷振军, 等. 珙桐 cpDNA 非编码序列引物反应条件优化与筛选[J]. 中南林业科技大学学报, 2011,31(3): 183-186.
[15] 王弦云 ,朱晓敏, 王 勤,等.杜仲 ISSR-PCR 反应体系的建立与引物筛选及其在遗传多样性研究中的应用[J].经济林研究,2013,31(1):30-34.
[16] 梁玉琴,李芳东,傅建敏,等.正交设计优化柿属植物 SSRPCR 反应体系[J].经济林研究,2011,29(4):17-22.
Optimization of PCR reaction system and primers screening of cpDNA non-coding regions forTsoongiodendron odorum
HE Chang-qing1, WANG Hong-xia2, FU Tian1, HUANG Fang-fang1, YAN Li-jun1, XU Gang-biao1
( 1. Lab. of Forest Genetics, Central South University of Forestry and Technology, Changsha 410004, Hunan, China;2. Planning and Design Institute of Forest Products Industry, State Forestry Administration, Beijing 100010, China)
With single factor orthogonal design method, the experiments were conducted to optimize the main elements affecting cpDNA-PCR amplif i cation system forTsoongiodendron odorum. An optimal cpDNA-PCR system forT. odorumwas set up as follows:50 ng DNA template, 1×PCR buffer, 0.3 μmol•L-1primer, 2.5 mmol•L-1MgCl2, 0.3 mmol•L-1dNTP and 1 UTaqDNA polymerase in a total volume of 20 μL. Five pairs of cpDNA non-coding regions primers which are appropriate for the study of molecular genealogy ofT.odorumwere screened out, they are:rpl32-trnL,psbJ-petA,3′rps16-5′trnK,atpI-atpHandpetL-psbE.
Tsoongiodendron odorum; cpDNA; non-coding sequence of PCR; system optimization; primers screening
S759.95;Q948
A
1673-923X(2014)09-0107-05
2014-02-10
国家林业公益性行业科研专项(201104033);十二五国家科技支撑计划项目(2012BAC01B07-2)
何长青(1990-),男,湖南永州人,硕士研究生,主要从事分子种群遗传学研究;E-mail:601867297@qq.com
徐刚标(1965-),男,安徽枞阳人,教授,主要从事植物种群遗传与林木遗传改良研究;E-mail:gangbiaoxu@163.com
[本文编校:谢荣秀]
