酸水解同位素稀释质谱法测量基因组DNA 含量*
2013-12-29张玲陈大舟武利庆高运华汤桦王晶
张玲,陈大舟,武利庆,高运华,汤桦,王晶
(中国计量科学研究院,北京 100013)
刘新海
(中国医学科学院整形外科医院,北京 100144)
含量测量是核酸测量的首要问题,世界工业先进国家计量机构均已针对核酸含量测量开展了基础研究和应用研究。核酸含量测量应用广泛,包括实时定量PCR 技术、基因芯片、电化学传感器和纳米技术等。这些技术领域质控体系的建立,需要依照权威测量和计量方法研制相应的标准物质,通过标准物质的使用保证核酸测量量值的溯源与传递。
目前国际上现有的具有明确溯源途径的高准确性核酸测量方法有酶解DNA 产物的液相色谱-串联同位素稀释质谱方法HPLC-IDMS[1-4]和DNA 中磷元素的电感耦合等离子体发射光谱法ICP-OES[5-7]。
DNA 酶解法可适用于长片段核酸,但依赖于DNA 水解酶的活性,而酶活性受酶解底物浓度的影响[8]。遇到核酸卷曲、核酸与蛋白质结合[9]、碱基修饰[10]等因素造成的特殊空间构象时,核酸水解酶无法进入而无法水解[8]。酶解反应使DNA 测量体系内混入大量的蛋白质、无机盐、两性电解质等物质,纯化过程容易导致DNA 损失。文献报道,DNA 酶解反应容易反应不完全,导致最终测量结果偏低[10]。DNA 中磷元素的电感耦合等离子体光谱法(ICP-OES)适用于短片段、纯度很高的DNA 样品。如果DNA 测量体系中混有其它元素状态的磷,如无机盐中的磷元素,则会造成DNA 测量结果偏高[5]。
加热可使得某些带修饰的碱基从DNA 骨架上解离出来成为游离碱基[11]。在强酸如HCl 条件下,DNA 上的碱基与核糖连接的糖苷键断裂,形成游离碱基[12],但是容易产生副产物。弱酸(如甲酸)在加热条件下可使得DNA 上腺嘌呤和鸟嘌呤及其修饰加合物解离为游离碱基[13-14],该过程也可解离出游离胞嘧啶和甲基胞嘧啶,用于DNA 甲基化水平研究[15]。在此基础上,笔者建立了DNA 水解产物的HPLC-IDMS 定量方法。
核酸的酸水解方法有以下优点:(1)酸解反应耗时较短,反应充分;(2)酸解切断的是碱基与核糖之间的糖苷键[13],不受DNA 长度、序列、DNA 上结合的蛋白质等因素的影响,水解过程进行较充分;(3)酸解反应不引入杂质,只有甲酸中携带的痕量金属离子,易于采用液相色谱串联同位素稀释质谱的分离分析。然而酸解法也有一定的缺点,高浓度强酸可能使DNA 的水解产生其它反应副产物,可能造成碱基进一步降解[16]。因此需要利用选择酸的类型、酸的浓度、酸解时间与温度,准确控制酸水解效率,以实现DNA 含量的准确测量。
噬菌体λDNA 是一种广泛应用的核酸含量标准品,常用于DNA 紫外或荧光定量方法的校准。噬菌体λDNA 的提取纯化过程需要先分离病毒颗粒,因此很少受到细菌本身基因组DNA 和RNA 的污染,可以获得较纯净的噬菌体DNA[17],适合作为标准物质候选物。
1 实验部分
1.1 主要仪器与试剂
水浴锅:DK-8D 型,上海一恒科学仪器有限公司;
pH 计:Origin 3 Star 型,美国Thermo Scientific公司;
旋涡振荡仪:MS3 型,德国IKA 公司;
真空干燥箱:DZF-6050 型,上海精宏实验设备有限公司;
恒温烘箱:UFE500 型,德国Memmert 公司;
离心机:3-15K 型,美国Sigma 公司;
纯水机:MilliQ 型,美国Millipore 公司;
分析天平:CP324S 型,感量为0.01 mg,德国Sartorius 公司;
精密分析天平:UMX2 型,感量为0.1μg,瑞士Mettler Toledo 公司;
电喷雾三重四级杆质谱仪:TSQ Vantage 型,美国Thermo Scientific 公司;
SDS、酚、氯仿、异丙醇、乙醇、琼脂糖、无机酸、无机盐:分析纯或优级纯;
乙腈、甲醇:色谱醇,美国J. T. Baker 公司;
纯度标准物质:自制,分别是腺嘌呤(Ade)[纯度 为(99.40±0.68)%,k=2],胞 嘧 啶(Cyt)[纯 度为(99.58±0.72)%,k=2],鸟嘌呤(Gua)[纯度为(99.96±0.18)%,k=2],胸 腺 嘧 啶(Thy)[纯 度 为(99.67±0.24)%,k=2]。其中腺嘌呤和胞嘧啶分别采用电位滴定法定值,量值溯源至酸碱国家基准,鸟嘌呤和胸腺嘧啶采用液相色谱面积归一化法定值;
同位素标记物:13C,15N 双标记碱基,同位素标记腺嘌呤(L-Ade)化学式为1,3-15N298%,同位素标记胞嘧啶(L-Cyt)化学式为2,4-13C2,15N3(99%C,98%N),同位素标记鸟嘌呤(L-Gua)化学式为8-13C,7,9-15N2(98%C,98%N),同位 素 标 记 胸 腺 嘧啶(L-Thy)化学式为1,3-15N2,98%,均购自剑桥同位素实验室(Cambridge Isotope Labotory)公司。
1.2 仪器工作条件
1.2.1 色谱条件
色 谱 柱:Acqurity UPLC HSS T3色 谱 柱(100 mm×2.1 mm,1.8 μm,美国Waters 公司);流动相:A 为5 mmol/L NH4Ac(0.1% HAc,pH 4.0),B 为乙腈,水相配制后用0.45 μm 滤膜抽滤,并超声脱气;柱温:30 ℃;进样体积:3 μL;流速:120 μL/min;梯 度 洗 脱:0~9 min 99%A,9~9.5 min 99%~80%A,9.5~12.5 min 80%A,12.5~13 min 80%~99%A,13~18 min 99%A。
1.2.2 质谱条件
正电模式;喷雾电压:3.0 kV;雾化气:N2;雾化气温度:200℃;鞘气压力传感器参数:20 arb;辅助气压力传感器参数:5 arb;毛细管温度:350℃;碰撞气:Ar;一级质谱扫描宽度(m/z):0.002;扫描时间:0.1 s;一级质谱半峰宽(FWHW):0.7;二级质谱半峰宽:0.7;微扫描时间:1 ms。
质谱多反应监测模式(MRM)的样品离子监测条件(包括同位素碱基标记物)见表1。
按质谱仪说明书配制含有咖啡因、MRFA(多聚酪氨酸短肽)和MLtramark l621 的质量轴校准液,完成质谱仪的校准。
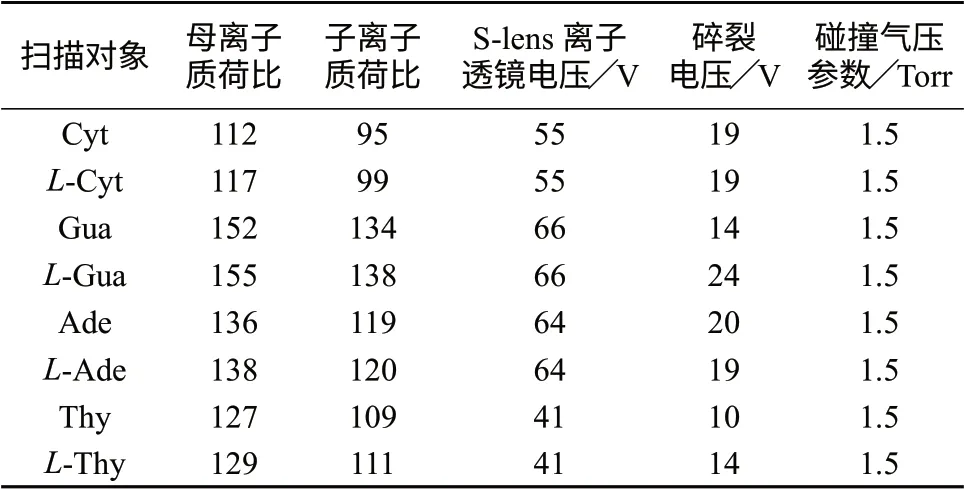
表1 质谱多反应监测模式(MRM)的样品离子监测条件
1.3 标准溶液和样品溶液的配制
称取约1 mg 碱基和同位素标记的碱基,分别溶于10 mL 去离子水中,配制成标准储备液。Gua 和L-Gua 不易溶于水,溶于体积分数3%的盐酸溶液中,然后配制成混合标准品和混合同位素标记内标品。根据λDNA 用HPLC 外标法测得的浓度,在溶液中添加约等量的混合同位素标记内标品,同时配制与样品浓度近似的碱基与同位素标记碱基质量比值约为0.90 和1.10 的高标和低标溶液。
1.4 DNA 水解
将基因组DNA 含量标准物质候选物冻干粉溶于100 μL 水,在λDNA 溶液中添加与其组成成分核苷酸含量相近的标记核苷酸混合物。量取100 μL 样品溶液于安瓿瓶中,真空干燥箱抽干。加入200 μL 甲酸水溶液(体积分数为88%),充氮气,封口。在170℃恒温烘箱内反应30 min,冷却后用真空干燥箱抽干,量取加入100 μL 0.1 mol/L 盐酸溶解,充分溶解混匀,溶液于4℃下保存。
1.5 IDMS 法碱基含量的计算
针对每一种碱基:
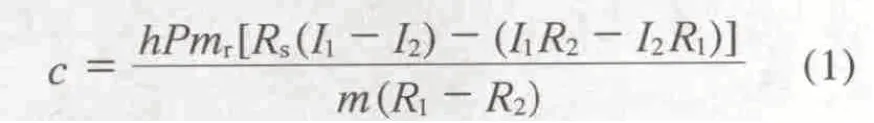
式中:c——碱基质量分数;
h——水解效率,%;
P——碱基标准物质的纯度,%;
mr——样品中碱基同位素标记物的质量,g;
Rs——样品中碱基与碱基标记物的峰面积比;
I1——高标溶液碱基与碱基标记物的质量比;
I2——低标溶液碱基与碱基标记物的质量比;
R1——高标溶液碱基与碱基标记物的峰面积比;
R2——低标溶液碱基与碱基标记物的峰面积比;
m——样品质量,g。
1.6 λDNA 含量的计算
由于λDNA 是一个具有49.5 kb 长度的双链闭环DNA。Cyt,Gua,Thy,Ade 4 种碱基质量分数分别为8.96%,12.19%,10.23%,10.97%,根据4 种碱基含量计算得λDNA 总质量,取平均值,得到同位素稀释碱基法测量DNA 质量的值。
2 结果与讨论
2.1 DNA 水解条件的优化
分别用5%盐酸溶液于110℃水解24~48 h,用88%甲酸水溶液于140℃水解2~16 h,用88%甲酸水溶液于170℃水解15 min~4 h[13-14]。试验结果表明,盐酸水解产物杂质较多;甲酸140℃水解胸腺嘧啶核苷一磷酸的水解不完全;甲酸170℃水解效果较好,水解反应完全。用88%甲酸水溶液于170℃水解30 min 回收率满足要求,水解产物中4种碱基保持稳定,因此水解条件优化为88%甲酸水溶液于170℃水解30 min。
2.2 水解效率
根据核苷酸和基因组DNA 水解实验的回收率计算酸水解回收率。微生物基因组DNA 提取样品中定量加入已知浓度的5 种碱基混合物质控品,以1.4 步骤水解样品并进行测定,重复测量7 个样品,计算回收率,结果表明4 种待测物碱基的回收率都在95%以上,水解效率不确定度不大于0.8%。经过t检验,4 种dNMP 溶液与λgDNA 溶液添加dNMP 溶液后水解测量的回收率无显著性差异。另外,经过Picogreen 荧光染料实验,水解产物没有出现荧光反应,说明水解产物中已不含有双链DNA,即说明水解反应完全。
2.3 色谱与质谱参数的优化
测试不同NH4Ac 溶液浓度和不同pH 值条件下4 种碱基的分离效果,结果表明,pH 值越大,碱基保留时间越长;有机相比例越高,碱基保留时间越短。考虑出峰时间、色谱柱的耐受性和流动相对质谱的影响,确定了1.2.1 色谱条件。
配制0.01 mg/g 同位素标记的碱基和非标记碱基的混合物,进行流动注射,选定合适的喷雾电压和毛细管温度,并且优化S-lens 和碎裂CID 电压,考虑质谱的扫描速度、分析时间,获得尽可能大的信号强度,确定了1.2.2 质谱条件。
利用样品与同位素标记物混合后水解的产物进行HPLC-IDMS 实验,样品总离子流图和子离子流图分别见图1~图5。

图1 总离子流图
图2 Cyt 和L-Cyt 子离子流图
图3 Gua 和L-Gua 子离子流图
图4 Ade 和L-Ade 子离子流图
图5 Thy 和L-Thy 子离子流图
2.4 样品中碱基含量的同位素稀释质谱法测定
取15 支λDNA 冻干粉样品,称量后溶于100 μL 去离子水中,然后量取100 μL 样品溶液,加入100 μL 同位素标记混合样,充分混匀。参照1.4 步骤进行实验,结果列于表2,由表2 可知,4 种碱基测量结果的相对标准偏差都在5%以内。
按照1.6λDNA 计算方法,依据不同核苷酸在整个λDNA 序列中的比例计算λDNA 含量,将4种核苷酸碱基推算所得DNA 含量取平均值,并与数字PCR(dPCR)测量结果相比较[18],结果见图6。λDNA的测量结果为每支(2.51 ±0.06)μg(k=2)。由图6可见,两种不同原理的测量方法所得结果近似,测量值在不确定度范围内。

表2 λDNA 样品中4 种碱基含量的同位素稀释质谱测量结果
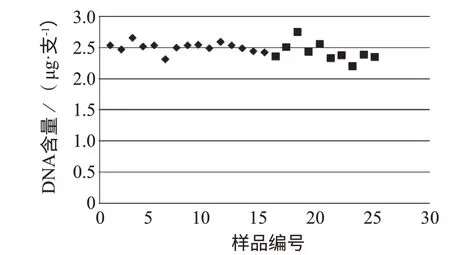
图6 λDNA 冻干粉样品用IDMS 法与数字PCR 法测量结果比较
3 结语
基因组DNA 水解-同位素稀释质谱法测量长片段核酸的含量,可以将DNA 质量溯源到碱基质量,利用电位滴定将碱基质量溯源到酸碱国家基准,继而溯源至国际标准单位SI,从而建立了核酸含量溯源的方式。在优化酸解方法的基础上,采用加标回收法测量酸解效率,通过同位素稀释质谱法测量长片段DNA 含量,线性范围约为1~1 000 μg/g,检出限可低至100 ng/g。共测量15 个样品,测得λDNA 的含量为每管2.51 μg,相对扩展不确定度为2.34%(k=2)。该方法可用于长片段核酸含量标准物质定值,改进后的方法适用性广、灵敏度高、准确度好。但是由于DNA 水解方法的缺点在于可能产生水解副产物,因此对烘箱的温度均匀性和稳定性均有较高的要求,并且烘箱水解时间也需要小心调整。
[1] O'Connor G, Dawson C, Woolford A, et al. Quantitation of oligonucleotides by phosphodiesterase digestion followed by isotope dilution mass spectrometry: proof of concept[J]. Anal Chem,2002,74(15): 3 670-3 676.
[2] Donald C E, Stoked P, O'Connor G, et al. A comparison of enzymatic digestion for the quantitation of an oligonucleotide by liquid chromatography-isotope dilution mass spectrometry[J].J Chromatogr B Analyt Technol Biomed Life Sci,2005,817(2):173-182.
[3] Kinumi T, Narukawa T, Fujii S I, et al. Quantification of oligonucleotide containing sequence failure product:comparison of isotope dilution mass spectrometry with other quantification methods[J]. Eur J Mass Spectrom (Chichester,Eng),2009,15(3): 399-407.
[4] Dong L, Zang C, Wang J, et al. Lambda genomic DNA quantification using ultrasonic treatment followed by liquid chromatography-isotope dilution mass spectrometry[J]. Anal Bioanal Chem,2012,402(6): 2 079-2 088.
[5] Dash K, Rastpqi L, Arunachalam J. DNA quantification via traceable phosphorus measurement through microwave-assisted UV digestion-ion chromatography[J]. Analyst, 2012,137(3):668-674.
[6] Holden M J, Rabb S A, Tewari Y B, et al. Traceable phosphorus measurements by ICP-OES and HPLC for the quantitation of DNA[J]. Anal Chem,2007,79(4): 1 536-1 541.
[7] Yang I, Han M S, Yim Y H, et al. A strategy for establishing accurate quantitation standards of oligonucleotides:quantitation of phosphorus of DNA phosphodiester bonds using inductively coupled plasma-optical emission spectroscopy[J]. Anal Biochem,2004,335(1): 150-161.
[8] Singer B, Grunberger D. Molecular Biology of Mutagens and Carcinogens[M]. New York: Plenum Press, 1983.
[9] Kuraoka I, Robins P, Masutani C, et al. Oxygen free radical damage to DNA.Translesion synthesis by human DNA polymerase eta and resistance to exonuclease action at cyclopurine deoxynucleoside residues[J]. J Biol Chem,2001,276(52): 49 283-49 288.
[10] Park S, Seetharaman M, Ogdie A, et al. 3'-Exonuclease resistance of DNA oligodeoxynucleotides containing O6-[4-oxo-4-(3-pyridyl)butyl]guanine[J]. Nucleic Acids Res,2003,31(7):1 984-1 994.
[11] Goggin M, Loeber R, Park S, et al. HPLC-ESI+-MS/MS analysis of N7-guanine-N7-guanine DNA cross-links in tissues of mice exposed to 1,3-butadiene[J]. Chem Res Toxicol,2007,20(5): 839-847.
[12] Nissim I, Starr S E, Sullivan K E, et al. Rapid method for determining the rate of DNA synthesis and cellular proliferation[J]. Anal Biochem,2000,278(2): 198-205.
[13] Goggin M, Anderson C, Park S, et al. Quantitative highperformance liquid chromatography-electrospray ionizationtandem mass spectrometry analysis of the adenine-guanine crosslinks of 1,2,3,4-diepoxybutane in tissues of butadiene-exposed B6C3F1 mice[J]. Chem Res Toxicol,2008,21(5): 1 163-1 170.
[14] Swarts S G, Smith G S, Miao L, et al. Effects of formic acid hydrolysis on the quantitative analysis of radiation-induced DNA base damage products assayed by gas chromatography/mass spectrometry[J]. Radiat Environ Biophys,1996,35(1): 41-53.
[15] Kok R M, Smith D E, Barto R, et al. Global DNA methylation measured by liquid chromatography-tandem mass spectrometry:analytical technique,reference values and determinants in healthy subjects[J]. Clin Chem Lab Med,2007,45(7): 903-911.
[16] Yang L H, Liu Q, Sun C J, et al. The formation of BPDE-DNA adduct in rat and its determination by high performance liquid chromatography[J]. Sichuan Da Xue Xue Bao:Yi Xue Ban,2008,39(2): 302-304.
[17] Joseph S, David W R. Molecular Cloing: A Laboratory Manual[M]. 3rd, New York: Cold Spring Harbor Laboratory Press, 2001.
[18] 柳方方,张玲,王晶,等.数字PCR 准确测定DNA 含量的方法及测量结果不确定度评定[J].化学分析计量,2013,22(1):18-22.
