水稻倒一节间生长发育调控基因OEUI的精细定位
2013-12-04方雪
方 雪
(合肥师范学院 生命科学系,安徽 合肥230601)
水稻是世界主要粮食作物之一,占世界粮食作物产量的40%[1]。近年来,水稻基因组研究进展迅速,随着水稻全基因组序列的测序的的完成,水稻分子生物学研究进入功能基因组时代[2-3]。水稻节间生长发育有着特定的时空模式,有多个基因参与调控水稻节间发育[4,5]。
Takeda曾将水稻矮杆突变体依据其对节间排布模式的影响而分为五类:dn、dm、d6、sh、nl。其中dn型水稻突变体中每个节间均匀缩短,其伸长形式跟野生型相似;dm型突变表现为第二节间特异缩短;sh和d6型突变体则是倒一节或是倒二节缩短[6]。随着近年来水稻功能基因组研究的迅猛发展,许多与这五类突变相关的基因已被鉴别和克隆。已克隆出的基因OSH15、OSBRI1的突变可以造成d6表型[7];本研究中克隆的OEUI的突变可以造成sh表型。多个EUI基因则特异调控倒一节的伸长模式。对这些基因的克隆以及相关的生物学研究将极大地促进人们理解水稻节间模式形成的分子机理[8,9]。
经典的植物学研究早已表明植物激素赤霉素对于节间的伸长是必须的,EUI基因还与赤霉素的合成代谢以及信号转导有关,许多与赤霉素合成、代谢、信号转导有关的突变体都表现出节间伸长的缺陷[10]。另外,油菜素内酯和生长素都对节间的伸长有着重要的作用[11]。因此,可以利用节间伸长探讨赤霉素、油菜素内酯和生长素信号转导途径之间的关系。水稻生产中,水稻雄性不育系中普遍存在的“包颈”现象(即倒一节间伸长受阻),印证了在顶端发育的穗与倒一节间的居间分生组织之间存在长距离信号的转导[12]。在已克隆的基因中,OSH15编码了一个同源异形盒(homeo-domain)蛋白;OSBRI1编码的蛋白是油菜素内酯的受体,OSH15、OSBRI1的突变可以造成相似的d6表型,提示激素BR信号转导与同源异形盒基因之间的互作关系[13,14]。
图位克隆是近年来随着各种植物的分子标记图谱的相继建立而发展起来的一种新的基因克隆技术。根据功能基因在基因组中都有相对较稳定的基因座,在利用分子标记技术对目的基因进行精细定位的基础上,用与目的基因紧密连锁的分子标记筛选DNA文库(包括 YAC、BAC、TAC、PAC或cosmid文库),从而构建目的基因区域的物理图谱,再利用此物理图谱通过染色体步移逐步逼近目的基因或通过染色体登陆的方法最终找到包含该目的基因的克隆,并通过遗传转化实验证实目的基因的功能[15]。2003年朱宏波用图位克隆的策略将EUI2基因定位于一个41.7kb的区间内,并成功克隆到该基因。仅知道EUI2同样与赤霉素有关,但对EUI2的功能研究尚未见报道[16]。
目前几乎所有重要农作物,如水稻、拟南芥、番茄、玉米、甜菜、小麦、大豆、马铃薯、棉花、油菜等的RFLP图谱均已构成,随着多种标记的不断添加与定位,分子遗传图谱正日渐趋于饱和。2001年Cornell大学发表的SSR遗传连锁图上SSR标记达到500多个,而2002年IRMI图谱上则整合了2240个新开发的SSR标记。水稻全基因组序列的完成,为水稻分子遗传学研究提供无穷的分子标记和序列信息,必将对水稻基因图位克隆技术的发展产生了巨大的推动作用。
本研究中的OEUI基因的表型为水稻倒一节伸长受阻,该表型由γ射线诱变水稻种子得到,为单基因隐性突变。为深入研究该基因的功能,采用图位克隆法,利用SSR分子标记技术,依靠PCR扩增进行全基因组扫描,确定OEUI基因在染色体中的位置,寻找与OEUI基因连锁的遗传标记,最终对OEUI基因进行精细定位,从而研究其功能。
1 材料与方法
1.1 定位群体的构建和总DNA的提取
通过对本实验室所构建的水稻突变体库进行大规模筛选,获得一稳定遗传的单基因隐性突变体oeui。oeui为辐射处理野生型中花11(ZH11)的种子产生诱变获得。以oeui突变体和龙特甫(LTF)为亲本构建F2群体,用于OEUI基因的初步定位。从F2代中选取30株显性正常株和54株突变株构成定位群体,分单株取叶片提取总DNA。在此基础上随机选取正常株和突变株各10株,取等量叶片,混合研磨后提取DNA,建成混合池。参照 Mc-Couch等的方法提取总DNA[17]。
1.2 筛选与目标基因连锁的SSR标记
利用日本水稻基因组计划(RGP)公布的1号染色体的有关信息,设计并合成SSR引物。对突变体oeui和正常株的基因组DNA分别进行扩增,PCR按常规方法进行。PCR产物用1%的琼脂糖凝胶电泳检测。如果PCR产物在亲本间具有多态性,直接用作分子标记;如果没有多态性,则将扩增产物用不同的限制性核酸内切酶进行酶切,并用1.5%的琼脂糖凝胶电泳检测,表现多态者则用作SSR分子标记。
1.3 基因初定位
用开发出的SSR标记对龙特甫/oeui突变体F2群体中的突变体进行分析,将OEUI基因所控制的性状按双亲表型分为2群,每一群中各个体DNA等量混合,形成2个DNA混合池(倒一节间伸长和倒一节间伸长受阻)。用OEUI基因附近的SSR标记对混合DNA样品池进行分析,根据基因池中包含有交换的DNA池的比例确定与OEUI基因连锁最紧密的分子标记和OEUI基因附近所有分子标记的顺序。SSR标记的分析结果利用Mapmaker 3.0作图软件进行连锁分析及遗传距离计算。
1.4 基因精细定位
以oeui突变体和广陆矮4号(GLA4)为亲本构建F2群体,用于OEUI基因的精细定位。把目标基因初步定位在2个标记之间后,从国际水稻基因组测序计划(IRGSP)公布的序列中下载这2个标记区域的部分SSR标记。对扩增良好并表现出多态性的引物分别在定位群体中的oeui突变株上作分析,将目标基因范围缩小到一个很小的基因组区域并应用于精细定位分析。
2 结果与分析
2.1 OEUI基因初步定位群体的构建:
OEUI基因初步定位群体来源于龙特甫(LTF)/oeui突变体的F2中的突变表型株,共64株。突变体植株矮化,倒一节间生长受阻,突变体不结实。形态特征如图1。
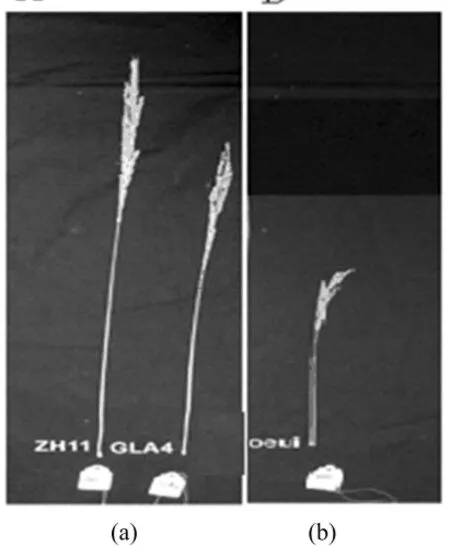
图1 突变体与其野生型亲本的植株形态
2.2 OEUI基因连锁引物的筛选
随机挑选10株突变体,构建DNA混合池,以中花11、龙特甫、F1、DNA混合样为模板,全基因组扫描,发现水稻第一染色体标记RM3252可能与基因OEUI连锁,用RM3252扩增64株龙特甫/oeui突变体的F2中的突变表型株,结果发现有三株表现F1带型,即为交换单株(图2),其余均与中花带型一致,因此认为SSR标记RM3252与OEUI基因连锁。
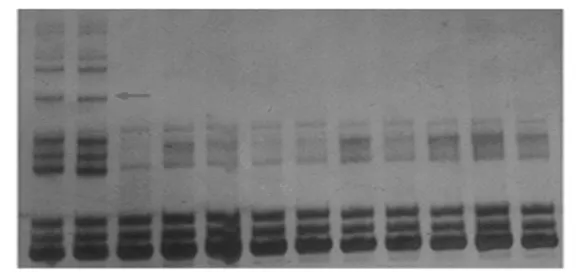
图2 标记RM3252对部分LTF/oeui的F2突变株DNA的PCR扩增产物的凝胶电泳图(含箭头所指带型的单株为交换株)
2.3 OEUI基因的初步定位
用64株LTF/oeui突变体将OEUI基因初步定位在RM4554与RM5552之间,它们之间的遗传距离约14.8cm。
2.4 OEUI基因的精细定位
OEUI基因的精细定位群体来源于广陆矮4号(GLA4)/oeui突变体的F2中的突变表型株,共504株。
在初定位的基础上进一步在RM4554与RM5552之间发展SSR分子标记,在亲本表现多态的有效引物有八对,分别为:RM3148,RSL0126/0127,RSL0114/0115,RSL0108/0109,RSL0160/0161,RSL0168/0169,RSL0172/0173与 RSL0152/0153。其中RSL0126/0127是在紧接RM3148后的BAC AP002867 中设计的,RSL0114/0115,RSL0108/0109是在紧接 AP002867后的 BAC AP002747中设计的,RSL0160/0161,RSL0168/0169,RSL0172/0173 是在 RM3148 与 RSL0152/0153中部的BAC AP002868中设计的(图3)。

图3 八对有效引物在染色体上的位置
通过扩增504株F2表型突变株发现:RM3148扩增504株F2突变株,交换单株编号为87,124,164,208,257,297,316,376,共八株(图4)。
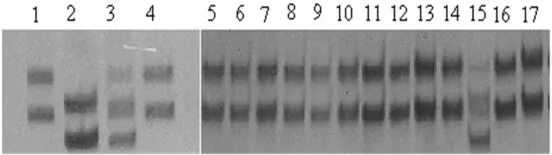
图4 RM3148对部分GLA4/oeui的F2突变株DNA的PCR扩增产物的凝胶电泳图
RSL0152/0153扩增504株F2突变株,交换单株编号为12,15,46,66,68,88,110,124,125,128,135,137,161,166,176,185,234,236,251,257,273,283,285,289,291,295,300,317,332,341,347,348,354,358,366,369,370,375,379,390,396,399,433,435,444,450,468,470,共48株。用前八对可用引物扩增RM3148的八株交换单株,结果如表1。
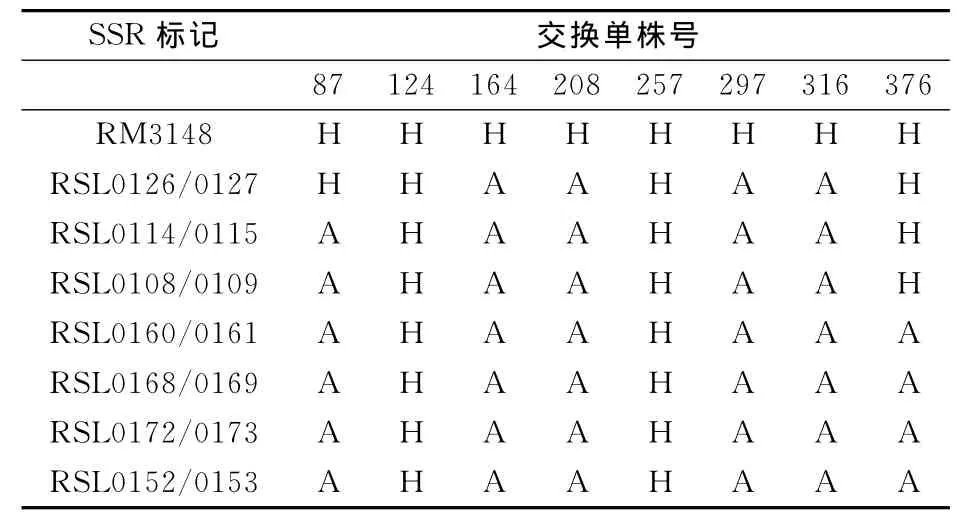
表1 八对可用引物扩增RM3148的八株交换单株
分析F2群体的504株倒一节间伸长受阻表型个体,检测出重组个体并验证区间走势,最终把OEUI基因定位在SSR标记RM3148和RSL0152/0153之间,它们之间的物理距离约为876kb,遗传距离为3.9cm。
3 讨论
本研究首先利用64株LTF/oeui突变体,采用集团分离分析法筛选到与OEUI基因连锁的标记RM3252。随后运用在亲本间具有多态性的SSR标记筛选F2定位群体,将目标基因定位在1号染色体上的标记RM3148和RSL0152/0153之间。根据已经公布的水稻全基因组序列,查找到标记RM3148与SL0152/0153之间的基因组序列,利用Primer5软件对查找到的系列进行分析,寻找可以利用的微卫星,设计合成新的SSR标记。最后利用新发展的6对可用标记扩增RSL0152/0153的48株交换单株,继续缩小两个侧翼标记间的距离,这进一步克隆该基因奠定了基础。当然,为了证实该候选基因就是OEUI,还需要进行遗传互补实验。进一步进行OEUI基因的功能分析,研究赤霉素的合成代谢及信号转导以及节间模式形成的分子机理。
一般而言,精细定位在图位克隆中时一个限速步骤。随着目标染色体区域的逐渐缩小,寻找分子标记的难度越来越大。因此,采取合适的方法来发掘多态标记是非常重要的。在本研究中,我们尝试了多种分子标记技术,包括CAPS、SNP和SSR。相对而言,SSR比较适用,但数量较少,开发费用较高。根据我们的经验,最有效的还是SSR。利用这种方法开发SSR标记效率较高,费用较低,适合于基因精细定位的需要。
[1]Khush G S.Origin,dispersal,cultivation and variation of rice[J].Plant Mol Biol.1997,35:15-34.
[2]Goff,S.,Ricke,D.,Lan,T-H.et al.A draft sequence of the rice genome(Oryza sativa L.spp.Japonica)[J].Science.2002,296:92-100.
[3]Yu,J.,Hu,S.,Wang,J.et al.A draft sequence of the rice genome(Oryza sativa L.ssp.Indica)[J].Science.2002,296:79-92.
[4]Akagi,H.,Yokozeki,Y.,Inagaki,A.et al.Microsatellite DNA markers for rice chromosomes,Theor[J].Appl.Genet.1996,94:61-67.
[5]Chen,X.,Temnykh,S.,Xu,Y.et al.Development of a microsatellite framework map providing genome-wide coverage in rice(Oryza sativa L.)[J].Theor.Appl.Genet.1997,95:553-567.
[6]Takeda K.Internode elongation and dwarfism in some gramineous plants[J].Gamma Field Symp,1977,16:1-18.
[7]McCouth,S.R.,Kochert,G.,Yu,Z.H.et al.,Molecular mapping of rice chromosomes[J].Theor Applied Genetics.1988,76:815-829.
[8]Yamamuro,C.,Ihara,Y.,Wu,X.,Noguchi,T.,Fujioka,S.,Takatsuto,S.,Ashikari,M.,Kitano,H.,and Matsuoka,M.(2000).Loss of functionof a rice brassinosteroid insensitive1homolog prevents internode elongation and bending of the lamina joint[J].Plant Cell.2000,12:1591-1605.
[9]Bleecker AB,Schuette JL,Kende H.Anatomical analysis of growth and developmental patterns in the internode of deep water rice[J].Planta.1986,169:490-497.
[10]Fu,X.et al.Gibberellin-mediated proteasome-dependent degradation of the barley DELLA proteinSLN1repressor[J].Plant Cell.2002,14:3191-3200.
[11]Ashikari,M.,Wu,J.,Yano,M.,Sasaki,T.,and Yoshimura,A.Rice gibberellin-insensitive dwarf mutant gene Dwarf1encodes the--subunit of GTP-binding protein[J].Proc.Natl.Acad.Sci.1996,96.
[12]Itoh,H.,Ueguchi-Tanaka,M.,Sentoku,N.,Kitano,H.,and Matsuoka,M.Cloning and functional analysis of two gibberellin 3--hydroxylasegenes that are differently expressed during the growth of rice[J].Proc.Natl.Acad.Sci.2001,98:8909-8914.
[13]Hedden P,Phillips AL.Gibberellin metabolism:new insights revealed by the genes[J].Trends Plant Sci.2000,5:523-530.
[14]Ueguchi-Tanaka M,Fujisawa Y,Kobayashi M,Ashikari M,Iwasaki Y,Kitano H,Matsuoka M:Rice dwarf mutant d1,which isdefective in the a subunit of the heterotrimeric G protein,affects gibberellin signal transduction[J].Proc Natl Acad Sci.2000,97:11638-11643.
[15]Georg Jander,Susan R.Norris et al.Arabidopsis Map-Based Cloning in the Post-Genome Era[J].Plant Physiol.2002,129:440-448.
[16]钱前,郭龙彪,杨长登.水稻基因设计育种[M].科学出版社,2007:12.
[17]McCouch SR,Kochert G,Yu ZH.Molecular mapping of rice chromosome[J].Theor Appl Genet,1998,76:815-829.
