长江下游鳙放流群体和天然捕捞群体遗传多样性的微卫星分析
2013-11-05张敏莹徐东坡段金荣周彦锋方第安施炜纲
张敏莹,刘 凯,徐东坡,段金荣,周彦锋,方第安,施炜纲
(农业部长江下游渔业资源环境科学观测实验站/内陆渔业生态环境和资源重点开放实验室/淡水渔业研究中心 中国水产科学研究院,江苏 无锡214081)
鳙(Aristichthys nobilis)作为“四大家鱼”之一,在我国淡水渔业中占有极其重要的地位。已有研究表明,长江四大家鱼的养殖性能明显优于其它水系[1]。近年来,由于水工建设、环境污染、酷鱼滥捕等诸多原因,长江渔业资源受到严重冲击,四大家鱼资源量急剧下降[2]。长江水系野生鳙群体的产量急剧下降,其生长表现、抗病抗逆性和遗传多样性等也有明显的降低[3]。
早期对长江水系鳙群体遗传多样性的研究主要集中在同工酶[4-7]、RAPD[8-10]、线粒体DNA(mtDNA)分析[11-13]等方面。近年来,微卫星(Microsatellite)分子标记由于具有突变快、多态信息含量丰富、共显性遗传等特点,在DNA 指纹分析、功能基因定位、种质资源鉴定和遗传多样性评估等方面得到广泛应用。近年已有少量关于鳙微卫星引物的筛选及分子标记的研究报道。鲁翠云等通过构建鳙基因组文库,利用微卫星探针筛选出82 个鳙微卫星序列[14];耿波等利用17 个微卫星分子标记对四川泸州和江西鄱阳湖2 个种群鳙鱼的遗传多样性进行了研究[15]。田华采用22 对鲢微卫星位点对鳙2 个群体的遗传多样性进行了研究[16]。
近年来长江中下游对以鲢鳙为主的重要经济鱼类实施了大规模的人工增殖放流。各江段(湖泊)鳙放流群体的种质如何,放流群体和长江天然捕捞群体(在江群体)遗传多样性是否有差异?为了寻求答案,2011 年我室(淡水渔业研究中心资源室,下同)在参加下游鱼类标志放流活动时现场采集了不同放流群体的样本。本文利用筛选的18 对微卫星引物,对鳙3 个放流群体(蠡湖、巢湖、长江无为段)和1个天然捕捞群体(长江常熟段)的种质资源特征进行了分析和研究,以期为长江水系鳙的增殖放流及种质资源保护提供一定的理论依据。
1 材料与方法
1.1 样品来源及基因组DNA 提取
本研究对鳙4 个群体的遗传多样性进行了研究。3 个群体为放流群体,分别于2011 年2—4 月采自蠡湖、巢湖和长江无为江段鳙增殖放流活动现场,均为1 龄鱼种。1 个群体为2011 年6 月长江常熟段天然捕捞群体。每个群体不分雌雄随机各取30 尾,剪取鳍条放人分析纯乙醇固定,带回实验室备用。
基因组DNA 采用常规苯酚/氯仿法抽提,灭菌双蒸水溶解,0.8%的琼脂糖凝胶(EB 染色)电泳检测DNA 的质量和浓度。
1.2 SSR 引物序列
引物设计参照文献[14],上海申能博彩生物工程公司合成。共合成25 对引物,最后筛选18 对鳙微卫星引物,引物编号及序列见表1。
1.3 PCR 反应、电泳及数据统计
PCR 反应程序参照耿波等[15],产物用9.0%非变性聚丙烯酰胺凝胶电泳分离,银染,拍照记录。
将电泳谱带中的每一条DNA 片段作为该座位的一个等位基因来处理,分析各位点的等位基因,依据等位基因片段长度从小到大标为A、B、C 等。微卫星位点的多态位点比例、观测等位基因数(A)、有效等位基因数(Ne)、基因观测杂合度(Ho)、基因期望杂合度(He)、遗传相似度(F)、遗传距离(D)、基因分化系数(Fst)、基因流(Nm)等参数使用Popgene 1.32 软件进行统计分析。其中,多态位点比例=多态位点数/位点总数×100%;遗传杂合度h=1为位点i 在某一种群中出现的频率;基因分化系数:Fst=(∑Ji/s–Ji)/(1 –Ji),s 为种群数目,Ji 是第i 个种群内的基因的一致性;Nei’s 遗传相似度:F=2Nxy/(Nx+Ny),D=1 – F,Nx 和Ny 分别为种x 和y 的位点总数,Nxy 为2 种群间共享位点数,D为遗传距离。多态信息含量按BOTSTEIN[17]等方法计算:
其中Pi和Pj分别为第i 和第j 个等位基因在群体中的频率,m 为等位基因数。
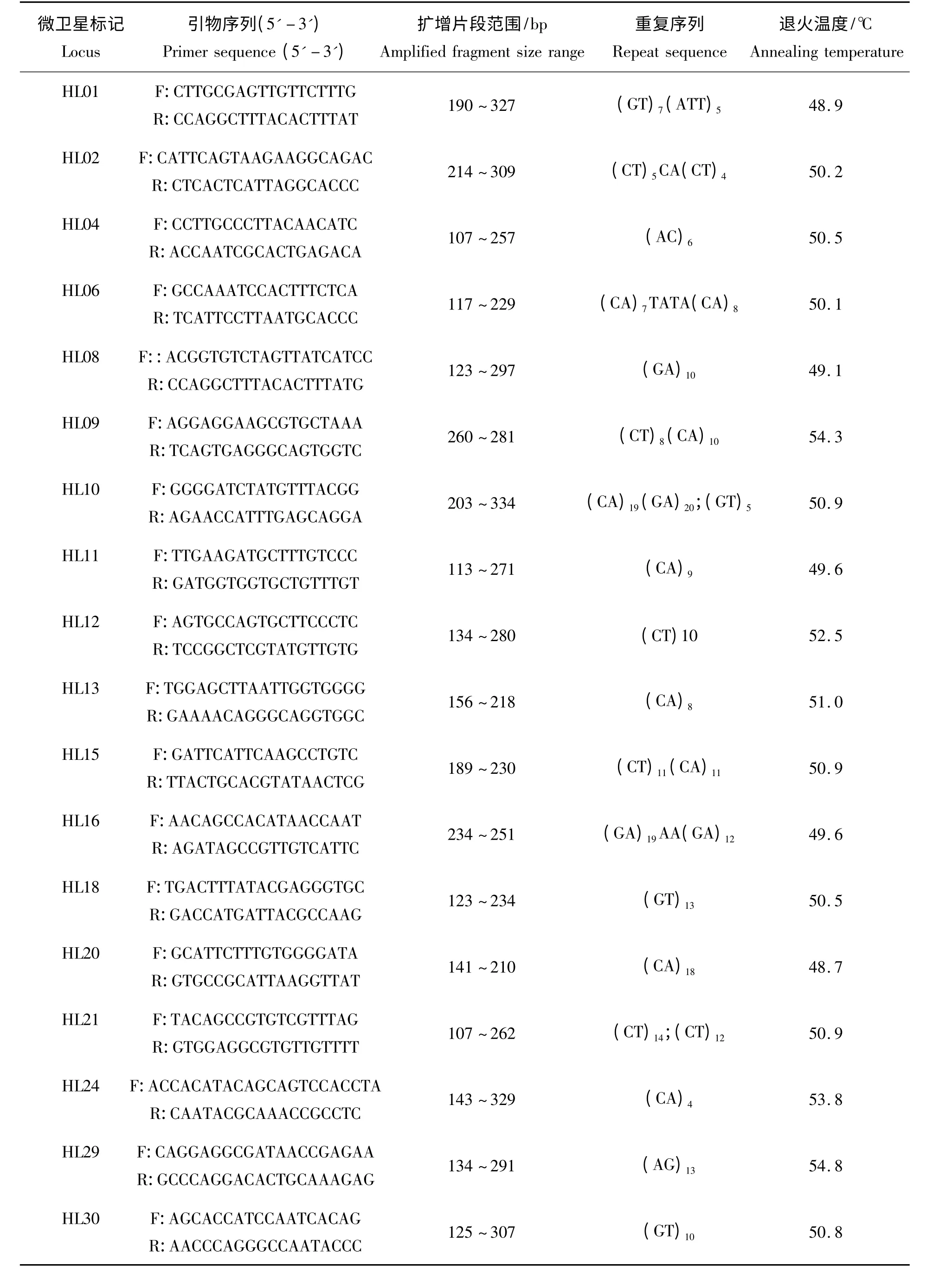
表1 本研究所用微卫星座位及其引物Tab.1 SSR loci and primers used in the paper
2 结果与分析
2.1 PCR 扩增结果
从合成的25 对引物中最后筛选出18 对微卫星引物,18 对引物在4 个鳙群体120 个个体中经特异扩增、非变性聚丙烯酰胺凝胶电泳后均可以表现出清晰的条带及一定的多态性(图1)。
2.2 微卫星标记的遗传多样性
18 个微卫星位点在4 个群体中扩增出的等位基因数(A)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)见表2。总共扩增出109 个等位基因,平均等位基因数为6.056,平均有效等位基因数为4.110。每个位点的观测杂合度为0.092 ~0.917,平均值为0.722,期望杂合度为0.512 ~0.888,平均值为0.708,多态信息含量为0.378 ~0.831,平均值为0.598。12 个位点表现为高度多态(PIC >0.5),6 个位点表现为中度多态(0.25 <PIC <0.5),由此说明本实验所选用的18 个微卫星位点具有丰富的多态性,可以用来进行鳙群体遗传多样性分析。
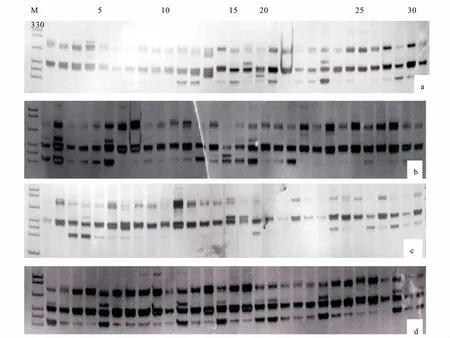
图1 微卫星基因座HL11 在4 个鳙群体的扩增结果图Fig.1 PCR result of HL11 locus in four bighead carp populations
4 个鳙群体遗传多样性参数统计结果见表3。18 个微卫星位点上共检测到109 个等位基因。其中无为放流群体、常熟捕捞群体、巢湖放流群体、蠡湖放流群体分别检测到96、102、94、91 个等位基因。每个座位检测到的等位基因个数3 ~10 个不等,18 对引物在4 个群体中均表现为多态,多态位点百分率均为100%。常熟捕捞群体的观测杂合度(0.753)、期望杂合度(0.721)和多态信息含量(0.633)最高。
2.3 群体间的Fst 值、遗传相似系数与遗传距离
群体间的Fst 值(表4)表明4 个群体间遗传分化程度较弱,蠡湖放流群体与巢湖放流群体间遗传分化最弱,蠡湖放流群体与常熟捕捞群体间遗传分化最强。
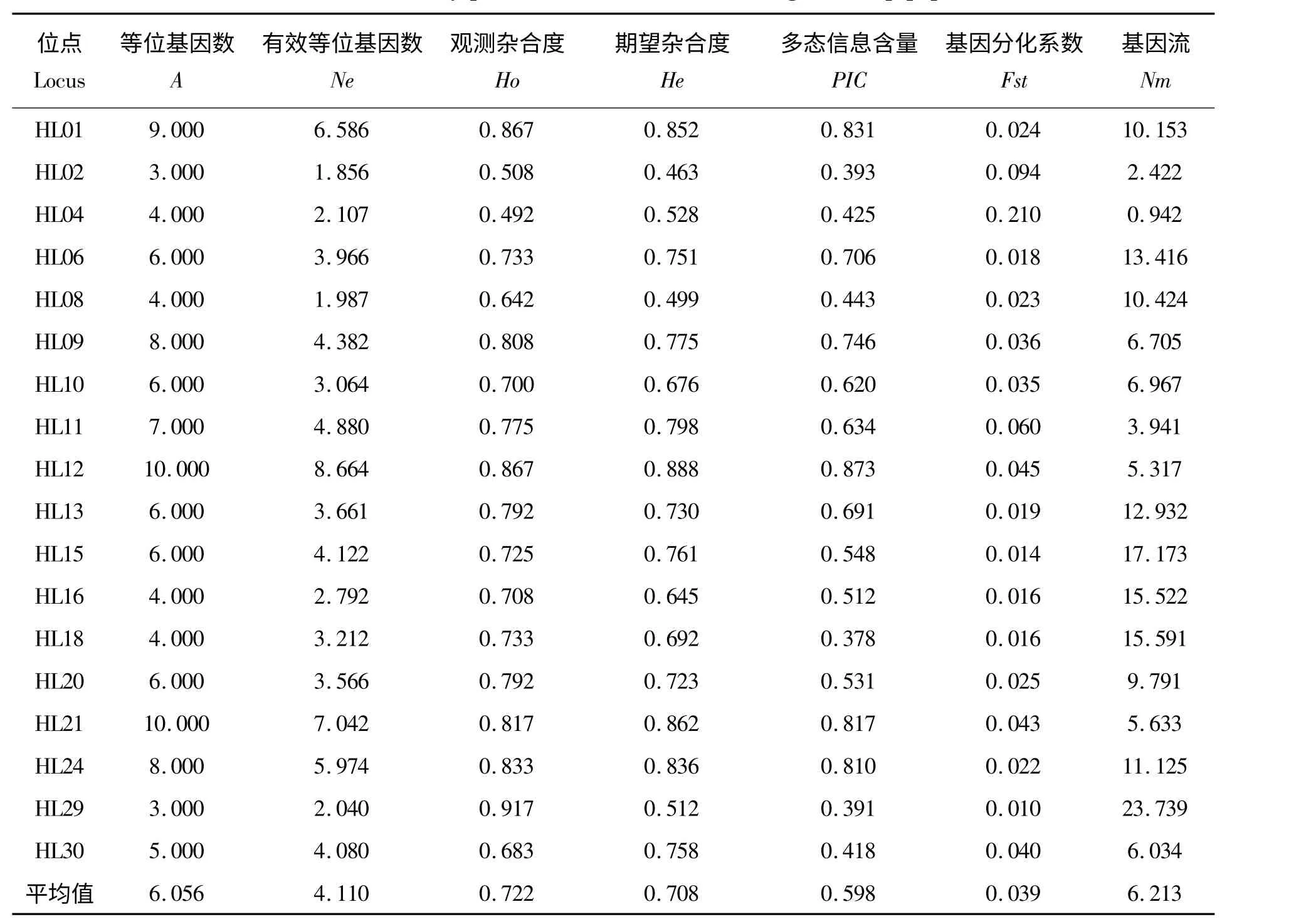
表2 鳙4 个群体在18 个微卫星位点遗传多样性参数Tab.2 Genetic diversity parameters in 18 loci of four bighead carp populations

表3 鳙4 个群体遗传多样性参数Tab.3 Genetic diversity parameters of four bighead carp populations

表4 鳙4 个群体间的Fst 值Tab.4 Fst value of four populations of bighead carp
利用Popgene 1.32 软件计算了4 个群体间的遗传相似系数与Nei,s 遗传距离,具体结果见表5。由表5 可以看出,蠡湖放流群体与常熟捕捞群体之间的遗传距离最大,与巢湖放流群体间的遗传距离最小。

表5 鳙4 个群体的遗传相似系数和遗传距离Tab.5 Genetic simi1arity coefficient and Nei,s genetic distance of four bighead carp populations
3 讨论
3.1 鳙群体的遗传多样性
遗传多样性是生物适应环境的基础和物种进化动力。对一个物种来说,其遗传多样性越高,则其对生存环境变化所产生的适应能力越强,进化的潜力也越大。等位基因数、杂合度被认为是度量物种遗传多样性的2 个较好指标。一般认为,杂合度越高,等位基因数越多,物种遗传多样性越丰富,对生活环境适应的潜能越大。廖小林[18]等利用6 对多态微卫星引物研究了长江水系的4 个草鱼群体的遗传结构,结果显示群体平均观测杂合度为0.400 0 ~0.574 1;姬长虹等[19]采用34 个微卫星分子标记对长江湘江、 监利和枞阳的3 个鲢群体遗传多样性进行了研究,得到3 个群体平均观测杂合度为0.416 9 ~0.461 9;DeWoody 等[20]通过微卫星标记研究得出13 种淡水鱼的平均观测杂合度约为0.460。本研究4个鳙群体的平均观测杂合度为0.678 ~0.753(表3),高于上述研究结果,表明所检测4 个鳙群体具有较高的遗传多样性。
吴力钊等[4]采用同工酶技术对池塘人工养殖4 ~5 龄鳙鱼群体(苗种来自长江南京江段)的遗传多样性进行了检测,结果表明该群体平均期望杂合度为0.057 7;赵金良等[5]采用同工酶技术分析从长江中游天鹅洲故道、汉阳、瑞昌和长江下游安庆4 个江段采集鱼苗饲养长大所得鳙鱼群体,得到平均期望杂合度为0.067 4;耿波等[15]采用17 个鳙鱼微卫星分子标记分析四川泸州和江西鄱阳湖2 个鳙鱼种群的遗传多样性,得到2 个群体平均期望杂合度分别为0.422 0 和0.452 0,平均观测等位基因数分别为3.325 和3.882;田华[16]对采自长江监利种质资源库的野生鳙群体和和长江所窑湾试验场养殖鳙群体的遗传多样性进行了研究,结果表明,野生和养殖鳙群体平均期望杂合度为0.550 2 和0.540 0,平均观测等位基因数为3.727 3 和3.636 4,2 个鳙群体都具有丰富的遗传多样性,野生群体的略丰富于养殖群体,但差异并不显著(P >0.05);本研究鳙4 个群体的平均期望杂合度为0.672 ~0.721,平均观测等位基因数为5.055 ~5.666,鳙4 个群体平均期望杂合度值明显高于吴力钊[4]和赵金良[5]的研究结果,推测由于采用的检测方法不同所致,说明基于DNA 分子的检测技术比基于蛋白质(酶)的检测技术更灵敏,SSR 检测到的信息比同工酶更丰富。与耿波等[15]的研究结果相比,本研究平均期望杂合度和平均观测等位基因数明显偏高,推测有2 个原因:一是电泳检测方法不同,耿波等[15]采用的是2%的琼脂糖凝胶,电泳分辨力不及聚丙烯酰胺凝胶,可能有些等位基因未检测到。二是由于检测的样本来源不同所致(笔者仔细查阅了耿波等的文章试图找出实验样品的来源,发现文章只交代了“DNA 样品为江西鄱阳湖鳙鱼群体24 尾个体DNA 及四川泸州鳙鱼群体24 尾个体DNA”,因此无法确认这2 个鳙群体为野生或养殖群体)。本研究结果和田华等[16]的研究结果最接近,只是平均期望杂合度和平均观测等位基因数略高于后者,可能和所检测的位点不同有关(本研究12 个位点表现为高度多态,后者只有4 个位点表现为高度多态)。具体原因有待于进一步的研究。
不少文献报道人工养殖群体的遗传多样性明显低于野生群体[21-23]。关于鳙养殖群体遗传多样性下降的研究已有报道[4,9-10]。这些研究认为,在人工养殖条件下封闭的群体比自然群体更容易发生瓶颈效应和近交衰退,进而造成了一些稀有等位基因的丢失,导致人工养殖群体平均等位基因数下降,人工养殖群体的遗传多样性明显低于天然(野生)群体。本研究中3 个放流群体的平均等位基因数与常熟捕捞群体相比,表现出一定程度的等位基因丢失现象(表3);但方差分析表明,3 个放流群体的遗传多样性与常熟捕捞群体没有显著差异(P >0.05),这和上述对鳙养殖群体和野生群体遗传多样性的研究结果不完全一致,笔者认为有以下2 个方面的原因:(1)本研究放流群体的亲本均来自长江,放流群体和长江野生群体的亲缘关系近,遗传多样性差异本身就很小。(2)由于近年大规模的增殖放流,加大了人工养殖种群和长江野生种群的基因交流,常熟江段捕捞群体可能混杂有以往人工放流群体,因而并不是真正意义上的“野生群体”。
3.2 群体内及群体间的遗传分化
基因分化系数Fst 是表征群体间遗传分化程度的重要参数,当该参数在0 ~0.05 时,群体间遗传分化较弱;在0.05 ~0.15 时,遗传分化中等;0.15 ~0.25 时,遗传分化较大;当大于0.25 时,表示群体间分化极大。张四明等[24]用RAPD 技术对长江水系的鲢和草鱼4 个地理群体的遗传分化进行了研究,发现鲢82.5%的遗传多样性来源于群体内,群体间的遗传分化为17.5%,草鱼87.7%的遗传多样性来源于群体内,群体间的遗传分化为12.3%;张志伟等[25]采用10 个微卫星标记对草鱼养殖和野生群体进行了研究,发现有14.99%的遗传变异来自于群体间,85.01%的遗传变异来于群体内。本研究结果表明4个鳙群体间的Fst 为0.023 ~0.053,群体间遗传分化小于上述研究结果,表明绝大多数的遗传多样性来源于群体内,群体间的遗传分化程度较弱。
3.3 增殖放流苗种的种质资源控制
现代杂种优势理论认为,杂种优势的大小在一定程度取决于亲本间遗传差异的大小,遗传距离愈大所产生的杂种优势愈大。本研究中放流群体的亲本均来自长江,虽然3 个放流群体的遗传多样性与长江常熟江段捕捞群体的遗传多样性没有显著差异,但3 个放流群体在一些位点的等位基因数明显低于常熟捕捞群体,存在一定程度的稀有等位基因丢失现象,应该引起足够重视。但值得注意的是,由于原(良)种场捕获的长江野生亲本数量有限,同时受生产条件、生产规模及资金的限制,原(良)种场保存亲本数量有限,因此人工养殖条件下的封闭群体比自然群体更容易发生瓶颈效应和近交衰退现象,从而加速种质的同质化,降低群体的遗传多样性。因此,有关部门应加强对原(良)种场的资金投入和监管力度,从源头上对放流苗种的种质资源把关。建议选择遗传多样性丰富、遗传距离较大而又具有良好性状的亲鱼作为增殖放流苗种的亲本。
[1]李思发.长江、珠江、黑龙江鲢、鳙、草鱼种质资源研究[M].上海:上海科学出版社,1990:25 -50.
[2]刘绍平,邱顺林,陈大庆,等.长江水系四大家鱼种质资源的保护和合理利用[J].长江流域资源与环境,1997,6(2):127 -131.
[3]廖亚明,刘金炉,汤学林.浅析“四大家鱼”性状退化的原因及重视种质保护的建议[J].水产科技情报,1994,21(2):62 -63.
[4]吴力钊,王祖熊.长江下游鳙鱼天然种群的生化遗传变异[J].水生生物学报,1991,15(1):94 -96.
[5]赵金良,李思发.长江中下游鲢、鳙、草鱼、青鱼种群分化的同工酶分析[J].水产学报,1996,20(2):104 -110.
[6]夏德全,杨弘,吴婷婷,等.天鹅洲通江型长江故道“四大家鱼”种群遗传结构研究[J].中国水产科学,1996,3(4):11 -18.
[7]蒋建国,熊全沫,姚汝华.青草鲢鳙四种鱼同工酶的比较研究[J].遗传,1998,20(2):19 -22.
[8]张锡元,杨建琪,张德春,等.白鲢和鳙鱼的随机扩增多态DNA 分析[J]. 生物化学与生物物理进展,1999,26(5):469 -472.
[9]张德春,张锡元,杨代淑,等.长江鳙遗传多样性的研究[J].武汉大学学报,1999,45(6):857 -886.
[10]张德春.鳙鱼人工繁殖群体遗传多样性的研究[J].三峡大学学报,2002,24(4):379 -381.
[11]李思发,吕国庆,贝纳切兹L. 长江中下游鲢鳙青草四大家鱼线粒体DNA 多样性分析[J]. 动物学报,1998,44(1):82 -93.
[12]樊连春,崔建勋,余其兴.鳙鱼线粒体DNA 的限制性内切酶图谱[J].武汉大学学报,1994,40(l):121 -125.
[13]单淇,董仕,吴海防,等. 三个群体鳙鱼mtDNA D -Loop 区段的限制性片段长度多态性分析[J]. 中国水产科学,2006,13(2):174 -179.
[14]鲁翠云,孙效文,梁利群.鳙鱼微卫星分子标记的筛选[J].中国水产科学,20O5,12(2):192 -196.
[15]耿波,孙效文,梁利群,等.利用17 个微卫星标记分析鳙鱼的遗传多样性[J].遗传,2006,28(6):683 -688.
[16]田华.鲢鳙长江野生群体和养殖群体微卫星的遗传多样性分析[D].武汉:华中农业大学,2008.
[17]Botstein D,White RL,Sckolnick M,et al.Construction of a genetic linkage map in mall using restriction fragment length polymorphism[J].Am J Human Genetic,1980,32(3):314 -331.
[18]廖小林,俞小牧,谭德清,等.长江水系草鱼遗传多样性的微卫星DNA 分析[J].水生生物学报,2005,29(3):113-119.
[19]姬长虹,谷晶晶,毛瑞鑫,等.长江、珠江、黑龙江水系野生鲢遗传多样性的微卫星分析[J].水产学报,2009,33(3):354 -371.
[20]De Woody J A,Avise J C.Microsatellite variation in marine freshwater and anadromous fishes compared with other animals[J].Journal of Fish Biology,2000,56:461 -473.
[21]LI Q,PARK C,ENDO T,et al.Loss of genetic variation at microsatellite loci in hatchery strains of the Pacific abalone (Haliotisdiscus hannai)[J].Aquaculture,2004,235(1 -4):207 -222.
[22]Skaala O,Hbyhein B,Glover K,et al.Microsatellite analysis in domesticated and wild Atlantic salmon (Salmo salar L.):Allelic diversity and identification of individuals[J].Aquaculture,2004,240(1/4):l31 -143.
[23]Norris A T,Bradley D G,Cunningham E P. Microsatellite genetic variation between and within farmed and wild Atlantic salmon (Salmo sala)populations[J].Aquaculture,1999,180(3/4):247 -264.
[24]张四明,汪登强,邓怀,等.长江中游水系鲢和草鱼群体mtDNA 遗传变异的研究[J].水生生物学报,2002,26(2):142-146.
[25]张志伟,曹哲明,杨弘,等.草鱼野生和养殖群体间遗传变异的微卫星分析[J].动物学研究,2006,27(2):189 -196.
