牛源性肠炎沙门氏菌的三型分泌系统基因分析
2013-10-22付红蕾杜冬冬庄燕飞常维山
付红蕾,李 彦*,杜冬冬,庄燕飞,常维山,王 涛
(1.青岛市饲料兽药检测站,山东 青岛 266071;2.山东农业大学,山东 泰安 271018;3.秦皇岛出入境检验检疫局,河北秦皇岛 066000)
肠炎沙门氏菌是一类重要的革兰氏阴性致病杆菌,可引起从轻度腹泻到全身性感染等一系列不同程度的疾病。其中肠炎沙门氏菌能够引起宿主发病与其三型分泌系统密切相关。三型分泌装置将细菌毒力蛋白运送至宿主细胞中,改变宿主细胞的功能如信号传导路径、细胞骨架结构、膜转运以及细胞因子的表达等。尽管转运的效应蛋白因病原体的不同而不同,但是最近的研究显示毒力相关的三型分泌装置在沙门氏菌、志贺氏菌和耶尔森菌中结构很保守[1,2,3]。其中SipA 编码分子量大小为87 kDa 的蛋白,与志贺氏菌IpaA 同源[4,5]。体外试验显示SipA和IpaA 都不是细菌侵袭细胞必需的蛋白[4,5,6],但有研究显示IpaA与细胞表面的纽带蛋白作用后参与侵袭作用[7]。SipA 的突变使肠黏膜炎性反应程度下降和腹泻程度减轻[9]。SipD 编码分子量为38kDa 的蛋白,SipD 与SipB、SipC 不同,不侵入真核细胞[8],是SipB、SipC和其它效应蛋白转移必需的蛋白[10,11]。SipD 突变后细菌的侵袭能力下降[4,5]。SipA 基因、SipD 基因,发生突变将会影响细菌的致病性。本次试验将该两个基因为研究对象,研究该株牛源性肠炎沙门氏菌与其它沙门氏菌间致病性差异。
1 材料和方法
1.1 材料
1.1.1 菌株来源 由本实验室从腹泻犊牛中分离保存的一株肠炎沙门氏菌。
1.1.2 主要试剂 细菌基因组DNA 提取试剂盒(北京天根生物技术公司);PCR 产物凝胶回收试剂盒(上海生工生物工程公司);ExTaqDNA 聚合酶、dNTP、MgCl2、Trans2K PlusⅡDNA Marker 购自北京全式金生物科技有限公司;TGL-16B 型微量离心机(上海安亭科学仪器厂);JunYi JY200C 型电泳仪(北京君意东方电泳设备有限公司)凝胶成像系统(美国UVP 公司,76-0312-02)。
1.2 方法
1.2.1 细菌DNA 提取及回收目的片段 严格按照细菌基因组DNA 提取试剂盒说明书要求提取细菌全基因组。
1.2.2 PCR 扩增SipA 基因目的片段 PCR 扩增体系:10×buffer 2.5μL;Mg2+2μL;上游引物0.5μL;下游引物0.5μL;模板DNA 0.5μL;dNTP 0.5μL;双蒸水18.3μL;ExTaq DNA 聚合酶0.2μL 。
扩增条件:94 ℃预变性10 min(94 ℃变性50 s,60 ℃退火50 s,72 ℃延伸2 min 50 s,30个循环)72℃延伸10 min。
PCR 产物按照PCR 产物凝胶回收试剂盒说明书进行胶回收,-20 ℃保存。
1.2.3 PCR 产物连接T 载体及转化DH5(感受态细胞回收纯化PCR 产物与克隆载体PEASY-T1 Cloning Vector 参照说明书进行连接,反应体系如下:
PCR 产物连接T 载体反应体系:PCR 产物0.5~4μL;PEASY-T1 Cloning Vector 1μL。
轻轻混合,室温(20 ℃~37 ℃)反应5 min。反应结束后,将离心管置于冰上。采用CaCl2法转化至DH5α 感受态细胞。
1.2.4 酶切鉴定 根据已发表的SipA 基因序列以及T 载体上的酶切位点,选用HindⅢ和EcoRⅤ双酶切转化成功的质粒,酶切体系如下:
酶切体系:质粒DNA 8.0μL;ddH2O 8.4μL;10×H Buffer 2μL;HindⅢ 0.8μL;EcoRⅤ0.8μL。
混匀后离心,37 ℃水浴酶切反应2 h,酶切产物于0.8% 琼脂糖凝胶中150 V 电泳30 min,在凝胶系统下记录结果。
1.2.5 SipD 基因PCR 扩增目的片段 PCR 扩增体系:10×Buffer 2.5μL;Mg2+2μL;上游引物0.5μL;下游引物0.5μL;模板DNA 0.5μL;dNTP 0.5μL;双蒸水18.3μL;ExTaq DNA 聚合酶0.2μL。
扩增条件:94 ℃预变性10 min (94 ℃变性50 s,55 ℃退火50 s,72 ℃延伸1 min 50 s,30个循环)72 ℃延伸10 min。
PCR 产物按照PCR 产物凝胶回收试剂盒说明书进行胶回收,-20 ℃保存。
1.2.6 PCR 产物连接转化及酶切 连接及转化体系同SipA 基因,根据已发表的SipD 基因序列以及T 载体上的酶切位点选用Apal和Notl 双酶切,酶切体系如下:
酶切体系:质粒DNA 8.0μL;ddH2O 8.4μL;10×H Buffer 2μL;Apal 0.8μL;Notl 0.8μL。
混匀后离心,37 ℃水浴酶切反应2 h,酶切产物于0.8% 琼脂糖凝胶中150 V 电泳30min,在凝胶系统下记录结果。
1.2.7 SipA 基因、SipD 基因测序分析 将连接转化成功的质粒送上海生工进行测序,测定好的SipA 基因、SipD 基因测序在GeneBank 上进行比对,以确定分离菌与其他菌株亲缘关系。
2 结果
2.1 SipA PCR 胶回收及酶切结果
SipA 基因经PCR 扩增后取20μL PCR 产物在0.8% 琼脂糖凝胶上电泳30 min,用凝胶回收试剂盒回收目的片段,回收目的片段在凝胶成像系统观察。SipA 基因PCR 扩增到与预期目标2058 bp 大小一致的目的片段(图1),PCR 产物回收纯化后,克隆至pEASY-T1 载体,构建重组质粒。重组质粒转化到E.coli DH5α 感受态细胞中,提取质粒,进行HindⅢ和EcoRⅤ双酶切鉴定。酶切得到两条片段,分别为载体片段约3928 bp、目的片段约2058 bp(图2),表明目的基因已经插入pEASY-T1 载体。
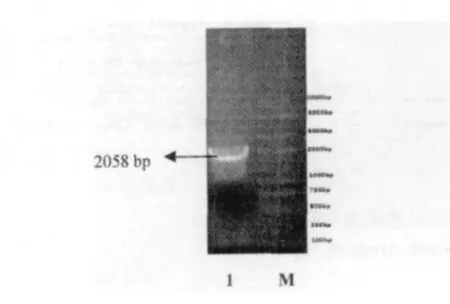
图1 SipA 基因PCR 扩增图Fig.1 PCR amplification of SipA gene
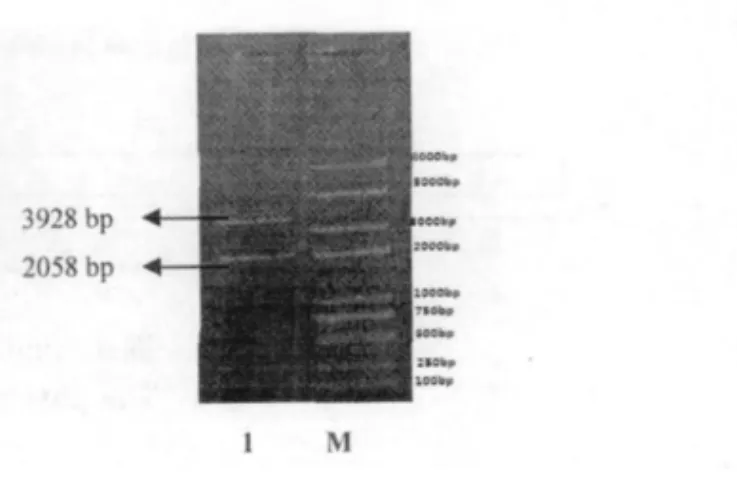
图2 SipA 基因酶切鉴定图Fig.2 EcoRΙ/HindⅢdigests of plasmid profile
2.2 SipD 胶回收及酶切结果
SipD 基因经PCR 扩增后取20μL PCR 产物在0.8% 琼脂糖凝胶上电泳30 min,用凝胶回收试剂盒回收目的片段,回收目的片段在凝胶成像系统观察。SipA 基因PCR 扩增到与预期目标1032 bp 大小一致的目的片段(图3)。PCR 产物回收纯化后,克隆至pEASY-T1 载体,构建重组质粒。重组质粒转化到E.coli DH5α 感受态细胞中,提取质粒,进行HindⅢ和EcoRⅤ双酶切鉴定。酶切得到两条片段,分别为载体片段约3928 bp、目的片段约1032 bp(图4),表明目的基因已经插入pEASY-T1 载体。
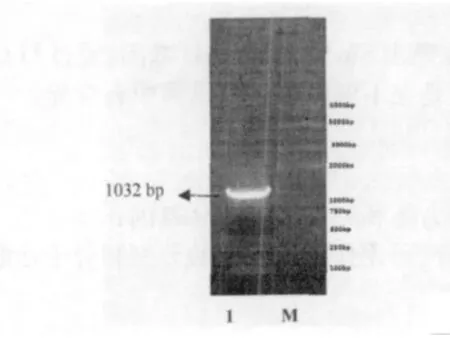
图3 SipD 基因PCR 扩增图Fig.3 PCR amplification of SipA gene
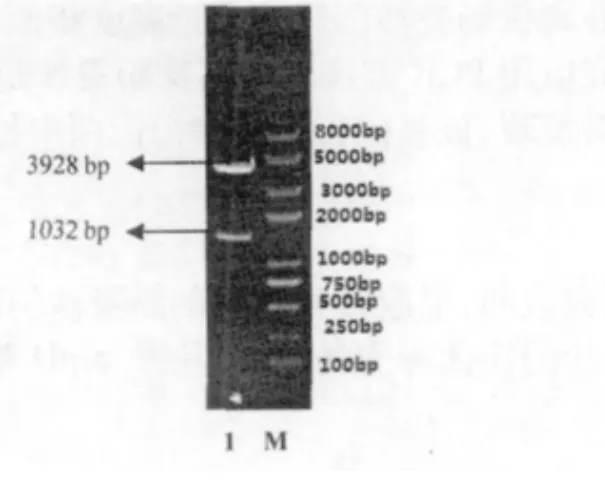
图4 SipD 酶切鉴定图Fig.4 Apal/Notl digests of plasmid profile
2.3 SipA 基因系统进化树分析
本次分离株的SipA 基因系统进化树分析结果表明,本次分离株与肠炎沙门氏菌P125109、都柏林沙门氏菌CT_02021853、新港沙门氏菌SL254 同源性高达100%。与鼠伤寒沙门氏菌、鸡白痢沙门氏菌等其它几株沙门氏菌的同源性相对较低(图5)。
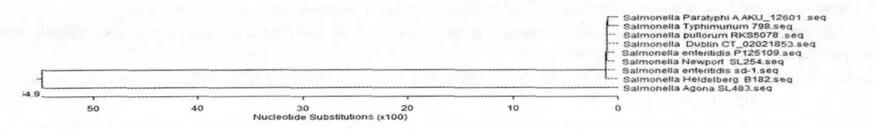
图5 SipA 基因系统进化树Fig.5 The phylogenetic trees of SipA gene
2.4 SipD 基因进化分析
本次分离株的SipD 基因系统进化树分析结果表明,本次分离株与肠炎沙门氏菌P125109、都柏林沙门氏菌CT_02021853、新港沙门氏菌SL254、海德堡沙门氏菌B182、鸡白痢沙门氏菌RKS5078 同源性高达100%。与鼠伤寒沙门氏菌等其它几株沙门氏菌的同源性相对较低(图6)。
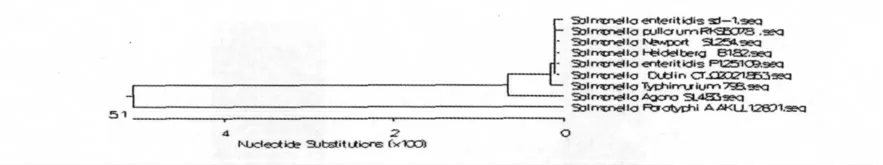
图6 SipD 基因系统进化树Fig.6 The phylogenetic trees of SipD gene
3 讨论
很多致病菌都拥有一个进化保守的三型分泌系统,这些致病菌通过三型分泌系统分泌一系列效应蛋白分子进入真核宿主体内,这些效应蛋白能够调节宿主细胞的正常信号通路并且发挥致病作用。Ⅲ型分泌系统通常由30~40 kbp 大小的基因组编码,以毒力岛的形式存在于细菌的大质粒或染色体。本试验研究了一株肠炎沙门氏菌毒力岛的SipA 基因、SipD 基因的结构和遗传变异。
研究发现,该株由腹泻犊牛体内分离的肠炎沙门氏菌与肠炎沙门氏菌P125109 同源性达100%,该株细菌最早在蛋鸡体内分离到。肠炎沙门氏菌P125109 临床上能够引起蛋鸡腹泻,与本次临床分离株肠炎沙门氏菌能够引起犊牛腹泻相似。通过SipA 基因、SipD 基因进化树分析发现,与临床分离株SipA 基因、SipD 基因同源性均为100%的菌株有肠炎沙门氏P125109、都柏林沙门氏CT_02021853、新港沙门氏菌SL254。研究发现肠炎沙门氏菌P125109、都柏林沙门氏菌CT_02021853、新港沙门氏菌SL254,在致病性上与临床分离株肠炎沙门氏菌保持高度相似。
本研究应用PCR 方法对分离株肠炎沙门氏菌成功克隆出SipA 基因、SipD 基因,通过与GenBank 提交的序列比对发现,该基因在肠炎沙门氏菌中稳定遗传,但是在不同型的沙门氏菌中有变异。
4 结论
4.1 本研究表明,引起犊牛腹泻的肠炎沙门氏菌含有毒力岛SipA 基因、SipD 基因;
4.2 肠炎沙门氏菌毒力岛SipA 基因、SipD 基因高度保守,可用作沙门氏菌的分型和分子诊断目标基因。
[1]Kubori T,Matsushima Y,Nakamura D,et al.Supramolecμlar structure of the Salmonella typhimurium type III protein secretion system.Science.1998,280:602–605
[2]Tamano K,Aizawa S,Katayama E,et al.Supramolecμlar structure of the Shigella type III secretion machinery:the needle part is changeable in length and essential for delivery of effectors.Embo J.2000,19:3876–3887
[3]Hoiczyk E,Blobel G.Polymerization of a single protein of the pathogen Yersinia enterocolitica into needles punctures eukaryotic cells.Proc Natl Acad Sci USA.2001,98:4669-4674
[4]Hueck CJ,Hantman MJ,Bajaj V,et al.Salmonella typhimurium secreted invasion determinants are homologous to Shigella Ipa proteins.Mol Microbiol.1995,18:479-490
[5]Kaniga K,Trollinger D,Galan JE.Identification of two targets of the type III protein secretion system encoded by the inv and spa loci of Salmonella typhimurium that have homology to the Shigella IpaD and IpaA proteins.J Bacteriol.1995,177:7078-7085
[6]Menard R,Sansonetti PJ,Parsot C.Non-polar mutagenesis of the ipa genes defines IpaB,IpaC,and IpaD as effectors of Shigella flexneri entry into epithelial cells.J Bacteriol.1993,175:5899-5906
[7]Tran Van Nhieu G,Ben-Ze'ev A,Sansonetti PJ.Modulation of bacterial entry into epithelial cells by association between vinculin and the Shigella IpaA invasion.EMBO J.1997,16:2717
[8]Collazo CM,Galan JE.The invasion-associated type III system of Salmonella typhimurium directs the translocation of the Sip proteins into the host cell.Mol Microbiol.1997,24:747-756
[9]Zhang S,Santos RL,Tsolis RM,et al.The Salmonella enterica serotype Typhimurium effector proteins SipA,SopA,SopB,SopD,and SopE2 act in concert to induce Diarrhea in Calves.2002,70:3843-3855
[10]Santos RL,Tsolis RM,B umler AJ,et al.Salmonella enterica serovar Typhimurium induces cell death in bovine monocyte-derived macrophages by early sipB-dependent and delayed sipB independent mechanisms.Infect Immun.2001,69:2293-2301
[11]Hersh D,Monack DM,Smith MR,et al.The salmonella invasin SipB induces macrophage apoptosis by binding to caspase-1.Proc Natl Acad Sci USA.1999,96:2396-2401
