基于COI和ITS-1基因对魁蚶野生群体和人工繁育子代的遗传分析
2013-10-13邱兆星王鸿霞张秀梅刘保忠
陈 桢 , 邱兆星, 王鸿霞, 张秀梅 刘保忠
(1. 中国海洋大学 海水养殖教育部重点实验室, 山东 青岛 266003; 2. 中国科学院 实验海洋生物学重点实验室, 山东 青岛 266071; 3. 山东省海水养殖研究所, 山东 青岛 266002)
魁蚶(Scapharca broughtoniiSchrenck)俗称赤贝、血贝、大毛蚶, 隶属软体动物门(Mollusca), 双壳纲(Bivalvia), 翼形亚纲(Pterimorphia), 蚶目(Arcoida), 蚶科(Arcidae), 是一种大型经济贝类, 主要分布于中国、日本、朝鲜半岛及俄罗斯东南部沿海。在我国以黄海北部较多, 主要在山东及辽宁沿海分布[1]。迄今为止,对魁蚶的研究工作已开展较多, 包括生殖生物学和苗种繁育[2-6]、资源生态[7-8], 群体遗传结构[9-12]等。
山东荣成濒临黄海北部, 沿岸浅海水域历来就有魁蚶这一优势种质资源的自然分布。20世纪80年代初, 海岸带生物资源调查结果发现, 魁蚶自然资源在荣成海域的集中分布面积约7000 ha, 其蕴藏量超过2万t , 平均生物密度达0.476个/m2, 是荣成近岸水域资源开发的重要经济种[13]。自 20世纪 90年代以来, 由于资源过度开发, 使原有的魁蚶资源受到极大破坏,自然种群生物量显著下降。目前, 荣成海域内魁蚶自然资源分布区域面积锐减, 按原始资源面积计算, 自然生物量少于0.01个/m2[13]。因此, 荣成市海洋与渔业局广泛组织、发动沿海魁蚶资源引进、增殖和修复技术。
近年来, 为满足魁蚶资源保护和增殖的需要,魁蚶的底播增养殖研究日益受到重视, 并取得一定的生态和经济效益[14-17]。在资源增殖过程中, 人们更多地关注资源量的增加, 而对人工苗种对本底生物资源可能带来的遗传影响重视不够。不合理的繁育和增殖方式可能导致近交快速积累、有效群体不断减少等负面效果, 引起近交衰退, 使种苗遗传多样性下降, 表现为生长减缓、抗病力减弱、品质退化等。有报道指出, 群体的遗传多样性每丧失 10%, 就会对其繁育能力、存活率、生长等重要性状产生很大的负面影响[18]。
本研究采用线粒体COI基因和核糖体ITS-1基因序列, 对荣成和即墨 2个魁蚶野生群体进行遗传多样性研究, 探讨了即墨群体人工繁育子一代群体的遗传多样性变化及其在荣成底播增殖的可行性。
1 材料与方法
1.1 实验材料
于 2009年 9月, 从荣成(编号 RC)采集 26个野生魁蚶个体; 从即墨采集一批野生魁蚶(数量>100个)作为亲本, 通过人工育苗的方式得到一批子代,从亲本群体中随机取37个个体(编号JM), 从子代中随机取40个个体(编号JZ)(子代生长到2010年5月进行采集), 共对103个魁蚶个体进行分析。
1.2 实验方法
1.2.1 基因组DNA提取及PCR扩增
采用TIANGEN海洋动物组织基因组DNA提取试剂盒提取魁蚶的基因组DNA后, 保存-20℃待用。
先用 COI通用引物(COI L1490: 5′-GGTCAACAAATCATAAAGATATTGG-3′和 COI H2198: 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′)扩增得到 2 个魁蚶个体的COI序列; 再根据得到的序列, 用Primer Premier 5.0软件设计COI特异引物, 序列为: COI-1:5′-CAAATGTTGGAATAGTAAAGGA-3′; COI-2: 5′-TATTTTCAGGATTCTGGGCA-3′。所用的ITS-1引物[19]为 : ITS-1A: 5′-GGTTCTGTAGGTGAACCTGC-3′;ITS-1B: 5′-CTGCGCCATCGACCC-3′。
PCR 反应体系 25 μL: ddH2O 17.25 μL; DNA 模板1 μL; 10×PCR buffer 2.5 μL; MgCl2(25 mmol/L)1.5 μL;dNTPs(10mM)0.5 μL; 引 物 F(10mM)1 μL; 引 物R(10mM)1 μL; Taq DNA Polymerase(5U/μL)0.25 μL。PCR反应程序为: 94℃变性5 min, 1个循环; 94℃变性20 s, 50℃/58℃退火30 s, 72 ℃延伸30 s, 34个循环;72℃延伸10 min, 1个循环; 4℃保存。
所有PCR扩增在Takara T650 PCR仪上进行。
1.2.2COI基因PCR产物纯化、克隆及测序
PCR产物经琼脂糖凝胶电泳确定为单一目的条带。用上海生工SanPrep柱式DNA胶回收试剂盒回收纯化。将纯化产物与pMD19-T 载体(大连宝生物)连接, 连接反应体系为 5 μL(solution I 2.5 μL, vector 0.4 μL, 纯化产物2.1 μL), 16℃连接过夜。连接产物42℃转化到大肠杆菌 TOP10感受态细胞中, 并涂布于 LB平板上 37℃过夜。挑取边缘清晰的菌落作模板, 用 M13 通用引物(M13 F: 5′-AGCGGATAACAATTTCACACAGG-3′; M13 R: 5′-CGCCAGGGTTTTCCCAGTCACGAC-3′)进行PCR扩增, 筛选阳性克隆。将阳性克隆扩大培养, 送交北京六合华大基因科技有限公司测序。
1.2.3ITS-1基因PCR产物纯化及测序
PCR产物经琼脂糖凝胶电泳确定为单一目的条带。用上海生工SanPrep柱式DNA胶回收试剂盒回收纯化。将纯化产物送交北京六合华大基因科技有限公司测序。
1.2.4 数据分析
所有序列均由Mega 4.0软件[20]进行编辑、校对和比对, 并对比对结果进行分析和手工校正, 并去除序列中不确定的部分。一共得到63个魁蚶野生个体(荣成26个和即墨37个)和40个即墨人工繁育子代的 605bp的 COI基因序列 (NCBI登录号为:KC588467- KC588503)和相应的354bp的ITS-1基因序列(NCBI登录号为: KC555313- KC555327)。
采用Mega 4.0软件按照Kimura等[21]双参数进化模型计算单倍型间的遗传距离, 构建单倍型的邻接关系树, 并采用1000次重抽样检验系统树分支的可靠性。
用DnaSP 5.0[22]软件计算序列中核苷酸组成、多态位点数(S)、核苷酸多样性(π)、单倍型数(h)和单倍型多样性指数(Hd), 分析群体的多样性水平。
利用 Arlequin3.11软件[23]计算单倍型间的关系和核苷酸替换数, 手工绘制最小跨度树(Minimum spanning tree)。采用Arlequin 3.11软件计算魁蚶群体之间的遗传分化指数Fst值来检验群体遗传结构。Fst值的显著水平通过 10000次重抽样来检验, 并用Bonferroni方法[24]对其进行校正。
2 结果
2.1 序列分析
实验共获得长度为605bp的COI基因序列片段和长度为354bp的ITS-1基因序列片段各103条。COI基因片段的T、C、A、G碱基平均含量分别为23.7%,21.4%, 40.1%, 14.8%, A+T含量明显高于C+G含量,而ITS-1基因片段的 T、C、A、G碱基平均含量分别为 25.6%, 26.9%, 20.1%, 27.4%, A+T含量低于C+G含量。
2.2 遗传多样性
在COI基因序列的605个位点中, 多态位点73个, 其中简约信息位点29个。这些多态位点共定义了 37个单倍型(表 1), 其中有 Hap1出现频率最高,为37.8%的即墨野生(亲代)群体和77.5%的即墨子代群体所共享, 在荣成野生群体中未检测到单倍型Hap1; Hap4为18.9%的即墨野生群体与61.5%的荣成野生群体共享, 而其他35个单倍型为各个群体私有且只在一个个体中发现。即墨野生群体中私有单倍型占总单倍型的 43.3%, 荣成野生群体的私有单倍型占总单倍型的 38.5%, 即墨子代群体的私有单倍型占总单倍型的22.5%。

表1 COI序列单倍型在各群体中分布Tab.1 Distribution of COI haplotypes of S. broughtonii among localities
在ITS-1基因序列的354个位点中, 检测到插入/缺失3处, 多态位点10个, 其中简约信息位点7个。这些多态位点共定义了15个单倍型(表2), 其中有5个单倍型为3个群体共有享, H1是出现频率最高的单倍型, 有 2个单倍型为即墨野生群体和即墨子代群体共享, 其他 8个单倍型为各个群体私有且只在一个个体中发现。

表2 ITS-1序列单倍型在各群体中分布Tab.2 Distribution of ITS-1 haplotypes of S. broughtonii among localities
基于多态位点得到各群体的核苷酸多样性指数(π),多态位点数(S), 单倍型数(h)和单倍型多样性指数(Hd)见表3和表4。分析COI基因序列发现, 荣成和即墨野生群体的单倍型多样性指数分别为0.631和0.832, 核苷酸多样性指数分别为0.0015和0.0252; 即墨人工培育子代群体的单倍型多样性指数为0.404, 核苷酸多样性指数为 0.0008, 显然比 2个野生群体的多样性指数低。分析ITS-1基因序列的结果, 发现野生群体和人工培育即墨子代群体间的单倍型多样性和核苷酸多样性指数差异不大。
2.3 系统发育关系
基于COI基因片段定义的37个单倍型的邻接关系树显示, 所有单倍型明显分为 2个类群(图 1), 并得到很高的置信度支持。全部荣成野生群体的单倍型和部分即墨野生群体单倍型聚成类群Ⅰ, 部分即墨野生群体的单倍型和全部即墨子代群体的单倍型聚成类群Ⅱ。而基于ITS-1基因片段定义的15个单倍型的邻接关系树置信度低, 单倍型之间的关系无法确定(图2)。
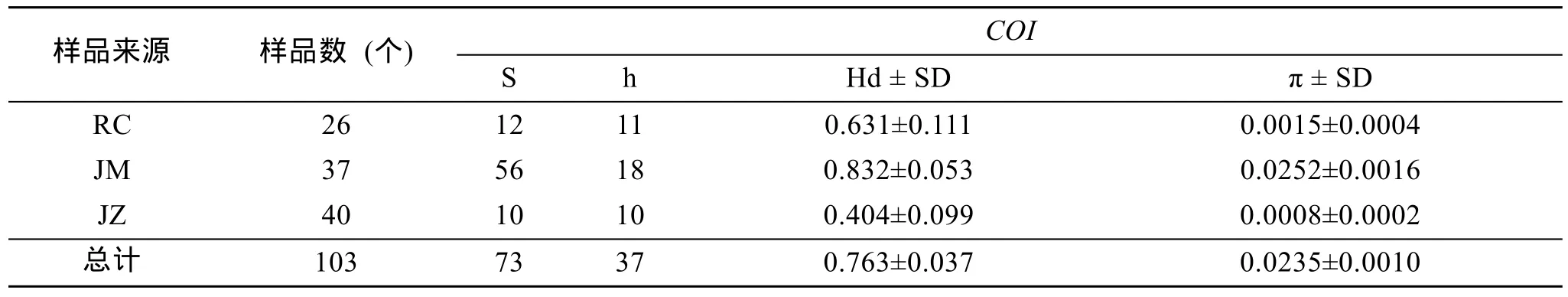
表3 魁蚶样品数和基于线粒体 COI基因的多态位点数(S), 单倍型数(h), 单倍型多样性(Hd)和核苷酸多样性(π)及其标准差(Standard deviation, SD)Tab.3 Sample size and number of variable sites (S), number of haplotype (h), haplotype diversity (Hd), nucleotide diversity (π)of S. broughtonii based on COI. SD: Standard deviation
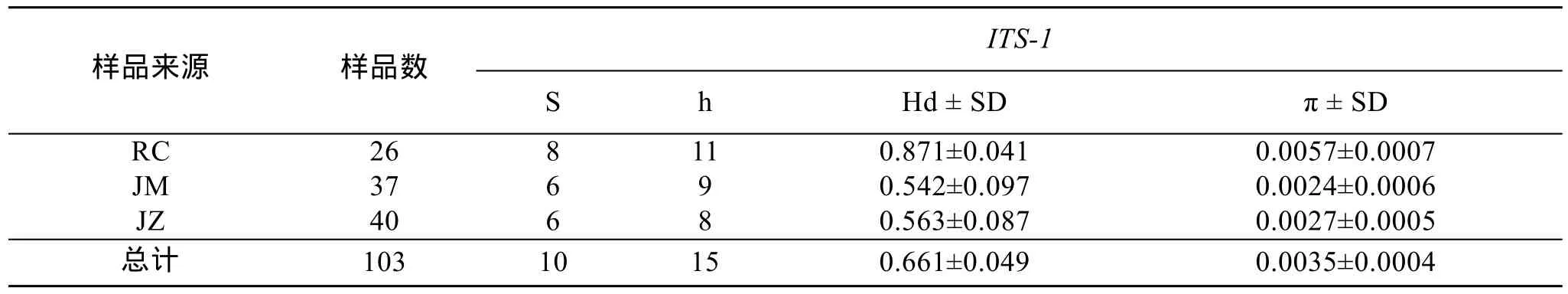
表4 魁蚶样品数和基于核糖体内转录间隔区ITS-1的多态位点数(S), 单倍型数(h), 单倍型多样性(Hd)和核苷酸多样性(π)及其标准差(Standard deviation, SD)Tab.4 Sample size and number of variable sites (S), number of haplotype (h), haplotype diversity (Hd), nucleotide diversity (π)of S. broughtonii based on ITS-1. SD: Standard deviation
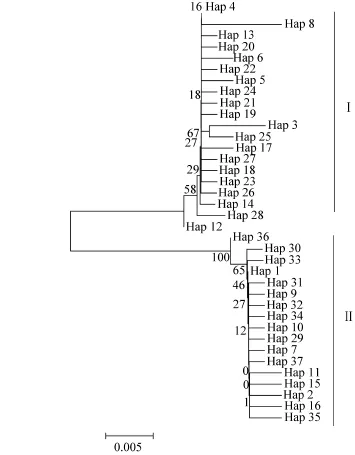
图1 用Mega 4.0构建的魁蚶COI基因单倍型间的邻接关系树Fig.1 Neighbor-joining tree of haplotypes of S. broughtonii based on COI sequences constructed by Mega 4.0
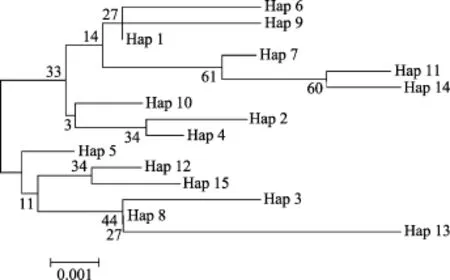
图2 用Mega 4.0构建的魁蚶ITS-1基因单倍型间的邻接关系树Fig.2 Neighbor-joining tree of haplotypes of S.broughtonii based on ITS-1 sequences constructed by Mega 4.0
基于COI和ITS-1基因构建的单倍型最小跨度树有相似的结构, 都呈星形辐射状。处在星状结构中心的都为出现频率最高的单倍型(基于COI和ITS-1基因分别为Hap1和H1), 如图3和图4与表1和表2。COI基因单倍型中, Hap1只在即墨野生和子代群体中出现, 荣成群体中未出现; 而ITS-1单倍型中,H1在3个群体中均有出现。
2.4 群体遗传结构
基于COI基因序列片段, AMOVA分析结果表明,魁蚶群体的群体间差异占 26.74%, 群体内差异占73.26%, 分子差异主要在群体内(表5)。基于ITS-1基因序列片段, AMOVA分析结果也表明分子差异主要在群体内, 群体间与群体内分子差异分别占 6.86%和93.14%(表5)。2个基因序列分析得到的固定指数分别为 0.2674和 0.0686, 统计检验结果都为极显著(P<0.01), 表明魁蚶群体中存在明显的遗传结构。表现群体间遗传分化的Fst值见表 6。基于COI基因得到的荣成和即墨 2个野生魁蚶群体之间的Fst值为 0.168,基于ITS-1基因得到的两群体间Fst值为0.1193, 表明2个野生群体之间存在一定程度的遗传分化。
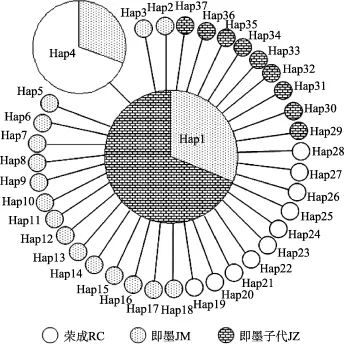
图3 魁蚶37个COI基因单倍型的最小跨度树Fig.3 Minimum spanning tree of the 37 haplotypes of the S.broughtonii based on COI sequences

图4 魁蚶15个ITS-1基因单倍型的最小跨度树Fig.4 Minimum spanning tree of the 15 haplotypes of the S.broughtonii based on ITS-1 sequences
3 讨论
从本研究的结果看,COI基因的碱基含量分布不均匀, A+T含量高于C+G含量, 表现出明显的碱基偏向性, 这与在其他无脊椎动物的COI基因中观察到的结果是一致的[25-26]。而ITS-1基因的碱基含量分布表现为A+T含量低于C+G含量, 陈琳琳[27]在日本、俄罗斯海参崴和中国威海 3个魁蚶地理群体中也发现类似结果, 这与其他贝类的ITS序列中观察到的结果相似[28]。
遗传多样性降低在人工繁殖群体中是一个较普遍的现象。方耀林等[29]在采用 mtDNA控制区序列,对野生大鲵及其人工繁殖后代的遗传多样性进行分析时发现, 两组亲本的单倍型多样性及核苷酸多样性均高于各自的子代。王鸿霞等[30]发现凡纳滨对虾人工繁育的子代的等位基因数比亲代低 11.11%, 平均期望杂合度、平均观测杂合度和平均多态性信息含量等指标均低于亲代。已有群体遗传学研究表明,遗传多样性的保持和有效群体大小密切相关。当有效群体较小时, 由于瓶颈效应, 野生或养殖群体倾向于近交水平的增加[31], 引起种群遗传多样性水平的下降。在本研究中, 虽然采用了100个个体以上规模的亲本群, 但基于COI基因序列片段的结果看,即墨人工繁育的子代群体的单倍型多样性和核苷酸多样性明显比即墨野生群体低。又有研究证实, 在水产动物的自然繁育中, 只有很少的亲本参与了子代基因库的构成, 有效群体比繁育亲本群体要小的多。如Sekino等[32]利用4个高度多态性的微卫星位点对牙鲆子代群体进行了家系鉴定, 结果表明不同的亲本对子代的贡献率不同, 18个亲本中仅一半对后代基因有贡献。Vandeputte等[33],王鸿霞等[30]在对子代群体进行家系鉴定时也发现相似的现象。也就是说,亲本数量和亲本对子代的不同贡献率可能是造成子代群体遗传多样性低于亲代群体的原因。
在对COI基因序列分析时发现, 即墨人工繁育群体的核苷酸多样性比野生亲代群体有明显降低,但基于ITS-1基因序列分析时发现, 两群体的多态位点数和核苷酸多样性指数相差不大。同样, 在即墨群体与荣成群体之间, 基于COI基因序列分析结果发现两者的多态位点数和核苷酸多样性指数差异显著,两群体的多态位点数分别为56和12, 核苷酸多样性分别为0.0252和0.0015, 呈现明显的倍数关系。而基于ITS-1基因序列分析却发现, 两群体的多态位点数和核苷酸多样性指数相差不大。这可能是由于线粒体基因比核基因的进化速率快[34]和ITS-1基因种内变异小[35]的特点造成的。ITS-1基因种内变异小的现象在其他研究中也有发现, 如Philips等[36]对不同地理种群的红点鲑的ITS-1进行测序分析, 发现种内不同种群之间的序列相似性较大; 林昕等[37]采用ITS-1基因序列片段, 发现福建沿海不同养殖区泥蚶群体具有较高的遗传相似性。因此, 作者认为基于COI基因序列检测魁蚶群体水平多态性比ITS-1基因更为敏感。
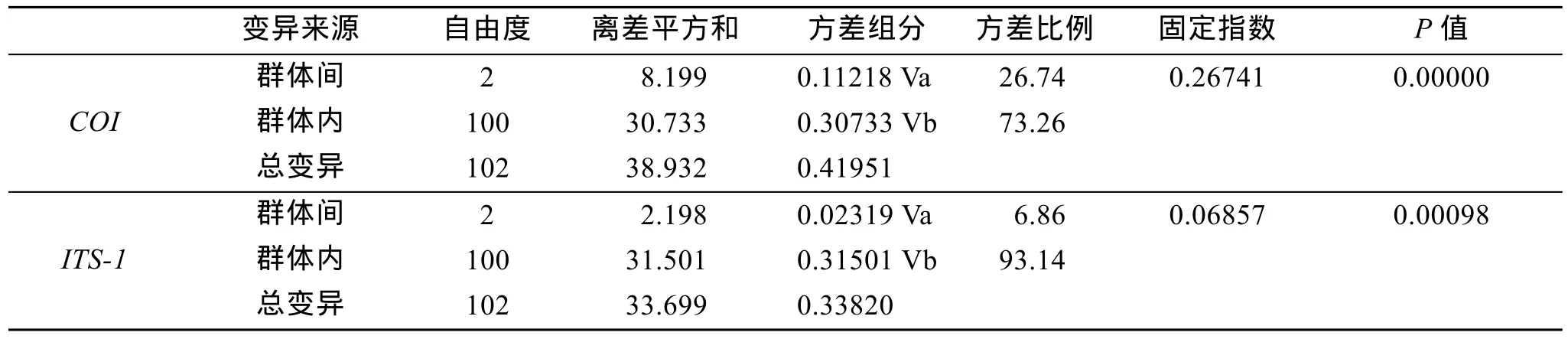
表5 3个魁蚶群体的分子方差分析Tab.5 Analysis of molecular variance(AMOVA)of COI and ITS-1 haplotypesof S. broughtonii populations

表6 基于COI(对角线以上)和ITS-1(对角线以下)基因序列单倍型分析得到的群体间遗传分化指数Fst值Tab.6 Population pairwise(Fst)between S.broughtonii populations based on COI(above diagonal)和ITS-1(below diagonal)sequences
在单倍型邻接关系树中, 基于COI基因序列得到的单倍型以高置信度聚成2个类群, 而基于ITS-1基因序列得到的单倍型邻接关系树置信度低, 单倍型之间的关系无法确定, 该结果可能也与ITS-1基因的种内变异小有关。在COI基因序列单倍型的邻接关系树中, 荣成野生群体和部分即墨野生群体聚在同一个类群(类群Ⅰ)中, 另一部分即墨野生群体与即墨子代聚在类群Ⅱ, 表明荣成和即墨野生群体之间存在某些程度的遗传分化。根据 Wright[38]对遗传分化程度划分的等级,Fst为 0.05~0.15, 说明群体间存在中等程度的遗传分化。而即墨和荣成野生群体之间的Fst值基于COI和ITS-1基因分别为0.1678和0.1193, 表明魁蚶2个野生群体之间的遗传分化程度为中等水平。在本研究中, 基于COI和ITS-1基因构建的单倍型最小跨度树都为星状结构, 这通常被认为是种群经历过种群扩张的结果[39], 处于星状结构中心的单倍型(COI和ITS-1基因序列中分别为Hap1和H1)推测为祖先单倍型。在COI单倍型分布中, 可能是祖先单倍型的 Hap1在荣成野生群体中未出现,这可能与分析样品数量有关。
2种基因序列片段的分子方差分析(AMOVA)结果都表明, 魁蚶 3个群体内的分子遗传变异明显大于群体间的遗传变异。研究中发现, 相对于ITS-1基因的AMOVA分析结果, 基于COI基因序列分析时群体间的遗传变异比例较大(26.74%)。进行Fst分析时同样发现这样的问题, 基于COI基因序列得到的群体间Fst值比ITS基因序列得到的Fst值大。我们认为这可能是由于2个基因的进化速率不同和ITS-1种内变异小的特性共同导致的结果。同时也注意到, 基于COI基因序列分析得到的荣成野生群体与即墨子代之间的Fst值最大(为 0.4958), 两群体间遗传差异较大, 这可能会对荣成魁蚶野生群体的遗传结构产生不可忽略的影响, 底播时应慎重。方耀林等[29]在研究野生大鲵及其人工繁殖后代的遗传多样性分析时,发现2个子代种群的Fst值较大, 认为不同大鲵人工繁殖的后代, 遗传分化有扩大的趋势。这提示人们也可进一步对人工繁育的荣成群体子代和即墨群体子代的遗传多样性和遗传结构进行研究, 以更准确地揭示魁蚶不同地理群体的遗传多样性和遗传分化关系, 为魁蚶的底播增殖工作提供科学的参考依据。
4 结论
荣成市魁蚶的底播增殖正逐步深入开展。底播苗种的选用是底播增殖能否取得显著成效的关键。人工增殖是一种重要的资源保护与修复手段。但选择适当的种苗和增殖地点是前提。若选择不当, 不仅达不到保护资源或修复资源的作用, 反而可能对种群本身的遗传特性造成破坏。在切实开展增殖工作之前, 应采取必要的调研等相关工作, 确保增殖活动能取得预期的成效。
本研究通过对即墨和荣成两地的野生群体以及人工繁育即墨群体获得的子代进行遗传多样性和遗传结构的分析, 探讨了即墨群体人工繁育的苗种在荣成海域底播增殖的可行性。研究结果发现, 荣成和即墨2个野生群体本身就存在中等程度的遗传分化。人工繁育的即墨子代群体遗传多样性比即墨野生群体有所降低。荣成群体与即墨子代之间的遗传差异,较之与即墨野生群体的差异更大。如果采用即墨子代在荣成底播, 可能会对荣成野生魁蚶野生群体的遗传结构产生一定影响, 因此, 采用即墨人工繁育子代作为荣成魁蚶底播增殖的苗种来源时必须十分慎重, 并在苗种繁育时尽量采用大群体有效亲本。
[1]王如才, 王昭萍, 张建中. 海水贝类养殖学[M]. 青岛: 青岛海洋大学出版社, 1993: 156.
[2]郭学武. 魁蚶生殖生物学初步研究[J]. 海洋水产研究, 1994, 15:55-63.
[3]于瑞海, 孙志刚, 王长竹, 等. 魁蚶大水体升温育苗技术研究[J]. 海洋科学, 1995, 2: 5-7.
[4]于瑞海, 王如才, 姜春丽, 等. 魁蚶生产性间断升温育苗技术的研究[J]. 黄渤海海洋, 1997, 15(3): 40-45.
[5]Numaguchi K. Gonad development of the ark shellScapharca broughtonii(Schrenck)broodstock in farming grounds of Japan [J]. Nippon Suisan Gakkaishi,1996, 62(3): 384-392.
[6]郑国富, 王晓波, 付卓. 魁蚶人工育苗生产中几项技术探讨[J]. 河北渔业, 2008, 1: 41-42.
[7]陈觉民, 王恩明, 李何. 海水中某些化学因子对魁蚶幼虫、稚贝及成体的影响[J]. 海洋与湖沼, 1989, 20(1):15-22.
[8]Park MS, Lim HJ, Kim PJ. Effect of environmental factors on the growth, glycogen and hemoglobin content of cultured ark shell,Scapharca broughtonii[J].J Korean Fish Soc, 1998, 31: 176-185.
[9]Lee JM, Park JW, Yoo MS, et al. Morphological characteristics and genetic diversity using the RAPD technique in the ark shell,Scapharca broughtonii(Schrenck)from Korea and China [J]. J Korean Fish Soc, 1997, 30: 297-304.
[10]Yokogawa K. Morphological and genetic differences between Japanese and Chinese red ark shellScapharca broughtonii[J]. Fish Sci, 1997, 63: 332–337.
[11]喻子牛, 孔晓喻, 杨锐, 等. 魁蚶(Scapharca broughtonii)等位基因酶遗传变异研究[J]. 青岛海洋大学学报, 1998, 28(1): 51-58.
[12]Cho ES, Jung CG, Sohn SG, et al. Population genetic structure of the ark shellScapharca broughtoniiSchrenck from Korea, China, and Russia based on COI gene sequences [J]. Marine Biotechnology, 2007, 9 (2):203-216.
[13]宋云杰, 郭旭强. 荣成市魁蚶底播增殖可行性探讨[J]. 齐鲁渔业, 2010, 27(8): 53-54.
[14]唐启升, 王俊, 邱显寅, 郭学武. 魁蚶底播增殖的试验研究[J]. 海洋水产研究, 1994, 15: 79-86.
[15]孙鹏飞, 刘杰, 王卫民, 等. 浅海底播养殖魁蚶试验[J]. 河北渔业, 2009, 10: 42.
[16]于瑞海, 李琪. 无公害魁蚶底播增养殖稳产新技术[J]. 海洋湖沼通报, 2009, 3: 87-90.
[17]林培振, 刘德坤, 杜萌萌, 等. 魁蚶海区底播增养殖技术[J]. 中国水产, 2010, 11:42.
[18]王晓伟, 牙鲆生长相关性状遗传力估计[D]. 青岛:中国科学院海洋研究所, 2008.
[19]陈琳琳, 孔晓瑜, 周立石, 等. 魁蚶核糖体DNA基因转录间隔区的序列特征[J]. 中国水产科学, 2005,12(1): 104-108.
[20]Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA)Software Version 4.0 [J]. Molecular Biology and Evolution, 2007,24 (8): 1596-1599.
[21]Kimura M, Yanagimoto T, Munehara H. Maternal identification of hybrid eggs inHexagrammosspp. by means of multiplex amplified product length polymorphism of mitochondrial DNA [J]. Aquatic Biology, 2007:187-192.
[22]Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data [J].Bioinformatics, 2009, 25 (11): 1451-1452.
[23]Excoffier L, Laval G, Schneider S. Arlequin ver. 3.0:An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online 1,2005, 47-50.
[24]Rice W. Analyzing tables of statistical tests [J].Evolution, 1989, 43: 223-225.
[25]郑文娟, 朱世华, 沈锡权, 等. 基于线粒体 COI基因序列探讨泥蚶的遗传分化[J]. 动物学研究, 2009,30(1): 17-23.
[26]陈丽梅, 孔晓瑜, 喻子牛, 等. 3种蛏类线粒体 16S rRNA和COI基因片段的序列比较及其系统学初步研究[J]. 海洋科学, 2005, 29(8): 27-32.
[27]陈琳琳. 几种双壳贝类ITS区序列分析与遗传多样性研究[D]. 青岛: 中国海洋大学, 2004.
[28]许志强, 葛家春, 李晓辉, 等. 基于rDNA ITS序列研究蚌科 6种类的系统发生关系[J]. 淡水渔业, 2009,39(1): 16-20.
[29]方耀林, 张燕, 肖汉兵, 等. 野生大鲵及其人工繁殖后代的遗传多样性分析[J]. 水生生物学学报, 2008,32(5): 783-786.
[30]王鸿霞, 吴长功, 相建海. 凡纳滨对虾繁殖中不同亲本对子代遗传贡献率的差异[J]. 动物学报, 2006,52(1): 175-181.
[31]Shikano T, Chiyokubo T, Taniguchi N. Effect of inbreeding on salinity tolerance in the guppy (Poeciliareticulata)[J]. Aquaculture, 2001, 202: 45-55.
[32]Sekino M, Saitoh K, Yamada T, et al. Microsatellite-based pedigree tracing in a Japanese flounderParalichthys olivaceushatchery strain: implications for hatchery management related to stock enhancement program [J].Aquaculture, 2003, 221: 255-263.
[33]Vandeputte M, Kocour M, Maugera S,et al.Heritability estimates for growth-related traits using microsatellite parentage assignment in juvenile common carp (Cyprinus carpioL.)[J]. Aquaculture, 2004, 335: 223-236.
[34]Brown W M. Evolution of Genes and Proteins [M].Sunderland: Sinauer, 1983, 62-88.
[35]王庆, 傅洪拓, 核rDNA ITS区序列在水产动物研究中的应用[J]. 安徽农学通报, 2007, 13(11): 99-101.
[36]Philips R B, Sajdak S L, Domanico M J. Relationships among charrs bases on DNA Sequences [J]. Nord J Frdabw Res, 1995, 71: 378-391.
[37]林昕, 王鹏, 杜琦, 李振华, 等. 福建沿海不同养殖区泥蚶的 ITS-1基因片段序列分析[J]. 福建水产,2008, 1: 61-65.
[38]Wright S. The genetical structure of populations [J].Annals of Eugenics, 1951, 15: 323-354.
[39]Slatkin M, Hudson R R. Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations [J]. Genetics, 1991, 129: 555-562.
