光滑河蓝蛤3个野生群体线粒体COI基因遗传多样性研究
2013-09-25刘志鸿杨爱国周丽青严加坤董春光
孙 超,刘志鸿,杨爱国,周丽青,吴 彪,严加坤,侯 丫,董春光
(1.农业部海洋渔业可持续重点实验室,中国水产科学研究院黄海水产研究所,山东 青岛 266071;2.上海海洋大学水产与生命学院,上海 201306)
光滑河蓝蛤(Potamocorbula laevis),俗称蓝蛤,隶属于抱蛤科(Corbulidae)河蓝蛤属(potamocorbula),系广温性底栖贝类,常栖息于江河口附近水域的软泥底质海底,在我国南北方均有分布。光滑河蓝蛤是虾蟹的可口饵料,具有很大的经济价值。随着虾蟹养殖业的大规模发展,人们在光滑河蓝蛤栖息地大规模捕捞,使光滑河蓝蛤种质资源遭受了极大破坏,亟需对光滑河蓝蛤的种质资源现状进行研究与保护。
线粒体DNA 是环状分子,长度在14~17 kb 之间[1],具有进化速度快、核苷酸替代率高[2]等特点,成为种类鉴别、群体遗传学等研究的有效遗传标记[3]。线粒体DNA 基因序列的差异在遗传多样性研究中占有非常重要的地位[4-5]。目前国内有关光滑河蓝蛤的相关研究资料较少,在分子方面研究尚未见有报道。对3 个地理群体的光滑河蓝蛤的线粒体细胞色素氧化酶(COI)基因片段进行了测序分析,以期为光滑河蓝蛤的种质资源保护和种群遗传学研究提供基础资料。
1 材料与方法
1.1 材 料
光滑河蓝蛤3 个群体样本分别于2012年采自东营,潍坊,日照相关海域,每个地理群体光滑河蓝蛤取样品数为10,保存于-80℃冰箱。
1.2 方 法
1.2.1 基因组DNA 提取 采用常规方法抽提DNA,取约20 mg 光滑河蓝蛤肌肉组织,加入400 μL TEN 细胞裂解缓冲液(50 mmol/L Tris-HCl,pH值9.0;100 mmol/L EDTA;200 mmol/L NaCl),剪碎后再加入40 μL SDS(10%)和8 μL 蛋白酶K(10 mg/mL),于56℃水浴锅内消化,直到溶液澄清,用酚、氯仿抽提,异戊醇沉淀,然后用去离子超纯水溶解。提取的基因组DNA 定量后配成20 ng/μL 的工作溶液备用。
1.2.2 PCR 扩增与电泳检测 所用COI 基因引物序列为AR:5′-GGTCAACAAATCATAAAGATATT GG-3′;BR:5′-TAAACTTCAGGGTGACCAAAAA ATCA-3′,由上海生工合成。PCR 反应在Eppendorf扩增仪上进行,30 μL 反应体系:3 μL PCR 10×buffer (Mg2+plus),4 μL 2 mmol/L dNTP ,2 μL 10 μmol/L 的AR 与BR 引物,0.5 μL 5 U/μL Taq 酶,2 μL 20 ng/μL DNA,其余为纯水。PCR 反应条件:94℃预变性3 min;94℃变性40 s,55℃退火40 s,72℃延伸1 min,35 个循环;72℃延伸10 min。反应多次重复,PCR 结束后取样用琼脂糖凝胶电泳检测(1×TBE,5V/cm 恒压),利用凝胶成像系统(UVP,Biolmaging Systems)进行观察和拍照。
1.2.3 测序和数据处理 PCR 产物由北京华大基因测序,将测序得到的序列用MegAlign 软件分析其同源性和相对遗传距离。并用MegAlign 软件对所获得的线粒体COI 基因片段序列进行序列比较。用DnaSp4.0 软件计算群体的单倍体型数(H)、平均核苷酸差异(K)及核苷酸多样性指数(Pi)。用MEGA5.0 软件计算序列的碱基组成、变异位点以及遗传距离,用NJ 法和UPGMA 法构建系统进化树,系统树各结点的支持率以序列数据集1 000 次重复抽样检验的自引导值(Boot-Strap value)表示。
2 结果与分析
2.1 线粒体COI 基因片段序列分析
测序得到的目的片段经过clustal W 比对后,去除引物及测序起始的部分序列,并经过在线BLAST 分析,确认得到665 bp 的COI 基因序列。利用MEGA 软件计算这段序列的碱基组成(表1),3 个群体碱基组成基本一致,COI 基因序列T、C、A、G 和A+T 的平均含量分别为46.2%、13.3%、21.7%、18.8%及67.9%,A+T 含量显著高于G+C 含量,这与线粒体DNA 碱基组成特点是相符的。
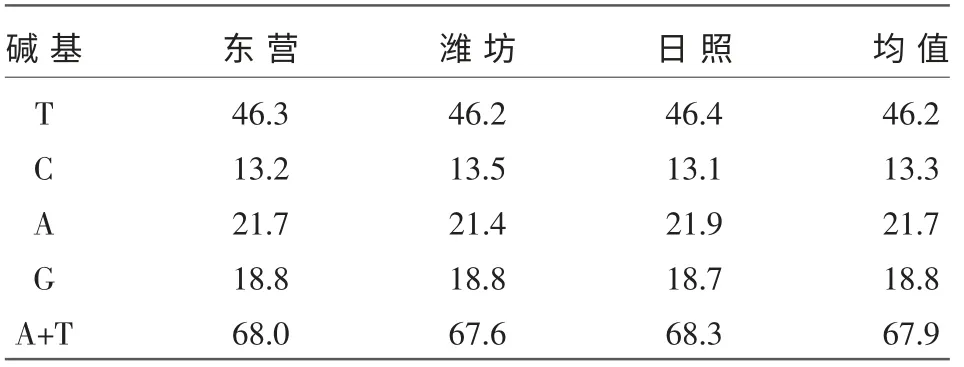
表1 光滑河蓝蛤COI 基因片段的碱基组成
对这3 个不同地理群体的30 个光滑河蓝蛤的COI 基因片段进行分析,检测到31 个多态性位点,32 个变异位点,其中包括19 个单一变异位点和13个简约信息位点。转换位点3 个,颠换位点1 个,7个插入/缺失位点。碱基转换与颠换的平均比值R=2.77。共检测到18 种单倍体型,3 个群体有一个共用单倍体型,东营群体和日照群体有2 个共用单倍体型,潍坊群体和日照群体有1 个共用单倍体型,其余单倍体型均为每个群体独有,单倍体型及其在3 个群体中的分布如表2 所示。

表2 COI 基因单倍体型片段序列及变异位点
2.2 光滑河蓝蛤群体多样性分析
利用DNAsp4.0 计算群体内遗传多样性参数,结果如表3 所示。3 个群体的COI 基因片段核苷酸多样性指数(Pi)为0.006 01,单倍体多态性(Hd)为0.890。由基于COI 基因片段可以看出,东营群体核苷酸多样性指数最高,为0.007 36,东营群体单倍体多态性最高,为0.934,潍坊群体核苷酸多样性指数最低,为0.004 46,潍坊群体的单倍体多态性最低,为0.867。
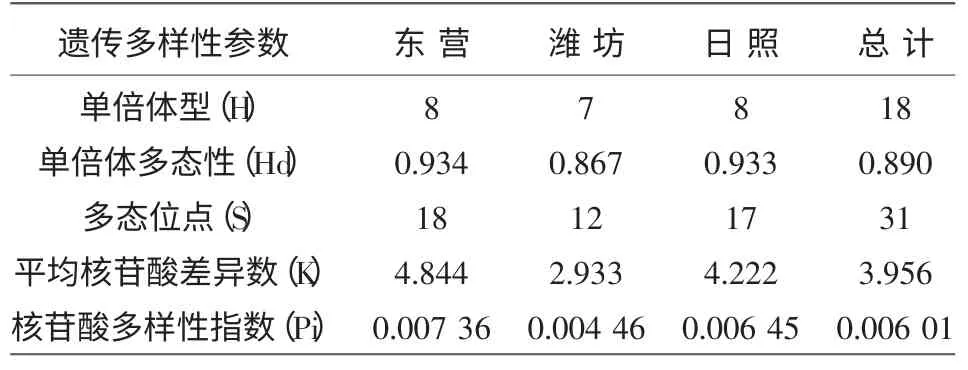
表3 光滑河蓝蛤COI 遗传多样性参数比较
对群体间的遗传多样性指数进行分析,如表4所示,群体间COI 的平均核苷酸差异数(K)在3.450~4.380 之间,最低值3.450 出现在潍坊与日照群体之间,而最高值则出现在东营与日照群体之间。根据群体间的平均遗传距离(D),东营群体和日照群体之间的平均遗传距离最大,潍坊群体和日照群体之间的平均遗传距离最小,在3 个群体中日照群体的核苷酸差异数最高。
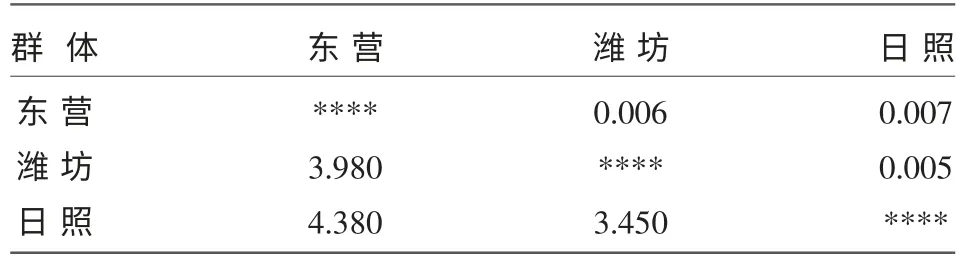
表4 光滑河蓝蛤群体间平均核苷酸差异数(K)和平均遗传距离(D)
基因流(Nm)及遗传分化系数(Fst)如表5 所示,数据显示遗传分化系数最高出现在东营与潍坊群体之间,基因流最大出现在日照与潍坊群体之间,但是基因流小于1,说明两个群体之间虽然有基因交流但是非常少。如表6 所示,群体间平均每位点核苷酸代替数(Dxy)最大出现在东营群体和日照群体之间,群体间每位点净核苷酸代替数(Da)最小值出现在东营群体和日照群体之间,且为负值,说明东营群体遗传背景在3 个群体中较丰富,群体间的分化很少。
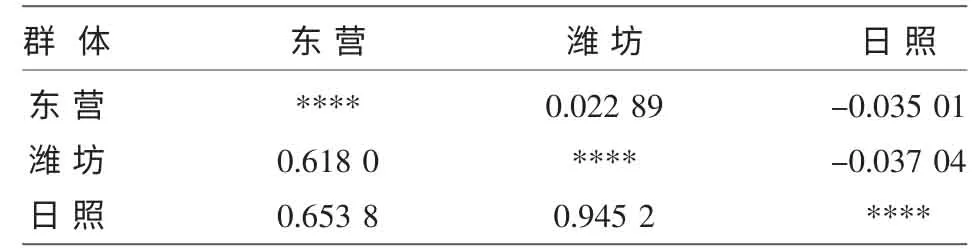
表5 基因流(Nm)和遗传分化系数(Fst)

表6 光滑河蓝蛤群体间遗传差异比较
构建基于COI 基因片段的系统发生NJ 树(图1)和UPGMA 树(图2),2 种进化树拓扑结构相似,显示出3 个群体之间的个体聚合在一起,没有出现群体内个体首先聚在一起的情况。
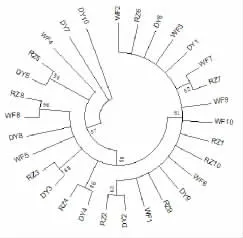
图1 3 个群体光滑河蓝蛤线粒体COI 基因片段的NJ 树

图2 3 个群体光滑河蓝蛤线粒体COI 基因片段的UPGMA 树
3 讨论与结论
细胞色素氧化酶(COI)基因进化速度快,是分析和检测群体遗传变异的一种有效的分子标记,在鱼类[6]、虾蟹类等[7-9]动物中都有应用。该研究对3个群体光滑河蓝蛤COI 基因进行PCR 扩增测序,得到665 bp 目的片段,A+T 含量明显高于G+C 含量,这与牛东红[10]、刘亚军[11]、苏天凤[12]等研究中的A+T 含量高于G+C 含量的结果一致。COI 基因30条序列中共检测到变异位点32 个,构成18 种单倍体型,单倍体型多态性为0.890,低于牛东红等[10]所研究的浙闽沿海海缢蛏(0.098 6)。基于COI 基因部分片段分析,3 个群体中有1 个共有单倍体型,除此以外,东营群体与日照群体有2 个共用单倍体型,潍坊群体和日照群体有1 个共用单倍体型,其他单倍体型均为每个群体所独有,这说明3 个群体都有各自的遗传优势。COI 基因核苷酸多样性指数东营群体最高,为0.007 36,日照群体的核苷酸多样性指数为0.006 45,这2 个群体的核苷酸多样性指数均低于缢蛏0.008 7[10],高于长蛸0.005 72[13],潍坊群体的核苷酸多样性指数最低,为0.004 46。东营群体核苷酸多样性指数较高,这可能是由于其样品采于自然保护区内,光滑河蓝蛤栖息地人为保护较好,没有遭到人为破坏,种群发展比较稳定,积累的变异较多。潍坊群体取样地点有捕捞船在当地大量采捕光滑河蓝蛤,种群在一定程度上遭到了破坏,没有足够时间积累核苷酸变异,因此核苷酸多样性指数较低,野生光滑河蓝蛤资源数量减少,遗传多样性水平降低。
Fst 值可以表示群体间的遗传分化程度,Fst 值在0~0.05 之间,表示分化较弱,在0.25 以上表示遗传分化极大。该研究中3 个群体之间的遗传分化较弱。通过2 种方法构建的系统发育树显示的拓扑结构是一致的,不同群体的个体聚在一起,没有出现明显的同一群体首先聚类的情况。从地理位置上来看,东营和潍坊地理距离较近,而这2 个群体之间的基因流却最小,遗传分化最大。潍坊群体和日照群体之间的基因流最大,遗传分化最小,分析其原因,可能是东营群体和潍坊群体虽然地理距离相对较近,但是都生活在封闭性较强的渤海,海水流动缓慢导致基因交流很少,从而产生了相对较大的遗传分化。日照地区虾蟹养殖比较多,做为虾蟹可口饵料的光滑河蓝蛤在潍坊海域被大量捕捞后运到日照地区,造成了一定程度上小范围内的基因交流,因此潍坊群体和日照群体之间的遗传分化比东营地区的遗传分化要小。孟学平等[14]在研究西施舌5 个地理群体间遗传分化也遇到了类似问题,广西北海群体与黄海的3 个群体遗传分化很弱,这也说明地理位置不是决定遗传分化程度的决定性因素。
研究中发现3 个群体的光滑河蓝蛤存在一定的遗传差异,但群体遗传多样性较小。因此,要提高检测的遗传多样性的水平,应再选择核DNA 和线粒体DNA 其他的基因区域,如16S rRNA、COⅡ等基因得到多组序列数据等,从而更全面地分析光滑河蓝蛤群体遗传水平。
[1]Wolstenholme D R.Animal mitochondrial DNA:structure and evolution[J].Interernational Review of Cytology,1992,141:173-216.
[2]Whitmoer D H,Thaith,Craftcm.The largemouth bass ctyochrome bgene[J].Fish Bio,1994,44(4):637-645.
[3]Hallerman E M.Population genetics:principle and applications for fisheries scientist[M].Bethesda,MD:American Fisheries Society,2003.
[4]Kappner I,BielerR.Phylogeny of venus clams(Bivalvia:Venerinae)as inferred from nuclear and mitochondrial gene sequences[J].Molecular Phylogenetics and Evolution,2006,40:317-331.
[5]Cannas R,Caul A,Deianal A M,et al.Discrimination between the Mediterranean spiny lobsters Palinuruselephasand P.mauritanicus(Crustacea:Decapoda)by mitochondrial sequence analysis[J].Hydrobiologia,2006,557:1-4.
[6]张凤英,马凌波,施兆鸿,等.3 种鲳属鱼类线粒体COI 基因序列变异及系统进化[J].中国水产科学,2008,15(3):392-399.
[7]杨学明,郭亚芬,蒋钦杨,等.三个群体罗氏沼虾线粒体COI 基因的遗传多样性分析[J].上海海洋大学学报,2006,15(2):144-149.
[8]林 琪,李少菁,黎中宝,等.中国东南沿海青蟹属不同种类的mtDNA COI 基因序列分析及其系统发育[J].厦门大学学报(自然科学版),2008,47(2):268-273.
[9]程汉良,夏德全,吴婷婷等,6 种帘蛤科贝类及4 种地理种群文蛤线粒体COI 基因片段序列分析[J].海洋学报,2007,29(5):109-116.
[10]牛东红,李家乐,汪桂玲,等.缢蛏六群体16S rRNA 基因片段序列的差异分析[J].上海水产大学学报,2007,(1):1-6.
[11]刘亚军,喻子牛,姜艳艳,等.栉孔扇贝16SrRNA 基因片段序列的多态性研究[J].海洋与湖沼,2002,33(5):477-483.
[12]苏天凤,黄建华,吴进锋,等.两种东风螺线粒体基因序列多态性研究[J].中国水产科学,2007,14(3):369-376.
[13]孙宝超,杨建敏,孙国华,等.中国沿海长蛸(Octopus variabilis)自然群体线粒体COI 基因遗传多样性研究[J].海洋与湖沼,2010,2(41):259-265.
[14]孟学平,高如承,申 欣,等.西施舌5 个地理群体ITS1 序列变异及系统发生分析[J].生态学报,2010,30(20):5556-5561.
